Command Palette
Search for a command to run...
Les Paramètres Du Modèle Dépassent La Diffusion RF De 5 Fois ! NVIDIA Et d'autres Lancent Proteina, Une Base Protéique Conçue À Partir De Zéro Avec Des Performances SOTA

Depuis le siècle dernier, les scientifiques se consacrent à l’exploration de la prédiction de la structure des protéines à partir des séquences d’acides aminés, avec la vision d’utiliser les acides aminés pour créer de nouvelles protéines et construire le plan de la vie. Cependant, cette grande mission a progressé lentement au fil du temps. Ce n’est que ces dernières années, avec le développement rapide de la technologie de l’IA, qu’elle a reçu un fort élan et est entrée dans la voie rapide du développement.
Depuis 2016, une révolution technologique initiée par Xu Jinbo, fondateur et scientifique en chef de Molecular Heart, et d’autres, change tranquillement ce domaine. Ils ont été les pionniers de l'introduction de l'architecture de réseau résiduel profond ResNet dans le domaine de la prédiction de structure.Une amélioration significative de la prédiction du contact des résidus protéiques a été obtenue avec succès.Cette avancée a jeté des bases solides pour l’intégration profonde de l’IA et de la conception des protéines. Depuis lors, de nombreuses équipes de recherche scientifique ont emboîté le pas et ont travaillé dur dans ce domaine. Un grand nombre d’algorithmes combinant coévolution et apprentissage profond ont vu le jour. Parmi elles, une série de réalisations majeures de David Baker, lauréat du prix Nobel de chimie 2024, et d’AlphaFold sont devenues encore plus célèbres, poussant la recherche dans ce domaine vers de nouveaux sommets.
Cependant, en examinant les études précédentes,Les modèles de génération de structures protéiques inconditionnelles ne sont souvent formés que sur des ensembles de données à petite échelle, avec un nombre de structures ne dépassant pas 500 000.De plus, lors du processus de synthèse, les réseaux neuronaux de ces modèles manquent de méthodes de contrôle efficaces, et il existe un écart important en termes d'échelle et de performances par rapport aux modèles génératifs dans des domaines tels que le langage naturel, la génération d'images ou de vidéos.
Dans le domaine du langage naturel, de la génération d’images et de vidéos, les gens ont été témoins de changements bouleversants et de percées majeures provoquées par des architectures de réseaux neuronaux évolutives, des ensembles de données de formation à grande échelle et un contrôle sémantique précis. Cela a amené les chercheurs à réfléchir profondément : pouvons-nous tirer les leçons des expériences réussies dans ces domaines et réaliser des extensions et des contrôles similaires sur les modèles de diffusion et de flux de la structure des protéines, afin de réaliser un saut qualitatif dans le domaine de la conception des protéines ?
Ce qui est gratifiant, c’est que NVIDIA a récemment collaboré avec Mila, l’Institut de recherche en intelligence artificielle du Québec, l’Université de Montréal et le Massachusetts Institute of Technology pour développer un nouveau type de générateur de protéines à flux à grande échelle, Proteina. Proteina possède cinq fois plus de paramètres que le modèle RFdiffusion et étend les données de formation à 21 millions de structures protéiques synthétiques.Nous obtenons des performances de pointe dans la conception de squelettes protéiques de novo et générons des protéines diverses et concevables d'une longueur sans précédent, jusqu'à 800 résidus.
Les résultats de recherche associés, intitulés « Proteina : Scaling Flow-based Protein Structure Generative Models », ont été sélectionnés pour l'ICLR 2025 Oral.

Adresse du document :
https://openreview.net/forum?id=TVQLu34bdw&nesting=2&sort=date-desc
Recommander un événement de partage académique. La dernière invitation à la diffusion en direct de Meet AI4S aura lieu à 12h00 le 7 mars.Huang Hong, professeur associé à l'Université des sciences et technologies de Huazhong, Zhou Dongzhan, jeune chercheur au Centre d'IA pour les sciences du Laboratoire d'intelligence artificielle de Shanghai, et Zhou Bingxin, chercheur adjoint à l'Institut des sciences naturelles de l'Université Jiao Tong de Shanghai,Présentez vos réalisations personnelles et partagez votre expérience de recherche scientifique.
L'IA renforce la conception des protéines : de la structure à la séquence, de la prédiction à la conception
Dans le processus de recherche en sciences de la vie, la conception des protéines a toujours occupé une position extrêmement critique. Pendant longtemps, l’apprentissage des règles et des modèles à partir de quantités massives de données de séquences protéiques a été un problème auquel les chercheurs scientifiques ont été confrontés. Heureusement, grâce au soutien de la technologie de l’IA, ce domaine a pris les devants en matière de redressement.
Par exemple,AlphaFold3 lancé par DeepMind améliore la modélisation des interactions entre l'ADN, l'ARN et les petites molécules.La capacité de prédire avec précision la structure des complexes protéiques fournit un support solide pour comprendre les interactions complexes des protéines au sein des cellules. Meta a lancé une fois ESMFold, qui combine un modèle de langage avec une prédiction de structure.Cela améliore considérablement la vitesse de prédiction, permettant aux chercheurs d’obtenir des informations sur la structure des protéines plus efficacement.Le dernier BioEMU-1 de Microsoft simule les changements dynamiques de conformation des protéines.Cela a ouvert une nouvelle voie pour une exploration approfondie du mécanisme de mouvement des protéines et de la conception de médicaments.
Grâce à ces bases, l’IA a progressivement commencé à pénétrer dans la conception de la structure des protéines.
La conception de la structure des protéines repose principalement sur des structures protéiques connues, qui sont modifiées et optimisées par diverses méthodes pour obtenir des protéines dotées de fonctions ou de propriétés spécifiques. Étant donné que la fonction d’une protéine est principalement déterminée par sa conformation tridimensionnelle,La méthode de modélisation directe de la distribution structurelle est progressivement devenue la tendance dominante, parmi laquelle les algorithmes basés sur des modèles de diffusion ou des modèles d'écoulement se distinguent particulièrement.Par exemple, le modèle Chroma développé par Generate Bio est la première application à grande échelle de la modélisation par diffusion pour la conception précise de protéines.Capable de produire des « protéines qui n’existent pas du tout dans la nature ».
aussi,La diffusion RF proposée par David Baker peut générer des squelettes de protéines avec des fonctions spécifiques en ajustant avec précision le réseau de prédiction de structure RoseTTAFold.Il fournit une base structurelle précise pour la conception de protéines fonctionnelles. Genie2 proposé par des chercheurs de l'Université de Columbia et de l'Université Rutgers étend les données de formation à l'AFDB.Capable de générer des protéines complexes avec plusieurs sites fonctionnels indépendants.
Il est bien connu que la structure et la séquence des protéines sont interdépendantes, la structure détermine la fonction et la séquence est la base de la structure. Lorsque la structure des protéines est modifiée par la technologie de l’IA, la séquence des protéines changera inévitablement également. La conception de séquences protéiques est principalement basée sur la structure connue des protéines, grâce à des méthodes de calcul et de prédiction, pour concevoir une séquence d'acides aminés correspondant à la structure.
À l’heure actuelle, la conception de séquences de protéines d’IA est principalement divisée en deux types :L'un est un outil de conception de séquences protéiques à squelette fixe,Par exemple, l'ESM-IF lancé par l'Université de Stanford adopte un paradigme qui combine la pré-formation et le réglage fin, intégrant intelligemment les connaissances structurelles dans la conception de protéines fonctionnelles, fournissant un support solide pour la conception de protéines avec des fonctions spécifiques. ProteinMPNN proposé par David Baker est basé sur un réseau neuronal graphique et peut générer des séquences d'acides aminés correspondantes en fonction de la structure de la chaîne principale, fournissant une méthode efficace et précise pour la conception de séquences de protéines.
L’autre est un outil de conception de séquences protéiques orienté fonction.Par exemple, ProGen lancé par Salesforce est un modèle de génération conditionnelle qui peut personnaliser les séquences de protéines en fonction d’exigences fonctionnelles spécifiques, offrant une solution très flexible pour la conception de protéines fonctionnelles. ZymCTRL, lancé par l'Université de Gérone en Espagne, permet une conception axée sur la fonction en affinant le modèle de langage pré-entraîné, fournissant un support solide pour la régulation précise de la fonction des protéines. La diffusion P450 proposée par l'Institut de biotechnologie industrielle de Tianjin, Académie chinoise des sciences, génère des variantes d'enzymes P450 avec des fonctions catalytiques spécifiques basées sur le modèle de diffusion, apportant de nouvelles opportunités de développement dans le domaine de l'ingénierie enzymatique.
Cependant, comparé aux trois autres types de modèles de protéines, l’échelle des modèles actuels de conception de la structure des protéines est généralement petite. Plus précisément, la taille de l'ensemble d'entraînement d'AlphaFold 3 est proche de 100 millions, BioEmu-1 utilise plus de 200 millions de séquences protéiques de la base de données AFDB dans la phase de pré-entraînement, et le nombre de paramètres de ProGen atteint 1,2 milliard. Cependant, RFdiffusion, un représentant exceptionnel dans le domaine de la conception de structures protéiques, utilise uniquement ses données de formation à partir de dizaines de milliers de structures protéiques réelles dans le référentiel Protein Data Bank (PDB), et la longueur totale de la structure qu'il peut produire ne peut atteindre que 600 résidus d'acides aminés. Le plus grand ensemble de données de Genie2 ne comprend qu'environ 600 000 protéines structurelles synthétiques.
Dans ce contexte,L'industrie attend avec impatience la naissance d'un modèle de conception de structure protéique avec un volume de données de formation plus important, une longueur de structure totale plus longue et une meilleure contrôlabilité - Proteina.
Modèle Proteina : une nouvelle avancée dans la conception des protéines grâce à la technologie de l'IA
Proteina, en tant que modèle de structure protéique basé sur le flux, utilise une architecture de transformateur non équivariante évolutive innovante, inspirée du transformateur de diffusion dans le champ visuel. Il peut atteindre des performances de pointe même sans s'appuyer sur la couche triangulaire coûteuse en termes de calcul.Cela permet à Proteina d'être formé sur jusqu'à 21 millions de structures protéiques, soit une augmentation de 35 fois des données de formation, générant finalement des squelettes allant jusqu'à 800 résidus.Tout en conservant la conception et la diversité, il est nettement meilleur que tous les travaux précédents.
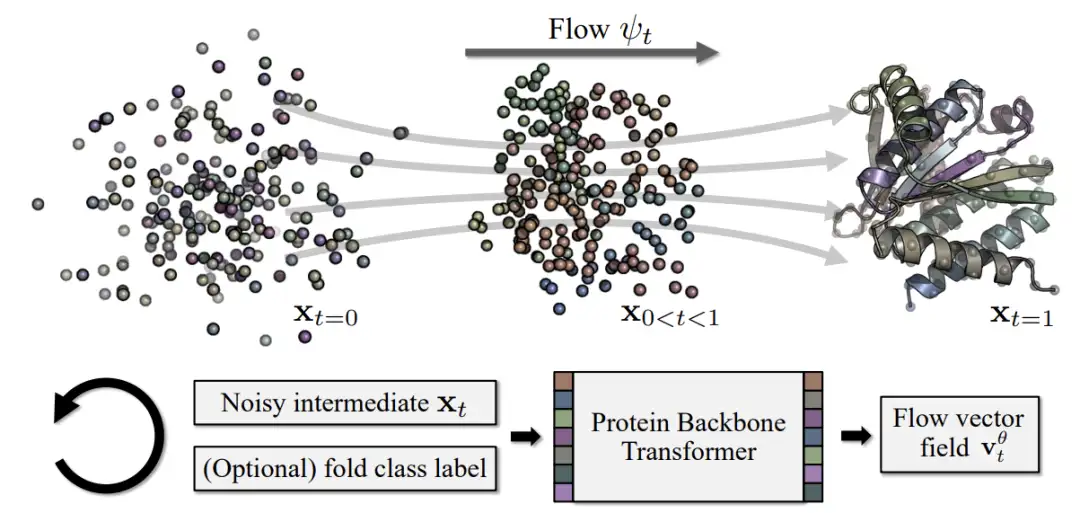
Comme le montre la figure ci-dessous, cette étude utilise principalement l’ensemble de données de clustering DFS Foldseek AFDB utilisé par Genie2.L'ensemble de données couvre environ 600 000 protéines structurelles synthétiques.Dans le même temps, l’étude a également utilisé le sous-ensemble AFDB filtré de haute qualité D21M filtré à partir d’environ 214 millions de structures AFDB.Ce sous-ensemble contient environ 21 millions de protéines structurelles synthétiques.
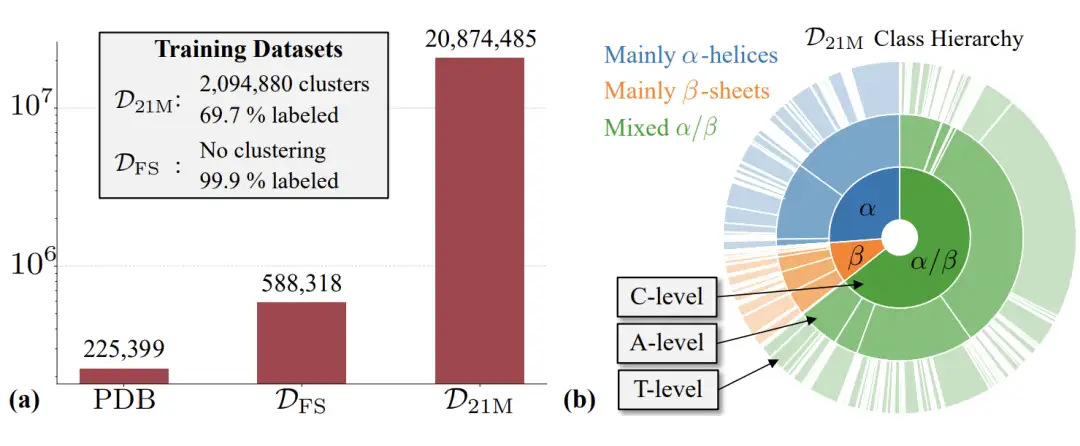
Sur la base des deux ensembles de données ci-dessus, les chercheurs ont formé trois modèles Proteina : le premier est le modèle MFS, qui contient un transformateur avec 200 millions de paramètres et une couche triangulaire avec 10 millions de paramètres ; le deuxième est le modèle Mno-triFS, qui ne contient qu'un transformateur avec 200 millions de paramètres, mais ne contient aucune mise à jour de couche triangulaire ou de représentation par paires ; le troisième est le modèle M21M, qui contient un transformateur avec 400 millions de paramètres et une couche triangulaire avec 15 millions de paramètres.
Dans le domaine de la génération de structures protéiques inconditionnelles, les méthodes isovariantes ont longtemps dominé, mais Proteina démontre que les modèles de flux non isovariants à grande échelle peuvent également être efficaces. Les paramètres de sa version d'entraînement dépassent les 400 millions.Plus de 5 fois plus grand que la diffusion RF, il s'agit actuellement du plus grand générateur de squelette protéique.Les résultats montrent également que les modèles formés sur DFS présentent une plus grande diversité, mais les chercheurs peuvent également créer des quantités beaucoup plus importantes de données de haute qualité à partir de structures entièrement synthétiques que DFS.
En termes d'indicateurs d'évaluation, Proteina ne se contente pas des évaluations traditionnelles de diversité, de nouveauté et de conception, mais introduit des indicateurs d'évaluation innovants - en saisissant directement les étiquettes empiriques du DFS dans le modèle. Cette démarche renforce la diversité entre les différentes structures de pliage, offrant un contrôle sans précédent sur les structures de protéines synthétiques grâce à de nouvelles contraintes de classe de pliage.
Comme le montre la figure ci-dessous, comparé à la génération inconditionnelle, le modèle conditionnel de Proteina atteint la diversité TM-Score la plus avancée tout en obtenant les meilleurs scores FPSD, fS et fJSD.Cela démontre pleinement son avantage en termes de diversité de structure de pliage « fS » et de meilleure correspondance de distribution entre la structure générée et les données de référence.
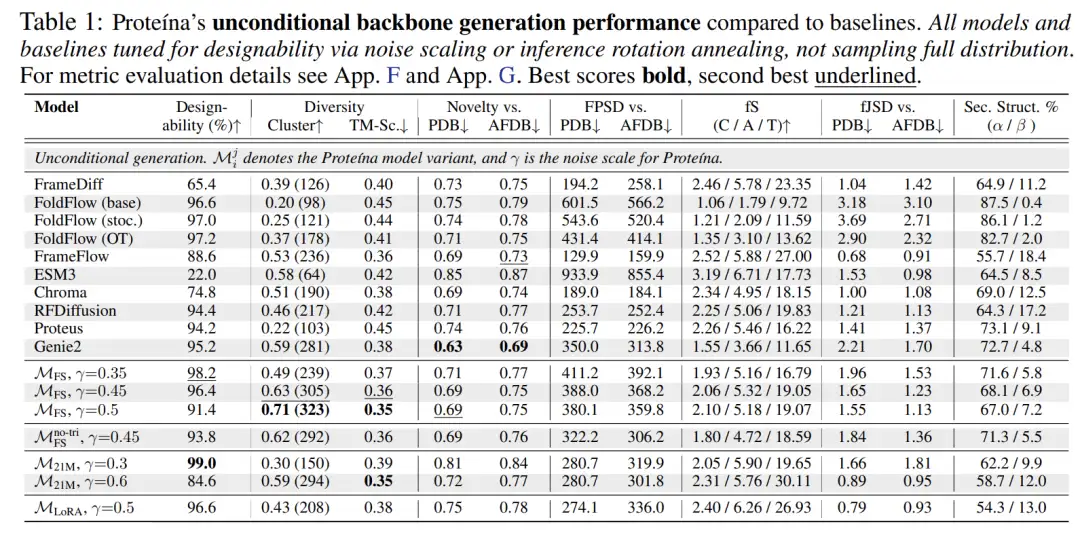
De plus, Proteina a adapté l'objectif de correspondance de flux pour s'adapter à la génération de structures protéiques et a exploré des stratégies de formation par phases, telles que l'utilisation de LoRA pour affiner le modèle afin de lui permettre de générer des protéines naturelles et concevables. Il a également développé de nouveaux schémas de guidage pour les contraintes conditionnelles de catégorie de pliage hiérarchique et a démontré avec succès une auto-orientation pour améliorer la concevabilité des protéines.En termes de performances de génération de squelette protéique, Proteina a atteint le niveau SOTA, en particulier dans la synthèse à longue chaîne.Il surpasse considérablement tous les modèles de base et démontre des capacités de contrôle supérieures aux modèles précédents grâce à de nouvelles contraintes de condition de catégorie de pliage.
Des innovations émergent dans le domaine de la conception de protéines IA en Chine
Actuellement, alors que DeepSeek relance une fois de plus le grand modèle linguistique, le domaine de la conception de protéines ouvrira sans aucun doute de nouvelles opportunités de développement, et de plus en plus de forces chinoises émergeront. En fait, à ce jour, les chercheurs et les entreprises chinois ont produit de nombreux résultats dans le seul domaine de la conception de la structure des protéines.
En 2022,Shanghai Tianrang XLab, piloté par l'IA, a lancé une nouvelle plateforme de conception de protéines - TRDesign. TRDesign peut explorer avec précision toutes les possibilités potentielles dans l'espace de repliement des protéines en apprenant beaucoup sur la relation entre la séquence et la structure des protéines, cartographier de manière inverse l'association séquence-structure-fonction apprise à partir du repliement des protéines et effectuer une conception, des tests, une stabilité et une optimisation de l'affinité des protéines de bout en bout à partir de zéro, concevant ainsi des structures protéiques qui répondent mieux à la demande.
En 2023,Le professeur Jinbo Xu, fondateur de Molecular Heart, a lancé le grand modèle NewOrigin lors de la Conférence mondiale sur l'intelligence artificielle 2023 « WAIC ».En apprenant à partir de centaines de milliards de données massives multimodales, le modèle peut réaliser une génération dirigée multimodale. Un seul modèle peut répondre à toutes les exigences du processus de génération de protéines, y compris la génération de séquences, la prédiction de structure, la prédiction de fonction et la conception de novo. Il peut résoudre le problème de la génération de protéines fonctionnelles spécifiques requises pour les applications industrielles et évaluer ses effets et sa valeur dans un environnement industriel réel.
En avril 2024, Wuxi Tushen Zhihe Artificial Intelligence Technology Co., Ltd. s'est associé à plusieurs instituts de recherche pourTourSynbio, le premier grand modèle de protéines textuelles en langage naturel publié conjointement en Chine. Le grand modèle TourSynbio ouvre le processus de conception des protéines et réalise « Protein Design AI in One ». Il peut fournir une représentation approfondie de n'importe quelle protéine, prendre en charge le dialogue et les invites en langage naturel et simplifier considérablement le processus de conception des protéines.
Août 2024L'équipe de Zhang Haicang de l'Institut de technologie informatique de l'Académie chinoise des sciences a proposé CarbonNovo.Ce résultat a été publié dans ICML2024. CarbonNovo conçoit conjointement la structure et la séquence de l'épine dorsale des protéines de manière de bout en bout. Il améliore efficacement l'efficacité et les performances de conception en établissant un modèle énergétique conjoint et en introduisant un modèle de langage protéique, présentant des avantages significatifs par rapport au modèle de conception en deux étapes existant.
Lien vers l'article :
https://openreview.net/pdf?id=FSxTEvuFa7 Lien du code :
https://github.com/zhanghaicang/carbonmatrix_public
En octobre 2024, l'équipe du professeur Liu Haiyan et du professeur Chen Quan de l'École des sciences de la vie et de la médecine de l'USTC,Nous avons développé un modèle de probabilité de diffusion de débruitage de squelette protéique SCUBA-D qui ne repose pas sur un réseau de prédiction de structure pré-entraîné.Il peut concevoir automatiquement la structure de la chaîne principale à partir de zéro, formant une chaîne d'outils complète capable de concevoir des protéines artificielles avec de nouvelles structures et séquences à partir de zéro. Il s'agit de la seule méthode de conception de protéines de novo qui a été entièrement vérifiée expérimentalement en plus de RosettaDesign. Des résultats similaires ont été publiés dans Nature Methods.
Lien vers l'article :
https://doi.org/10.1038/s41592-024-02437-W
En 2025, l'équipe de Lu Peilong à l'Université Westlake a combiné l'apprentissage profond et les méthodes basées sur l'énergie pourLa protéine transmembranaire activée par fluorescence tmFAP, qui peut se lier spécifiquement à des ligands fluorescents, a été conçue avec succès.Des algorithmes d’apprentissage profond ont été utilisés pour résoudre les problèmes fondamentaux de la conception de protéines transmembranaires. Pour la première fois, la conception précise de novo d'interactions non covalentes entre des protéines transmembranaires et des molécules ligandes au sein de la membrane a été réalisée, et leur capacité d'activation de la fluorescence dans les cellules vivantes a été démontrée, ouvrant de nouvelles voies pour la conception et l'application de protéines transmembranaires. Cette recherche a été publiée dans Nature, une revue universitaire internationale de premier plan.
Lien vers l'article :
https://www.nature.com/articles/s41586-025-08598-8
À l’heure actuelle, la Chine a formé un écosystème technologique unique dans le domaine de la conception de protéines basée sur l’IA. Ses progrès révolutionnaires ne se reflètent pas seulement dans le niveau d’innovation des algorithmes, mais également dans la construction d’une chaîne d’innovation complète, de la théorie de base à l’application industrielle. L’émergence de ces réalisations démontre pleinement la profondeur et l’ampleur des avancées technologiques de la Chine dans le domaine de la conception des protéines. Avec le développement continu de la technologie de l’IA, je pense qu’il y aura davantage de réalisations remarquables à l’avenir, ce qui entraînera un changement de paradigme dans le développement de la recherche mondiale en sciences de la vie et de l’industrie biopharmaceutique.








