Command Palette
Search for a command to run...
S’appuyant Sur Des Milliards De Gènes Provenant D’un Million D’espèces, NVIDIA Et D’autres Ont Construit La Série De Modèles EDEN, Atteignant Des Capacités De Prédiction De Génome Et De Protéines De Pointe (SOTA).

L'objectif fondamental de la biologie programmable est de parvenir à une conception rationnelle et à un contrôle précis des systèmes vivants, permettant ainsi des thérapies révolutionnaires pour les maladies complexes. Cependant,Ce processus est depuis longtemps limité par la complexité inhérente aux systèmes biologiques.Les réseaux de régulation à différentes échelles, les dépendances cachées à longue séquence et la grande adaptabilité des organismes aux changements environnementaux ont conduit la recherche et le développement traditionnels par « essais et erreurs » à un dilemme de personnalisation, de faible débit et de coût élevé.
En définitive, les données d'entraînement sur lesquelles reposent les modèles informatiques actuels, tant en termes d'échelle que de diversité, sont loin de couvrir l'immense espace de conception que la vie a développé au cours de milliards d'années d'évolution. Ces modèles peinent donc à saisir les principes de conception universels.Face à des modèles thérapeutiques innovants, multimodaux et à différentes échelles, la capacité de généralisation fait cruellement défaut.
Pour surmonter cette limitation fondamentale,Basecamp Research, NVIDIA et plusieurs institutions universitaires de premier plan ont développé conjointement la série EDEN de modèles métagénomiques de base.En exploitant d'immenses quantités de données d'évolution naturelle, communes à plusieurs espèces et liées à l'environnement, la « grammaire » profonde et les principes universels de la conception biologique ont été systématiquement extraits pour la première fois. Ce modèle, doté de 28 milliards de paramètres, a obtenu des résultats exceptionnels lors de nombreux tests de référence. Son avancée majeure réside dans sa capacité remarquable à comprendre et à générer des séquences interspécifiques, faisant ainsi passer le génie biologique du simple criblage à une nouvelle étape de « programmation prévisible ».
Pour valider les capacités d'EDEN en tant que moteur de bioconception unifié, l'équipe de recherche a mené des tests systématiques sur plusieurs modalités de traitement. En thérapie génique, EDEN, à partir d'une simple séquence de 30 bases indiquant un site cible, peut concevoir de novo des recombinases actives capables d'intégrer précisément de grands fragments dans le génome humain. Concernant la conception de peptides antimicrobiens…La bibliothèque de peptides générée par le même modèle a montré une activité allant jusqu'à 97% contre des agents pathogènes multirésistants.Elle possède également une puissance de l'ordre du micromolaire. À l'échelle de l'écosystème, EDEN a réussi à construire un microbiome synthétique contenant des dizaines de milliers de génomes artificiels, des voies métaboliques précises et des relations entre les espèces réalistes.
Les résultats de recherche associés, intitulés « Conception de thérapies programmables par l'IA avec la famille de modèles de base EDEN », ont été publiés en tant que prépublication sur bioRxiv.
Points saillants de la recherche :
* Elle a été pionnière dans un nouveau paradigme d'apprentissage des principes de conception universels directement à partir de l'histoire évolutive, et grâce à l'entraînement avec BaseData, une base de données métagénomiques couvrant la biodiversité mondiale, elle a atteint des capacités exceptionnelles de compréhension et de génération de séquences inter-espèces.
* Cette étude valide la grande polyvalence d'un modèle fondamental unique pour la conception de thérapies multimodales et multi-échelles, démontrant qu'un seul modèle peut répondre de manière uniforme à des défis de conception complexes allant des molécules aux écosystèmes.
* EDEN, utilisant uniquement des indices d'ADN, peut concevoir des recombinases fonctionnelles pour de multiples sites liés à la maladie, atteignant un taux de réussite fonctionnelle de 63,21 TP3T sur des cibles non entraînées.

Adresse du document :
https://doi.org/10.64898/2026.01.12.699009
Suivez notre compte WeChat officiel et répondez « EDEN » en arrière-plan pour obtenir le PDF complet.
Autres articles sur les frontières de l'IA :
https://hyper.ai/papers
Jeu de données BaseData : redéfinir les critères de référence des données d’IA biologique grâce à des séquences longues de haute qualité.
L'ensemble de données BaseData utilisé dans cette étude s'affranchit fondamentalement des limitations des bases de données biologiques traditionnelles. Ces dernières s'appuient généralement sur un nombre limité de génomes de référence et de courtes séquences fragmentées, tandis que BaseData vise à capturer systématiquement des signaux évolutifs complets, construisant ainsi une chaîne d'approvisionnement de données génomiques évolutives couvrant la biodiversité mondiale.
La valeur fondamentale de BaseData réside principalement dans son envergure et sa composition stratégique. Comme le montre le diagramme ci-dessous,Il contient 9,7 billions de marqueurs nucléotidiques pour l'entraînement, couvrant plus d'un million de nouvelles espèces et 100 milliards de nouveaux gènes.Plus important encore, les données ne sont pas collectées aléatoirement, mais délibérément enrichies de séquences à haute densité d'information, telles que les données métagénomiques environnementales, les bactériophages et les éléments génétiques mobiles. Ces données enregistrent naturellement des dynamiques évolutives clés, comme les interactions bactériophage-hôte et les transferts horizontaux de gènes, fournissant ainsi un matériel essentiel aux modèles pour l'apprentissage de règles fonctionnelles universelles entre les espèces.
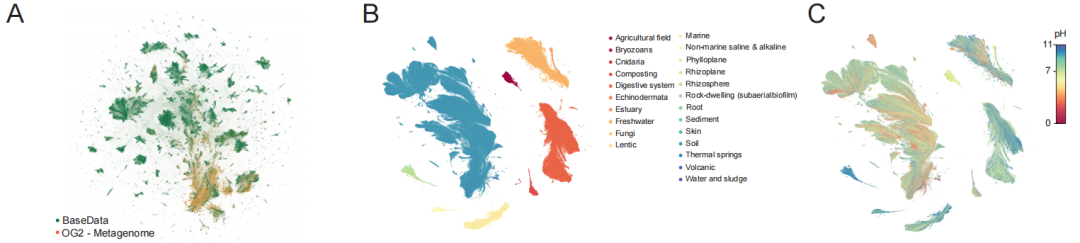
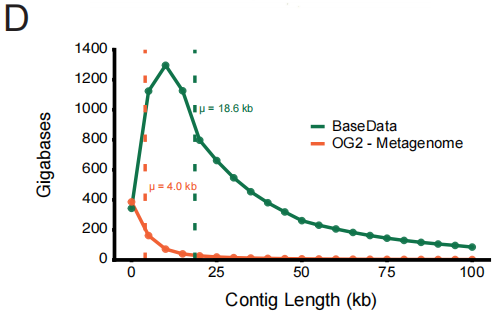
En termes de qualité des données, BaseData présente une amélioration qualitative, notamment grâce à une meilleure complétude du contexte de séquence. Comparée à OpenGenome-2 (OG2), largement utilisée, sa longueur médiane de fragments de séquence contigus (chevauchements) atteint 18,6 kpb (contre 4,0 kpb pour OG2), et chaque assemblage contient un nombre significativement plus élevé de gènes. Ce contexte contigu plus étendu est essentiel pour que le modèle puisse comprendre la régulation intergénique et les voies métaboliques.
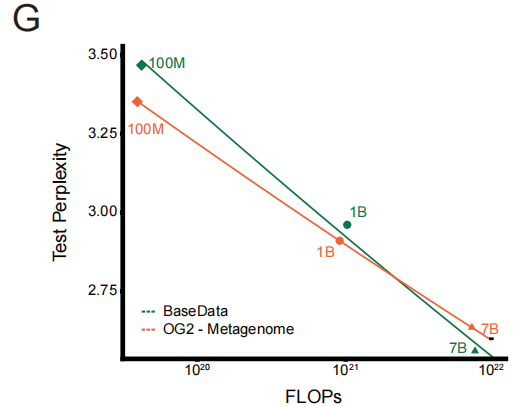
Pour quantifier cet avantage qualitatif, l'équipe de recherche a mené une expérience contrôlée : l'entraînement d'une série de modèles sur des ensembles de données de taille identique provenant de BaseData et d'OG2. Les résultats ont clairement validé la « loi de mise à l'échelle sensible à la qualité ». À charge de calcul égale, le modèle entraîné sur BaseData a connu une diminution plus rapide de sa perplexité sur les données de test. Un résultat clé est que les grands modèles (par exemple, 7 milliards de paramètres) peuvent exploiter pleinement les informations séquentielles longues de BaseData, surpassant ainsi les modèles similaires entraînés sur OG2.Cela démontre directement l'impact décisif du contexte à long terme sur la performance du modèle.
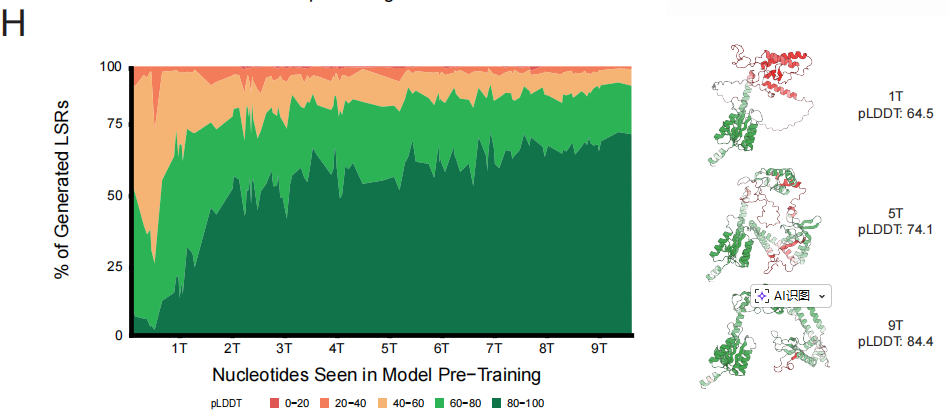
D'après ce modèleL'équipe de recherche a entraîné le modèle EDEN-28B avec 28 milliards de paramètres en utilisant l'ensemble des données de base.Ce modèle a non seulement atteint la plus faible perplexité de test, mais sa trajectoire d'amélioration des performances correspond également parfaitement aux prédictions d'échelle issues de modèles à petite échelle. Lors du suivi des tâches en aval, l'indice de confiance structurale des protéines générées par le modèle pendant le pré-entraînement a montré une augmentation monotone et continue au cours de l'entraînement, démontrant ainsi que des données de haute qualité améliorent directement et durablement la capacité à générer des données pour un traitement pratique.
De plus, toutes les données ont été obtenues grâce à des accords juridiques standardisés couvrant 28 pays et 208 licences, établissant un cadre de traçabilité et de partage des avantages de la source à l'utilisation, et définissant les normes éthiques et de gouvernance nécessaires à la recherche en IA biologique à grande échelle.
EDEN, un moteur de conception biologique à usage général
La famille de modèles EDEN est conçue avec « l'évolutivité, l'universalité et l'évolutivité » comme principes de conception fondamentaux, avec des paramètres de modèle allant de 100 millions à 28 milliards.Parmi eux, EDEN-28B, en tant que modèle de travail principal, possède une architecture et une stratégie d'entraînement profondément adaptées aux propriétés uniques des données métagénomiques.
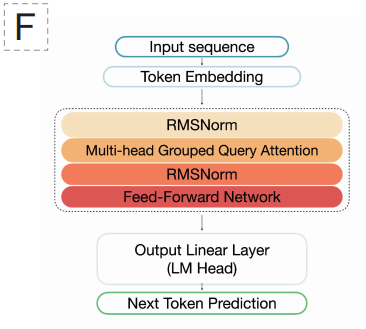
En termes d'architecture du modèle,EDEN adopte une architecture Transformer uniquement basée sur le décodeur, validée par des modèles de langage à grande échelle, et plus précisément sur le style de conception de Llama 3.1.Ce choix est rendu possible par la capacité supérieure du Transformer à modéliser les dépendances à longue portée. EDEN-28B est un réseau à 48 couches, comprenant 6 144 couches cachées, 48 nœuds d'attention et utilisant la fonction d'activation SwiGLU avec un encodage positionnel RoPE. Le modèle emploie une méthode de tokenisation à résolution mononucléotidique avec un vocabulaire de 512 caractères, ce qui lui permet de comprendre et de générer des séquences d'ADN au niveau le plus élémentaire, celui des « lettres ».
L'un des principaux atouts technologiques réside dans sa capacité à générer de longues séquences. Bien que la fenêtre de contexte du modèle soit fixée à 8 192 étiquettes, dans les applications pratiques,Il peut générer de manière stable et assembler avec précision des séquences génomiques cohérentes de plus de 13 000 paires de bases tout en conservant l'ordre correct des gènes, les cadres de lecture et les structures des éléments régulateurs.Cela indique que le modèle apprend bien plus qu'une simple correspondance de motifs locaux ; il peut inférer et appliquer une « grammaire » plus profonde de l'organisation génomique, qui dépasse la longueur de la fenêtre physique. L'entraînement complet a été réalisé sur 1 008 GPU H100, permettant un apprentissage efficace à partir d'une quantité massive de données évolutives grâce au calcul distribué à grande échelle.
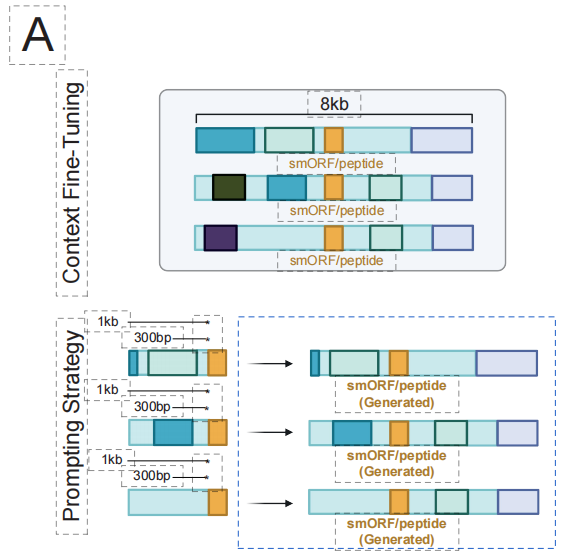
La philosophie de conception fondamentale d'EDEN repose sur un paradigme de « pré-entraînement et d'ajustement fin ». Dans un premier temps, le modèle est pré-entraîné à grande échelle sur une base de données couvrant l'histoire évolutive de différentes espèces, intégrant ainsi les principes généraux de la conception biologique tels que le repliement des protéines et l'assemblage des voies métaboliques.
En nous appuyant sur ces bases solides, nous pouvons ensuite cibler des tâches de conception thérapeutique spécifiques, telles que la conception de recombinases ciblant des sites d'ADN spécifiques ou la génération de nouveaux peptides antimicrobiens.Avec seulement une petite quantité de données de haute qualité appariées à la tâche pour un réglage fin léger, le modèle peut rapidement maîtriser le « dialecte » de la tâche.Cette conception permet à un seul modèle EDEN de servir de « moteur de séquence biologique » universel, s'adaptant et pilotant avec souplesse diverses modalités thérapeutiques allant de l'insertion de gènes et de la conception de peptides à l'ingénierie du microbiome, réalisant ainsi véritablement la vision de la biologie programmable « un modèle, de multiples capacités ».
Stimuler l'innovation thérapeutique aux niveaux moléculaire, cellulaire et écosystémique
Afin de vérifier systématiquement l'universalité et l'efficacité du modèle EDEN dans la conception réelle des traitements, l'équipe de recherche a sélectionné quatre directions clés très différentes en termes d'échelle, de modèle et de complexité biologique pour une vérification expérimentale.
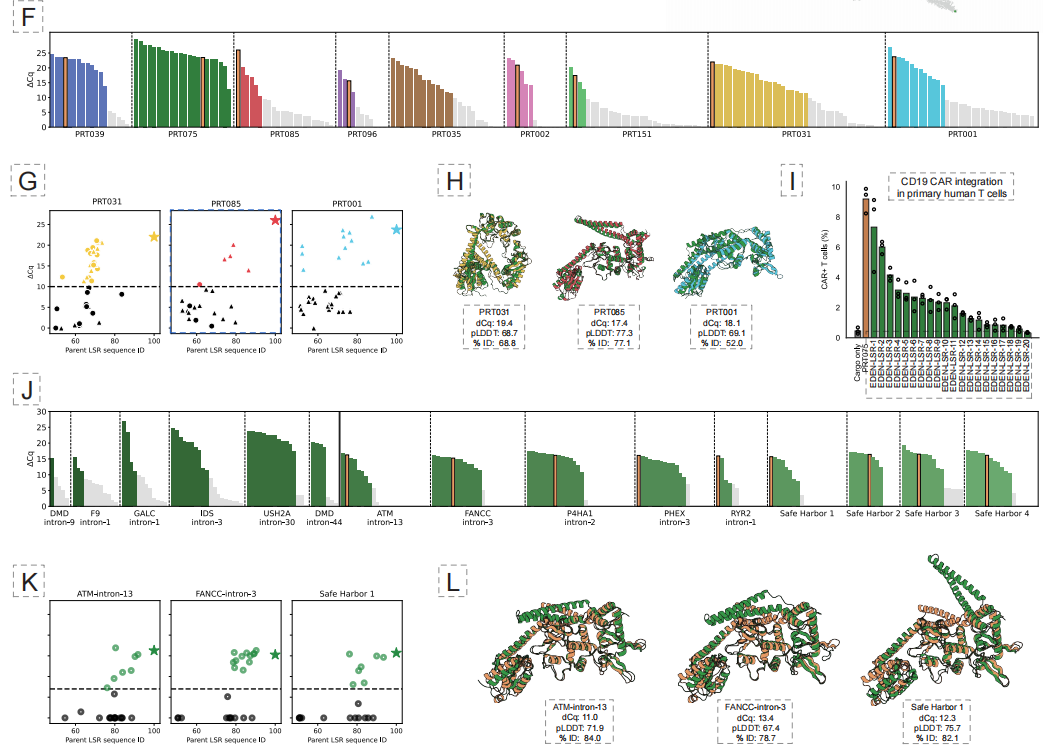
Dans le domaine de l'insertion de gènes programmable par l'IA (aiPGI), l'équipe s'est concentrée sur le dépassement du goulot d'étranglement de longue date que représente « l'intégration précise de grands fragments d'ADN ».La technologie CRISPR traditionnelle repose sur la création de cassures double brin, or les grandes recombinases à sérine naturelles ne peuvent pas reconnaître les séquences du génome humain. Comme illustré ci-dessous, la solution d'EDEN consiste à construire le modèle EDEN-LSR, capable de comprendre la relation de correspondance « séquence d'ADN cible → recombinase correspondante », grâce à un ajustement précis des millions d'appariements LSR-site d'attachement contenus dans le modèle.
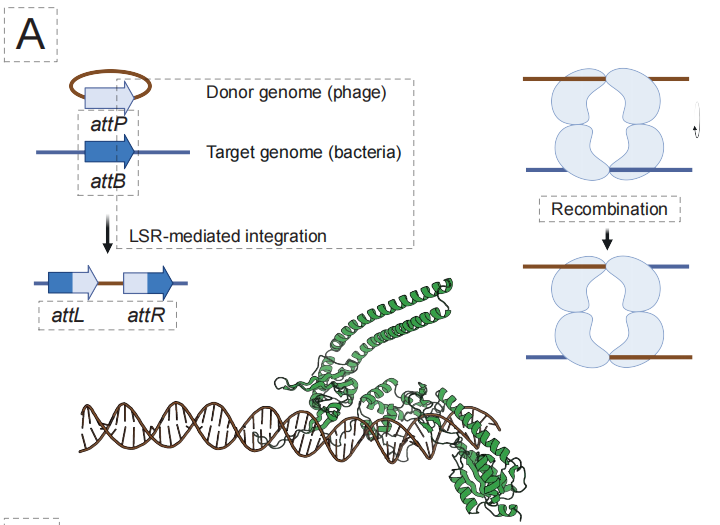
Les résultats expérimentaux ont montré que cette approche a permis de générer avec succès des LSR actifs pour 10 loci géniques différents liés à des maladies et 4 loci potentiels de « refuge sûr », avec un taux de succès fonctionnel global de 53,61 TP3T. Plus important encore,L'enzyme de conception 50% peut réaliser l'insertion du gène CAR lié au traitement dans les cellules T humaines primaires, et certaines variantes ont atteint des efficacités d'intégration allant jusqu'à 40% dans les lignées cellulaires.Cela démontre son potentiel d'application clinique.
Dans le domaine des nouvelles recombinases de pontage (BR),Les capacités du modèle EDEN ont été étendues à un système d'édition génique plus programmable : la recombinase de pontage.Comme le montre la figure ci-dessous, afin d'optimiser la conception, l'équipe a construit le modèle spécifique EDEN-BR en affinant le modèle sur des millions de régions génomiques contenant des BR.
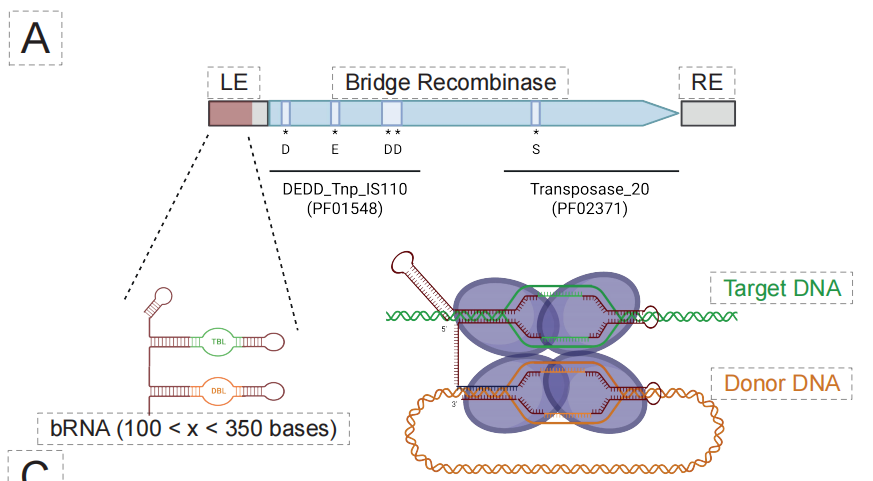
Des expériences biochimiques clés ont validé la faisabilité de ce processus de conception. Comme illustré dans la figure ci-dessous, lors de tests préliminaires acellulaires, deux des 49 séquences candidates générées par EDEN-BR ont présenté une activité recombinase confirmée. Ces deux protéines de synthèse, nommées DF3843 et DF3881, présentent une similarité maximale de seulement 851 et 65,81 TP3T, respectivement, avec toute séquence BR naturelle connue. Leur similarité de séquence avec une protéine de référence bien caractérisée, ISCro4, est encore plus faible (351 TP3T), mais leur structure tridimensionnelle est très similaire.Cela prouve qu'EDEN ne se contente pas d'imiter des séquences, mais qu'il maîtrise plutôt la logique structurelle fondamentale qui détermine la fonction et le repliement des protéines.
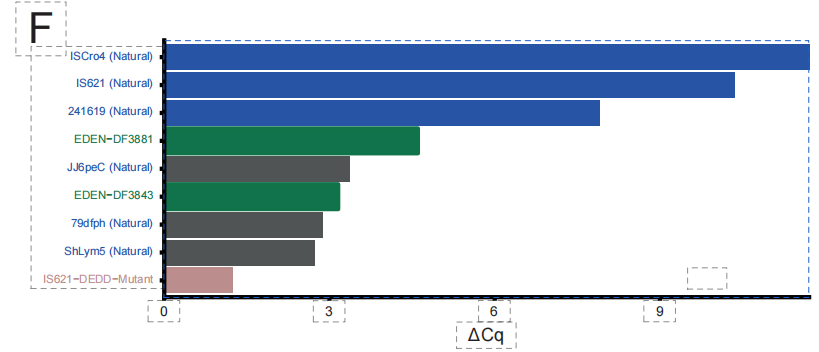
Dans le domaine des nouveaux peptides antimicrobiens (PAM), l'équipe de recherche a validé la capacité d'EDEN à concevoir de nouveaux peptides antimicrobiens. Comme le montre la figure ci-dessous,En employant une stratégie de réglage fin qui intègre des informations contextuelles génomiques, le modèle est capable de générer de nouvelles séquences de peptides antimicrobiens.
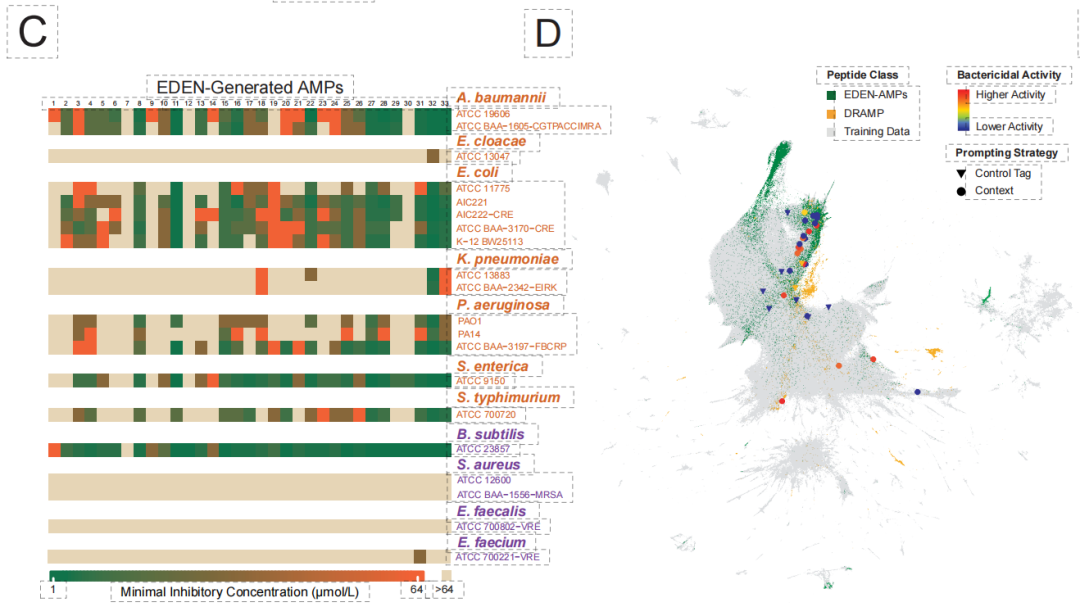
La vérification expérimentale a donné des résultats révolutionnaires. Comme le montre la figure ci-dessous,Dans une bibliothèque AMP composée de 33 peptides générateurs, jusqu'à 971 séquences TP3T ont présenté une activité antibactérienne.Parmi ces candidats, les plus performants ont atteint des concentrations inhibitrices micromolaires contre des bactéries Gram négatif multirésistantes (telles qu'Acinetobacter baumannii), démontrant ainsi une forte capacité de pénétration de la membrane externe. Les séquences générées présentaient généralement une faible similarité avec les bases de données connues, confirmant que le modèle peut surmonter les limitations traditionnelles de l'homologie et permettre une véritable conception de novo.
Enfin, au niveau des écosystèmes les plus complexes, la recherche a remis en question la conception de « microbiomes synthétiques ». Les méthodes traditionnelles peinent à coordonner les interactions métaboliques et l'équilibre écologique entre de multiples espèces. Comme le montre la figure ci-dessous, EDEN, après un ajustement précis à l'aide de données sur le microbiome du système digestif,En se basant uniquement sur des gènes fonctionnels ou des indices de niche, une métagénomique synthétique contenant plus de 90 000 espèces et couvrant des gigabases a été générée avec succès.
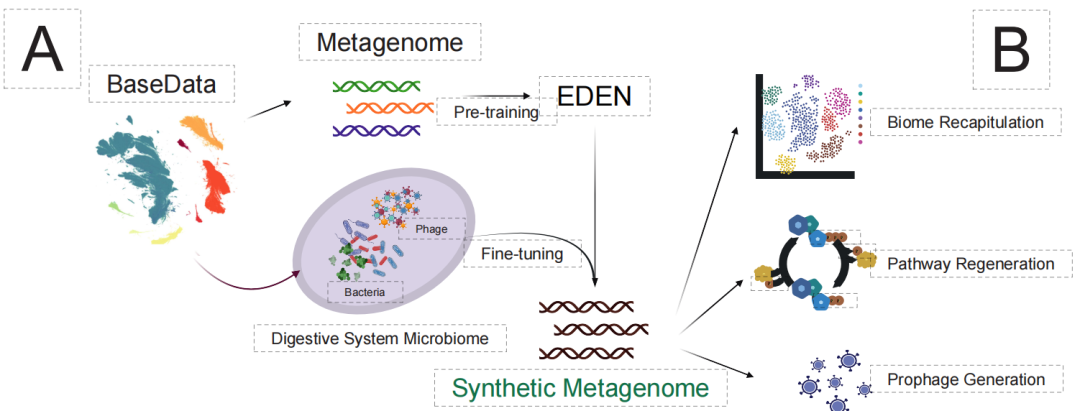
Les résultats obtenus témoignent d'un haut degré de réalisme écologique :L'espèce 99% a été correctement classée dans le biote lié au système digestif et ses voies métaboliques interspécifiques ont été entièrement préservées.De plus, le modèle peut même générer avec précision des structures de prophages intégrées au génome de l'hôte, démontrant ainsi qu'il a saisi la logique d'interaction complexe entre l'hôte et le virus.
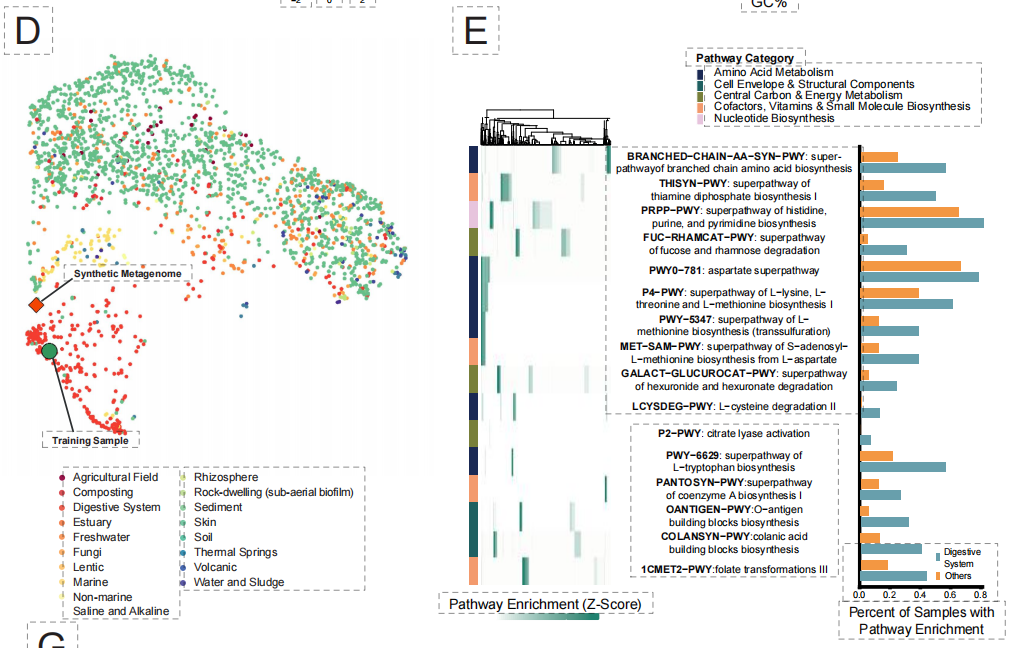
Ces quatre expériences à différentes échelles démontrent collectivement que le modèle EDEN, pré-entraîné sur des données évolutives unifiées, peut servir de moteur de conception biologique général. Il permet de piloter rapidement et de manière fiable des innovations thérapeutiques aux niveaux moléculaire, cellulaire et écosystémique avec un minimum de données spécifiques à la tâche, jetant ainsi les bases pratiques d'une biologie programmable.
L'innovation par l'intégration de l'IA et de la biologie synthétique
Ces dernières années, l'intégration et l'innovation entre le monde universitaire et l'industrie dans le domaine de la biologie programmable se sont considérablement accélérées, et une série d'avancées majeures redéfinissent les frontières de la bioconception.
Les plus grandes institutions académiques du monde transforment les connaissances sur l'évolution en modèles informatiques d'une ampleur et d'une précision sans précédent. Par exemple, début 2024, une équipe conjointe de DeepMind, d'Isomorphic Labs et de plusieurs universités a publié le modèle AlphaFold 3, capable de prédire simultanément la structure et les interactions des protéines, et de générer de nouvelles protéines aux fonctions spécifiques. Ce modèle est le premier à intégrer l'interaction complexe des biomolécules dans un cadre unifié pour la simulation de haute précision.La revue Nature l'a salué comme « un bond en avant dans la cartographie des mécanismes internes des machines moléculaires de la vie ».
L'industrie accélère la transformation de ces avancées en plateformes et thérapies. Dans le domaine du développement de médicaments piloté par l'IA, NVIDIA et Recursion Pharmaceuticals ont lancé BioNeMo, une bibliothèque de modèles d'IA en biochimie visant à faire évoluer la recherche de médicaments, d'une approche fastidieuse et répétitive à une approche guidée. La société de biologie synthétique Ginkgo Bioworks exploite sa plateforme automatisée pour concevoir systématiquement des communautés microbiennes destinées à la capture du carbone et à la production de composés chimiques, favorisant ainsi l'ingénierie d'écosystèmes synthétiques.
Cette nouvelle vague, portée par les données et les algorithmes, propulse la biologie d'une science observationnelle et descriptive vers une discipline d'ingénierie programmable, déboguable et prévisible. Cela signifie non seulement que nous pouvons programmer le vivant avec plus de précision pour vaincre les maladies, mais aussi que nous préfigurons notre capacité à concevoir systématiquement des systèmes biologiques pour relever les défis mondiaux liés aux ressources, à l'environnement et à la santé.
Liens de référence :
1.https://nvidianews.nvidia.com/news/nvidia-announces-broad-expansion-of-its-biomedicine-platform
2.https://www.ginkgobioworks.com/2024/01/04/ginkgo-bioworks-and-pfizer-expand-collaboration-to-advance-rna-based-therapeutics/








