Command Palette
Search for a command to run...
Inspiriert Von DeepSeek Engram, Dem „externen Gehirn“ Des Genom-Basismodells Gengram, Wurde Eine Leistungssteigerung Von Bis Zu 22,61 TP3T erzielt.

Grundlegende Genommodelle (GFMs) sind zentrale Werkzeuge zur Entschlüsselung des Codes des Lebens und ermöglichen es, durch die Analyse von DNA-Sequenzen wichtige biologische Informationen wie Zellfunktionen und die Entwicklung von Organismen zu erschließen. Bestehende Transformer-basierte GFMs weisen jedoch einen entscheidenden Mangel auf: Sie benötigen umfangreiches Vortraining und rechenintensive Prozesse, um Polynukleotidmotive indirekt abzuleiten. Dies ist nicht nur ineffizient, sondern auch bei der Erkennung motivbasierter funktioneller Elemente eingeschränkt.
kürzlich,Das vom Genos-Team, bestehend aus Mitgliedern des BGI Life Sciences Research Institute und des Zhejiang Zhijiang Laboratory, vorgeschlagene Gengram-Modell (Genomisches Engramm),Dies bietet eine revolutionäre Lösung für dieses Problem. Dieser Entwurf vermeidet die feste Kodierung biologischer Regeln und verleiht dem Modell gleichzeitig ein explizites Verständnis der genomischen „Grammatik“.
Als leichtgewichtiges, bedingtes Speichermodul, das speziell für die Modellierung von Genommotiven entwickelt wurde, liegt die Kerninnovation von Gengram in seinem k-Mer-Hash-Speichermechanismus, der ein hocheffizientes Multi-Basen-Motivspeicher-Repository erstellt. Im Gegensatz zu traditionellen Modellen, die Motive indirekt ableiten,Es speichert direkt k-Mere mit Längen von 1-6 Basen und deren Einbettungsvektoren und erfasst die lokalen Kontextabhängigkeiten funktionaler Motive durch einen lokalen Fensteraggregationsmechanismus.Die Motivinformationen werden anschließend über ein gate-gesteuertes Modul mit dem Backbone-Netzwerk fusioniert. Das Forschungsteam gab an, dass Gengram, integriert in das derzeit modernste Genommodell Genos, unter denselben Trainingsbedingungen signifikante Leistungsverbesserungen bei verschiedenen Aufgaben der funktionellen Genomik erzielt, mit einer maximalen Verbesserung von 22,61 TP3T.

Papieradresse:https://arxiv.org/abs/2601.22203
Codeadresse:https://github.com/BGI-HangzhouAI/Gengram
Modellgewichte:https://huggingface.co/BGI-HangzhouAI/Gengram
Die Trainingsdaten umfassen menschliche und nicht-menschliche Primatengenome.
Der Trainingsdatensatz enthält 145 hochwertige, haplotypgeparste und assemblierte Sequenzen, die menschliche und nicht-menschliche Primatengenome abdecken.Die menschlichen Sequenzen stammen primär aus dem Human Pangenome Reference Consortium (HPRC, 2. Auflage) und wurden durch die Referenzgenome GRCh38 und CHM13 ergänzt. Sequenzen nicht-humaner Primaten wurden aus der NCBI RefSeq-Datenbank integriert, um die evolutionäre Diversität zu berücksichtigen. Alle Sequenzen wurden mittels One-Hot-Encoding verarbeitet. Das Vokabular umfasst die vier Standardbasen (A, T, C, G), mehrdeutige Nukleotide (N) und Dokumentenendmarkierungen.
Finale,Das System erstellte drei Datensätze zur Unterstützung der Ablationsexperimente und des formalen Vortrainings.
50 Milliarden Token zu 8.192 (Ablation)
200 Milliarden Token à 8.000 (10 Milliarden formale Vorschulung)
100 Milliarden Token à 32.000 (10 Milliarden formale Vorschulung)
Und ein Datenmischungsverhältnis von Mensch zu Nicht-Mensch = 1:1 beibehalten.
Die Genommodellierung verlagert sich von der „Aufmerksamkeitsableitung“ hin zur „Gedächtnisverbesserung“.
Inspiriert vom Speichermechanismus von DeepSeek Engram entwickelte und implementierte das Genos-Team schnell Gengram.Dieses Modul bietet explizite Funktionen zur Speicherung und Wiederverwendung von Motiven für grundlegende Genommodelle und überwindet damit die Einschränkungen gängiger GFMs, denen ein strukturierter Motivspeicher fehlt und die sich lediglich auf die Erweiterung des „impliziten Speichers“ der Trainingsdaten stützen können. Dies führt dazu, dass die Genommodellierung von der „Aufmerksamkeitsableitung“ zur „Speichererweiterung“ übergeht. Die Modularchitektur ist in der folgenden Abbildung dargestellt:
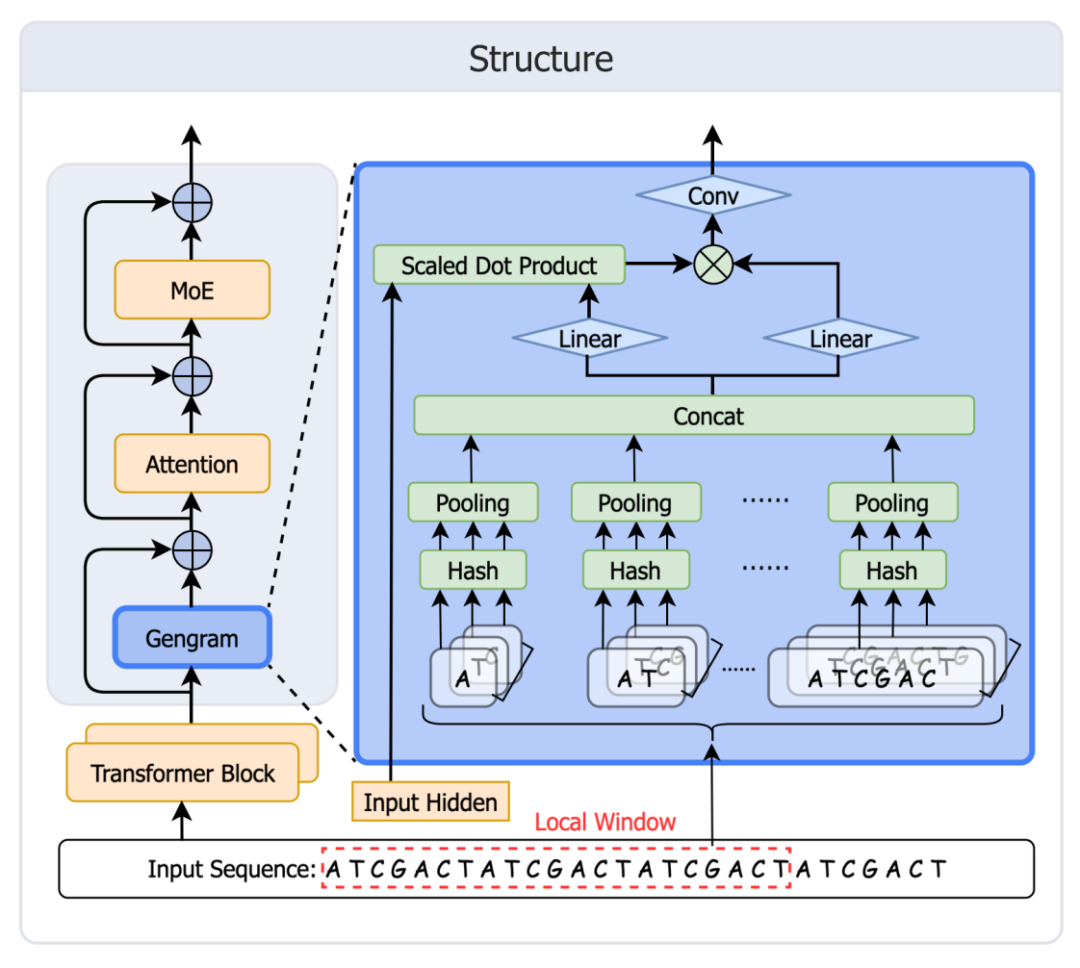
Tabellenerstellung: Erstellen eines Hash-Speichers (statischer Schlüssel + lernbarer Einbettungswert) für alle k-Mer-Werte von k=1 bis 6.
Abruf: Ordne alle im Fenster angezeigten k-Mer-Werte Tabelleneinträgen zu.
Aggregation: Zuerst werden die Ergebnisse für jedes k aggregiert, dann werden sie über alle k hinweg verkettet.
Gating: Das Gate steuert die Aktivierung, schreibt Motivbeweise in den Reststrom und gelangt dann in die Aufmerksamkeit.
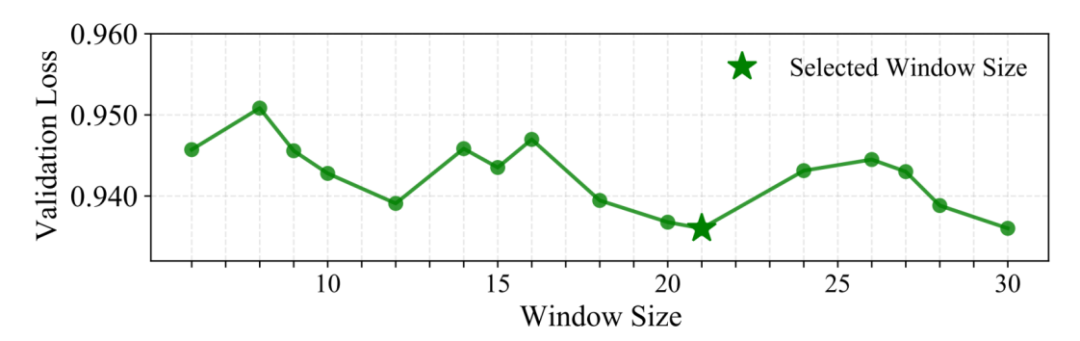
Ein wichtiges Designmerkmal: Lokale Fensteraggregation (W=21bp)
Anstatt an jeder Position ein einzelnes n-Gramm abzurufen, aggregiert Gengram mehrere k-Mer-Einbettungen innerhalb eines festen Fensters, um zuverlässiger „lokale, strukturell konsistente“ Motivnachweise einzufügen. Forscher validierten dies durch die Suche mit einer Fenstergrößenstrategie.Wir haben festgestellt, dass 21 bp auf dem Validierungsdatensatz die optimale Leistung erzielt.Eine mögliche biologische Erklärung ist, dass ein typischer DNA-Doppelhelixzyklus etwa 10,5 Basenpaare pro Umdrehung umfasst, sodass sich 21 Basenpaare genau zweimal drehen. Dies bedeutet, dass zwei Basen, die 21 Basenpaare voneinander entfernt sind, im dreidimensionalen Raum auf derselben Seite der Helix liegen und ähnlichen biochemischen Umgebungen ausgesetzt sind. Eine Betrachtung in diesem Maßstab könnte die Ausrichtung der Phasenkonsistenz lokaler Sequenzsignale begünstigen.
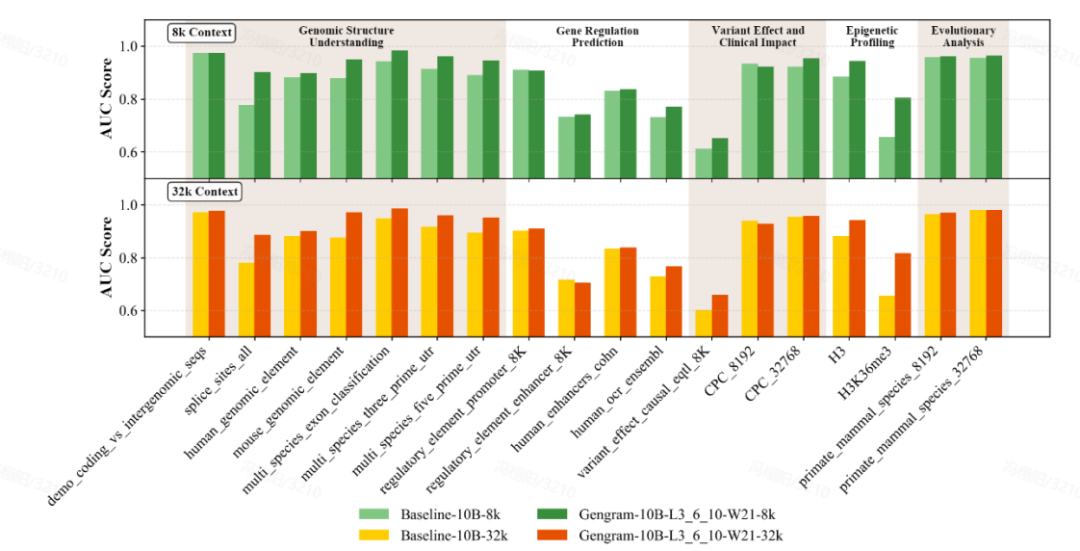
Wesentliche Verbesserungen bei der Auswertung: Kleine Parameter, große Veränderungen
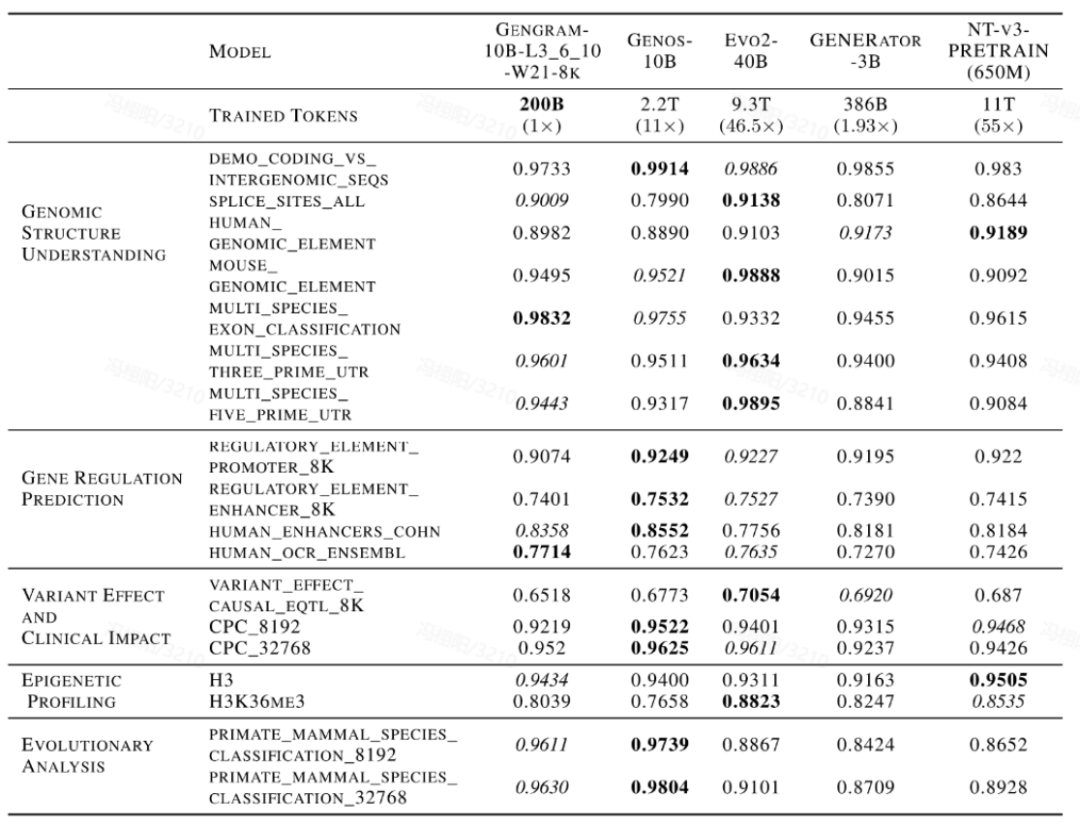
Das Team führte eine umfassende Evaluierung des Modells anhand von Multi-Standard-Benchmark-Datensätzen durch, die Genomic Benchmarks (GB), Nucleotide Transformer Benchmarks (NTB), Long-Range Benchmarks (LRB) und Genos Benchmarks (GeB) umfassen.Es wurden achtzehn repräsentative Datensätze ausgewählt, die fünf Hauptaufgabenkategorien abdecken:Genomstrukturverständnis, Genregulationsvorhersage, epigenetische Profilierung, Variantenwirkung und klinische Auswirkungen sowie evolutionäre Analyse.
Gengram, ein schlankes Plugin mit nur etwa 20 Millionen Parametern, repräsentiert einen Bruchteil der Parameter eines Basismodells mit Hunderten von Milliarden Parametern, erzielt aber dennoch deutliche Leistungsverbesserungen. Unter denselben Trainingsbedingungen, mit Kontextlängen von 8.000 und 32.000...In den meisten Aufgaben schnitten die mit Gengram integrierten Modelle besser ab als die nicht integrierten Versionen.Hinsichtlich spezifischer ErscheinungsformenDer AUC-Wert für die Vorhersage der Spleißstelle verbesserte sich von 0,776 auf 0,901, eine Steigerung um 16,11 TP3T;Der AUC-Wert der epigenetischen Vorhersageaufgabe (H3K36me3) verbesserte sich von 0,656 auf 0,804, eine Steigerung um 22,61 TP3T.
Darüber hinaus geht diese Leistungssteigerung mit einem signifikanten „Datennutzungseffekt“ einher. Im direkten Vergleich mit gängigen DNA-basierten Modellen wie Evo2, NTv3 und GENERATOR-3B,Modelle, die Gengramme integrieren, benötigen nur eine sehr geringe Menge an Trainingsdaten und weniger Aktivierungsparameter, um mit öffentlich verfügbaren Modellen mithalten zu können, deren Trainingsdaten bei Kernaufgaben um ein Vielfaches bis Zehnfache größer sind.Es beweist eine hohe Effizienz beim Datentraining.
Detaillierte Analyse von Gengram
Warum kann Gengram das Training beschleunigen?
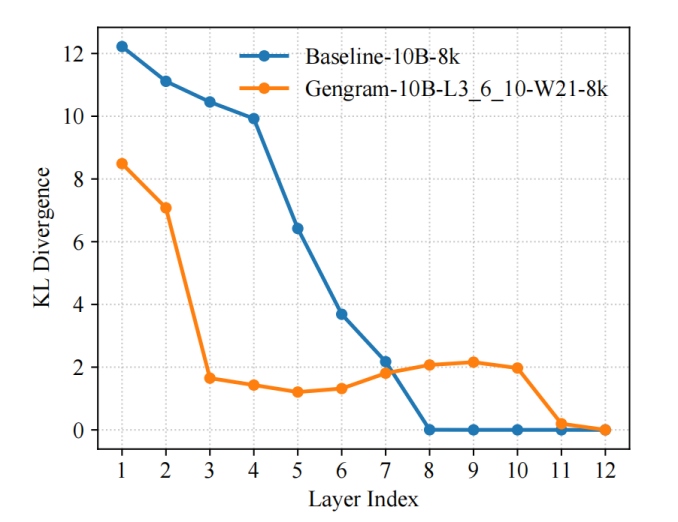
Das Team führte die KL-Divergenz als repräsentatives Diagnosekriterium für den Trainingsprozess ein und nutzte LogitLens-KL, um die „Vorhersagebereitschaft“ verschiedener Schichten zu quantifizieren und zu verfolgen. Die Ergebnisse zeigten, dass…Durch die Einführung von Gengrammen kann das Modell bereits in den flacheren Schichten eine stabile Vorhersageverteilung bilden:Im Vergleich zum Basismodell sinken die KL-Werte zwischen den Schichten schneller und erreichen früher den niedrigen Wertebereich. Dies deutet darauf hin, dass effektive Überwachungssignale früher in nutzbare Repräsentationen umgewandelt werden, wodurch Gradientenaktualisierungen direkter und Optimierungspfade glatter werden, was letztendlich zu einer schnelleren Konvergenz und einer höheren Trainingseffizienz führt.
Dieses Phänomen entstand nicht aus dem Nichts, sondern war eine direkte Folge von Gengrams Konstruktionskonzept:
Der explizite Abruf von Motivspeichern verkürzt den Weg von der Evidenz zur Repräsentation. In genomischen Aufgaben werden Überwachungssignale häufig durch kurze und spärliche Motive ausgelöst (z. B. gespleißte Konsensussequenzen, Promotor-bezogene Fragmente, Abschnitte geringer Komplexität usw.). Herkömmliche Transformer müssen diese lokalen Evidenzen schrittweise durch mehrere Aufmerksamkeits-/MLP-Schichten „ableiten und festigen“. Gengramme hingegen stellen diese informationsreichen lokalen Muster durch expliziten Zugriff auf k-Mere dem Netzwerk direkt als Speicher zur Verfügung. Dadurch muss das Modell nicht warten, bis tiefe Schichten schrittweise Motivdetektoren bilden, und befindet sich von Anfang an in einem besser vorhersagbaren Zustand.
Fensteraggregation und dynamisches Gating machen die eingebrachten Beweise „stabil und kontrollierbar“. Gengramme führen keine positionsgenaue harte Injektion durch; stattdessen aggregieren sie mehrere k-Mer-Einbettungen innerhalb eines festen Fensters.Darüber hinaus wird selektives Schreiben in den Residualstrom eingesetzt: Die Informationssuche wird in funktionalen Regionen eher aktiviert und in großen Hintergrundregionen unterdrückt. Diese Methode des Schreibens „sparsamer, ausgerichteter funktionaler Elemente“ reduziert einerseits Rauschstörungen und ermöglicht dem Netzwerk andererseits, Trainingssignale mit hohem Signal-Rausch-Verhältnis früher zu erhalten, wodurch die Optimierung vereinfacht wird.
Woher kommen Motiv-Erinnerungen? Eine detaillierte Erklärung des Schreibmechanismus von Gengram.
Das Forschungsteam beobachtete zunächst ein klares und konsistentes Phänomen bei allen Aufgaben in den nachfolgenden Auswertungen:Unter denselben Trainingsbedingungen verbesserte die Einführung von Gengrammen das Modell bei typischen motivbasierten Aufgaben signifikant, insbesondere in Szenarien mit kurzen Programmsequenzen, wie der Identifizierung von Spleißstellen und der Vorhersage epigenetischer Histonmodifikationsstellen. Beispielsweise stieg die AUC für die Spleißstellenvorhersage bei repräsentativen Aufgaben von 0,776 auf 0,901 und die AUC für die H3K36me3-Vorhersage von 0,656 auf 0,804, was stabile und deutliche Verbesserungen belegt.
Um die Frage „Woher kommen diese Verbesserungen?“ weiter zu beantworten, begnügte sich das Team nicht mit der Metrikebene, sondern extrahierte die Restschreibvorgänge des Gengramms aus der Vorwärtsausbreitung des Modells und visualisierte deren Intensitätsverteilung in der Sequenzdimension als Heatmap zur Analyse.Die Ergebnisse zeigen, dass das geschriebene Signal eine sehr spärliche und kontrastreiche Struktur aufweist: Die meisten Stellen liegen nahe der Grundlinie, und nur wenige Stellen bilden scharfe Spitzen.Wichtiger noch: Diese Peaks sind nicht zufällig, sondern signifikant angereichert und ausgerichtet auf funktionell relevante Regionen und Grenzen, darunter TATA-Box-Fragmente in der Nähe von Promotoren, Poly-T-Fragmente geringer Komplexität und Schlüsselpositionen in der Nähe der Grenzen funktioneller Regionen wie Gene/Exons.Das bedeutet, dass das Schreiben in ein Gengramm eher einem „Erfassen lokaler Hinweise auf seine entscheidende Funktion“ gleicht, als einem wahllosen Einfügen von Informationen in die gesamte Sequenz.
Ausgehend von den oben genannten Phänomenen und der Beweiskette,Forscher können Gengrams Motivgedächtnismechanismus wie folgt zusammenfassen: „Abruf auf Abruf – selektives Schreiben – strukturierte Ausrichtung“:Das Modul steuert die Intensität des Abrufs und Schreibens durch gezielte Filterung, indem es wiederverwendbare Motivinformationen in Regionen mit höherer funktionaler Informationsdichte aktiver einfügt und das Schreiben in Hintergrundregionen unterdrückt, um Störungen durch Rauschen zu reduzieren. Dadurch beruht die Beherrschung von Motiven durch das Modell nicht mehr primär auf dem „impliziten Gedächtnis“, das durch umfangreichere Datenmengen entsteht, sondern vielmehr auf der strukturierten Fähigkeit, explizit auf Repräsentationen zuzugreifen und diese interpretierbar zu schreiben.

Abschluss
In den letzten Jahren hat sich das Gebiet der Genommodellierung grundlegend gewandelt: vom „sequenzstatistischen Lernen“ hin zur „strukturorientierten Modellierung“.
Bedingte Motivspeichermechanismen, wie sie beispielsweise von Gengram demonstriert werden, eröffnen einen technischen Ansatz, der sich deutlich vom traditionellen intensiven Rechnen unterscheidet: Durch die explizite Modellierung von Multibasen-Funktionsmotiven als abrufbare strukturierte Speicher kann das Modell eine effizientere und stabilere Nutzung funktionaler Informationen erreichen und gleichzeitig die allgemeine architektonische Kompatibilität wahren.Dieser Ansatz hat nicht nur signifikante Leistungsvorteile bei verschiedenen Aufgaben der funktionellen Genomik gezeigt, sondern auch eine einheitliche technische Lösung für spärliche Berechnungen, die Modellierung langer Sequenzen und die Interpretierbarkeit der Modelle bereitgestellt.
Aus industrieller Sicht senkt das von Gengram verkörperte Paradigma „strukturierte Vorinformationen + modulare Erweiterung“ die Grenzkosten groß angelegter Genommodelle hinsichtlich Rechenleistung, Daten und Trainingszyklen erheblich. Dadurch wird der großflächige Einsatz in anspruchsvollen Anwendungsbereichen wie der Arzneimittelentwicklung, dem Variantenscreening und der Genregulationsanalyse praktisch realisierbar. Zukünftig könnten diese wiederverwendbaren und erweiterbaren Architekturkomponenten zum Standard für Genommodelle der nächsten Generation werden und die Branche von „größeren Modellen“ zu „intelligenteren Modellen“ führen. Dies beschleunigt die kontinuierliche Übertragung akademischer Forschungsergebnisse in industrielle Plattformen und klinische Anwendungen.