Command Palette
Search for a command to run...
Auf Der Grundlage Von Milliarden Von Genen Aus Einer Million Arten Haben NVIDIA Und Andere Die EDEN-Modellreihe Entwickelt Und Damit Modernste (SOTA) Fähigkeiten Zur Genom- Und Proteinvorhersage erreicht.

Das grundlegende Ziel der programmierbaren Biologie ist die rationale Gestaltung und präzise Steuerung lebender Systeme, um dadurch revolutionäre Therapien für komplexe Krankheiten zu entwickeln.Dieser Prozess wird seit langem durch die inhärente Komplexität biologischer Systeme eingeschränkt.Regulatorische Netzwerke über verschiedene Skalen hinweg, verborgene Abhängigkeiten über lange Sequenzen und die vielfältige Anpassungsfähigkeit von Organismen an Umweltveränderungen haben die traditionelle Forschung und Entwicklung nach dem Prinzip „Versuch und Irrtum“ in ein Dilemma aus Anpassungsfähigkeit, geringem Durchsatz und hohen Kosten geführt.
Letztlich reichen die Trainingsdaten, auf denen aktuelle Computermodelle basieren, hinsichtlich Umfang und Vielfalt bei Weitem nicht aus, um den enormen Gestaltungsspielraum abzubilden, den das Leben im Laufe von Milliarden von Jahren Evolution entwickelt hat. Daher fällt es diesen Modellen schwer, universelle Gestaltungsprinzipien zu erfassen.Bei innovativen Therapiekonzepten, die multimodal und skalenübergreifend sind, mangelt es stark an der Fähigkeit zur Generalisierung.
Um diese grundlegende Einschränkung zu überwinden,Basecamp Research, NVIDIA und mehrere führende akademische Einrichtungen haben gemeinsam die EDEN-Serie metagenomischer Basismodelle entwickelt.Durch die Analyse riesiger Mengen natürlicher Evolutionsdaten, die Artenübergreifend sind und Umweltinformationen berücksichtigen, wurden die tiefgreifende „Grammatik“ und die universellen Prinzipien des biologischen Designs erstmals systematisch extrahiert. Das Modell verfügt über 28 Milliarden Parameter und erzielte in zahlreichen Benchmark-Tests herausragende Ergebnisse. Sein zentraler Durchbruch liegt in seiner außergewöhnlichen Fähigkeit, artenübergreifende Sequenzen zu verstehen und zu generieren. Dadurch wird die Bioingenieurwissenschaft vom „Screening“ zu einer neuen Stufe der „vorhersagbaren Programmierung“ weiterentwickelt.
Um die Leistungsfähigkeit von EDEN als einheitliche Biodesign-Engine zu validieren, führte das Forschungsteam systematische Tests in verschiedenen Behandlungsmodalitäten durch. In der Gentherapie kann EDEN anhand eines nur 30 Basen langen Hinweises auf eine Zielsequenz aktive Rekombinasen neu entwickeln, die große Fragmente präzise in das menschliche Genom integrieren können. Im Hinblick auf das Design antimikrobieller Peptide…Die mit demselben Modell erzeugte Peptidbibliothek zeigte eine Aktivität von bis zu 97% gegen multiresistente Krankheitserreger.Es besitzt zudem eine Wirksamkeit im mikromolaren Bereich. Auf Ökosystemebene hat EDEN erfolgreich ein synthetisches Mikrobiom konstruiert, das Zehntausende künstlicher Genome, präzise Stoffwechselwege und plausible Artenbeziehungen enthält.
Die zugehörigen Forschungsergebnisse mit dem Titel „Designing AI-programmable therapeutics with the EDEN family of foundation models“ wurden als Preprint auf bioRxiv veröffentlicht.
Forschungshighlights:
* Es entwickelte ein neues Paradigma, um universelle Designprinzipien direkt aus der Evolutionsgeschichte zu lernen, und durch das Training mit BaseData, einer metagenomischen Datenbank, die die globale Biodiversität abdeckt, erreichte es herausragende Fähigkeiten im artenübergreifenden Sequenzverständnis und in der Sequenzgenerierung.
* Diese Studie bestätigt die hohe Vielseitigkeit eines einzigen fundamentalen Modells bei der Entwicklung multimodaler Therapien auf verschiedenen Skalen und zeigt, dass ein einziges Modell komplexe Designherausforderungen von Molekülen bis hin zu Ökosystemen einheitlich bewältigen kann.
* EDEN kann, ausschließlich anhand von DNA-Hinweisen, funktionelle Rekombinasen für mehrere krankheitsrelevante Stellen entwerfen und erzielt dabei eine Trefferquote von 63,21 TP3T bei untrainierten Zielen.

Papieradresse:
https://doi.org/10.64898/2026.01.12.699009
Folgen Sie unserem offiziellen WeChat-Konto und antworten Sie im Hintergrund mit „EDEN“, um das vollständige PDF zu erhalten.
Weitere Artikel zu den Grenzen der KI:
https://hyper.ai/papers
BaseData-Datensatz: Neugestaltung biologischer KI-Daten-Benchmarks mit hochwertigen langen Sequenzen.
Der in dieser Studie verwendete BaseData-Datensatz überwindet grundlegend die Beschränkungen traditioneller biologischer Datenbanken. Während traditionelle Datenbanken typischerweise auf begrenzten Referenzgenomen und fragmentierten kurzen Sequenzen basieren, zielt BaseData darauf ab, vollständige evolutionäre Signale systematisch zu erfassen und so eine Lieferkette für evolutionäre Genomdaten aufzubauen, die die globale Biodiversität abdeckt.
Der Kernwert von BaseData spiegelt sich vor allem in seiner Größe und strategischen Ausrichtung wider. Wie das folgende Diagramm zeigt,Es enthält 9,7 Billionen Nukleotidmarker für das Training und deckt mehr als 1 Million neue Arten und 100 Milliarden neue Gene ab.Noch wichtiger ist, dass die Daten nicht zufällig erhoben, sondern gezielt mit Sequenzen hoher Informationsdichte angereichert werden, wie etwa Umwelt-Metagenomik, Bakteriophagen und mobilen genetischen Elementen. Diese Daten erfassen auf natürliche Weise wichtige evolutionäre Dynamiken wie Bakteriophagen-Wirt-Interaktionen und horizontalen Gentransfer und liefern so das Kernmaterial für Modelle, um universelle Funktionsregeln über verschiedene Arten hinweg zu erlernen.
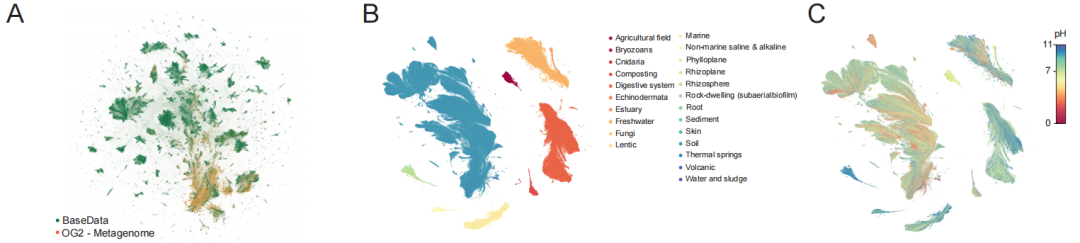
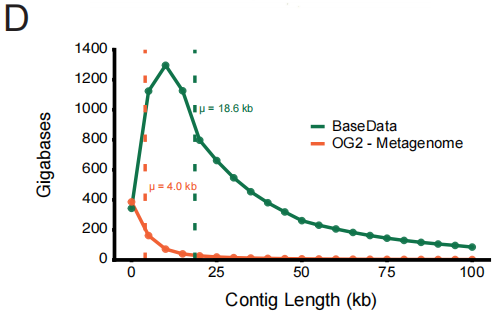
Hinsichtlich der Datenqualität erzielt BaseData eine qualitative Verbesserung, vor allem in der Vollständigkeit des Sequenzkontexts. Verglichen mit dem weit verbreiteten OpenGenome-2 (OG2) erreicht die mittlere Länge zusammenhängender Sequenzfragmente (Überlappungen) 18,6 kbp (OG2: 4,0 kbp), und jede Assemblierung enthält eine deutlich größere Anzahl an Genen. Dieser längere zusammenhängende Hintergrund ist entscheidend für das Verständnis intergenetischer Regulation und Stoffwechselwege durch das Modell.
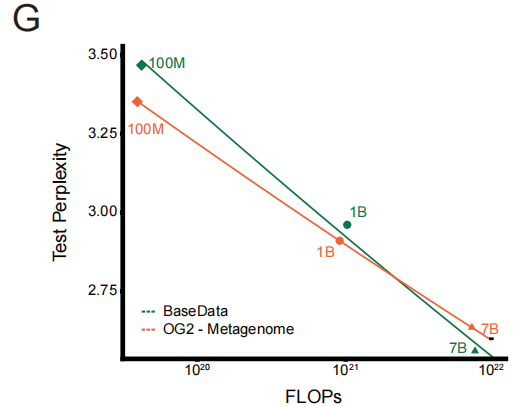
Um diesen Qualitätsvorteil zu quantifizieren, führte das Forschungsteam ein kontrolliertes Experiment durch: Es trainierte eine Reihe von Modellen mit gleich großen Datensätzen aus BaseData und OG2. Die Ergebnisse bestätigten eindeutig das „qualitätsabhängige Skalierungsgesetz“. Bei gleichem Rechenaufwand sank die Testperplexität des mit BaseData trainierten Modells schneller. Ein zentrales Ergebnis ist, dass große Modelle (z. B. mit 7 Milliarden Parametern) die Informationen langer Sequenzen aus BaseData vollständig nutzen und dadurch vergleichbare, mit OG2 trainierte Modelle übertreffen können.Dies beweist unmittelbar den entscheidenden Einfluss des langfristigen Kontextes auf die Modellleistung.
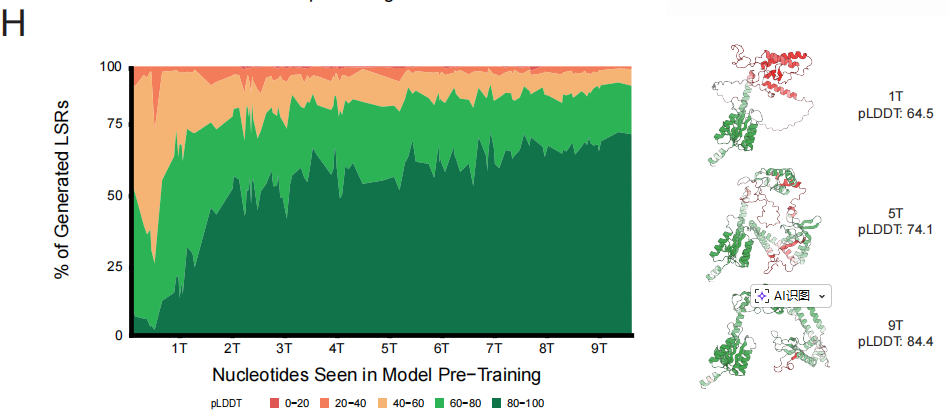
Basierend auf diesem MusterDas Forschungsteam trainierte das EDEN-28B-Modell mit 28 Milliarden Parametern unter Verwendung der vollständigen Basisdaten.Dieses Modell erzielte nicht nur die niedrigste Testperplexität, sondern seine Leistungssteigerung entsprach auch perfekt den Skalierungsvorhersagen kleinerer Modelle. Bei der Überwachung nachfolgender Aufgaben zeigte der Strukturkonfidenzindex der vom Modell während des Vortrainings generierten Proteine einen kontinuierlichen, monotonen Anstieg im Trainingsprozess. Dies belegt, dass qualitativ hochwertige Daten die Fähigkeit zur Generierung von Daten für die praktische Anwendung direkt und stetig verbessern.
Darüber hinaus wurden alle Daten durch standardisierte Rechtsvereinbarungen mit 28 Ländern und 208 Lizenzen gewonnen, wodurch ein Rahmen für Rückverfolgbarkeit und Vorteilsausgleich von der Quelle bis zur Nutzung geschaffen und die notwendigen ethischen und Governance-Standards für die groß angelegte biologische KI-Forschung festgelegt wurden.
EDEN, eine universelle biologische Design-Engine
Die EDEN-Modellfamilie wurde mit den Kernprinzipien „Skalierbarkeit, Universalität und Skalierbarkeit“ entwickelt, wobei die Modellparameter zwischen 100 Millionen und 28 Milliarden liegen.Unter ihnen verfügt EDEN-28B als zentrales Arbeitsmodell über eine Architektur und eine Trainingsstrategie, die optimal auf die einzigartigen Eigenschaften metagenomischer Daten abgestimmt sind.
Im Hinblick auf die Modellarchitektur,EDEN verwendet eine Transformer-Architektur, die ausschließlich aus Decodern besteht und durch groß angelegte Sprachmodelle validiert wurde, insbesondere basierend auf dem Designstil von Llama 3.1.Diese Wahl wird durch die überlegene Fähigkeit des Transformers ermöglicht, Langzeitabhängigkeiten zu modellieren. EDEN-28B besteht aus einem 48-schichtigen Netzwerk mit 6.144 verborgenen Schichten und 48 Aufmerksamkeitsköpfen und verwendet die SwiGLU-Aktivierungsfunktion mit RoPE-Positionskodierung. Das Modell nutzt eine Tokenisierungsmethode mit Einzelnukleotidauflösung und einem Vokabular von 512, wodurch es DNA-Sequenzen auf der grundlegendsten Ebene („Buchstaben“) verstehen und generieren kann.
Ein zentrales technologisches Highlight ist die Fähigkeit, lange Sequenzen zu generieren. Obwohl das Kontextfenster des Modells auf 8.192 Labels eingestellt ist, …Es kann stabil zusammenhängende Genomsequenzen von mehr als 13.000 Basenpaaren generieren und präzise zusammensetzen, wobei die korrekte Genreihenfolge, die Leserahmen und die Strukturen der regulatorischen Elemente erhalten bleiben.Dies deutet darauf hin, dass das Modell weit mehr lernt als nur lokale Mustererkennung; es kann eine tiefere „Grammatik“ der genomischen Organisation ableiten und anwenden, die über die Länge des physikalischen Fensters hinausgeht. Das gesamte Training wurde auf 1.008 H100-GPUs durchgeführt, wodurch durch großflächiges verteiltes Rechnen ein effizientes Lernen aus riesigen Mengen evolutionärer Daten ermöglicht wurde.
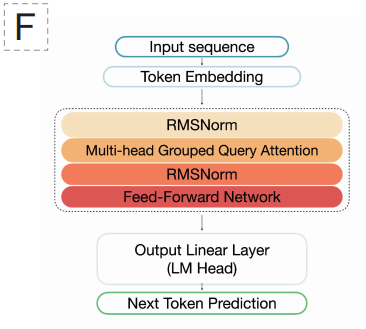
EDENs zentrale Designphilosophie folgt einem „Vortraining-Feinabstimmungs“-Paradigma. In der ersten Phase wird das Modell in großem Umfang mit Basisdaten vortrainiert, die die Evolutionsgeschichte über verschiedene Arten hinweg abdecken, wodurch allgemeine Prinzipien des biologischen Designs wie Proteinfaltung und der Aufbau von Stoffwechselwegen verinnerlicht werden.
Auf dieser soliden Grundlage können wir uns dann gezielten Aufgaben im Bereich der therapeutischen Entwicklung widmen – wie der Entwicklung von Rekombinasen, die auf spezifische DNA-Stellen abzielen, oder der Erzeugung neuartiger antimikrobieller Peptide.Mit nur wenigen, qualitativ hochwertigen, aufgabenbezogenen Daten für eine einfache Feinabstimmung kann das Modell den "Dialekt" der Aufgabe schnell beherrschen.Dieses Design ermöglicht es, dass ein einzelnes EDEN-Modell als universelle "biologische Sequenzmaschine" dient, die sich flexibel an verschiedene therapeutische Modalitäten anpasst und diese antreibt, von der Geninsertion und dem Peptiddesign bis hin zum Mikrobiom-Engineering, und damit die Vision der programmierbaren Biologie von "ein Modell, vielfältige Fähigkeiten" wahrhaft verwirklicht.
Förderung therapeutischer Innovationen von der molekularen und zellulären Ebene bis hin zur Ökosystemebene
Um die Universalität und Effektivität des EDEN-Modells bei der Gestaltung tatsächlicher Behandlungen systematisch zu überprüfen, wählte das Forschungsteam vier zentrale Richtungen aus, die sich hinsichtlich Umfang, Muster und biologischer Komplexität stark unterscheiden, um sie experimentell zu verifizieren.
Im Bereich der KI-programmierbaren Geninsertion (aiPGI) konzentrierte sich das Team auf die Überwindung des seit langem bestehenden Engpasses der „präzisen Integration großer DNA-Fragmente“.Die traditionelle CRISPR-Technologie beruht auf der Erzeugung von Doppelstrangbrüchen, und natürliche große Serin-Rekombinasen können menschliche Genomsequenzen nicht erkennen. Wie in der Abbildung unten dargestellt, besteht EDENs Lösung darin, das EDEN-LSR-Modell zu konstruieren, welches die Zuordnung von „Ziel-DNA-Sequenz → entsprechender Rekombinase“ durch Feinabstimmung der Millionen von LSR-Bindungsstellenpaarungen im Modell versteht.
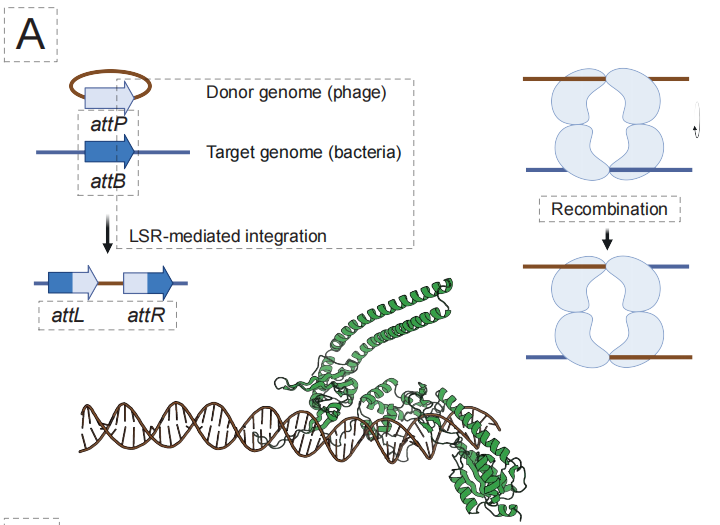
Experimentelle Ergebnisse zeigten, dass dieser Ansatz erfolgreich aktive LSRs für 10 verschiedene krankheitsrelevante Genloci und 4 potenzielle „Safe Harbor“-Loci generierte, mit einer Gesamt-Trefferrate von 53,61 TP3T. Wichtiger noch,Das 50%-Designenzym ermöglicht die Insertion von therapiebezogenen CAR-Genen in primäre menschliche T-Zellen, und einige Varianten haben Integrationseffizienzen von bis zu 40% in Zelllinien erreicht.Dies beweist sein Potenzial für die klinische Anwendung.
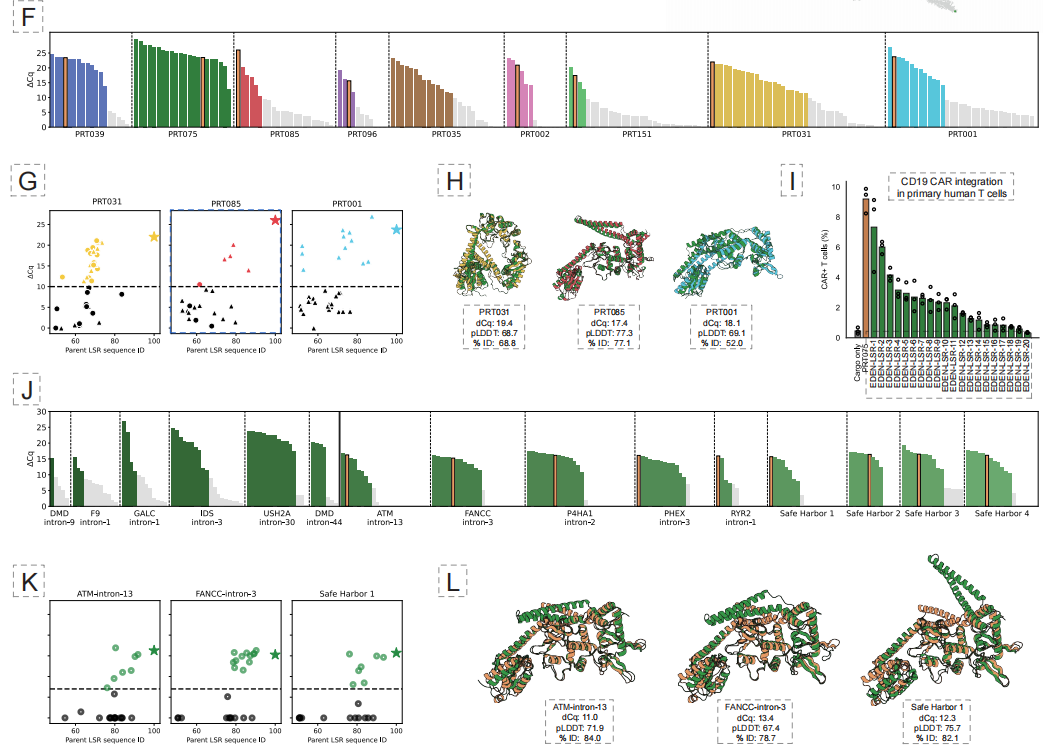
Im Bereich neuartiger Brückenrekombinasen (BRs),Die Fähigkeiten des EDEN-Modells wurden weiter zu einem besser programmierbaren Gen-Editing-System – der Brückenrekombinase – ausgebaut.Wie in der folgenden Abbildung dargestellt, hat das Team zur Optimierung des Designs das EDEN-BR-spezifische Modell erstellt, indem es das Modell anhand von Millionen von BR-haltigen genomischen Regionen feinabgestimmt hat.
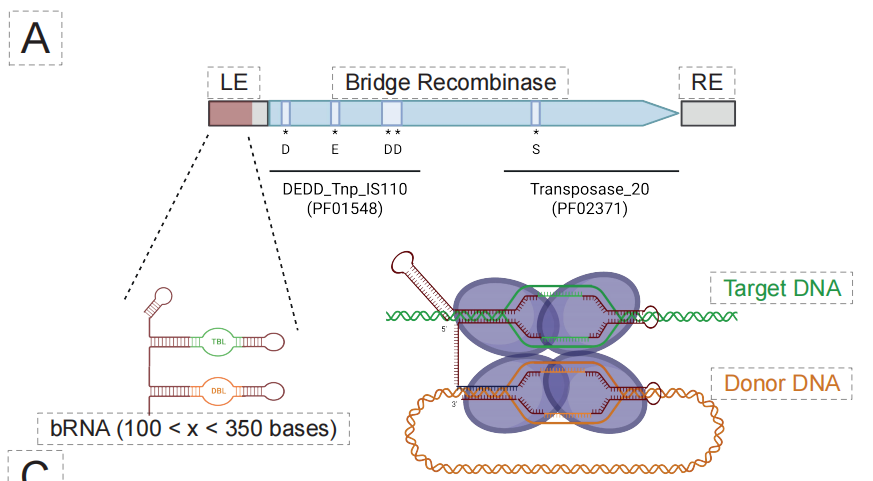
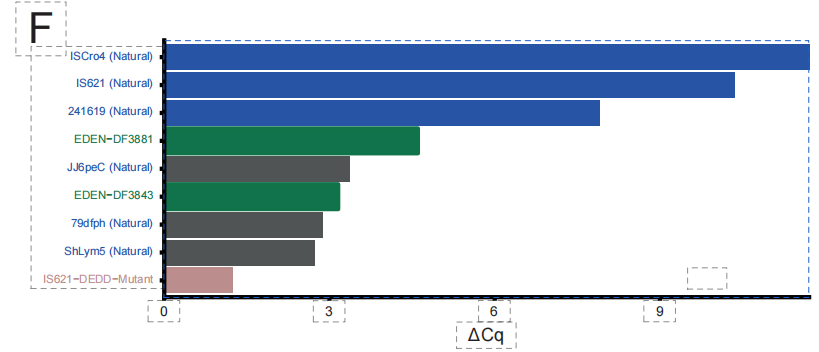
Wichtige biochemische Experimente bestätigten die Machbarkeit dieses Designprozesses. Wie in der Abbildung unten dargestellt, wiesen zwei der 49 von EDEN-BR generierten Kandidatensequenzen in ersten zellfreien Tests eine eindeutige Rekombinaseaktivität auf. Diese beiden künstlich hergestellten Proteine, DF3843 und DF3881, weisen eine maximale Ähnlichkeit von lediglich 851 TP3T bzw. 65,81 TP3T zu bekannten natürlichen BR-Sequenzen auf. Ihre Sequenzähnlichkeit zu dem gut untersuchten Referenzprotein ISCro4 ist sogar noch geringer als 351 TP3T, jedoch zeigen sie eine hohe Ähnlichkeit in ihrer dreidimensionalen Struktur.Dies beweist, dass EDEN nicht einfach nur Sequenzen nachahmt, sondern vielmehr die grundlegende Strukturlogik beherrscht, die die Funktion und Faltung von Proteinen bestimmt.
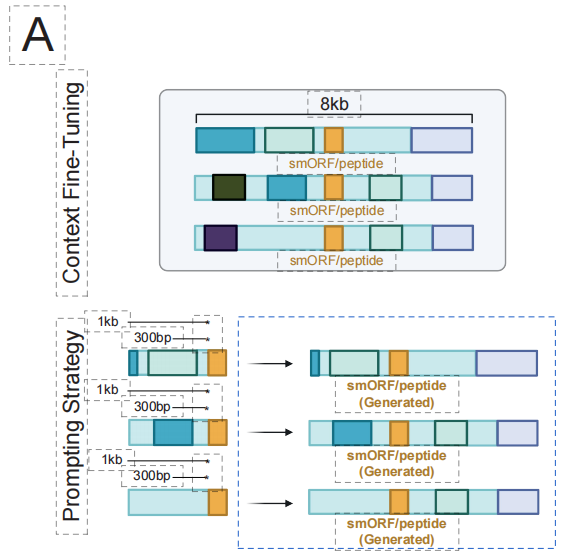
Im Bereich neuartiger antimikrobieller Peptide (AMPs) bestätigte das Forschungsteam die Fähigkeit von EDEN, neuartige antimikrobielle Peptide zu entwickeln. Wie in der folgenden Abbildung dargestellt,Durch den Einsatz einer Feinabstimmungsstrategie, die genomische Kontextinformationen einbezieht, ist das Modell in der Lage, neuartige antimikrobielle Peptidsequenzen zu generieren.
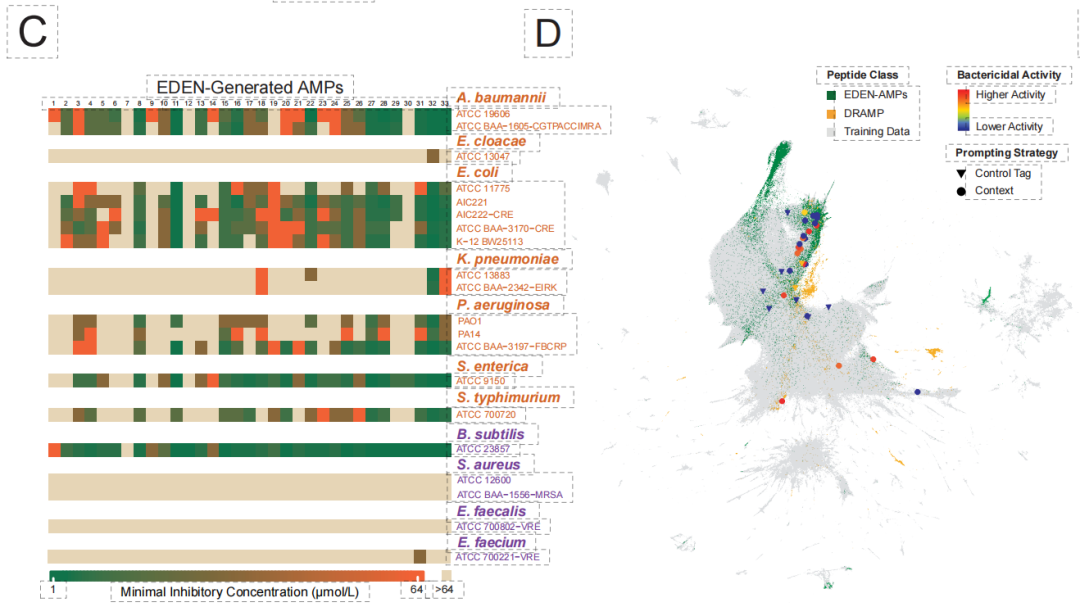
Die experimentelle Überprüfung hat bahnbrechende Ergebnisse geliefert. Wie in der folgenden Abbildung dargestellt,In einer AMP-Bibliothek, die aus 33 generierenden Peptiden besteht, zeigten bis zu 971 TP3T-Sequenzen antibakterielle Aktivität.Die vielversprechendsten Kandidaten erreichten mikromolare Hemmkonzentrationen gegen multiresistente gramnegative Bakterien (wie Acinetobacter baumannii) und demonstrierten damit ein starkes Penetrationsvermögen in die äußere Membran. Die generierten Sequenzen wiesen generell eine geringe Ähnlichkeit zu bekannten Datenbanken auf, was bestätigt, dass das Modell traditionelle Homologiebeschränkungen überwinden und ein echtes „De-novo-Design“ ermöglichen kann.
Schließlich stellte die Forschung auf der komplexesten Ebene des Ökosystems die Entwicklung „synthetischer Mikrobiome“ in Frage. Traditionelle Methoden haben Schwierigkeiten, metabolische Interaktionen und das ökologische Gleichgewicht zwischen mehreren Arten zu koordinieren. Wie die Abbildung unten zeigt, wurde EDEN nach der Feinabstimmung mithilfe von Mikrobiomdaten des Verdauungssystems optimiert.Ausschließlich auf der Grundlage von Funktionsgenen oder Nischenhinweisen wurde erfolgreich eine synthetische Metagenomik mit über 90.000 Arten und einem Umfang von Gigabasen generiert.
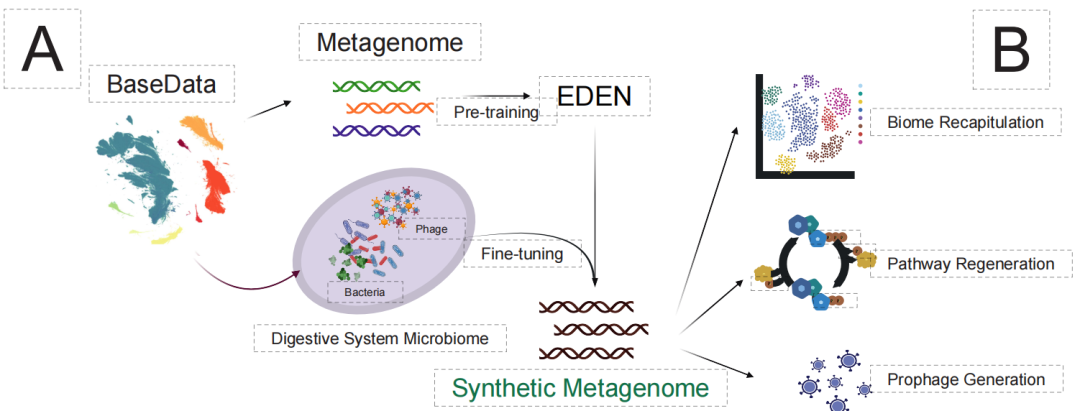
Die generierten Ergebnisse weisen einen hohen Grad an ökologischem Realismus auf:Die Spezies 99% wurde korrekt in die mit dem Verdauungssystem verbundene Biota eingeordnet, und ihre speziesübergreifenden Stoffwechselwege blieben vollständig erhalten.Darüber hinaus kann das Modell sogar präzise Prophagenstrukturen generieren, die in das Wirtsgenom integriert sind, was beweist, dass es die komplexe Interaktionslogik zwischen Wirt und Virus erfasst hat.
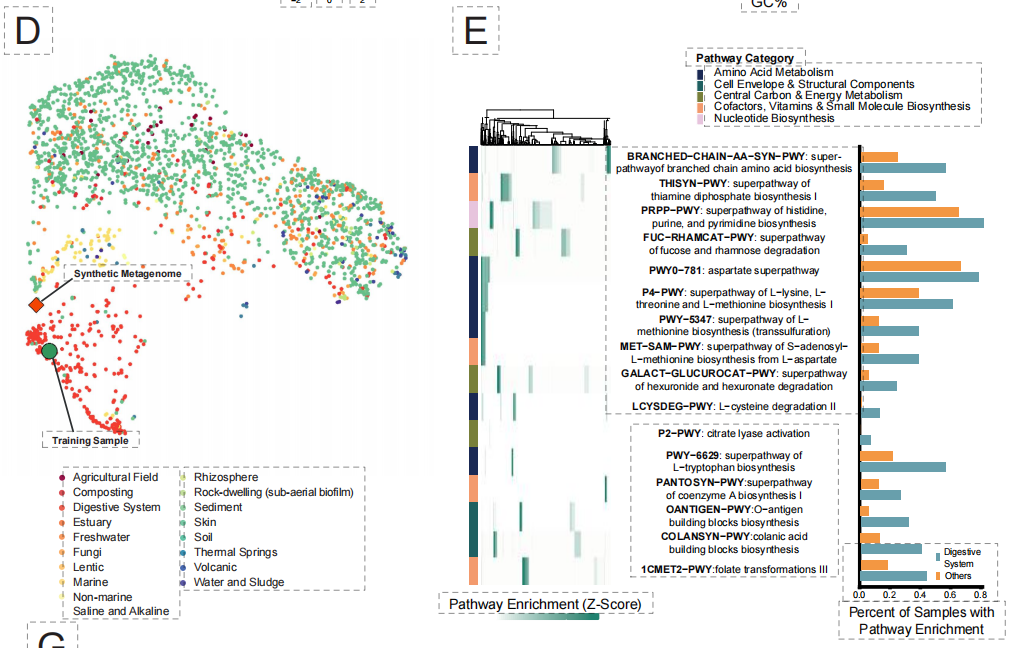
Diese vier skalenübergreifenden Experimente zeigen gemeinsam, dass das mit einheitlichen Evolutionsdaten vortrainierte EDEN-Modell als allgemeine biologische Design-Engine dienen kann. Es ermöglicht die schnelle und zuverlässige Entwicklung therapeutischer Innovationen auf molekularer, zellulärer und ökologischer Ebene mit minimaler aufgabenspezifischer Datenführung und legt damit eine solide praktische Grundlage für die programmierbare Biologie.
Innovation durch die Integration von KI und synthetischer Biologie
In den letzten Jahren haben sich die Integration und Innovation von Wissenschaft und Industrie auf dem Gebiet der programmierbaren Biologie deutlich beschleunigt, und eine Reihe bedeutender Fortschritte definiert die Grenzen des Biodesigns neu.
Führende akademische Einrichtungen weltweit übertragen Erkenntnisse der Evolution in computergestützte Modelle von beispielloser Skalierbarkeit und Präzision. So veröffentlichte beispielsweise Anfang 2024 ein gemeinsames Team von DeepMind, Isomorphic Labs und mehreren Universitäten das AlphaFold-3-Modell. Dieses Modell kann gleichzeitig Proteinstrukturen und -interaktionen vorhersagen sowie neue Proteine mit spezifischen Funktionen generieren. Es ist das erste Modell, das das komplexe Zusammenspiel von Biomolekülen in einen einheitlichen Rahmen für hochpräzise Simulationen integriert.Das Magazin Nature feierte es als „einen Quantensprung bei der Erforschung der inneren Abläufe der molekularen Maschinerie des Lebens“.
Die Branche beschleunigt die Umsetzung dieser bahnbrechenden Innovationen in Plattformen und Therapien. Im Bereich der KI-gestützten Arzneimittelentwicklung haben NVIDIA und Recursion Pharmaceuticals BioNeMo veröffentlicht, eine KI-Modellbibliothek für die Biochemie, die die Wirkstoffforschung von der Suche nach der Nadel im Heuhaufen hin zu einem zielgerichteten Ansatz verändern soll. Das Unternehmen für synthetische Biologie, Ginkgo Bioworks, nutzt seine automatisierte Plattform, um mikrobielle Gemeinschaften systematisch für die Kohlenstoffabscheidung und die chemische Produktion zu entwickeln und so die Entwicklung synthetischer Ökosysteme voranzutreiben.
Diese neue, daten- und algorithmengetriebene Welle transformiert die Biologie von einer beobachtenden und beschreibenden Wissenschaft hin zu einer programmierbaren, fehleranalysierbaren und vorhersagbaren Ingenieurdisziplin. Das bedeutet nicht nur, dass wir Lebensprozesse präziser steuern können, um Krankheiten zu besiegen, sondern lässt auch unsere Fähigkeit erahnen, biologische Systeme systematisch zu entwickeln, um globale Herausforderungen in den Bereichen Ressourcen, Umwelt und Gesundheit zu bewältigen.
Referenzlinks:
1.https://nvidianews.nvidia.com/news/nvidia-announces-broad-expansion-of-its-biomedicine-platform
2.https://www.ginkgobioworks.com/2024/01/04/ginkgo-bioworks-and-pfizer-expand-collaboration-to-advance-rna-based-therapeutics/








