Command Palette
Search for a command to run...
モデルパラメータは RFdiffusion を 5 倍超えています。 NVIDIA などが SOTA パフォーマンスを考慮してゼロから設計されたタンパク質バックボーン Proteina をリリース

前世紀以来、科学者たちは、アミノ酸を使って新しいタンパク質を作り、生命の設計図を構築するというビジョンを掲げ、アミノ酸配列に基づいてタンパク質の構造を予測する研究に専念してきました。しかし、この壮大なミッションは長い時間をかけてゆっくりと進められ、近年の AI 技術の急速な発展によってようやく大きな勢いが注入され、開発の急速な軌道に乗ることができました。
2016年以来、Molecular Heartの創設者兼主任科学者であるXu Jinbo氏らが始めた技術革命が、この分野に静かに変化をもたらしている。彼らは、深層残差ネットワーク ResNet アーキテクチャを構造予測の分野に導入した先駆者です。タンパク質残留物の接触予測の大幅な改善に成功しました。この画期的な進歩により、AIとタンパク質設計の深い統合のための強固な基盤が築かれました。それ以来、多くの科学研究チームがこれに倣い、この分野で懸命に研究を重ねてきました。共進化とディープラーニングを組み合わせたアルゴリズムが数多く登場しており、その中でも2024年のノーベル化学賞受賞者であるデイビッド・ベイカー氏とAlphaFoldによる一連の大きな成果はさらに有名になり、この分野の研究を新たな高みへと押し上げています。
しかし、これまでの研究を振り返ると、無条件タンパク質構造生成モデルは、構造の数が 500,000 を超えない小規模データセットでのみトレーニングされることがよくあります。さらに、合成プロセス中、これらのモデルのニューラル ネットワークには効果的な制御方法が欠けており、自然言語、画像、ビデオ生成などの分野の生成モデルと比較して、規模とパフォーマンスの両方で大きなギャップがあります。
自然言語、画像、ビデオ生成の分野では、スケーラブルなニューラル ネットワーク アーキテクチャ、大規模なトレーニング データセット、細かいセマンティック制御によってもたらされた、世界を揺るがすような変化と大きな進歩を人々は目撃してきました。これにより、研究者たちは深く考えるようになりました。これらの分野での成功体験から学び、タンパク質構造の拡散と流動モデルに同様の拡張と制御を加えることで、タンパク質設計の分野で質的な飛躍を達成できるだろうか?
喜ばしいことに、NVIDIA は最近、Mila、ケベック人工知能研究所、モントリオール大学、マサチューセッツ工科大学と連携し、新しいタイプの大規模フロー タンパク質バックボーン ジェネレーターである Proteina を開発しました。 Proteina は RFdiffusion モデルの 5 倍のパラメータ数を持ち、トレーニング データを 2,100 万の合成タンパク質構造に拡張します。当社は、de novo タンパク質バックボーン設計において最先端のパフォーマンスを達成し、最大 800 残基というこれまでにない長さの多様で設計可能なタンパク質を生成します。
「Proteina: フローベースのタンパク質構造生成モデルのスケーリング」と題された関連研究成果が、ICLR 2025 Oral に選出されました。

用紙のアドレス:
https://openreview.net/forum?id=TVQLu34bdw&nesting=2&sort=date-desc
学術共有イベントを推奨します。最新の Meet AI4S ライブ ブロードキャスト招待は、3 月 7 日の正午です。華中科技大学の黄紅准教授、上海人工知能研究所AI科学センターの若手研究員周東展氏、上海交通大学自然科学研究所の助手研究員周秉馨氏、個人の業績を紹介し、科学研究の経験を共有します。
AIがタンパク質設計を強化: 構造から配列、予測から設計まで
生命科学の研究過程において、タンパク質設計は常に極めて重要な位置を占めてきました。膨大な量のタンパク質配列データからルールやパターンを学習することは、長い間、科学研究者が直面する悩みの種でした。幸いなことに、AI技術の支援により、この分野は好転の先頭に立っています。
例えば、DeepMind がリリースした AlphaFold3 は、DNA、RNA、小分子の相互作用のモデリングを改善します。タンパク質複合体の構造を正確に予測する能力は、細胞内のタンパク質の複雑な相互作用を理解するための強力なサポートとなります。 Meta はかつて、言語モデルと構造予測を組み合わせた ESMFold をリリースしました。これにより予測速度が大幅に向上し、研究者はタンパク質構造情報をより効率的に取得できるようになります。Microsoft の最新の BioEMU-1 は、タンパク質構造の動的な変化をシミュレートします。これにより、タンパク質の運動メカニズムの詳細な研究と薬剤設計への新たな道が開かれました。
これらの基盤により、AI は徐々にタンパク質構造設計に浸透し始めています。
タンパク質構造の設計は主に既知のタンパク質構造に基づいており、さまざまな方法を通じて変更および最適化され、特定の機能または特性を持つタンパク質が得られます。タンパク質の機能は主にその3次元構造によって決定されるため、構造分布を直接モデル化する手法が徐々に主流となり、その中でも拡散モデルやフローモデルに基づくアルゴリズムが特に優れています。たとえば、Generate Bio が開発した Chroma モデルは、正確なタンパク質設計のための拡散モデリングの最初の大規模なアプリケーションです。「自然界には全く存在しないタンパク質」を生成する能力。
また、David Baker が提案した RFdiffusion は、RoseTTAFold 構造予測ネットワークを微調整することで、特定の機能を持つタンパク質骨格を生成できます。機能性タンパク質の設計のための正確な構造的基礎を提供します。コロンビア大学とラトガース大学の研究者によって提案された Genie2 は、トレーニング データを AFDB に拡張します。複数の独立した機能部位を持つ複雑なタンパク質を生成できます。
タンパク質の構造と配列は相互に関連しており、構造が機能を決定し、配列が構造の基礎となることはよく知られています。 AI技術によってタンパク質の構造が変化すると、必然的にタンパク質の配列も変化します。タンパク質配列設計は、主に既知のタンパク質構造に基づいて、計算と予測の方法を通じて、その構造に一致するアミノ酸配列を設計します。
現在、AI タンパク質配列設計は主に 2 つのタイプに分かれています。1つは固定バックボーンタンパク質配列設計ツールであり、例えば、スタンフォード大学が立ち上げたESM-IFは、事前トレーニングと微調整を組み合わせたパラダイムを採用し、構造知識を機能性タンパク質設計に巧みに統合し、特定の機能を持つタンパク質の設計を強力にサポートします。 David Baker が提案した ProteinMPNN はグラフニューラルネットワークに基づいており、主鎖構造に従って一致するアミノ酸配列を生成できるため、タンパク質配列設計のための効率的で正確な方法を提供します。
もう 1 つは、機能指向のタンパク質配列設計ツールです。たとえば、Salesforce がリリースした ProGen は、特定の機能要件に応じてタンパク質配列をカスタマイズできる条件付き生成モデルであり、機能性タンパク質の設計に非常に柔軟なソリューションを提供します。スペインのジローナ大学が立ち上げた ZymCTRL は、事前トレーニング済みの言語モデルを微調整することで機能指向の設計を実現し、タンパク質機能の正確な制御を強力にサポートします。中国科学院天津工業バイオテクノロジー研究所が提案したP450Diffusionは、拡散モデルに基づいて特定の触媒機能を備えたP450酵素変異体を生成し、酵素工学の分野に新たな発展の機会をもたらします。
※詳細レポートはこちら:触媒能力が3.5倍に向上!中国科学院の研究チームは、拡散モデルP450Diffusionに基づいてP450酵素の新規設計法を開発した。
しかし、他の 3 種類のタンパク質モデルと比較すると、現在のタンパク質構造設計モデルの規模は一般的に小さいです。具体的には、AlphaFold 3 のトレーニング セット サイズは 1 億に近く、BioEmu-1 は事前トレーニング段階で AFDB データベースから 2 億を超えるタンパク質配列を使用し、ProGen のパラメーターの数は 12 億に上ります。しかし、タンパク質構造設計の分野で優れた代表的存在である RFdiffusion は、Protein Data Bank (PDB) リポジトリ内の数万の実際のタンパク質構造からのトレーニング データのみを使用しており、生成できる構造の全長は 600 アミノ酸残基にしか達しません。 Genie2 の最大のデータセットは、合成構造タンパク質約 600,000 個のみです。
この文脈では、業界では、より大きなトレーニングデータ量、より長い総構造長、より強力な制御性を備えたタンパク質構造設計モデル「Proteina」の誕生を心待ちにしています。
Proteinaモデル: AI技術を用いたタンパク質設計の新たなブレークスルー
フローベースのタンパク質構造モデルである Proteina は、視野内の拡散トランスフォーマーにヒントを得た、革新的でスケーラブルな非等価トランスフォーマー アーキテクチャを使用しています。計算コストの高い三角形レイヤーに頼らなくても、最高のパフォーマンスを実現できます。これにより、Proteina は最大 2,100 万個のタンパク質構造でトレーニングできるようになり、トレーニング データが 35 倍増加し、最終的に最大 800 残基のバックボーンを生成できるようになります。デザイン性と多様性を維持しながら、これまでのすべての作品よりも大幅に優れています。
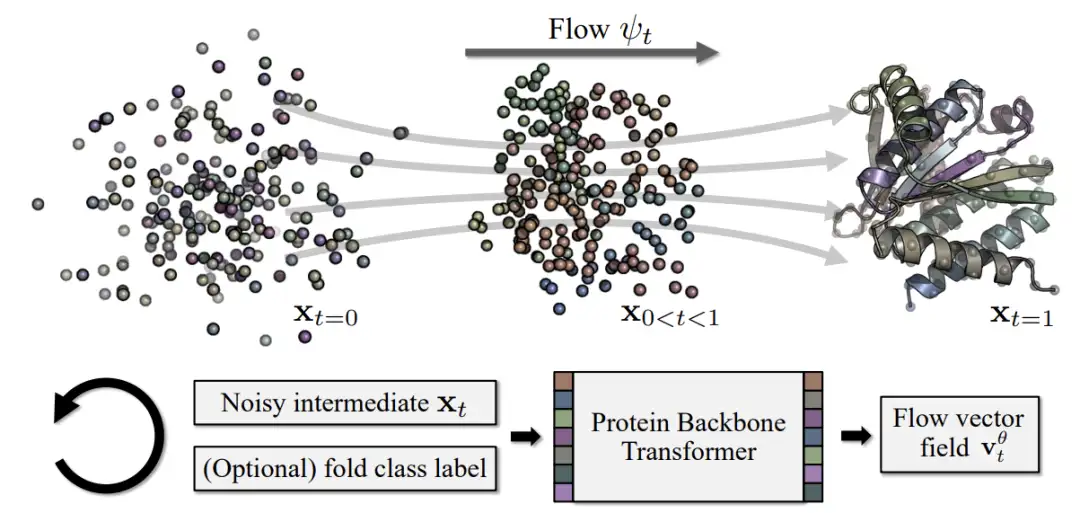
下の図に示すように、この研究では主に Genie2 で使用される Foldseek AFDB クラスタリング DFS データセットを使用します。データセットは約 600,000 個の合成構造タンパク質をカバーしています。同時に、この研究では約 2 億 1,400 万の AFDB 構造からフィルタリングされた高品質の AFDB サブセット D21M も使用しました。このサブセットには、約 2,100 万個の合成構造タンパク質が含まれています。
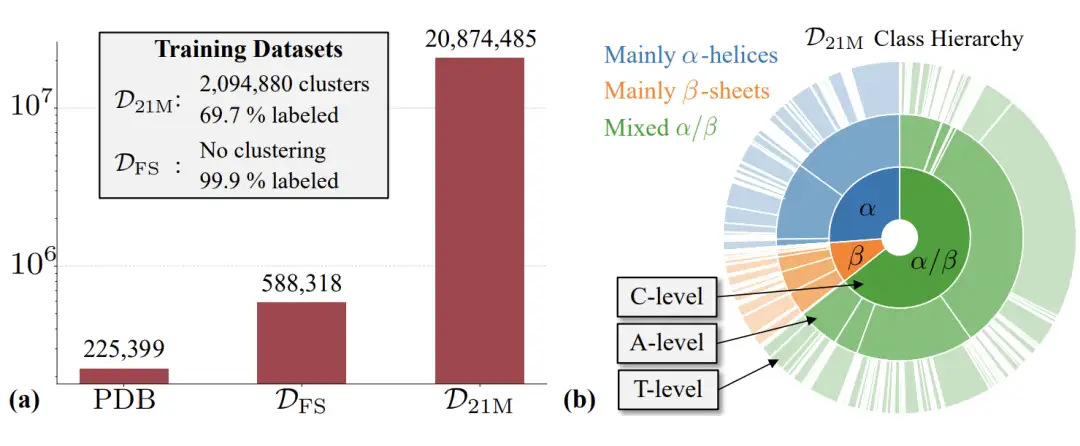
研究者らは、上記の 2 つのデータセットに基づいて、さらに 3 つの Proteina モデルをトレーニングしました。1 つ目は MFS モデルで、2 億のパラメータを持つ Transformer と 1,000 万のパラメータを持つ三角形のレイヤーが含まれています。2 つ目は Mno-triFS モデルで、2 億のパラメータを持つ Transformer のみを含み、三角形のレイヤーやペアワイズ表現の更新は含まれていません。3 つ目は M21M モデルで、4 億のパラメータを持つ Transformer と 1,500 万のパラメータを持つ三角形のレイヤーが含まれています。
無条件タンパク質構造生成の分野では、長い間、アイソバリアント法が主流でしたが、Proteina は大規模な非アイソバリアントフローモデルも成功できることを実証しています。トレーニングバージョンのパラメータは4億を超えます。RFdiffusion の 5 倍以上の大きさで、現在最大のタンパク質バックボーンジェネレータです。結果はまた、DFS でトレーニングされたモデルはより高い多様性を示すが、研究者は DFS よりも完全に合成された構造からはるかに大量の高品質データを作成することもできることを示しています。
評価指標に関して、Proteina は従来の多様性、新規性、設計可能性の評価に満足せず、DFS の実証ラベルをモデルに直接入力するという革新的な評価指標を導入しています。この動きにより、さまざまな折り畳み構造間の多様性が強化され、新しい折り畳みクラスの制約を通じて合成タンパク質構造に対する前例のない制御が可能になります。
下の図に示すように、無条件生成と比較すると、Proteina の条件付きモデルは、最高の FPSD、fS、fJSD スコアを達成しながら、最も高度な TM スコア多様性を実現します。これにより、折り畳み構造の多様性「fS」における利点と、生成された構造と参照データ間の分布の一致が改善されることが十分に実証されます。
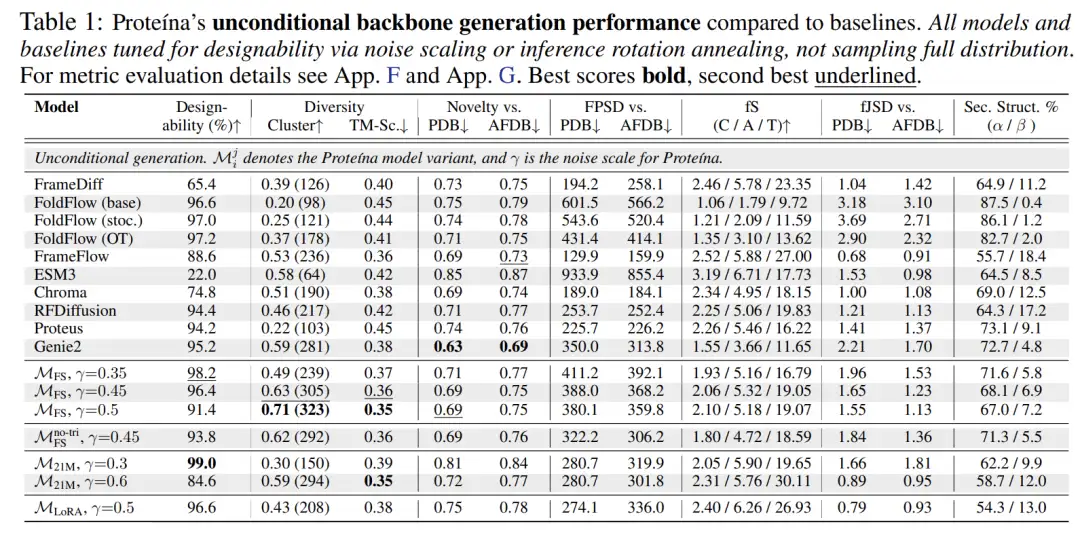
さらに、Proteina はフロー マッチングの目的をタンパク質構造の生成に適応させ、LoRA を使用してモデルを微調整し、自然で設計可能なタンパク質を生成できるようにするなど、段階的なトレーニング戦略を検討しました。また、階層的なフォールディング カテゴリの条件付き制約に対する新しいガイダンス スキームを開発し、タンパク質の設計可能性を高めるための自己ガイダンスを実証することに成功しました。タンパク質バックボーン生成性能の面では、Proteina は特に長鎖合成において SOTA レベルに達しています。このモデルは、すべてのベースライン モデルを大幅に上回り、新しい折りたたみカテゴリ条件制約により、以前のモデルよりも優れた制御機能を発揮します。
中国のAIタンパク質設計分野で出現するイノベーション
現在、DeepSeekが再びビッグ言語モデルに火をつけているため、タンパク質設計の分野は間違いなく新たな発展の機会を迎え、ますます多くの中国勢力が出現するだろう。実際、現時点では、中国の研究者や企業は、タンパク質構造設計の分野だけでも多くの成果を上げています。
2022年、AI を活用した上海天朗 XLab が、新しいタンパク質設計プラットフォーム TRDesign を立ち上げました。 TRDesign は、タンパク質の配列と構造の関係について多くのことを学習することで、タンパク質フォールディング空間におけるすべての潜在的な可能性を正確に探索し、タンパク質フォールディングから学習した配列-構造-機能の関連性を逆にマッピングし、エンドツーエンドのタンパク質設計、テスト、安定性、親和性の最適化をゼロから実行して、需要をよりよく満たすタンパク質構造を設計できます。
2023年分子心創始者の徐金波教授が2023年世界人工知能大会「WAIC」でNewOriginビッグモデルを発表した。このモデルは、数兆個のマルチモーダルビッグデータから学習することで、マルチモーダル指向生成を実現できます。単一のモデルで、配列生成、構造予測、機能予測、de novo設計など、タンパク質生成の全プロセス要件を満たし、産業用途に必要な特定の機能タンパク質を生成する問題を解決し、実際の産業環境での効果と価値を評価できます。
2024年4月、無錫土神志和人工知能技術有限公司はいくつかの研究機関と協力し、中国初の自然言語テキスト-タンパク質大規模モデルTourSynbioを共同リリース。 TourSynbio の大規模モデルは、タンパク質設計プロセスを開放し、「タンパク質設計 AI を 1 つに」実現します。あらゆるタンパク質の詳細な表現を提供し、自然言語の対話とプロンプトをサポートし、タンパク質設計プロセスを大幅に簡素化します。
2024年8月CarbonNovo を提案したのは、中国科学院計算技術研究所の張海滄氏のチームです。この結果はICML2024に掲載されました。 CarbonNovo は、エンドツーエンド方式でタンパク質バックボーンの構造と配列を共同で設計します。ジョイントエネルギーモデルを確立し、タンパク質言語モデルを導入することで、設計効率とパフォーマンスが効果的に向上し、既存の 2 段階設計モデルに比べて大きな利点が示されます。
論文リンク:
https://openreview.net/pdf?id=FSxTEvuFa7 コードリンク:
https://github.com/zhanghaicang/carbonmatrix_public
2024年10月、USTC生命科学医学部の劉海燕教授と陳泉教授のチームが、事前にトレーニングされた構造予測ネットワークに依存しない、タンパク質バックボーンのノイズ除去拡散確率モデル SCUBA-D を開発しました。主鎖構造をゼロから自動設計し、新しい構造と配列を持つ人工タンパク質をゼロから設計できる完全なツールチェーンを形成します。RosettaDesign 以外で完全に実験的に検証された唯一のタンパク質 de novo 設計方法です。関連する結果はNature Methodsに掲載されました。
論文リンク:
https://doi.org/10.1038/s41592-024-02437-W
2025年、ウェストレイク大学のルー・ペイロンのチームは、ディープラーニングとエネルギーベースの手法を組み合わせて、蛍光リガンドに特異的に結合できる膜貫通型蛍光活性化タンパク質 tmFAP の設計に成功しました。ディープラーニングアルゴリズムは、膜貫通タンパク質設計における中核的な問題を解決するために使用されました。膜貫通タンパク質と膜内のリガンド分子間の非共有結合相互作用の正確な新規設計が初めて達成され、生細胞内での蛍光活性化能力が実証され、膜貫通タンパク質の設計と応用に新たな道が開かれました。この研究は、世界トップクラスの学術誌「ネイチャー」に掲載されました。
論文リンク:
https://www.nature.com/articles/s41586-025-08598-8
現在、中国はAI駆動型タンパク質設計の分野で独自の技術エコシステムを形成しており、その画期的な進歩はアルゴリズムの革新レベルだけでなく、基礎理論から産業応用までの完全なイノベーションチェーンの構築にも反映されています。これらの成果の出現は、タンパク質設計の分野における中国の技術的進歩の深さと幅広さを十分に証明しています。 AI技術の継続的な発展により、将来的にはさらに目覚ましい成果が生まれ、世界の生命科学研究とバイオ医薬品産業の発展にパラダイムシフトをもたらすと信じています。








