Command Palette
Search for a command to run...
タンパク質構造の不均一性という原子レベルのモデリング課題を解決する!David BakerチームによるPLACERフレームワーク解析

分子の世界では、タンパク質と核酸、小さな有機・無機分子、そして金属イオン間の相互作用は生命機能にとって極めて重要です。これらの相互作用の認識と結合は、生物学的機能に影響を与え、薬効を決定し、さらには新規酵素設計の成否を左右する可能性があります。しかしながら、これらの相互作用とその構造的不均一性を原子レベルでモデリングすることは、産業界にとって依然として極めて困難な課題です。
DiffDock などのディープラーニング (DL) ベースの小分子ドッキング ツールは、以前の方法に比べて精度が向上しています。ただし、高精度のタスクではパフォーマンスの違いはそれほど大きくありません。さらに、目に見えない受容体に遭遇するとパフォーマンスが大幅に低下します。また、化学構造から小分子の立体配座を生成するためのさまざまなディープラーニングベースの手法が開発されています。ただし、これらの方法では通常、相互作用するオブジェクトの特定のカテゴリのみをモデル化するため、タンパク質機能の全範囲を特徴付ける能力が制限されます。
これを基に、ワシントン大学のノーベル賞受賞者デビッド・ベイカー教授率いる研究チームは、PLACER(Protein-Ligand Atomistic Conformational Ensemble Resolver)と呼ばれるグラフニューラルネットワークを開発しました。小分子の原子構成と結合情報に基づいて、さまざまな有機小分子の構造を正確に生成できます。また、タンパク質のマクロな構造環境が与えられれば、タンパク質と小分子のドッキングタスクのために小分子とタンパク質側鎖の詳細な構造を構築できます。酵素設計研究において、本研究チームは、PLACERを用いて設計された活性部位の精度と事前組織化の程度を評価することで、設計成功率と酵素活性を大幅に向上できることを発見しました。例えば、研究者らは、kcat/KM = 11,000 M⁻¹·min⁻¹という事前組織化された抗アルドラーゼを得ました。これは、ディープラーニングの登場以前のすべての設計結果をはるかに上回ります。
「PLACER によるタンパク質-小分子立体配座アンサンブルのモデリング」と題された関連研究成果が、米国科学アカデミー紀要 (PNAS) に掲載されました。
研究のハイライト:
* PLACER は高速かつランダムであるため、構造異質性の分布を描写するための予測サンプルを大量に迅速に生成できます。
* すべての相互作用に対して統一された原子レベルの表現を使用することで、PLACER は大環状分子やその他の複雑な小分子などの生体分子以外にも簡単に拡張できます。
* PLACER は計算による酵素設計や小分子複合体設計に非常に価値があり、設計された活性部位の再構築の精度や、主要な触媒/相互作用側鎖官能基の事前構成を迅速に評価できます。

用紙のアドレス:
https://www.biorxiv.org/content/10.1101/2024.09.25.614868v2
完全な PDF を取得するには、当社の公式 WeChat アカウントをフォローし、バックグラウンドで「enzyme design」と返信してください。
データセット: 多階層かつ多様なデータ構成により、優れた一般化能力が実証されています。
低分子のコンフォメーション予測のために、研究チームはケンブリッジ構造データベース(CSD)から226,000件を超える有機非ポリマー低分子結晶構造をトレーニングセットとして選択し、7,116個のサンプルを検証セットとして選択しました。各分子は完全な原子組成と化学結合情報を提供し、原子座標はランダムに初期化されるため、モデルはノイズの多い条件下でも正確な構造を復元する能力を学習できます。このトレーニング戦略により、モデルがさまざまな立体配座にある小さな分子の微妙な変化を捉えられるようになるだけでなく、複数回の実行を通じて多様な分子立体配座のセットを生成することも可能になります。
タンパク質-小分子システムに関しては、研究チームはタンパク質データバンク(PDB)からタンパク質-小分子複合体を含む高解像度(<2.5Å)構造を選択し、合計約113,000のトレーニングサンプルと7,090の検証サンプルを使用しました。特に、研究チームは水分子のみを除外しましたが、潜在的に非生物学的な小分子(溶媒など)に関する情報は、分子界面の物理化学的嗜好に関する貴重な手がかりとなるため、保持しました。トレーニング データは、最大 600 個の重原子を含むように切り取られ、ランダムに選択された原子中心の周囲にガウス ノイズが加えられ、現実のタンパク質や小分子の複雑な動的環境をシミュレートします。
この多層的で多様なデータ構造により、PLACER は単一の小分子から複雑なタンパク質小分子システムまであらゆるものを処理する際に優れた一般化能力を発揮します。
ニューラル ネットワーク PLACER は、原子レベルの側鎖と小分子の立体配座に重点を置いた 3 トラック アーキテクチャを採用しています。
PLACERは、部分的に摂動を受けたタンパク質構造と、相互作用する分子の化学構造情報(座標を除く)を入力とするノイズ除去ニューラルネットワークです。出力は、複合体の完全な原子構造と、予測モデルにおける各原子の位置の不確実性です(下図A参照)。
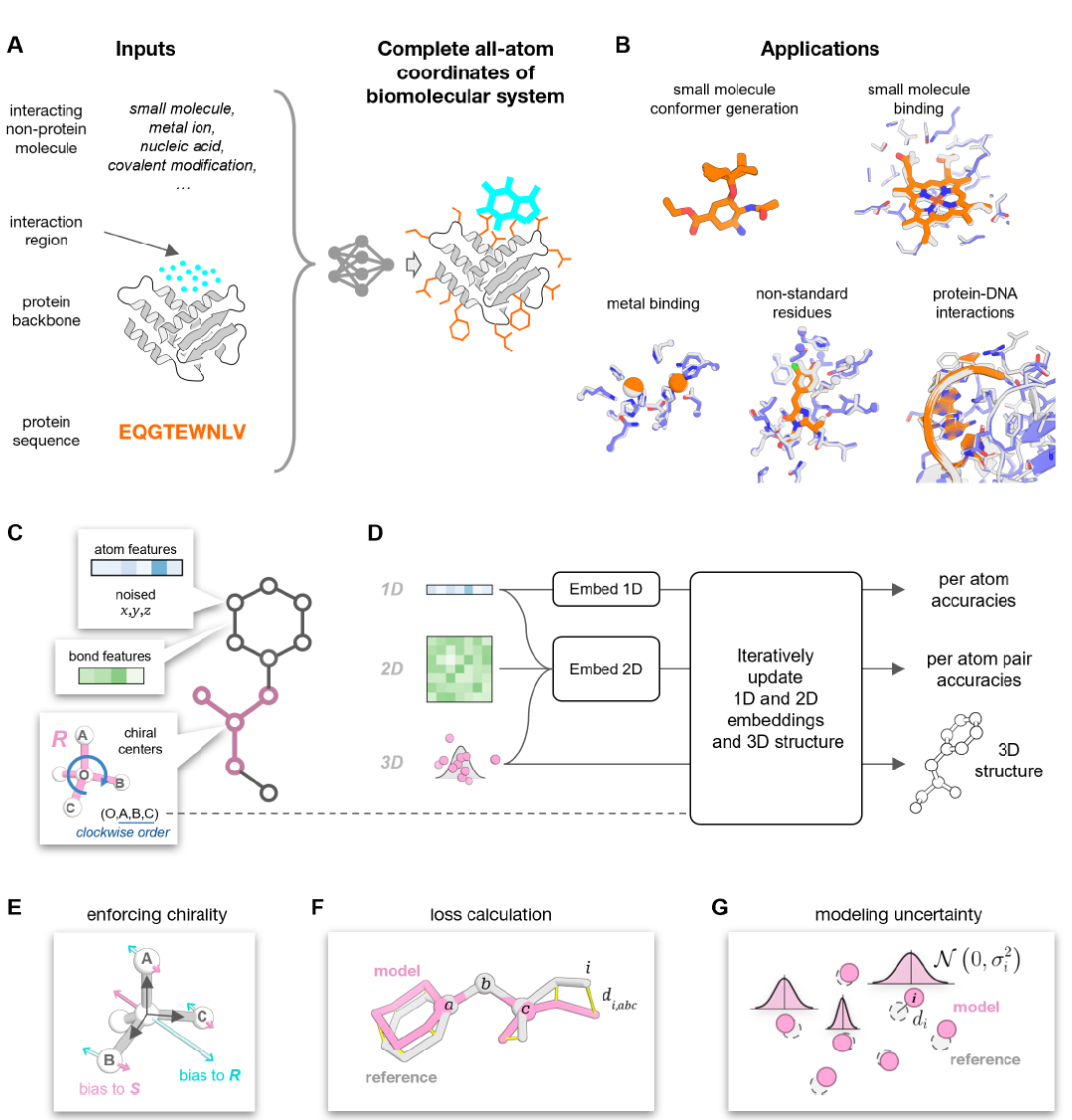
入力フェーズでは、分子系は化学グラフに変換されます。このグラフでは、ノードは個々の重原子(計算コストを削減するため水素原子はモデル化されません)を表し、エッジは原子間の化学結合を表します(上図C参照)。この表現は、異なる種類の分子間で一貫しています。ネットワーク内の各ノードには、原子の種類の情報と、初期に摂動された3D座標が含まれています。ネットワークの役割は、入力座標のノイズを反復的に除去すると同時に、出力モデル構造内の原子位置の不確実性を推定することです(上図D参照)。
PLACER は RoseTTAFold (RF) にヒントを得た 3 トラック アーキテクチャを採用しており、全体的なネットワーク アーキテクチャは次のとおりです。
* 3 軌道設計 (1D、2D、3D): 1D 軌道は原子の特徴情報を処理します。2D 軌道は原子間のペア関係 (化学結合や空間的近接性など) を処理します。3D 軌道は原子座標の更新を担当します。
* 反復最適化:1Dおよび2D特徴量の初期埋め込みが完了すると、これらの特徴量は反復ブロックに渡され、埋め込みベクトルと3D構造が反復的に更新されます。反復ブロックでは、まず原子近傍グラフが構築されます。各原子について、空間的近接性と化学グラフ近接性のそれぞれ半分が選択され、合計32個の最近傍原子が生成されます。次に、2D特徴量ペアはフィードフォワードアダプター層を介してエッジ埋め込みに投影され、1D特徴量、原子近傍グラフ、および現在の3D原子構造とともに、SE3-Transformerネットワークへの入力として使用され、3D座標と1D埋め込みベクトルが更新されます。
* キラル中心処理:キラル中心情報はタイプ1(ベクトル)特徴量を介してネットワークに渡されます(上図E参照)。2次元軌道上の特徴量はペアごとに更新され、構造バイアスと組み合わされます。原子とそのペアの信頼度予測ヘッドは、それぞれ1次元軌道と2次元軌道から分岐し、反復ブロックの計算を完了します。完全に学習されたネットワークには、共有重みを持つ8つの反復ブロックが含まれます。
* 損失関数設計:PLACERの学習では、各反復処理後に構造損失と信頼性予測損失を組み合わせて適用します。主要な構造損失は全原子FAPE(Frame-Aligned Point Error)であり、モデル構造の信頼性は原子レベルと原子ペアレベルの両方で評価されます。
この慎重に設計されたネットワークアーキテクチャを通じてPLACER は、ランダムに初期化された座標から、多様かつ原子レベルで正確なタンパク質小分子立体配座のセットを生成できます。これは、その後の分析と設計のための信頼できる基盤となります。AlphaFoldなどのタンパク質構造予測手法とは異なり、PLACER はタンパク質の主鎖構造を予測するのではなく、原子レベルの側鎖と小分子の立体配座に焦点を当てているため、計算速度が大幅に向上し、多様な立体配座セットの生成が可能になります。
成果のショーケース:小分子から複雑なタンパク質システムまで、精密エンジニアリングをサポート
小分子の立体構造予測
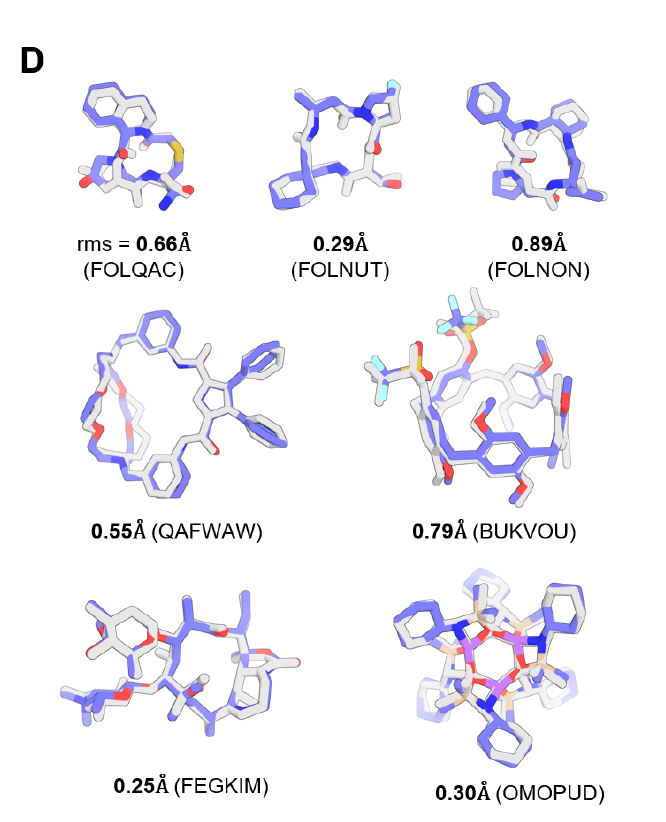
CSD 小分子データセットのテストでは、完全にトレーニングされた PLACER がサブÅ の精度で複雑な分子の 3 次元構造を正しく生成できることが実証されています。例としては、50個以上の原子を含むマクロサイクル(下図D参照)やペプチドマクロサイクル(下図D上段参照)が挙げられます。アブレーション実験では、結合距離情報の欠落や反復回数の減少によって予測精度が大幅に低下することが示されており、PLACER向けに設計された反復および特徴戦略の重要性が実証されています。
タンパク質-小分子相互作用
研究者らは PLACER を使用して、入力座標を毎回異なるランダム初期化しながらネットワークを複数回実行することで、ターゲットタンパク質のポケット内に一連の小分子立体配座を生成しました (下の図 A)。生成されたコンフォメーション セットを分析すると、PLACER はリガンドの初期位置に依存しないことがわかります。複数の異なる開始位置によって、自然なコンフォメーションに近い予測が生成され (下の図 B)、これらの位置が入力サンプリングの空間全体をカバーします (下の図 D)。研究者らはまた、リガンド原子に基づいて計算された予測RMSDスコア(pRMSD)を使用して、サンプリングプールからより正確なモデルを選択できることを観察しました(下の図E)。最高スコアのモデルは、実験構造と高い一致度を示しました(下の図C)。
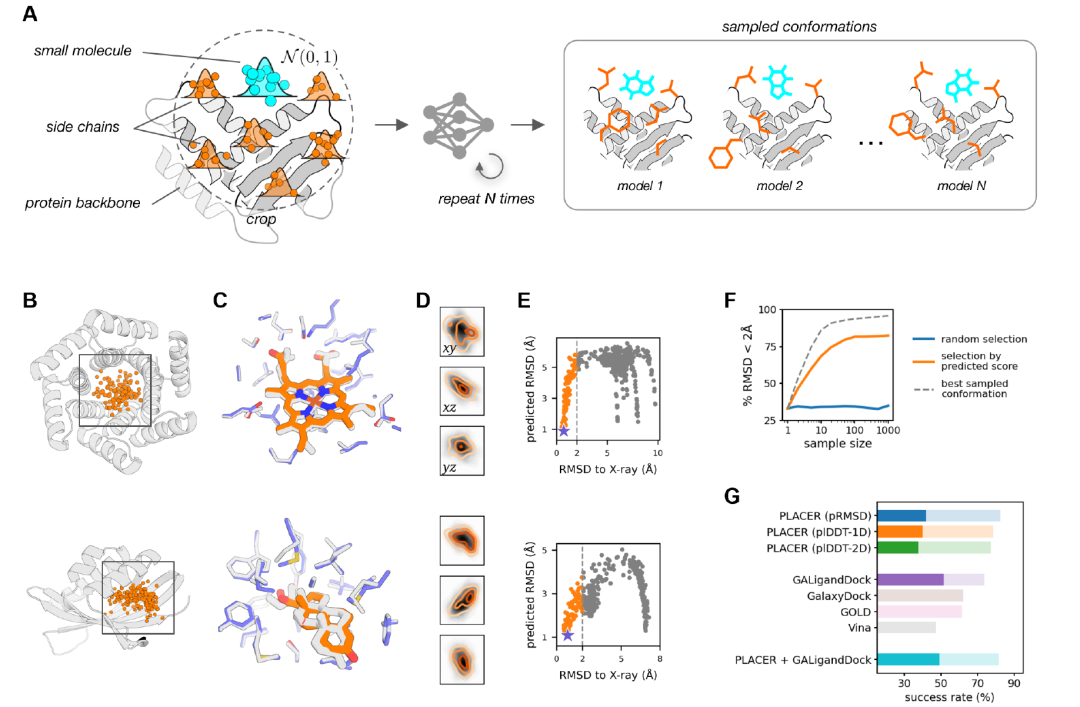
上図Gに示すように、65種類の薬物標的に対する非天然型コンフォメーション試験において、PLACERは天然型に近いコンフォメーションの生成と選択において優れた性能を示し、pRMSDスコアリングを用いた成功率はVina、GOLD、GalaxyDockといった従来のドッキングツールを上回りました。最高性能のRosetta GALigandDock法と比較すると、PLACERは低精度領域(リガンドRMSD < 2Åの複合体の割合)では優れた性能を示しました(82.4% vs 73.6%)。しかし、高精度領域(RMSD < 1Å)ではわずかに劣りました(41.8% vs 51.6%)。
しかし、PLACERのパフォーマンスは依然として注目に値します。他の手法とは異なり、非天然タンパク質と低分子のドッキングタスク向けに特別にトレーニングされていないためです。他のテスト手法は主に入力タンパク質の座標に依存しますが、PLACERは低分子とタンパク質側鎖の立体配座をゼロから再構築できます。
酵素活性部位の設計
PLACERのレトロアルドラーゼ設計への応用は特に注目に値します。研究チームは、RA95シリーズのレトロアルドラーゼとその進化型改良版について50回の反復シミュレーションを実施し、活性部位のリジンとその共有結合中間体の構造多様性を解析しました。その結果、初期の計算設計において活性の低い酵素では、PLACERは非常に多様な構造セットを生成し、事前組織化が欠如していることが示されました。一方、活性の高い進化型では、構造セットは次第に秩序化されました。これは…事前組織化の欠如は初期の酵素設計における大きな欠点ですが、PLACER は酵素設計作業をガイドするために使用できる迅速な評価ツールを提供します。
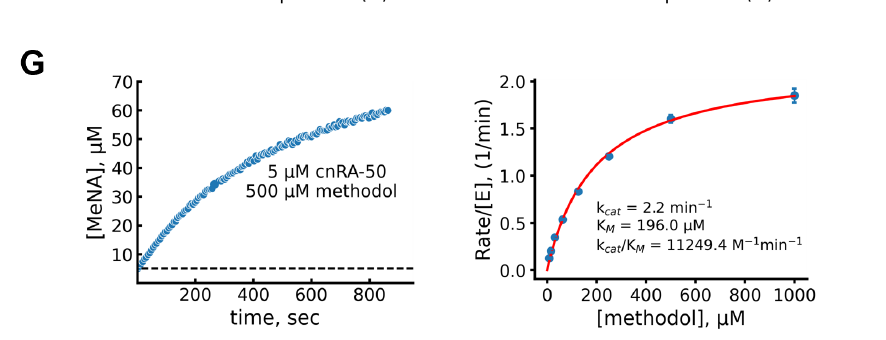
さらに、研究チームはNTF2類似のフォールディング構造に基づく新しいタイプのアルドステロン拮抗酵素を設計し、PLACERを用いて前組織化度とkcat/KM値の相関関係を評価した。その結果、PLACERは、高度に前組織化された設計は概して高い触媒効率を示すと予測した。最も活性の高いcnRA-50は、kcat/KM値が11,000 M⁻¹min⁻¹に達した。これは、従来の計算設計よりも大幅に高く、最新のRF拡散法およびタンパク質MPNN法を用いて作成された設計の活性に迫る値であった。
研究チームは次のように予測している。PLACER ベースのコンフォメーション セット生成法は、分離された状態またはタンパク質環境における複雑な非タンパク質分子の構造モデリング、および酵素設計とタンパク質-小分子複合体設計の評価に広く使用されます。
デビッド・ベイカー教授: 長年にわたり計算タンパク質設計に焦点を当ててきた先駆者
2024年10月9日、著名なタンパク質設計の先駆者であるデビッド・ベイカー教授が、AlphaFold2の開発者であるDeepMindのデミス・ハサビス氏とジョン・M・ジャンパー氏とともに、2024年のノーベル化学賞を受賞しました。
デイビッド・ベイカー教授は長年にわたり計算タンパク質設計に注力し、RoseTTAFold、RFdiffusion、ProteinMPNNといったディープラーニングツールをオープンソース化することで、新規タンパク質の設計を支援してきました。また、企業を設立することでこれらの技術の産業化を推進し、この分野における真の世界クラスの巨匠となりました。最新の研究において、ベイカー教授のチームは多くの新たな分野で大きな進歩を遂げています。
例えば、新薬開発においては、研究者はタンパク質を主要な創薬標的として用いることが多く、構造的に安定したタンパク質に薬剤を結合させることで疾患の進行を抑制します。しかし、構造、配列、および立体配座の好みが明確に定義されていない天然に存在する不規則タンパク質(IDP)を標的とすることは、依然として困難です。こうした背景から、2025年8月、David Baker氏のチームは、誘導適合結合戦略に基づくLogosと呼ばれるタンパク質設計戦略を提案しました。この戦略では、39種類の標的不規則アミノ酸配列に適応できる結合タンパク質を設計します。これにより、より多くのタンパク質が新薬開発の標的となり、がんやアルツハイマー病の研究を加速させる可能性が示唆されます。
論文のタイトル:本質的に無秩序な領域に結合するタンパク質の設計
用紙のアドレス:https://www.science.org/doi/10.1126/science.adr8063
2025年9月18日、David Baker率いるチームは、全原子拡散モデル「RFdiffusion3(RFD3)」を提案しました。このモデルは、全原子生体分子相互作用のde novo設計を可能にします。このモデルは、リガンド、核酸、その他の非タンパク質クラスターのコンテキストにおいてタンパク質構造を生成でき、従来の手法よりもシンプルで効率的です。一連のコンピュータシミュレーションベンチマークにおいて、RFdiffusion3は従来の手法を凌駕する性能を示し、計算コストは従来の10分の1に抑えられました。
論文のタイトル:RF拡散による全原子生体分子相互作用のde novo設計3
用紙のアドレス:https://www.biorxiv.org/content/10.1101/2025.09.18.676967v1
天然イオンチャネルは生物系において重要な役割を果たしており、人工的に改変されたチャネルは化学遺伝学的ツールやセンサーに広く利用されてきました。タンパク質設計は多孔質構造を持つ膜貫通タンパク質の構築に用いられてきましたが、天然イオンチャネルのように特定のイオンを標的とする精密なアミノ酸側鎖を持つ「選択フィルター」の設計は技術的な制約がありました。2025年10月、デイビッド・ベイカー氏のチームは最新の研究で、初めて人工知能を用いて新規カルシウムイオンチャネルをゼロから設計しました。この研究は、私たちが部分的にしか理解していない複雑な生化学的機能でさえ、AIを用いることで原理から構築できることを実証しています。
論文のタイトル:定義された選択性フィルター形状からのCa²⁺チャネルのボトムアップ設計
用紙のアドレス:https://www.nature.com/articles/s41586-025-09646-z
デイビッド・ベイカー教授率いるチームは、近年の成果を振り返ると、タンパク質科学のあり方を驚異的なスピードで変革しています。自然変性タンパク質に結合できるLogos戦略、原子レベルの分子相互作用設計を可能にするRFdiffusion3、そしてカルシウムイオンチャネルの初のde novo構築に関する画期的な研究など、その成果は多岐にわたります。ベイカー教授のチームは、計算によるタンパク質設計を理論から現実へと絶えず推し進めています。彼らの研究は、生体分子設計の限界を広げるだけでなく、「アルゴリズムで生命機能を構築する」という未来をますます明確にしています。
参考リンク:
1.https://www.biorxiv.org/content/10.1101/2024.09.25.614868v2
2.https://www.thepaper.cn/newsDetail_forward_31663354
3.https://www.biorxiv.org/content/10.1101/2025.09.18.676967v1
4.https://www.nature.com/articles/s41586-025-09646-z








