Command Palette
Search for a command to run...
Inspiré Par DeepSeek Engram, Le « Cerveau Externe » Du Modèle De Base Du Génome, Gengram a Réalisé Une Amélioration Des Performances Allant jusqu'à 22,61 TP3T.

Les modèles de génome de base (GFM) sont des outils essentiels pour décrypter le code de la vie, permettant d'accéder à des informations biologiques clés telles que la fonction cellulaire et le développement des organismes grâce à l'analyse des séquences d'ADN. Cependant, les GFM existants basés sur l'architecture Transformer présentent un défaut majeur : ils nécessitent un pré-entraînement à grande échelle et des calculs intensifs pour inférer indirectement les motifs polynucléotidiques, ce qui est non seulement inefficace, mais aussi limité pour les tâches de détection d'éléments fonctionnels basées sur les motifs.
récemment,Le modèle Gengram (engramme génomique) proposé par l'équipe Genos, composée de membres du BGI Life Sciences Research Institute et du Zhejiang Zhijiang Laboratory,Cela apporte une solution révolutionnaire à ce problème. Cette conception évite de coder en dur les règles biologiques tout en donnant au modèle une compréhension explicite de la « grammaire » génomique.
Conçu spécifiquement pour la modélisation de motifs génomiques, Gengram est un module de mémoire conditionnelle léger dont l'innovation majeure réside dans son mécanisme de mémoire de hachage k-mer, qui construit un référentiel de motifs multi-bases extrêmement efficace. Contrairement aux modèles traditionnels qui infèrent les motifs indirectement,Il stocke directement les k-mers de 1 à 6 bases et leurs vecteurs d'intégration, et capture les dépendances contextuelles locales des motifs fonctionnels grâce à un mécanisme d'agrégation de fenêtre locale.Les informations relatives aux motifs sont ensuite fusionnées avec le réseau principal via un module contrôlé par une porte logique. L'équipe de recherche a indiqué que, lorsqu'il est intégré au modèle génomique de pointe Genos, dans les mêmes conditions d'entraînement, Gengram permet d'obtenir des améliorations significatives des performances pour de multiples tâches de génomique fonctionnelle, avec une amélioration maximale de 22,61 points TP3T.

Adresse du document :https://arxiv.org/abs/2601.22203
Adresse du code :https://github.com/BGI-HangzhouAI/Gengram
Poids des modèles :https://huggingface.co/BGI-HangzhouAI/Gengram
Les données d'entraînement couvrent les génomes humains et ceux des primates non humains.
L'ensemble de données d'entraînement contient 145 séquences de haute qualité, analysées et assemblées par haplotype, couvrant les génomes humains et de primates non humains.Les séquences humaines proviennent principalement du Human Pangenome Reference Consortium (HPRC, 2e édition), complétées par les génomes de référence GRCh38 et CHM13. Les séquences de primates non humains ont été intégrées à partir de la base de données NCBI RefSeq afin de prendre en compte la diversité évolutive. Toutes les séquences ont été traitées par encodage one-hot. Le vocabulaire comprend quatre bases standard (A, T, C, G), les nucléotides ambigus (N) et les marqueurs de fin de document.
final,Le système a constitué trois ensembles de données pour soutenir les expériences d'ablation et la pré-formation formelle.
50 milliards de jetons à 8 192 (ablation)
200 milliards de jetons à 8k (10 milliards de jetons pré-entraînés formellement)
100 milliards de jetons à 32 000 (10 milliards de jetons pré-entraînés formellement)
Et maintenir un ratio de mélange de données humain:non-humain = 1:1.
La modélisation du génome passe de la "dérivation de l'attention" à l'"amélioration de la mémoire".
S'inspirant du mécanisme de mémoire de DeepSeek Engram, l'équipe Genos a rapidement développé et déployé Gengram.Ce module offre des fonctionnalités de stockage et de réutilisation explicites des motifs pour les modèles de génomes de base, surmontant ainsi les limitations des modèles de génomes classiques, qui ne disposent pas d'une mémoire structurée des motifs et ne peuvent s'appuyer que sur l'extension de la « mémoire implicite » des données d'entraînement. Il fait évoluer la modélisation du génome d'une approche basée sur la « dérivation de l'attention » vers une approche basée sur l'« amélioration de la mémoire ». L'architecture du module est illustrée dans la figure ci-dessous :
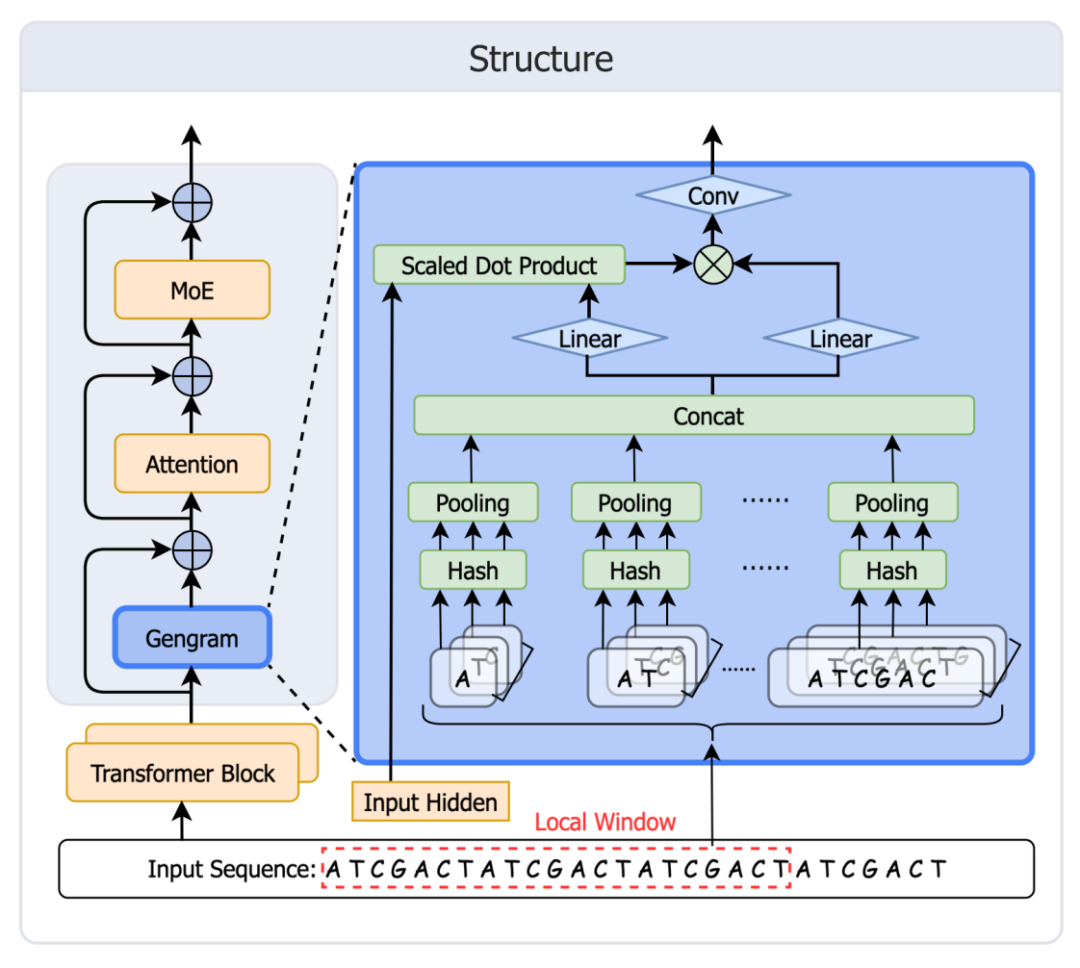
Création de la table : Construisez une mémoire de hachage (clé statique + valeur d'intégration apprenable) pour toutes les valeurs k-mer de k=1 à 6.
Récupération : Associer toutes les valeurs k-mer apparaissant dans la fenêtre aux entrées du tableau.
Agrégation : Commencez par agréger à chaque k, puis concaténez sur l'ensemble des k.
Contrôle d'accès : Le mécanisme de contrôle l'activation, inscrit les preuves du motif dans le flux résiduel, puis capte l'attention.
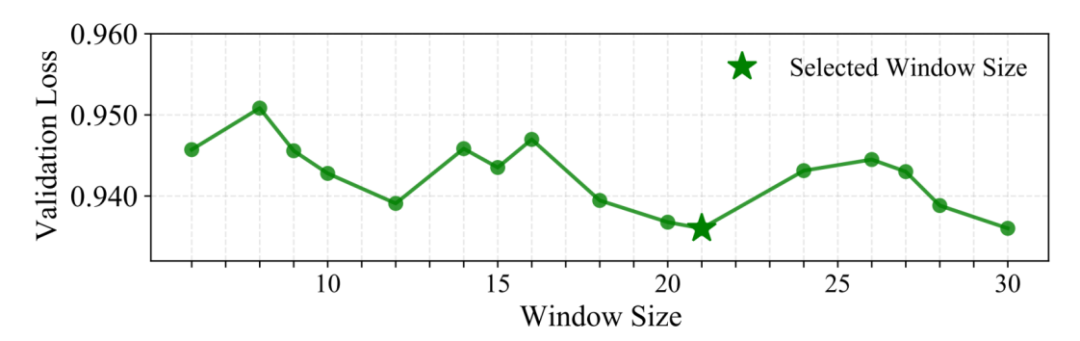
Une caractéristique de conception clé : l’agrégation de fenêtres locales (W=21 pb)
Au lieu de récupérer un seul n-gramme à chaque emplacement, Gengram utilise l'agrégation de plusieurs plongements k-mer dans une fenêtre fixe afin d'injecter de manière plus fiable des motifs « locaux et structurellement cohérents ». Les chercheurs ont validé cette approche en effectuant une recherche avec une stratégie de taille de fenêtre variable.Nous avons constaté que 21 pb permet d'obtenir des performances optimales sur l'ensemble de validation.Une explication biologique possible est que le cycle typique de la double hélice d'ADN comprend environ 10,5 paires de bases par rotation, soit 21 paires de bases qui effectuent exactement deux rotations. Cela signifie que deux bases distantes de 21 pb se trouvent du même côté de l'hélice dans l'espace tridimensionnel, face à des environnements biochimiques similaires. Un fenêtrage à cette échelle pourrait favoriser l'alignement de la phase des signaux de séquence locaux.
Améliorations significatives en matière d'évaluation : petits paramètres, grands changements
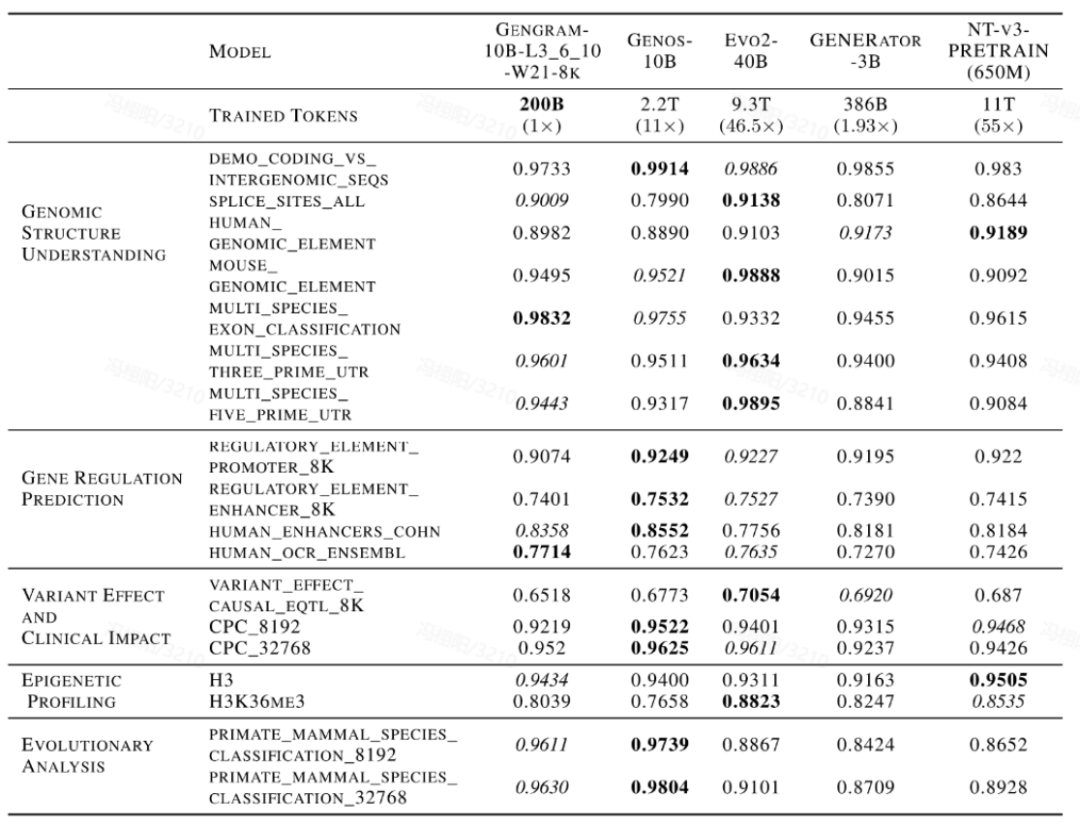
L'équipe a mené une évaluation complète du modèle en utilisant des ensembles de données de référence multi-standards, couvrant Genomic Benchmarks (GB), Nucleotide Transformer Benchmarks (NTB), Long-Range Benchmarks (LRB) et Genos Benchmarks (GeB).Dix-huit ensembles de données représentatifs ont été sélectionnés, couvrant cinq grandes catégories de tâches :Compréhension de la structure génomique, prédiction de la régulation des gènes, profilage épigénétique, effet des variants et impact clinique, et analyse évolutive.
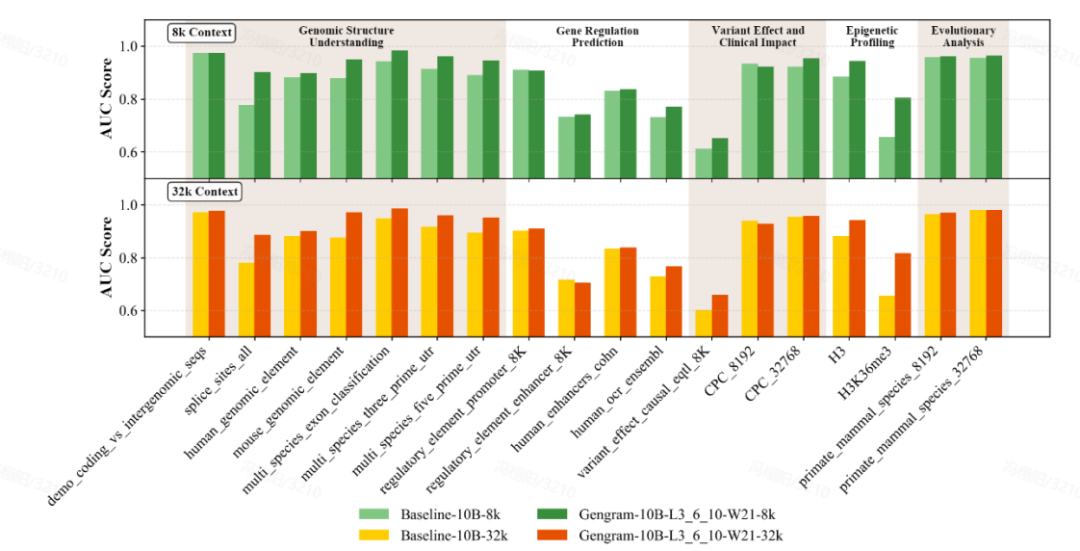
Gengram, un plugin léger ne comptant qu'environ 20 millions de paramètres, représente une infime fraction des paramètres d'un modèle de base qui en compte des centaines de milliards, tout en offrant des gains de performance significatifs. Dans les mêmes conditions d'entraînement, avec des longueurs de contexte de 8 000 et 32 000…Les modèles intégrés à Gengram ont surpassé les versions non intégrées dans la grande majorité des tâches.En termes de manifestations spécifiquesLe score AUC pour la tâche de prédiction du site d'épissage s'est amélioré de 0,776 à 0,901, soit une augmentation de 16,11 TP3T ;Le score AUC de la tâche de prédiction épigénétique (H3K36me3) est passé de 0,656 à 0,804, soit une augmentation de 22,61 TP3T.
De plus, cette amélioration des performances s'accompagne d'un important effet de « valorisation des données ». Dans une comparaison horizontale avec les modèles classiques basés sur l'ADN tels que Evo2, NTv3 et GENERATOR-3B,Les modèles intégrant des génogrammes ne nécessitent qu'une très petite quantité de données d'entraînement et moins de paramètres d'activation pour rivaliser avec les modèles disponibles publiquement dont les données d'entraînement sont plusieurs à des dizaines de fois plus importantes sur les tâches principales.Cela démontre une grande efficacité d'apprentissage des données.
Analyse approfondie de Gengram
Pourquoi Gengram peut-il accélérer l'entraînement ?
L'équipe a introduit la divergence de Kullback-Leibler comme métrique de diagnostic représentative pour le processus d'entraînement et a utilisé LogitLens-KL pour quantifier et suivre la capacité de prédiction des différentes couches. Les résultats ont montré que…En introduisant des génogrammes, le modèle peut former une distribution de prédiction stable plus tôt dans les couches superficielles :Comparativement au modèle de référence, ses valeurs KL inter-couches diminuent plus rapidement et atteignent plus tôt la plage des faibles valeurs, ce qui indique que les signaux de supervision efficaces sont organisés plus tôt en représentations utilisables, rendant ainsi les mises à jour de gradient plus directes et les chemins d'optimisation plus fluides, ce qui se traduit finalement par une vitesse de convergence plus rapide et une efficacité d'entraînement plus élevée.
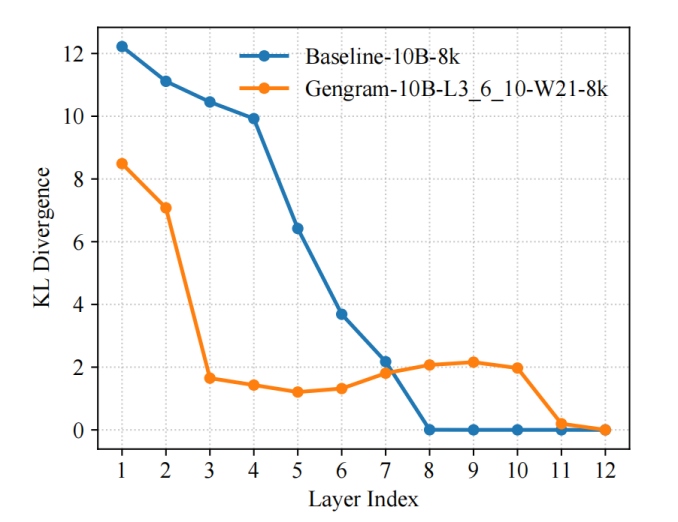
Ce phénomène n'est pas apparu par hasard, mais a été directement induit par la conception structurelle de Gengram :
La récupération explicite de motifs en mémoire raccourcit le chemin entre l'information et la représentation. Dans les tâches génomiques, les signaux de supervision sont souvent déclenchés par des motifs courts et épars (tels que des séquences consensus d'épissage, des fragments liés aux promoteurs, des séquences de faible complexité, etc.). Les Transformers classiques doivent progressivement « dériver et consolider » ces informations locales à travers plusieurs couches d'attention/MLP ; tandis que les Gengrams, grâce à un accès explicite aux k-mers, fournissent directement ces motifs locaux à haute densité d'information au réseau sous forme de mémoire. Ainsi, le modèle n'a pas à attendre que les couches profondes forment progressivement des détecteurs de motifs et se rapproche d'un état prévisible dès le départ.
L'agrégation de fenêtres et le contrôle dynamique rendent les preuves injectées « stables et contrôlables ». Les gengrammes n'effectuent pas d'injection dure position par position ; ils agrègent plutôt plusieurs plongements k-mer dans une fenêtre fixe.De plus, une écriture sélective contrôlée est utilisée dans le flux résiduel : la récupération est plus susceptible d'être activée dans les régions fonctionnelles et inhibée dans les grandes régions de fond. Cette méthode d'écriture d'« éléments fonctionnels clairsemés et alignés » réduit d'une part les interférences dues au bruit, et d'autre part, elle permet au réseau d'obtenir plus tôt des signaux d'entraînement à rapport signal/bruit élevé, réduisant ainsi la difficulté d'optimisation.
D’où proviennent les souvenirs Motif ? Une explication détaillée du mécanisme d’écriture de Gengram.
L'équipe de recherche a d'abord observé un phénomène clair et constant dans toutes les tâches lors des évaluations en aval :Dans les mêmes conditions d'entraînement, l'introduction des gengrammes a considérablement amélioré le modèle pour les tâches typiques basées sur les motifs, notamment dans les scénarios reposant sur de courtes séquences de programme, comme l'identification des sites d'épissage et la prédiction des sites de modification épigénétique des histones. Par exemple, sur des tâches représentatives, l'AUC pour la prédiction des sites d'épissage est passée de 0,776 à 0,901, et l'AUC pour la prédiction de H3K36me3 de 0,656 à 0,804, démontrant des gains stables et substantiels.
Pour répondre plus précisément à la question « D'où viennent ces améliorations ? », l'équipe ne s'est pas arrêtée au niveau des métriques, mais a extrait les écritures résiduelles du Gengram de la propagation avant du modèle et a visualisé leur distribution d'intensité dans la dimension de séquence sous forme de carte thermique pour analyse.Les résultats montrent que le signal écrit présente une structure très clairsemée et à contraste élevé : la plupart des emplacements sont proches de la ligne de base, et seuls quelques emplacements forment des pics nets ;Plus important encore, ces pics ne sont pas aléatoires, mais sont significativement enrichis et alignés avec des régions et des limites fonctionnellement pertinentes, notamment des fragments de boîte TATA près des promoteurs, des fragments poly-T de faible complexité et des emplacements clés près des limites de régions fonctionnelles telles que les gènes/exons.Cela signifie qu'écrire dans un gengramme revient davantage à « saisir des preuves locales de sa fonction décisive » qu'à injecter des informations sans discernement dans toute la séquence.
Sur la base des phénomènes susmentionnés et de la chaîne de preuves,Les chercheurs peuvent résumer le mécanisme de mémoire des motifs de Gengram comme suit : « récupération à la demande – écriture sélective – alignement structuré » :Le module contrôle l'intensité de la récupération et de l'écriture par un mécanisme de sélection, en injectant plus activement des motifs réutilisables dans les régions à forte densité d'informations fonctionnelles et en supprimant l'écriture dans les régions d'arrière-plan afin de réduire les interférences. De ce fait, la maîtrise des motifs par le modèle ne repose plus principalement sur la « mémoire implicite » apportée par les données à grande échelle, mais s'appuie désormais sur une capacité structurée à accéder explicitement aux représentations et à les écrire de manière interprétable.

Conclusion
Ces dernières années, le domaine de la modélisation du génome a connu un changement majeur, passant de « l'apprentissage statistique des séquences » à la « modélisation prenant en compte la structure ».
Les mécanismes de mémoire de motifs conditionnels, illustrés par Gengram, révèlent une voie technique distincte du calcul intensif traditionnel : en modélisant explicitement les motifs fonctionnels multi-bases comme des mémoires structurées récupérables, le modèle peut parvenir à une utilisation plus efficace et stable des informations fonctionnelles tout en maintenant une compatibilité architecturale générale.Cette approche a non seulement démontré des avantages significatifs en termes de performances dans de multiples tâches de génomique fonctionnelle, mais a également fourni une solution d'ingénierie unifiée pour le calcul parcimonieux, la modélisation de longues séquences et l'interprétabilité des modèles.
De plus, du point de vue industriel, le paradigme « a priori structuré + amélioration modulaire » incarné par Gengram réduit considérablement les coûts marginaux des modèles génomiques à grande échelle en termes de puissance de calcul, de données et de cycles d'entraînement, rendant ainsi leur déploiement à grande échelle dans des scénarios à forte valeur ajoutée tels que le développement de médicaments, le criblage de variants et l'analyse de la régulation génique tout à fait envisageable. À plus long terme, ces composants architecturaux réutilisables et modulaires pourraient devenir la configuration standard des modèles génomiques de nouvelle génération, faisant évoluer l'industrie de « modèles plus volumineux » vers des « modèles plus intelligents » et accélérant la transformation continue des résultats de la recherche académique en plateformes industrielles et applications cliniques.