Command Palette
Search for a command to run...
OBIA : Plus De 900 Patients, Plus De 193 Images, l'Institut De Génomique De l'Académie Chinoise Des Sciences a Publié La Première Base De Données De Partage d'images Biologiques De Mon Pays

Il est courant de consulter un médecin et de prendre des radiographies. Les données de tomodensitométrie, d'IRM, de radiographie et d'autres données d'imagerie peuvent être transmises à travers le corps humain de manière non invasive, rendant l'état des organes et des tissus internes clairement visible, fournissant une base fiable pour le diagnostic clinique et le traitement des maladies.
Avec le développement généralisé de la technologie d'imagerie médicale, les données d'imagerie représentent plus de 80% de données médicales nationales.Les problèmes tels que la pénurie de radiologues, les différences dans les résultats diagnostiques entre les hôpitaux à tous les niveaux et la répartition inégale des ressources médicales deviennent de plus en plus importants.
Il y a beaucoup de place pour l’imagination lorsque l’on combine l’IA avec l’imagerie médicale. Les technologies de cognition sensorielle et d’apprentissage profond présentent des avantages inégalés par rapport aux humains dans l’identification des résultats de diagnostic d’imagerie médicale. Ils peuvent aider les médecins à réduire les taux d’erreurs de diagnostic et à améliorer l’efficacité du travail.
Cependant,Les algorithmes d’IA de haute qualité nécessitent des ensembles de données d’images suffisamment grands et représentatifs.Ces images médicales contiennent souvent une grande quantité d’informations confidentielles sensibles. De plus, il existe des « îlots de données » entre les hôpitaux à tous les niveaux, et le système de partage incomplet rend les ressources disponibles pour l’IA d’imagerie médicale limitées.
Auteur | Tour
Rédacteur | Sanyang, Xuecai
De nombreux pays à travers le monde ont créé diverses bases de données de partage de données d’imagerie médicale. Mon pays est encore à la traîne par rapport à la communauté internationale dans ce domaine. Afin de favoriser le partage de données d'imagerie biologique médicale de haute qualité,L'Institut de génomique de l'Académie chinoise des sciences (Centre national de bioinformation, Chine) a créé l'Open Biomedical Imaging Archive (OBIA).
En tant que premier référentiel ouvert de données d'imagerie biomédicale et de données cliniques associées en Chine,L'OBIA est ouvert gratuitement aux praticiens et aux universitaires médicaux du monde entier. La version pré-imprimée des résultats associés a été publiée sur « bioRxiv » le 25 septembre 2023.

Lien vers l'article :https://www.nature.com/articles/s42256-023-00704-7
Suivez le compte public « HyperAI Super Neural » et répondez « OBIA » pour obtenir le PDF complet de l'article
Processus de construction et de mise en œuvre de la base de données OBIA
En tant que ressource de base de données principale du Centre national chinois de bioinformation, l'OBIA accepte les soumissions d'images du monde entier et offre un accès libre et gratuit à toutes les données publiques.Il prend en charge la désidentification, la gestion et le contrôle qualité des données d'image.La fourniture de services de données tels que la navigation, la récupération et le téléchargement peut favoriser la réutilisation des données d’image et des données cliniques existantes.
OBIA utilise cinq types d'objets de données (Collection, Individu, Étude, Série, Image) pour organiser les données.Accepte les soumissions d'images biomédicales multimodales, multi-organes et multi-maladies.
Pour protéger votre vie privée,L'OBIA a développé un processus unifié de désidentification et de contrôle qualité.Il fournit également une interface Web intuitive et conviviale pour la soumission, la navigation et la récupération de données, ainsi que la récupération d'images. Dans l’ensemble, OBIA fournit une plate-forme fiable pour la gestion des données d’imagerie biomédicale nationales, contribuant ainsi à soutenir la recherche biomédicale mondiale.
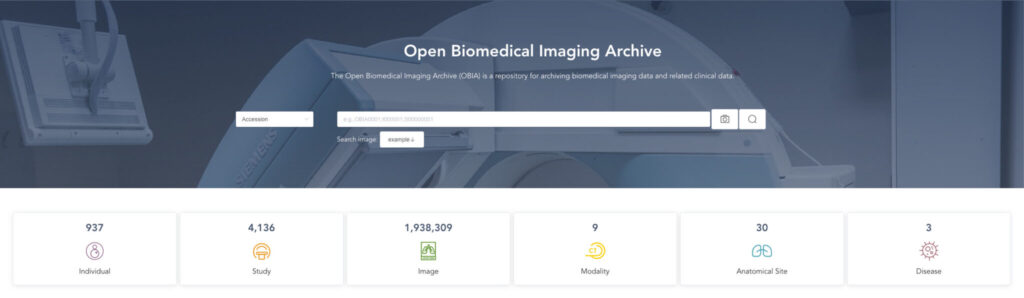
Figure 1 : Interface d'accès OBIA
Visitez l'URL :https://ngdc.cncb.ac.cn/obia
Détails de mise en œuvre——Récupération d'images
Les réseaux neuronaux profonds sont efficaces pour extraire des caractéristiques avantageuses.Il peut être utilisé pour récupérer des images médicales multimodales de divers organes du corps humain et améliorer les performances de classement dans les cas de petits échantillons. Par rapport aux méthodes traditionnelles, les méthodes basées sur l'apprentissage profond telles que la transformation de caractéristiques invariantes à l'échelle (SIFT), les modèles binaires locaux (LBP) et l'histogramme de gradients orientés (HOG) peuvent afficher de meilleures performances.
Dans l'OBIA, les chercheurs ont utilisé EfficientNet comme extracteur de caractéristiques basé sur des données multimodales sur le cancer provenant de la base de données d'imagerie du cancer TCIA, ont formé le modèle à l'aide d'un réseau de triplets et d'un module d'attention, et ont compressé l'image en valeurs de hachage discrètes (Figure 2). Par la suite, pour accélérer les performances d'inférence et réduire la latence d'inférence, le modèle formé est converti au format TensorRT et Faiss est utilisé pour stocker les codes de hachage.
Les chercheurs ont utilisé la distance de Hamming pour calculer la similarité des images et ont renvoyé l’image la plus similaire.Les résultats montrent que la valeur de précision moyenne (MAP) du modèle proposé dépasse les performances des modèles avancés de récupération d'images existants sur l'ensemble de données TCIA.
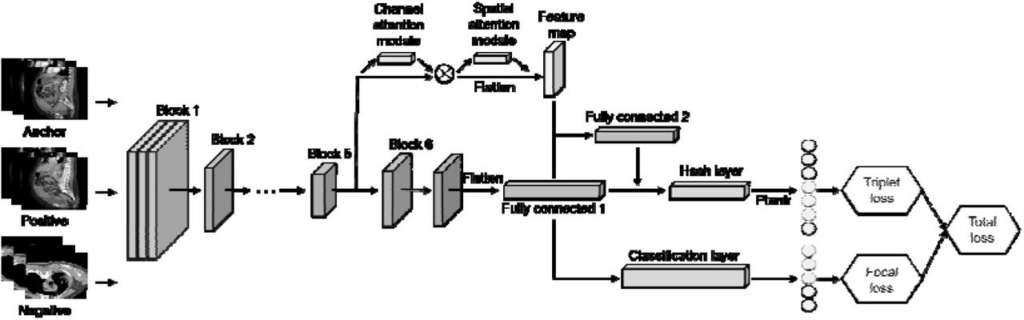
Figure 2 : Hachage de triplet profond basé sur les modules d'attention et de fusion de couches
Ce modèle utilise EfficientNet-B6 comme réseau principal et utilise le module d'attention CBAM dans Block5 pour obtenir des cartes de fonctionnalités. La fusion de couches est adoptée dans les couches entièrement connectées pour générer des codes de hachage et des intégrations de classes à l'aide de la perte focale et de la perte de triplet.
Note:
● CBAM : module d'attention par blocs convolutionnels
● EfficientNet : Un nouveau type de réseau CNN proposé par Google en 2019, qui présente une efficacité et une vitesse de paramètres extrêmement élevées et qui fonctionne bien dans le domaine de la classification d'images
● Faiss : une bibliothèque de recherche de similarité haute performance développée par Facebook AI Research, couramment utilisée dans l'apprentissage profond
Contenu et utilisation de la base de données - Modèle de données
Comme le montre la figure 3,Les données d'imagerie dans OBIA sont divisées en cinq types d'objets :Collection, Individu, Étude, Série, Image, désignent respectivement :
• Collections:Préfixé par « OBIA » pour fournir une description globale de la soumission complète ;
• Individuel:Les numéros d’enregistrement sont précédés d’un « I » et définissent les caractéristiques de l’organisme humain ou non humain qui reçoit ou est inscrit pour recevoir des services de soins de santé ;
• Étude:Le numéro d'accès est préfixé par « S » et contient des informations descriptives sur l'examen radiologique de l'individu ;
• Série:L'étude peut être divisée en une ou plusieurs séries selon différentes logiques (comme la partie du corps ou la direction) ;
• Image:Décrit les données de pixels d'un seul fichier DICOM (imagerie numérique et communications en médecine). Une image est associée à une seule série dans une seule étude.
Remarque : DICOM est une norme internationale largement utilisée dans le domaine de l’imagerie médicale. Il définit un ensemble de spécifications et de protocoles pour le stockage, la transmission, le partage et l'impression de données d'imagerie médicale, afin que les équipements et logiciels médicaux produits par différents fabricants puissent être compatibles et communiquer entre eux.
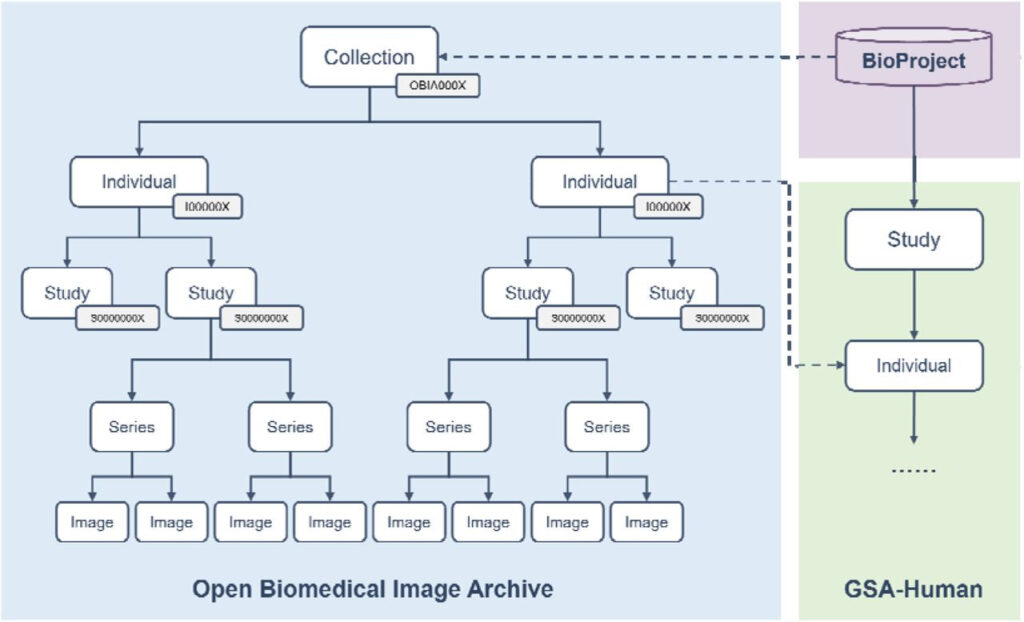
Figure 3 : Modèle de données OBIA
Sur la base de ces objets de données standardisés,OBIA relie la structure d'image définie par la norme DICOM à des projets de recherche réels.Le partage et l’échange de données sont réalisés.
De plus, chaque collection d'OBIA est liée à BioProject pour fournir des métadonnées descriptives sur le projet de recherche ;
Lorsque disponible, l'individu de l'OBIA peut être lié via le numéro d'accès individuel à GSA-Human, qui relie les données d'imagerie aux données génomiques pour que les chercheurs puissent effectuer des analyses multi-omiques.
URL du BioProject :
https://ngdc.cncb.ac.cn/bioproject/
Adresse du lien GSA-Human :
https://ngdc.cncb.ac.cn/gsa-human/
Contenu et utilisation de la base de données——Désidentification et contrôle qualité
Les images biomédicales peuvent contenir des informations de santé protégées (PHI) et doivent être traitées correctement pour minimiser le risque de violation de la vie privée. Afin de préserver autant d'informations scientifiques précieuses que possible tout en supprimant les PHI,OBIA fournit un mécanisme de désidentification et de contrôle qualité conforme à la norme DICOM (Figure 4).
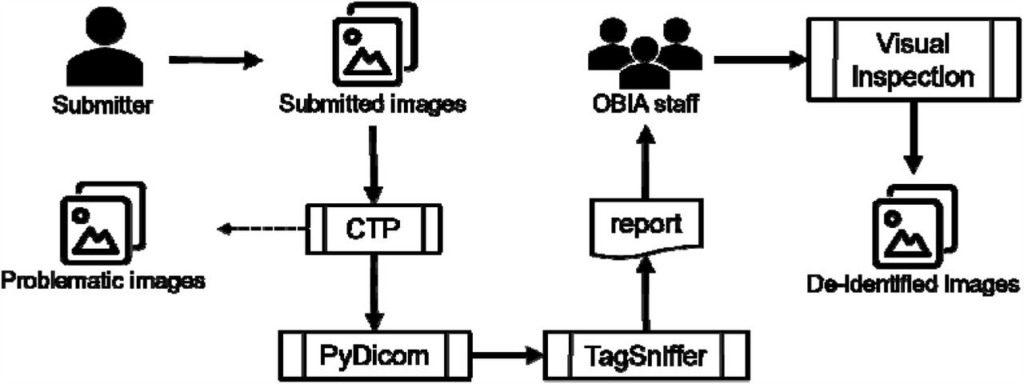
Figure 4 : Mécanismes de désidentification et de contrôle qualité de l'OBIA
L'OBIA utilise le processeur d'essais cliniques (CTP) MIRC de la Radiological Society of North America (RSNA) pour effectuer une grande partie du travail de désidentification :
• Pour les balises standard,Les chercheurs ont construit un CTP et développé un script de désidentification de base universel pour supprimer ou anonymiser certains marqueurs standard qui contiennent ou peuvent contenir des PHI ;
• Pour les tags privés,Utilisez PyDicom pour le traiter, en préservant son caractère purement numérique.
Une fois le processus de désidentification terminé, l'OBIA commence à exécuter les procédures de contrôle qualité :
• L'image en question :Isoler les images, où les soumissionnaires peuvent fournir des informations pertinentes pour réparer l'image ou la supprimer complètement (ces images sont celles avec des titres vides ou des identifiants de patients manquants, endommagées, mélangées avec d'autres images de patients, etc.) ;
• Image en double :Gardez-en un seul.
OBIA génère ensuite un rapport pour toutes les images à l'aide de TagSniffer où tous les éléments DICOM sont soigneusement examinés pour garantir qu'ils ne contiennent pas de PHI et que certaines valeurs (par exemple, l'ID du patient, la date de l'étude) sont modifiées comme prévu.
aussi,Le personnel de l’OBIA effectue également des inspections visuelles des pixels de l’image.pour garantir qu'aucun PHI n'est inclus dans les valeurs de pixels et que l'image est visible et non corrompue.
Contenu et utilisation de la base de données——Statistiques
En septembre 2023, l'OBIA a collecté 937 « Individus », 4 136 « Études », 24 701 « Séries » et 1 938 309 « Images », couvrant 9 modalités et 30 parties anatomiques.
Les modalités d'imagerie représentatives comprennent la tomodensitométrie (TDM) à rayons X, la résonance magnétique (IRM) et la radiographie numérique (DX), et les sites anatomiques comprennent l'abdomen, la poitrine, le thorax, la tête, le foie, le bassin, etc.
Le premier lot de données soumises à l’OBIA provenait de l’hôpital 301.Comprend des données d’imagerie pour 3 tumeurs gynécologiques majeures (cancer de l’endomètre, cancer de l’ovaire et cancer du col de l’utérus).
Comme le montre le tableau 1, ces données sont divisées en quatre « Collections », répertoriant le nombre d'« Individuels », le nombre d'« Études », le nombre de « Séries » et le nombre d'« Images ». aussi,OBIA collecte également des métadonnées cliniques pertinentes.Tels que les données démographiques, les antécédents médicaux, les antécédents familiaux, le diagnostic, le type de pathologie et la méthode de traitement.

Tableau 1 : Premier lot d'informations soumises à l'OBIA
Briser les barrières des données,Créer des plateformes de partage de données médicales au pays et à l'étranger
Les données ne génèrent de la valeur que lorsqu’elles sont diffusées. Afin d'améliorer le niveau de partage des données d'imagerie biologique,De nombreux pays à travers le monde se sont engagés à créer des bases de données médicales ouvertes :
• Instituts nationaux de la santé (NIH) :A sponsorisé plusieurs bases de connaissances, telles que MIDRC, une plateforme en libre accès pour les images et données médicales liées au COVID-19, IDA, NITRC-IR, FITBIR, OpenNeuro et NDA, qui collectent des images neuronales et cérébrales, et TCIA et IDC, bases de données d'imagerie du cancer (TCIA fournit des images localement et IDC fournit des images dans un environnement cloud de partage de données de recherche sur le cancer) ;
• Recherche sur le cancer au Royaume-Uni :a parrainé la base de données d'images de mammographie OPTIMAM (OMI-DB) ;
• Université de Porto, Portugal :a parrainé le Breast Cancer Digital Repository (BCDR), qui fournit des images annotées du cancer du sein et des détails cliniques ;
Dans les référentiels ci-dessus, à l'exception de NITRC-IR et IDC,La plupart des autres prennent en charge la désidentification des données et le contrôle de la qualité.De plus, certaines universités ou institutions fournissent également des ensembles de données open source, tels que OASIS, EchoNet-Dynamic, le projet CAMUS, etc.
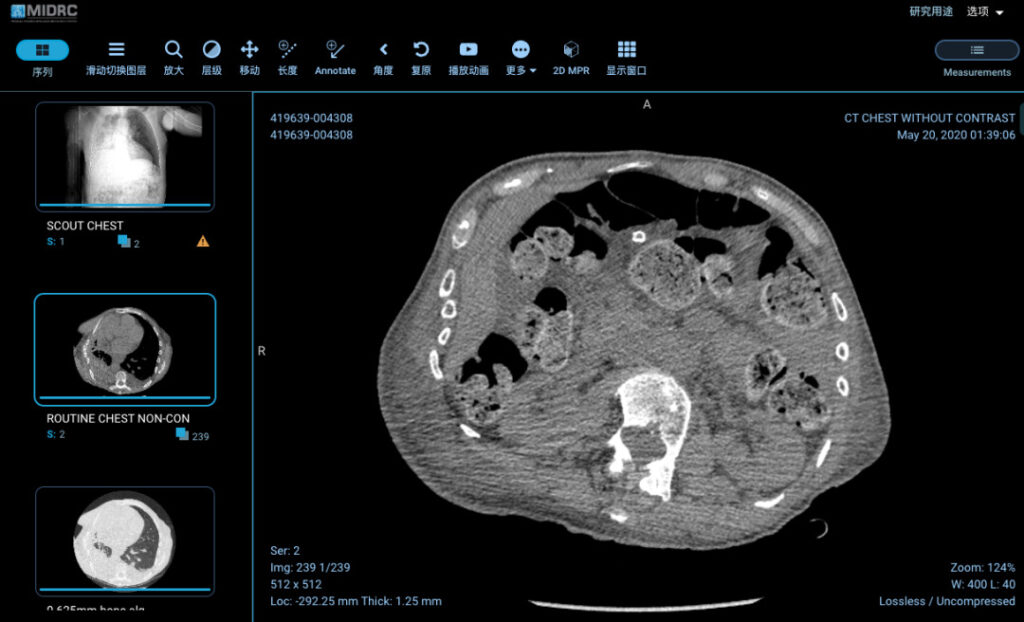
Figure 5 : TDM thoracique d'un patient de 79 ans dans la base de données MIDRC
dans le pays,L'Université des sciences et technologies de Huazhong fournit des ressources ouvertes d'images CT intégrées et de CF du COVID-19.Il comprend des images CT et des caractéristiques cliniques des patients atteints de pneumonie (y compris la COVID-19), mais se limite à une seule maladie et les ressources de recherche disponibles sont limitées. Par conséquent, il existe toujours en Chine un manque de bases de données spécialisées dans le stockage et l’acceptation des soumissions de données sur diverses maladies et modalités.
L'OBIA, créée par l'Académie chinoise des sciences, comble le vide laissé par le partage ouvert des données d'imagerie biomédicale nationales., qui permet aux chercheurs de différentes institutions de partager des données d'imagerie cliniquement pertinentes et peut combler efficacement le fossé dans le domaine des bases de données d'imagerie biomédicale en Chine.
Les chercheurs ont déclaré dans le document qu'ils continueraient à mettre à niveau l'infrastructure de l'OBIA et à renforcer les mesures de sécurité à l'avenir. Ils collecteront également davantage de types de données d’imagerie biomédicale et élargiront les sources de données.Nous prenons de nombreuses mesures pour progresser vers l’objectif de « conserver autant de métadonnées d’image valides que possible et de fournir des données d’imagerie de haute qualité aux chercheurs scientifiques ».
-- sur--








