Command Palette
Search for a command to run...
Online-Tutorial | Das Team Von David Baker Veröffentlicht RFdiffusion3 Als Open Source Und Erzielt Damit Einen Generativen Durchbruch Im Bereich Des All-Atom-Protein-Designs.

In den letzten Jahren wurden mithilfe generativer Deep-Learning-Methoden bedeutende Fortschritte beim Design neuartiger funktioneller Proteine erzielt. Aktuell nutzen die meisten Methoden, darunter RFdiffusion (RFD1) und BindCraft, eine Proteinrepräsentation auf Aminosäurerestebene und konnten erfolgreich Proteinmonomere, -komplexe und Protein-Protein-Interaktionssysteme entwerfen. Ihre Auflösung ist jedoch noch nicht ausreichend, um Strukturen präzise zu entwerfen, die spezifisch mit Nicht-Protein-Komponenten (wie niedermolekularen Liganden und Nukleinsäuren) über Seitenkettenwechselwirkungen interagieren.
Während RFdiffusion2 (RFD2) diese Einschränkung bis zu einem gewissen Grad überwunden hat, ist sein Diffusionsprozess immer noch auf die Restebene beschränkt und es ist schwierig, ihn weiter auszudehnen, um zusätzliche Seitenkettenwechselwirkungen mit Nicht-Protein-Komponenten zu bilden.Bisherige Forschungsergebnisse deuten darauf hin, dass Diffusionsprozesse auf atomarer Ebene zur Erzeugung von Proteingerüsten genutzt und auf die Modellierung von Seitenketten erweitert werden können. Allerdings ist es diesen Versuchen bisher nicht gelungen, Wechselwirkungen zwischen Nicht-Protein-Komponenten effektiv zu modellieren.
Auf dieser GrundlageDas Team des Nobelpreisträgers David Baker hat RFdiffusion3 (RFD3) entwickelt, das dreidimensionale Konformationen von Proteinen in Strukturen erzeugen kann, die aus Liganden, Nukleinsäuren und anderen Nicht-Protein-Atomen bestehen.Da dieses Modell alle Polymeratome explizit modelliert, kann es komplexe atomare Randbedingungen in Aufgaben wie dem Enzymdesign einfacher und effizienter handhaben. Die native All-Atom-Architektur von RFD3 vereinfacht zudem die Spezifikation atomarer Randbedingungen erheblich und ermöglicht eine präzise Kontrolle über Wasserstoffbrückenbindungen, Ligandenkontakte und Nukleinsäureinteraktionen.
Im Gegensatz zu AlphaFold3 (AF3), das auf einem rechenintensiven Pairformer-Modul basiert, um Informationen wie die Distanz zur Eingabesequenz zu extrahieren, hat das Forschungsteam ein leichteres Informationsextraktionsmodul entwickelt.Dadurch kann RFD3 die Anzahl der Schichten im Pairformer drastisch von 48 auf nur 2 reduzieren und so den Rechenaufwand erheblich senken. Das finale Modell enthält lediglich 168 Millionen trainierbare Parameter.Das Forschungsteam demonstrierte die breite Anwendbarkeit von RFD3 durch die Entwicklung und experimentelle Charakterisierung von DNA-bindenden Proteinen und Cysteinhydrolasen. RFD3 kann schnell Proteinstrukturen generieren, die komplexen atomaren Beschränkungen unterliegen und auf beliebiger nicht-proteinartiger atomarer Umgebung basieren. Dies erweitert das funktionelle Spektrum des Proteindesigns erheblich.
„RFdiffusion3: Protein Design Model“ ist jetzt auf der HyperAI-Website (hyper.ai) im Bereich „Tutorials“ verfügbar. Mit einem Klick herunterladen und ausprobieren!
Zur Feier des neuen Jahres bietet HyperAI allen Nutzern Rechenleistungsvorteile.Neukunden erhalten nach der Registrierung mit dem Einlösecode „2026 Happy New Year“ 2 Stunden NVIDIA-Guthaben. GeForce RTX 5090 NutzungsdauerNur solange der Vorrat reicht, sichern Sie sich jetzt Ihre Urlaubsvorteile!
Link zum Tutorial:
Demolauf
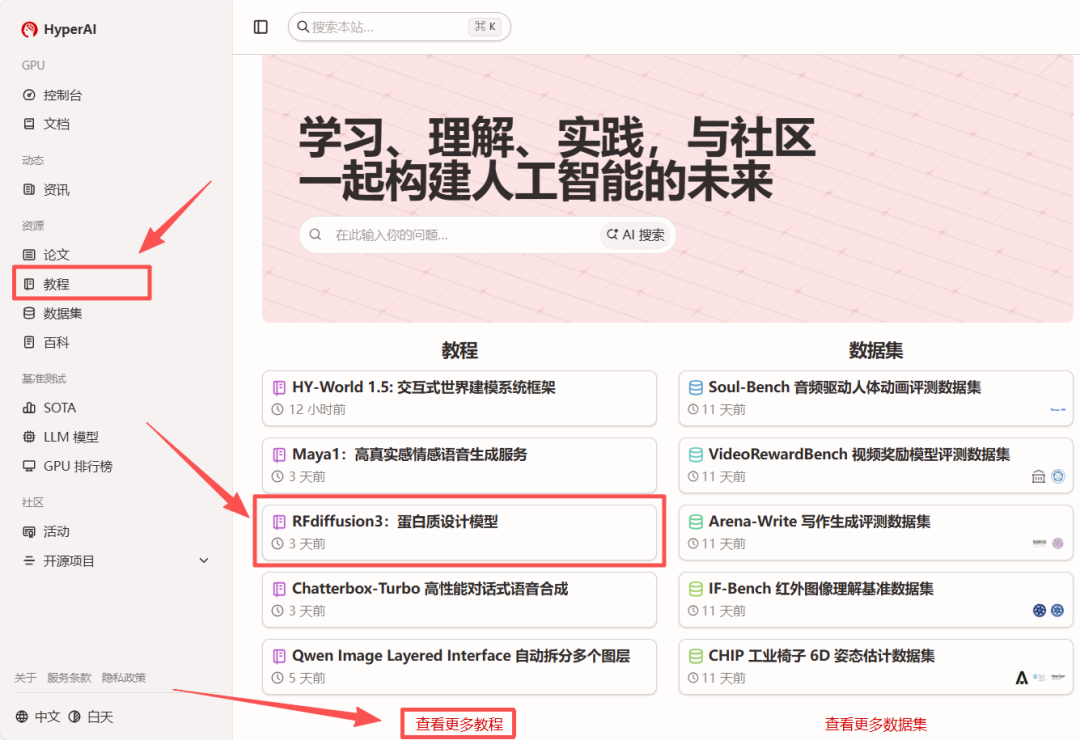
1. Nachdem Sie die Startseite von hyper.ai aufgerufen haben, wählen Sie „RFdiffusion3: Protein Design Model“ aus oder gehen Sie zur Seite „Tutorials“, um es auszuwählen. Klicken Sie anschließend auf „Dieses Tutorial online ausführen“.
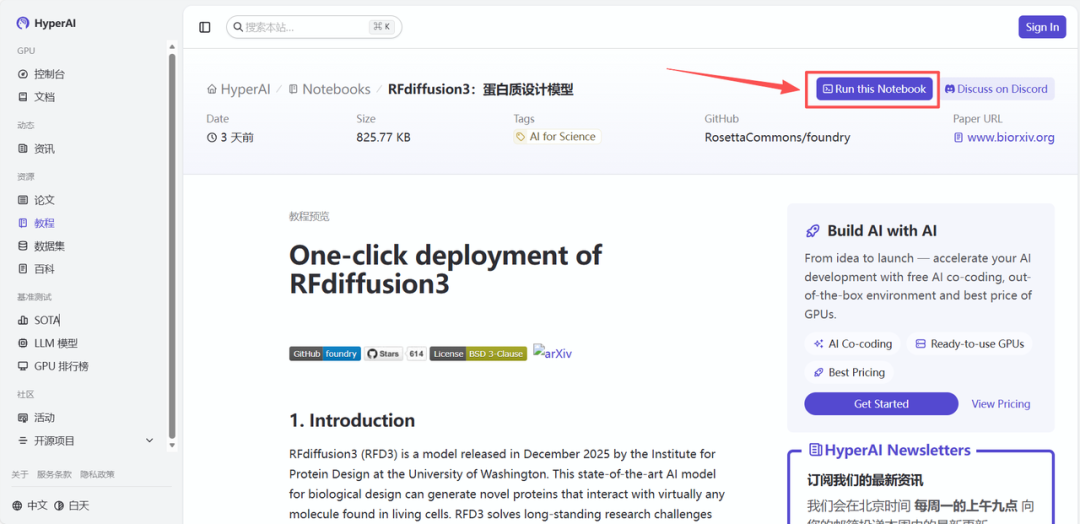
2. Nachdem die Seite weitergeleitet wurde, klicken Sie oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
Hinweis: Sie können die Sprache oben rechts auf der Seite ändern. Derzeit sind Chinesisch und Englisch verfügbar. Dieses Tutorial zeigt die Schritte auf Englisch.
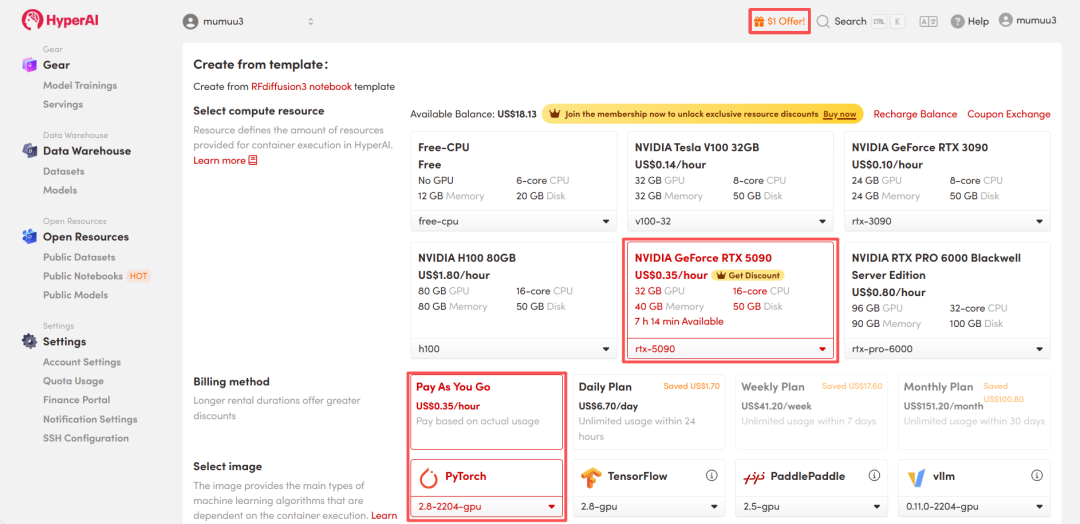
3. Wählen Sie die Images „NVIDIA GeForce RTX 5090“ und „PyTorch“ aus und wählen Sie je nach Bedarf „Pay As You Go“ oder „Tagesplan/Wochenplan/Monatsplan“. Klicken Sie anschließend auf „Auftragsausführung fortsetzen“.
HyperAI bietet Neukunden einen Registrierungsbonus: Für nur $1 erhalten Sie 20 Stunden RTX 5090 Rechenleistung (ursprünglich $7), und die Ressourcen sind unbegrenzt gültig.
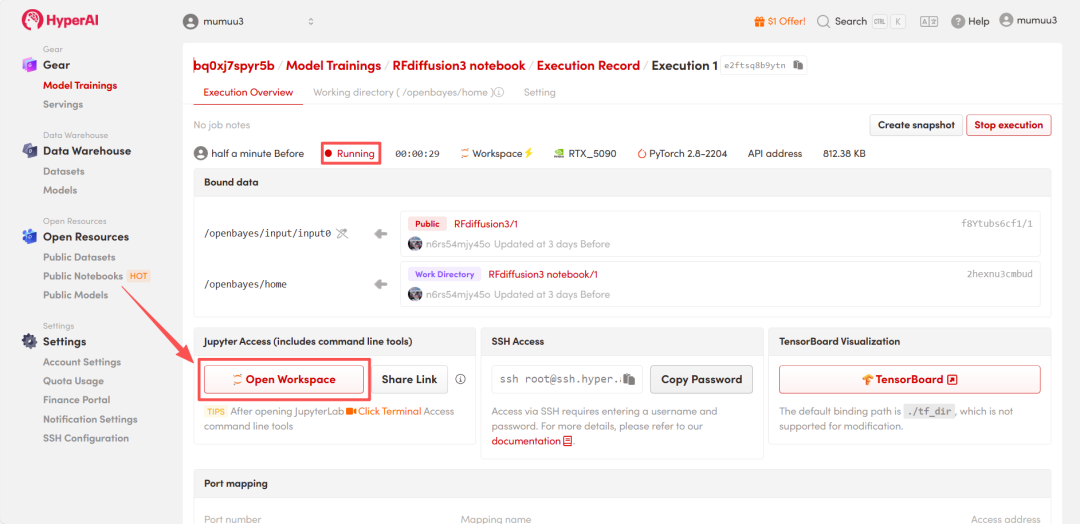
4. Warten Sie, bis die Ressourcen zugewiesen wurden. Sobald sich der Status auf „Wird ausgeführt“ ändert, klicken Sie auf „Arbeitsbereich öffnen“, um den Jupyter-Arbeitsbereich zu betreten.
Effektdemonstration
Nachdem die Seite weitergeleitet wurde, klicken Sie links auf die README-Seite und anschließend oben auf Ausführen.
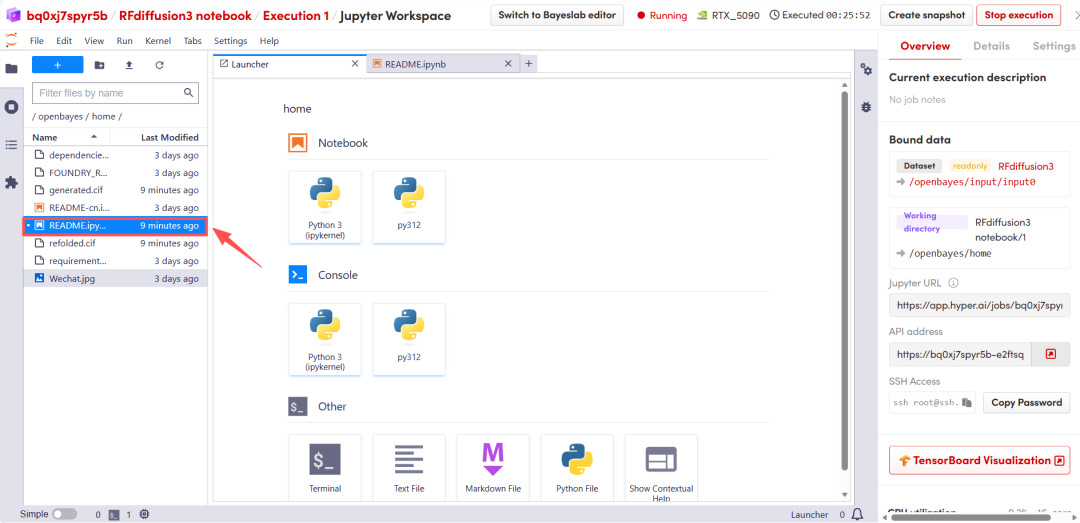
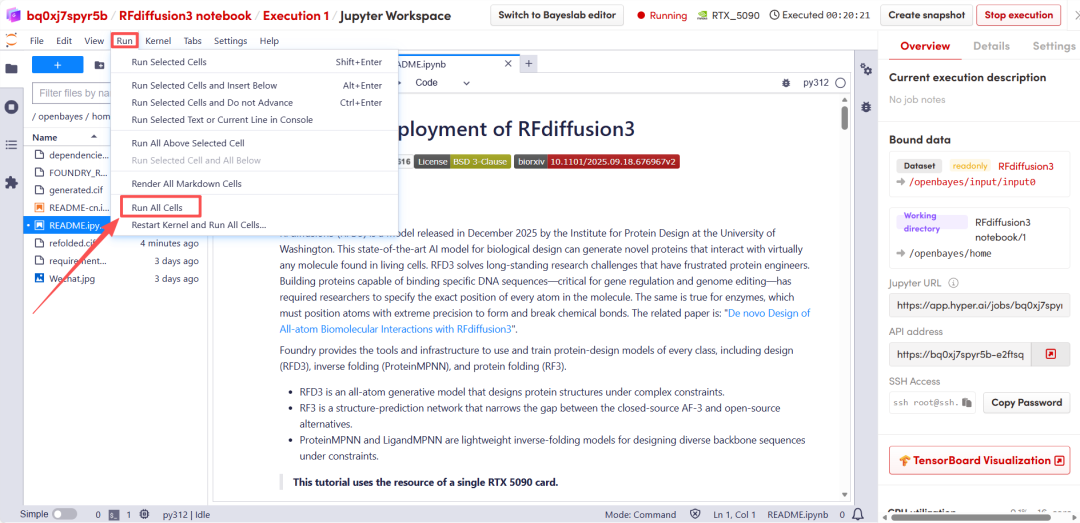
Nach kurzer Wartezeit scrollen Sie nach unten, um die Strukturvorhersageergebnisse von RFD3 anzuzeigen.
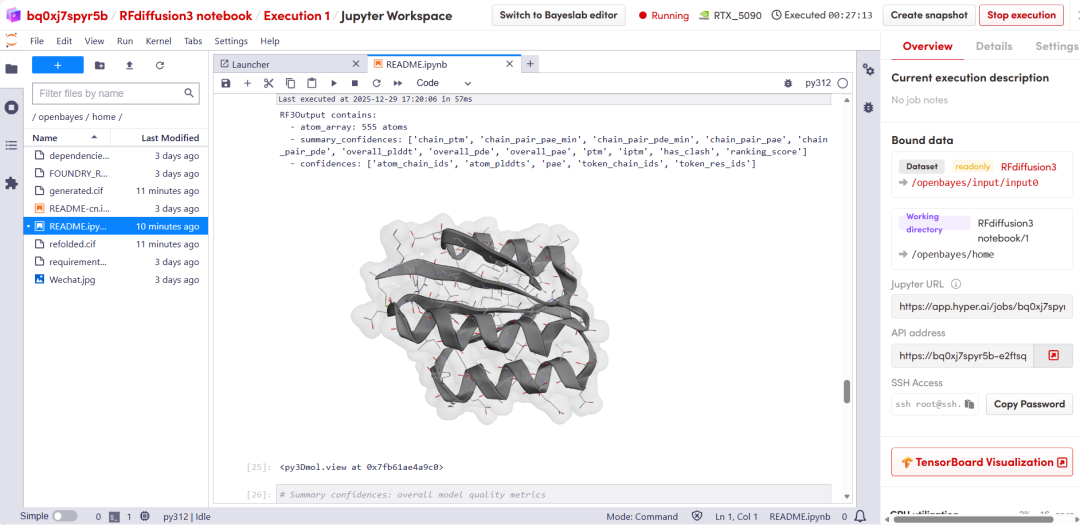
Das Obige ist das diesmal von HyperAI empfohlene Tutorial. Jeder ist herzlich eingeladen, vorbeizukommen und es auszuprobieren!
Link zum Tutorial: