Command Palette
Search for a command to run...
DeepMind Veröffentlicht AlphaGenome, Das Die Auswirkungen Von Mutationen Auf Alle Modalitäten Und Zelltypen in 1 Sekunde Vorhersagt

Die Alpha-Reihe von Google DeepMind erhält ein neues Mitglied – AlphaGenome,Damit können umfassendere und genauere Vorhersagen darüber getroffen werden, wie sich eine einzelne Variation oder Mutation in einer menschlichen DNA-Sequenz auf eine Reihe biologischer Prozesse auswirkt, die die Gene regulieren.
Das AlphaGenome-Modell verwendet eine DNA-Sequenz mit bis zu 1 Million Basenpaaren als Eingabe und prognostiziert Tausende von molekularen Eigenschaften, die mit ihrer regulatorischen Aktivität zusammenhängen.Gleichzeitig können die Auswirkungen von Genvariationen oder -mutationen durch den Vergleich der Vorhersageergebnisse von varianten und nicht-varianten Sequenzen bewertet werden. Dieses Modell basiert auf DeepMinds vorherigem Genommodell Enformer und ergänzt das AlphaMissense-Modell, das sich auf die Klassifizierung von Variationen proteincodierender Regionen konzentriert.
Jun Cheng, Co-Erstautor der Studie, erklärte auf seinem persönlichen X-Account: „RNA-Spleißfehler sind eine häufige Ursache vieler Krankheiten. Erstmals haben wir ein einheitliches Modell entwickelt, das gleichzeitig die RNA-Sequenzabdeckung, die Spleißstellen, die Nutzung der Spleißstellen und die von ihnen gebildeten spezifischen Spleißverbindungen vorhersagen kann und so das Gesamtbild der Spleißergebnisse umfassender darstellt.“ Er wies außerdem darauf hin, dassEiner der wichtigsten Durchbrüche von AlphaGenome ist „die Fähigkeit, Spleißstellen direkt aus Sequenzen vorherzusagen und sie zur Vorhersage von Varianteneffekten zu verwenden.“
„Dies ist ein Meilenstein auf diesem Gebiet“, sagte Dr. Caleb Lareau vom Memorial Sloan Kettering Cancer Center.Zum ersten Mal verfügen wir über ein Modell, das einen langen Kontext, Einzelbasisgenauigkeit und modernste Leistung kombiniert.DeepMind hat über die AlphaGenome-API eine Vorschauversion für nichtkommerzielle Forschungsnutzer geöffnet und plant, das Modell in Zukunft offiziell zu veröffentlichen.
* Link zur Forschungsarbeit:
Basierend auf 1 Million DNA-Sequenzen und Arteninformationen, unter Verwendung eines U-Net-ähnlichen Designs
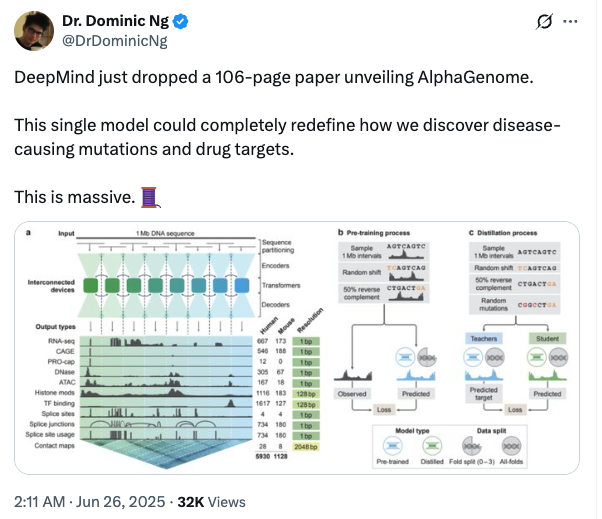
Wie in Abbildung a unten dargestellt, verwendet das Deep-Learning-Modell AlphaGenome 1 MB (Million Basen) DNA-Sequenz- und Speziesinformationen (Mensch/Maus) als Eingabe.5.930 menschliche Genomloci oder 1.128 Mausgenomloci zur Vorhersage verschiedener ZelltypenDeckt 11 Ausgabetypen ab, darunter:
* Genexpression (RNA-seq, CAGE, PRO-cap)
* Detaillierte Spleißmuster (Spleißstellen, Häufigkeit der Spleißstellennutzung, Spleißverbindungen)
* Chromatinzustand (DNase, ATAC-seq, Histonmodifikation, Transkriptionsfaktorbindung)
* Chromatin-Kontaktkarte
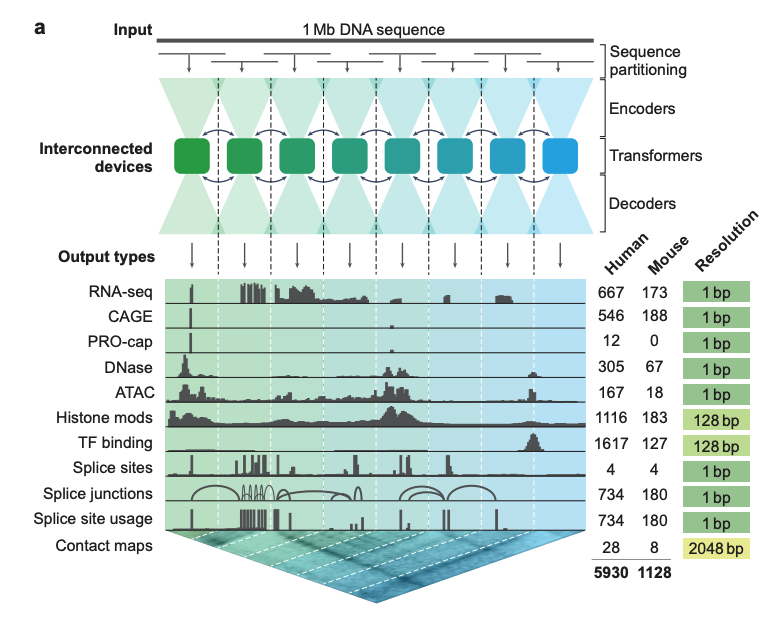
In Bezug auf die ModellarchitekturAlphaGenome verwendet ein U-Net-ähnliches Backbone-Architekturdesign.Wie in Abbildung a unten dargestellt, kann die Eingabesequenz effizient in zwei Arten von Sequenzdarstellungen verarbeitet werden:
1-dimensionale Einbettungen (1 bp und 128 bp Auflösung): stellen lineare Genomsequenzen dar und werden zur Generierung von Vorhersagen genomischer Trajektorien verwendet.
* 2-dimensionale Einbettungen (Auflösung 2048 bp): stellen die räumlichen Interaktionen zwischen Genomfragmenten dar und werden zur Vorhersage paarweiser Kontaktkarten verwendet.
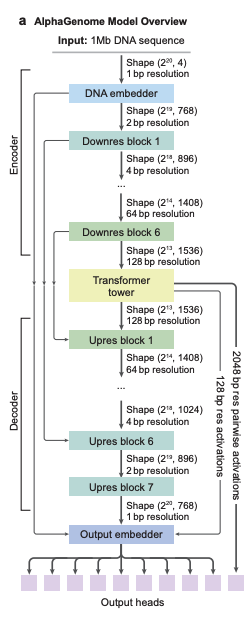
Die Faltungsschichten des Modells dienen zur Modellierung lokaler Sequenzmuster und unterstützen so feingranulare Vorhersagen, während die Transformer-Module zur Modellierung längerfristiger Abhängigkeiten, wie beispielsweise Interaktionen zwischen Enhancern und Promotoren, dienen. Dank verteilter Sequenzparallelität kann das Modell auf einer einzigen Basis einer vollständigen 1-MB-Sequenz trainiert werden und ist auf acht miteinander verbundenen TPUv3-Geräten lauffähig.
In Bezug auf das ModelltrainingDie Forscher wählten ein zweistufiges Training, nämlich Vortraining und Destillation.In der Vortrainingsphase werden die vorhandenen experimentellen Daten verwendet, um zwei Arten von Modellen zu trainieren, wie in Abbildung b unten dargestellt:
* Faltspezifische Modelle:Das Training erfolgte mit einem vierfachen Kreuzvalidierungsansatz, d. h., drei Viertel der Segmente des Referenzgenoms wurden für das Training verwendet, das verbleibende Viertel für Validierung und Tests. Diese Modelle wurden verwendet, um die Generalisierungsfähigkeit von AlphaGenome zur Vorhersage genomischer Trajektorien auf unbekannten Referenzgenomsegmenten zu bewerten.
* All-Fold-Modelle:Das Lehrermodell wird als nächste Destillationsstufe an allen verfügbaren Segmenten des Referenzgenoms trainiert, wie in Abbildung c unten dargestellt.
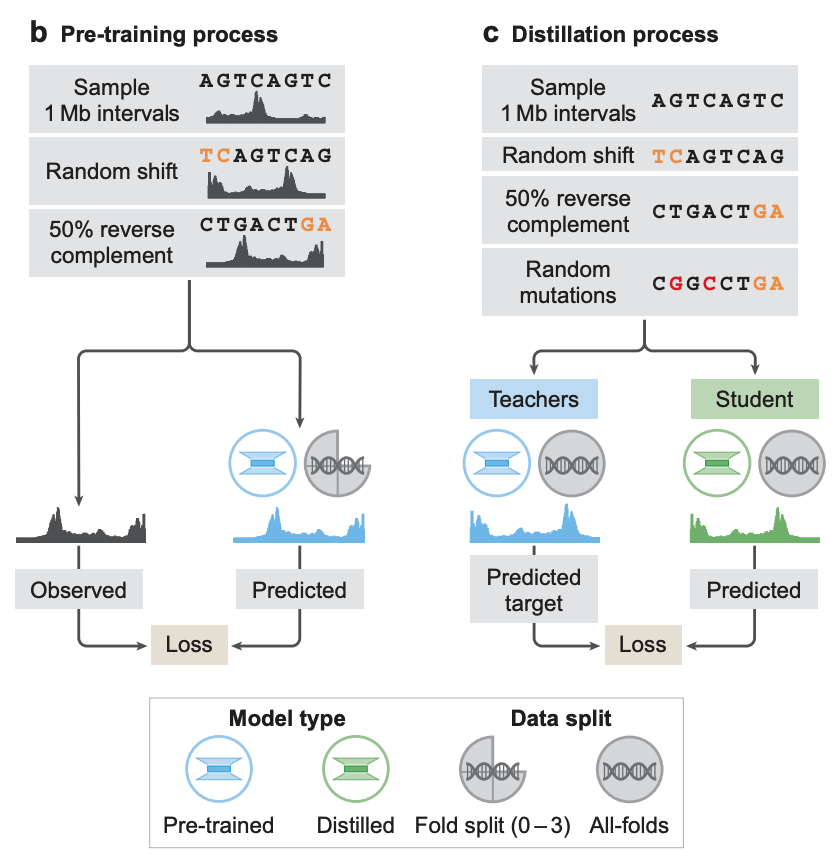
In der Destillationsphase trainierten die Forscher ein Student-Modell, das eine vortrainierte Architektur teilte.Das Ziel besteht darin, die kombinierte Ausgabe mehrerer vollständig gefalteter Lehrermodelle mithilfe zufällig erweiterter Eingabesequenzen vorherzusagen.Frühere Studien haben gezeigt, dass dieses destillierte Modell in einer Modellinstanz gleichzeitig eine stärkere Robustheit und eine höhere Genauigkeit der Variational Effect Prediction (VEP) erreichen kann.
Dank dieses Designs kann das Student-Modell die Aufgabe der Vorhersage der Variationseffekte aller Modalitäten und Zelltypen mit einem einzigen Geräteaufruf erledigen.Auf einer NVIDIA H100-GPU dauern Vorhersagen für jede Variante weniger als eine Sekunde.Dadurch ist es im Vergleich zu herkömmlichen Methoden der Multi-Modell-Integration äußerst effizient bei der Vorhersage großräumiger Variationseffekte.
AlphaGenome ist führend bei verschiedenen Aufgaben zur Genomvorhersage
Laut DeepMind bietet AlphaGenome gegenüber bestehenden Methoden folgende einzigartige Vorteile:
Langer Sequenzkontext + Einzelbasenauflösung
AlphaGenome kann DNA-Sequenzen mit einer Länge von bis zu einer Million Basen analysieren und Vorhersagen auf Einzelbasenebene treffen. Dadurch deckt es entfernte Bereiche regulatorischer Gene ab und erfasst gleichzeitig feine biologische Details. Frühere Modelle konzentrierten sich oft auf die Balance zwischen Sequenzlänge und Vorhersagegenauigkeit, was die Bandbreite und Genauigkeit der modellierbaren Modalitäten einschränkte. Der technologische Durchbruch von AlphaGenome überwindet diese Einschränkung: Das Training benötigt nur die Hälfte der Rechenressourcen des ursprünglichen Enformer-Modells und eine Trainingssitzung dauert nur vier Stunden.
Umfassende multimodale Prognosefunktionen
Durch die Kombination aus hoher Auflösung und langen Eingabesequenzen ist AlphaGenome in der Lage, beispiellos vielfältige Regulationsmuster vorherzusagen und den Forschern so systematischere Informationen zur Genregulation zu liefern.
Effiziente Mutationsbewertung
AlphaGenome kann die Auswirkungen von Varianten innerhalb einer Sekunde bewerten. Durch den Vergleich der vorhergesagten Unterschiede in den Sequenzen vor und nach der Variante und die Verwendung der jeweils am besten geeigneten Zusammenfassungsmethode für verschiedene Modalitäten kann es die potenziellen Auswirkungen genetischer Varianten auf molekulare Mechanismen schnell und präzise bewerten.
Neuartige Modellierung von Spleißstellen
AlphaGenome ermöglicht die innovative Vorhersage der Position von RNA-Spleißstellen und ihrer Expressionsniveaus direkt anhand von Sequenzen. Viele seltene genetische Erkrankungen (wie spinale Muskelatrophie und bestimmte Formen der Mukoviszidose) werden mit Spleißfehlern in Verbindung gebracht. Diese Fähigkeit bietet ein neues Instrument für die damit verbundene Ätiologieforschung.
Hervorragende Leistung in Benchmarks
AlphaGenome ist führend bei verschiedenen Genomvorhersageaufgaben, beispielsweise bei der Vorhersage der DNA-Strukturnähe, der Auswirkungen von Mutationen auf die Genexpression und von Veränderungen in Spleißmustern. Es übertraf die besten bestehenden Modelle in 22 der 24 DNA-Sequenzvorhersagebewertungen und erreichte oder übertraf die aktuell besten Modelle in 24 der 26 Mutationseffektaufgaben. Noch wichtiger ist, dass es auch das einzige Modell ist, das gemeinsame Vorhersagen für alle Bewertungsmodalitäten treffen kann und damit seine große Vielseitigkeit beweist.
Um die Leistung der Modelle von AlphaGenome zu bewerten,Die Forscher untersuchten zunächst die Generalisierungsfähigkeit auf unbekannte Genomsegmente, die eine Voraussetzung für die qualitativ hochwertige Vorhersage von Varianteneffekten ist.Sie führten insgesamt 24 Bewertungen der Vorhersage genomischer Trajektorien durch, die alle elf vom Modell vorhergesagten Modalitäten abdeckten. In der Kreuzvalidierung von Out-of-Fold-Bewertungen verwendeten die Forscher vorab trainierte, faltenspezifische AlphaGenome-Modelle und verglichen deren Vorhersagen mit dem aktuell stärksten externen Modell in jeder Aufgabe.
Die Ergebnisse zeigen, dassAlphaGenome übertraf die entsprechenden externen Modelle in 22 der 24 Bewertungen.Wie in Abbildung d unten dargestellt. Es ist erwähnenswert, dass AlphaGenome bei der Vorhersage zelltypspezifischer Veränderungen der Genexpression (log-fold change, LFC) eine relative Leistungsverbesserung von +17,4% im Vergleich zu einem anderen multimodalen Sequenzmodell, Borzoi, zeigte, wie in Abbildung e unten dargestellt.
Darüber hinaus übertraf AlphaGenome auch spezialisierte Modelle, die sich bei verschiedenen Aufgaben auf eine einzige Modalität konzentrierten. Zum Beispiel:
Bei der Vorhersage der Chromatin-Kontaktkarte,AlphaGenome übertrifft das Orca-Modell, wie ein Anstieg des Pearson-Korrelationskoeffizienten der Kontaktkarte um +6,3% und der zelltypspezifischen Unterschiede um +42,3% zeigen (siehe Abbildung d unten).
Bei der Vorhersage von Transkriptionsstartstellen-TracksAlphaGenome übertrifft ProCapNet mit einer Verbesserung des Pearson-Korrelationskoeffizienten von insgesamt +15%;
Bei der Vorhersage der ChromatinzugänglichkeitAlphaGenome übertrifft ChromBPNet und verbessert sich um +8% bei ATAC-seq und um +19% bei DNase-seq.
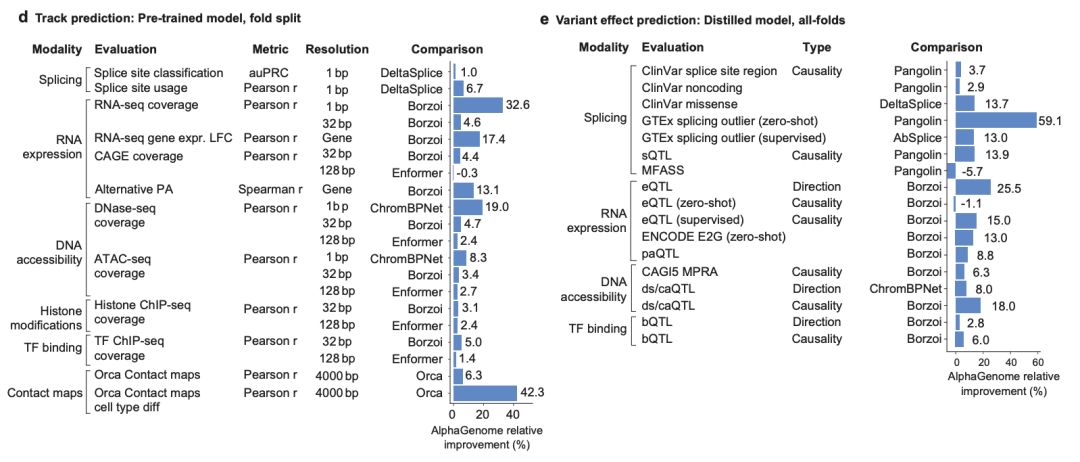
* Abbildung e: Relative Leistungsverbesserung von AlphaGenome bei der Vorhersage partieller Varianteneffekte.
Branchenmeilenstein hoch gelobt
Die Einführung dieses Blockbuster-Modells durch AlphaGenome hat seit der Veröffentlichung der Nachricht weiterhin hitzige Diskussionen auf Twitter ausgelöst.
Pushmeet Kohli, Vizepräsident für Forschung bei DeepMind, sagte: „AlphaGenome bietet einen umfassenden Überblick über das nicht-kodierende Genom des Menschen, indem es die Auswirkungen von DNA-Varianten vorhersagt.Es wird unser Verständnis der Krankheitsbiologie vertiefen und neue Forschungswege eröffnen.“Im Kommentarbereich geht es neben Ausrufen und Lob vor allem um die Verwendung.
Ein Doktorand der Genetik von der Universität Edinburgh sagte:„Dieses Modell könnte die Art und Weise, wie wir krankheitsverursachende Mutationen und Wirkstoffziele entdecken, völlig neu definieren, was von großer Bedeutung ist.“
Ein Kommentator aus dem Bereich der Biowissenschaften erklärte: „AlphaGenome ist nicht nur ein einzelnes Gen, sondern das gesamte regulatorische Genom. Wenn man DNA mit Code vergleicht, dann ist AlphaGenome die aus Code bestehende Software.“
Im Hinblick auf praktische Anwendungen verfügt AlphaGenome über ein breites wissenschaftliches Forschungspotenzial.Zum Beispiel gegenüberErforschung von Krankheitsmechanismen,Damit lassen sich die Auswirkungen genetischer Variationen auf regulatorische Prozesse präziser vorhersagen, potenzielle pathogene Variationen identifizieren und neue Angriffspunkte aufdecken. Das System eignet sich besonders für die Untersuchung seltener Variationen mit erheblichen Auswirkungen.existierenIm Bereich der synthetischen BiologieLenken Sie die Gestaltung der DNA für spezifische regulatorische Funktionen, beispielsweise die Aktivierung eines Zielgens nur in Nervenzellen.existierenIn der genomischen Grundlagenforschung kann esBeschleunigen Sie die Lokalisierung und Rollendefinition wichtiger Funktionselemente und helfen Sie bei der Identifizierung der „Kernanweisungen“, die zur Regulierung der Funktion bestimmter Zelltypen erforderlich sind.
Professor Marc Mansour vom University College London kommentierte: „AlphaGenome liefert wichtige Puzzleteile für die groß angelegte Identifizierung der Rolle nicht-kodierender Varianten und ermöglicht uns so ein besseres Verständnis komplexer Erkrankungen wie Krebs.“ AlphaGenome steht derzeit für nichtkommerzielle Forschung offen, und wir freuen uns auf weitere Erfolge der wissenschaftlichen Gemeinschaft auf dieser Grundlage.








