Command Palette
Search for a command to run...
Die Universität Cambridge Hat Einen Bildklassifikator Für Blutzellen Entwickelt; Sein Diffusionsmodell Hilft Bei Der Erkennung Von Leukämie Und Übertrifft Die Fähigkeiten Klinischer Experten.

Die Bildanalyse von Blutzellen spielt eine entscheidende Rolle in der klinischen Diagnostik und der wissenschaftlichen Forschung. Die morphologischen Merkmale von weißen und roten Blutkörperchen sowie Blutplättchen spiegeln nicht nur den Zustand des Blutsystems wider, sondern können auch frühe Krankheitszeichen wie Leukämie und myelodysplastische Syndrome aufdecken. Die traditionelle manuelle Mikroskopie ist jedoch auf die manuelle Klassifizierung durch erfahrene Experten angewiesen, was ineffizient, zeitaufwendig und anfällig für subjektive Verzerrungen ist.
In den letzten Jahren hat die Anwendung von Deep-Learning-Technologien im Bereich der medizinischen Bildanalyse zugenommen, und einige Studien haben versucht, diskriminative Modelle, insbesondere Convolutional Neural Networks (CNNs), zur Beurteilung der Morphologie von Blutzellen einzusetzen. Obwohl die leistungsstärksten diskriminativen ML-Klassifikationsmodelle die menschliche Leistung bei der Klassifizierung von Zellen in vordefinierte Kategorien annähernd erreichen können, lernen sie Entscheidungsgrenzen primär auf Basis von Expertenklassifizierungen. DaherSie sind von Natur aus nicht darauf ausgelegt, eine vollständige Datenverteilung der Zellmorphologie zu erfassen.Diese Einschränkung mindert ihre Leistungsfähigkeit, insbesondere angesichts der inhärenten Komplexität und Variabilität klinischer hämatologischer Daten.
In diesem ZusammenhangEin Forschungsteam der Universität Cambridge in Großbritannien hat CytoDiffusion vorgeschlagen, eine Methode zur Klassifizierung von Blutzellbildern, die auf einem Diffusionsmodell basiert.Es kann die morphologische Verteilung der Blutzellen getreu modellieren, eine genaue Klassifizierung erreichen und verfügt über leistungsstarke Fähigkeiten zur Erkennung von Anomalien, Resistenz gegenüber Verteilungsverschiebungen, Interpretierbarkeit, hohe Dateneffizienz und Unsicherheitsquantifizierungsfähigkeiten, die die von klinischen Experten übertreffen.
Das Modell übertrifft die modernsten Diskriminanzmodelle bei der Anomalieerkennung (AUC: 0,990 vs. 0,916), der Robustheit gegenüber Verteilungsverschiebungen (Genauigkeit: 0,854 vs. 0,738) und der Leistung in Szenarien mit geringen Datenmengen (ausgewogene Genauigkeit: 0,962 vs. 0,924).Der in dieser Studie entwickelte umfassende Bewertungsrahmen etabliert einen multidimensionalen Maßstab für die hämatologische medizinische Bildanalyse, von dem erwartet wird, dass er die diagnostische Genauigkeit im klinischen Umfeld verbessert.
Die entsprechenden Forschungsergebnisse mit dem Titel „Deep generative classification of blood cell morphology“ wurden in Nature veröffentlicht.
Forschungshighlights:
* Anwendung eines latenten Diffusionsmodells zur Klassifizierung von Blutzellbildern.
* Wir schlagen einen Bewertungsrahmen vor, der über Standardmetriken wie Genauigkeit hinausgeht und Robustheit gegenüber Verteilungsverschiebungen, Anomalieerkennungsfähigkeiten und Leistungsfähigkeit in Szenarien mit geringen Datenmengen einbezieht.
* Erstellung eines neuen Bilddatensatzes von Blutzellen, der Bildartefakte und die Konfidenz der Annotatoren berücksichtigt und damit wichtige Einschränkungen bestehender Datensätze behebt.

Papieradresse:
https://www.nature.com/articles/s42256-025-01122-7
Folgen Sie dem öffentlichen Konto und antworten Sie auf „CytoDiffusionVollständiges PDF herunterladen
Datensatzadresse:
Weitere Artikel zu den Grenzen der KI:
https://hyper.ai/papers
Datensätze: Kombination von öffentlich verfügbaren und selbst erstellten Datensätzen
Daten sind die Grundlage der Blutzellbildanalyse und eine zentrale Garantie für die Leistungsfähigkeit und Generalisierungsfähigkeit von KI-Modellen. Das CytoDiffusion-Team verwendete fünf Datensätze, von denen vier öffentlich zugänglich sind und der fünfte ein selbst erstellter Datensatz namens CytoData ist.
Der CytoData-Datensatz ist ein anonymisierter Datensatz mit 2.904 Blutausstrichen des Addenbrooke’s Hospital in Cambridge, der insgesamt 559.808 Einzelzellbilder umfasst. Davon sind 4.996 Bilder mit zehn Blutzelltypen, darunter Erythroblasten, Eosinophile, Monozyten und unreife Zellen, annotiert. Die Bilder wurden mit einem CellaVision DM9600-System aufgenommen, und die Annotationen enthalten Konfidenzwerte der jeweiligen Annotationsexperten, die eine wichtige Referenz für die nachfolgende Unsicherheitsquantifizierung darstellen. CytoData beinhaltet außerdem Artefaktkategorien, um häufige nicht-zelluläre strukturelle Störungen in Blutausstrichen zu erfassen, was für klinische Anwendungen von großem Wert ist.
Raabin-WBC, PBC, Bodzas, LISC als 4 öffentliche DatensätzeDer Datensatz umfasst Blutzellbilder, die mit verschiedenen Mikroskopen, Färbemethoden und Geräten aufgenommen wurden. Raabin-WBC stellt die Partitionen Test-A und Test-B bereit; Test-B verwendet andere Aufnahmegeräte als der Trainingsdatensatz, um eine Domänenverschiebung zu simulieren. Aufgrund der Unterschiede in den Geräten und Färbemethoden legt der LISC-Datensatz einen stärkeren Fokus auf die Generalisierungsfähigkeit des Modells.
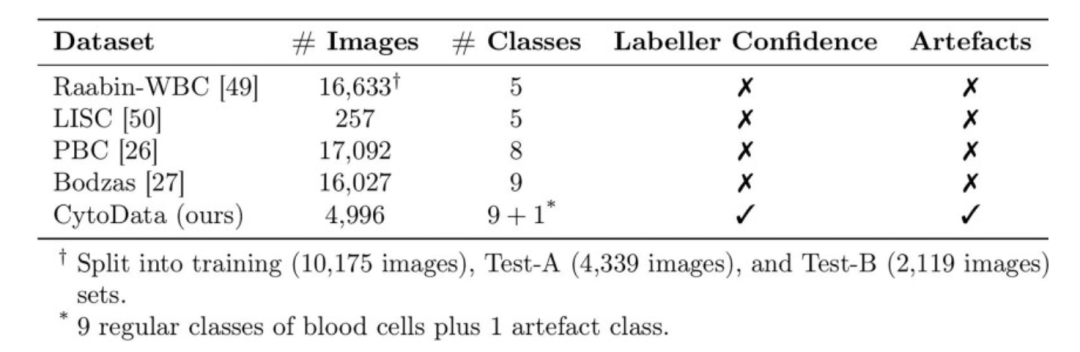
Durch die Kombination dieser Datensätze aus verschiedenen Quellen stellte das Team nicht nur die Diversität des Modelltrainings sicher, sondern schuf auch eine vollständige Grundlage für die domänenübergreifende Leistungsbewertung, die Erkennung abnormaler Zellen und das Testen des Modells unter datenarmen Bedingungen.
Datensatzadresse:
Rahmenkonzept: Anwendung eines Diffusionsmodells zur Klassifizierung von Blutzellbildern
Die Kerninnovation von CytoDiffusion besteht in der Anwendung eines Diffusionsmodells zur Klassifizierung von Blutzellbildern.Im Gegensatz zu traditionellen diskriminativen Modellen besitzen Diffusionsmodelle generative Eigenschaften, die es ihnen ermöglichen, die vollständige Verteilung eines Bildes zu erlernen und es mithilfe eines Rauschvorhersagemechanismus zu klassifizieren.
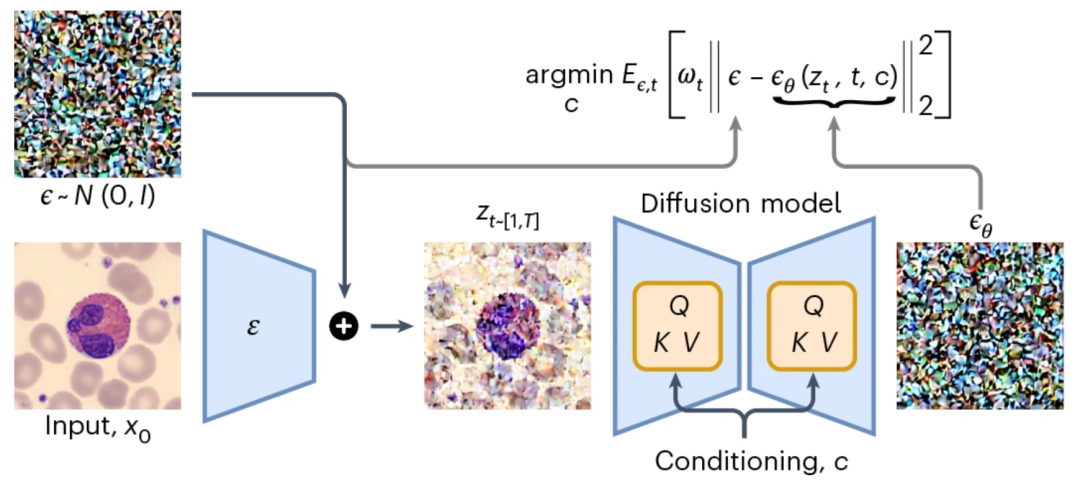
Modellprinzipien
Das Kernprinzip des Diffusionsmodells besteht darin, einen Vorwärtsdiffusionsprozess zu definieren, der die Daten durch schrittweises Hinzufügen von Rauschen in eine rauschähnliche Verteilung transformiert. Anschließend lernt das Modell einen inversen Prozess, um die Daten zu entrauschen und so die ursprüngliche Datenverteilung effektiv zu rekonstruieren.
Kodierung des latenten Raums:Das Eingabebild wird zunächst durch einen Encoder in den latenten Raum abgebildet, anschließend wird Gaußsches Rauschen hinzugefügt, um eine verrauschte latente Repräsentation zu erzeugen;
Bedingte Diffusion:Das Modell generiert Rauschvorhersagen für jeden Zelltyp und erreicht eine Klassifizierung, indem es den Fehler zwischen dem vorhergesagten Rauschen und dem tatsächlichen Rauschen minimiert.
Nach und nach eliminieren:Es wird eine iterative Stichprobenziehung auf allen Kandidatenkategorien durchgeführt, und unmögliche Kategorien werden schrittweise mithilfe des gepaarten t-Tests nach Student eliminiert, bis die endgültige Kategorie bestimmt ist.
Allgemeine Trainingsumgebungen
Die Forscher verwendeten Stable Diffusion 1.5 als Basismodell.Bei kategoriebasierten Bedingungen umgeht das Verfahren den Tokenizer und den Text-Encoder und liefert direkt One-Hot-kodierte Vektoren für jede Kategorie. Diese Vektoren werden vertikal kopiert und horizontal aufgefüllt, um der erwarteten 77 × 768-dimensionalen Matrix zu entsprechen. Es wurden eine Batchgröße von 10, eine Lernrate von 10⁻⁵ und ein lineares Warm-up von 1000 Schritten verwendet. Das Training erfolgte auf einer A100-80GB-GPU.
Ausbildung und logisches Denken
Die Forscher verwendeten während des Trainings eine Vielzahl von Datenerweiterungsmethoden, darunter zufällige diagonale Spiegelungen, zufällige Drehungen (gleichmäßige Abtastung zwischen 0 und 359 Grad), Farbjitter (Helligkeit = 0,25, Kontrast = 0,25, Sättigung = 0,25, Farbton = 0,125), Mixup (α = 0,3, angewendet auf bedingte Eingaben anstatt auf das Ziel) und RandAugment (unter Verwendung der Standardparameter).
Das Training erfolgte mit dem AdamW-Optimierer (β1 = 0,9, β2 = 0,999, ϵ = 10⁻⁸, Gewichtungsabfall 0,01), gemischter Präzision (fp16) und exponentiellem gleitenden Durchschnitt (0,9999). Alle Bilder wurden einheitlich auf 360 × 360 Pixel skaliert.
Während der Inferenzphase wurden dieselben Datenaugmentierungsmethoden wie in der Trainingsphase angewendet, Mixup wurde jedoch ausgeschlossen. Da sich weiße Blutkörperchen typischerweise im Bildzentrum befinden und um Störungen durch die Augmentierung von Bildrandbereichen zu minimieren, wurde der Inferenzfehler nur innerhalb eines Radius von 20 Pixeln um das Bildzentrum im latenten Raum berechnet. Die Dimension des latenten Raums betrug 45 × 45 × 4.
Ergebnispräsentation: CytoDiffusion kann dazu beitragen, zentrale Herausforderungen bei der klinischen Anwendung zu bewältigen.
Bildgenerierung und Echtheitsprüfung
Die klinische Anwendung von Systemen der künstlichen Intelligenz erfordert nicht nur hohe Leistungsfähigkeit, sondern auch zuverlässige Repräsentationsfähigkeiten der Modelle. Um zu demonstrieren, dass CytoDiffusion die tatsächliche morphologische Verteilung von Blutzellen erlernt und nicht auf „Abkürzungen“ wie Artefakte zurückgreift, führten die Forscher einen Realismustest durch.
Basierend auf 32.619 Trainingsbildern sind die von CytoDiffusion generierten Blutzellbilder praktisch nicht von realen Bildern zu unterscheiden. Zehn Hämatologen führten Identifizierungstests an 2.880 Bildern durch und erzielten eine Gesamtgenauigkeit von 0,523 (entspricht etwa einem Zufallstreffer), eine Sensitivität von 0,558 und eine Spezifität von 0,489. Diese Ergebnisse liegen nahe am Zufallstreffer und zeigen, dass selbst erfahrene Fachleute die von CytoDiffusion generierten Blutzellbilder praktisch nicht von realen Bildern unterscheiden können.
Die Fähigkeit, Bilder zu erzeugen, die von realen Bildern kaum zu unterscheiden sind, zeigt, dass CytoDiffusion die wahre Verteilung der Blutzellmorphologie erfolgreich erlernt hat, wie in der folgenden Abbildung dargestellt:
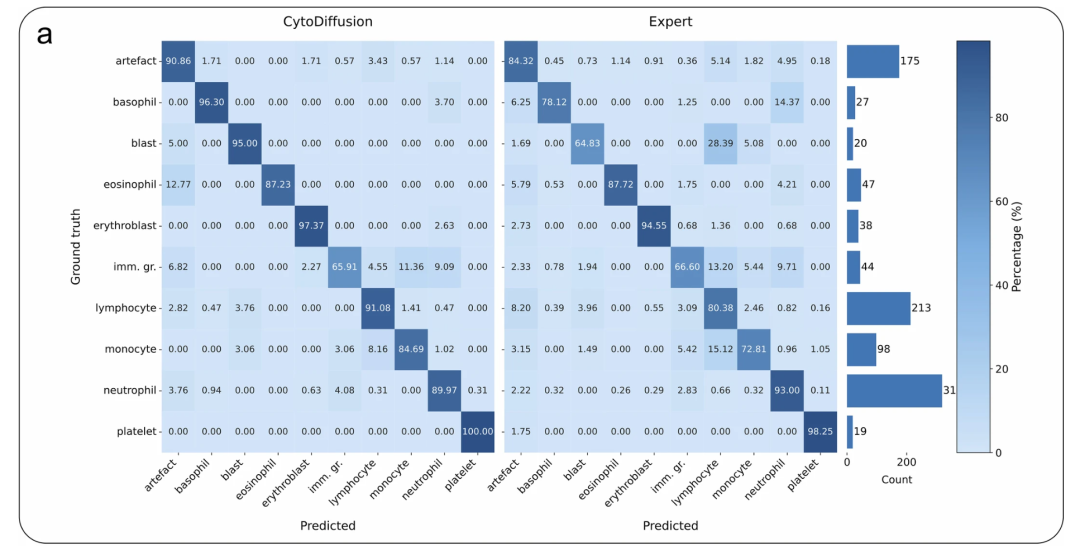
CytoData-Vergleich: Die linke Matrix zeigt die Ergebnisse von CytoDiffusion, die rechte Matrix die durchschnittliche Leistung menschlicher Experten.
Klassifizierungsleistung
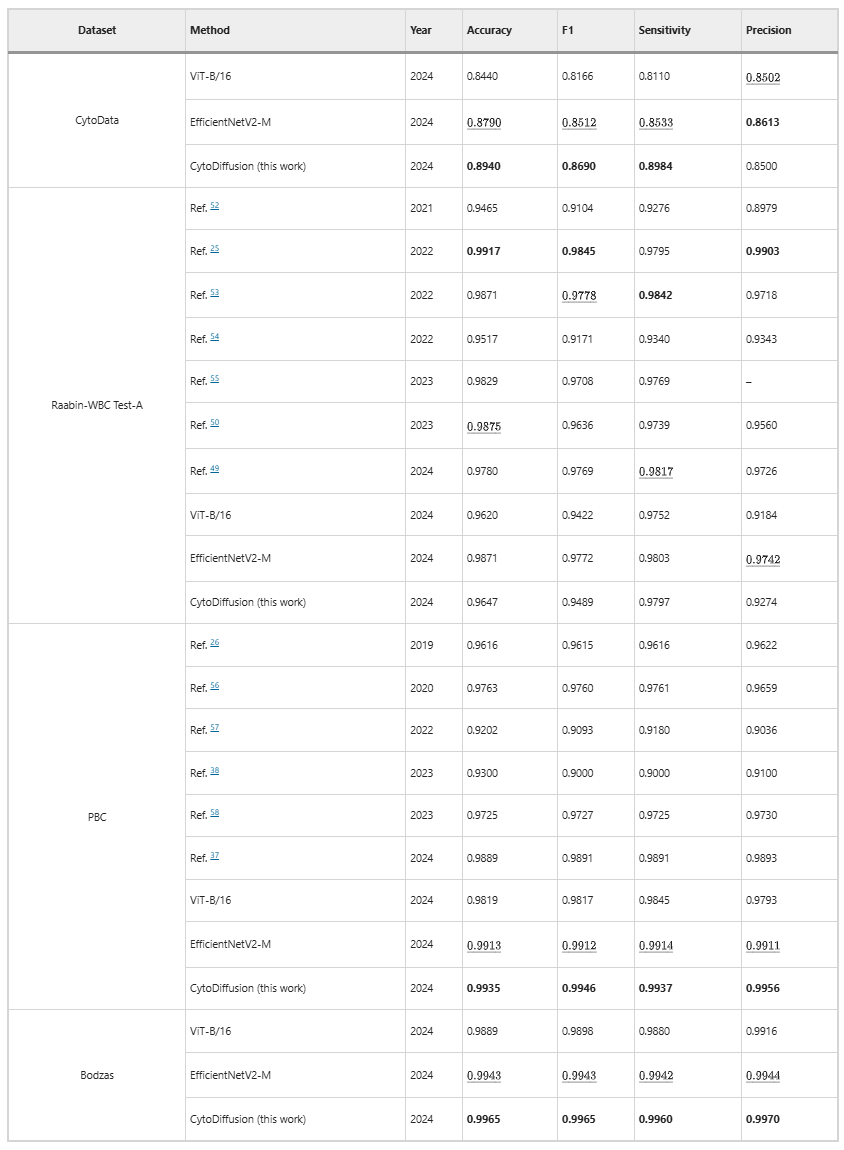
Auf vier Datensätzen (CytoData, Raabin-WBC, PBC und Bodzas) erreicht oder übertrifft CytoDiffusion die Leistung traditioneller diskriminativer Modelle. Insbesondere auf CytoData, PBC und Bodzas erzielt das Modell Spitzenleistungen und beweist damit, dass diffusionsbasierte Methoden mit traditionellen diskriminativen Modellen mithalten oder diese sogar übertreffen können, wie in der folgenden Tabelle dargestellt:
Die Unsicherheitsquantifizierung ist derjenigen menschlicher Experten überlegen.
Biologische Systeme weisen naturgemäß eine nicht reduzierbare Unsicherheit auf. Bei jeder analytischen Aufgabe sollte die Messung nicht nur die Genauigkeit der Vorhersagen, sondern auch die Unsicherheit des Akteurs (ob Mensch oder Maschine) berücksichtigen.
Die Forscher verwendeten etablierte Bayes'sche psychometrische Modellierungstechniken, um die psychometrische Funktion von CytoDiffusion abzuleiten, wie in der folgenden Abbildung dargestellt.Die Ergebnisse zeigen, dass das Modell sehr gut passt und die A-posteriori-Verteilungen der wichtigsten Schwellenwert- und Breitenparameter sehr kompakt sind (eingebettete Koordinatenachsen in der Abbildung unten).Obwohl sie nicht direkt messbar ist, legen diese Ergebnisse nahe, dass die Unsicherheit von CytoDiffusion hauptsächlich von einer Zufallskomponente dominiert wird und ihr Verhalten dem eines idealen Beobachters sehr ähnlich ist.
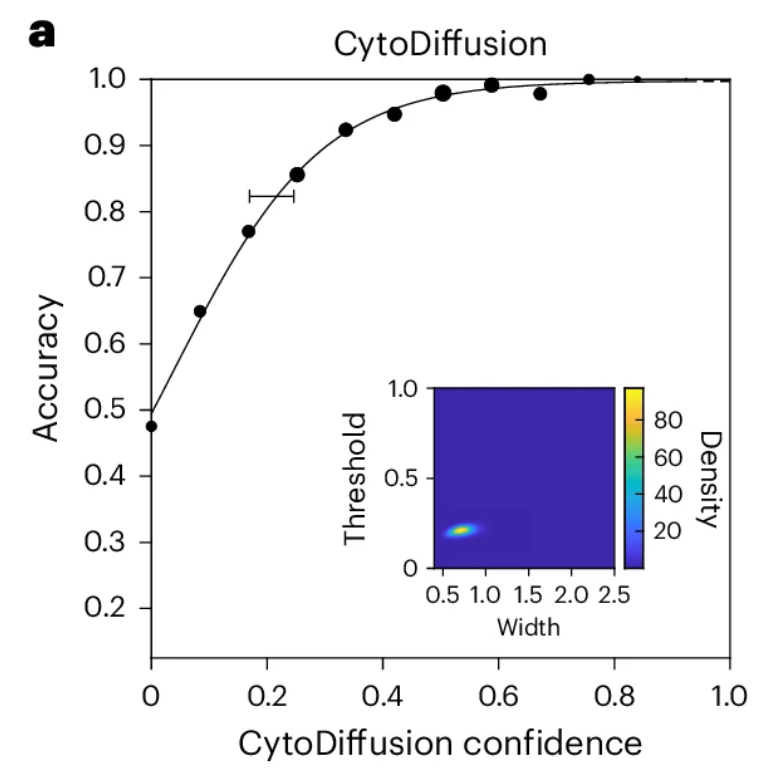
Anomalienerkennung und Leistung in Umgebungen mit geringer Datenmenge
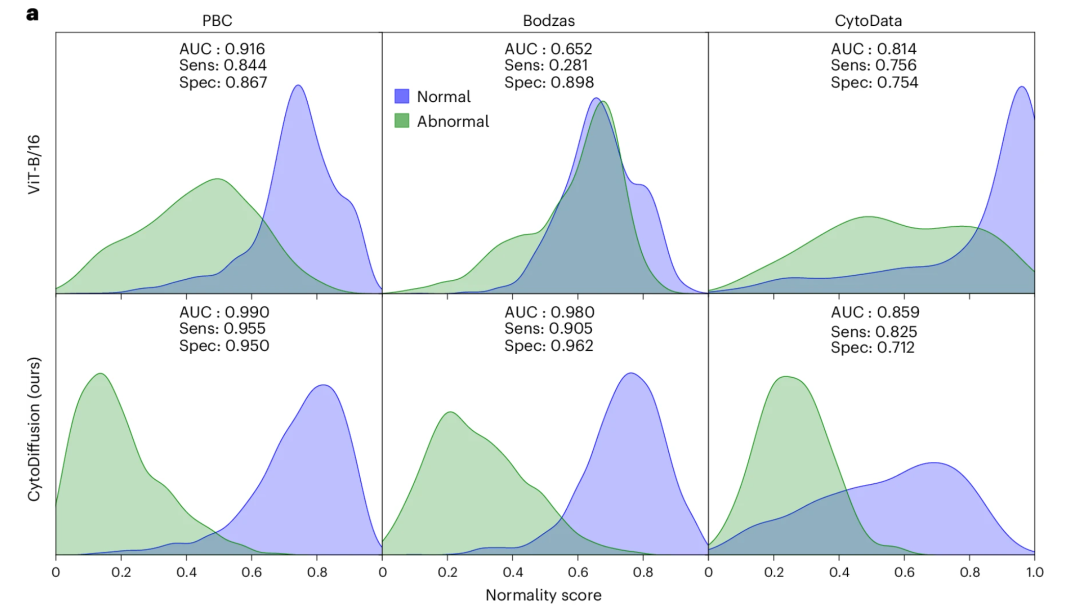
Für die Erkennung abnormaler Zellen erreichte CytoDiffusion eine hohe Sensitivität (0,905) und Spezifität (0,962), wenn primitive Zellen als Kategorie für Anomalien im Bodzas-Datensatz verwendet wurden. Im Gegensatz dazu zeigte ViT eine extrem niedrige Sensitivität (0,281) und erfüllte damit eindeutig nicht die Anforderungen klinischer Anwendungen, wie in der folgenden Abbildung dargestellt:
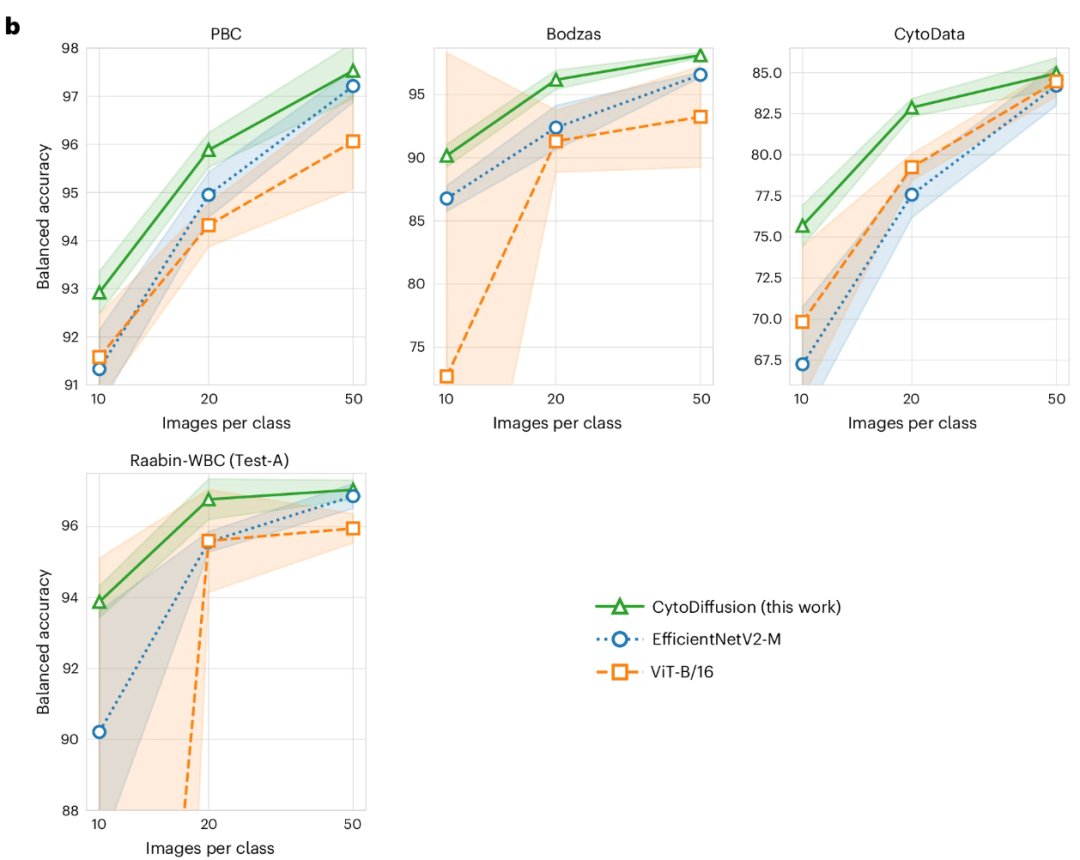
In Umgebungen mit wenigen Daten, mit nur 10–50 Trainingsbildern pro Klasse, übertrifft CytoDiffusion EfficientNetV2-M und ViT-B/16 deutlich und demonstriert damit seine effiziente Lernfähigkeit unter datenarmen Bedingungen, wie in der folgenden Abbildung dargestellt:
Generalisierungsfähigkeit des Modells
Um die Generalisierungsfähigkeit des Modells zu evaluieren, testeten die Forscher seine Leistungsfähigkeit anhand verschiedener Datenbereiche. Modelle, die mit Raabin-WBC trainiert wurden, wurden mit den Datensätzen Test-B (unter Verwendung verschiedener Mikroskope und Kameras) und LISC (unter Verwendung verschiedener Mikroskope, Kameras und Färbemethoden) getestet; Modelle, die mit CytoData trainiert wurden, wurden mit PBC und Bodzas getestet. CytoDiffusion erzielte auf allen vier Datensätzen eine herausragende Genauigkeit.Dieser Konsistenzvorteil über verschiedene Grade von Domänenabweichungen hinweg beweist, dass CytoDiffusion robust gegenüber Datensatzvariationen ist und eine gute Generalisierungsfähigkeit in realen klinischen Szenarien besitzt.
Erklärbarkeitsprüfung
Mithilfe einer kontrafaktischen Heatmap-Analyse kann das Modell wichtige morphologische Merkmale innerhalb von Zellen identifizieren, wie in der Abbildung unten dargestellt. Beispielsweise hebt das Modell beim Übergang von Monozyten zu unreifen Granulozyten Unterschiede in der Zytoplasma-Azidität und der Vakuolenfüllung hervor. Diese Visualisierung bestätigt nicht nur die Lernfähigkeit des Modells, sondern kann auch zur Erkennung potenzieller Verzerrungen genutzt werden, um sicherzustellen, dass die Klassifizierungskriterien mit der klinisch-wissenschaftlichen Logik übereinstimmen.
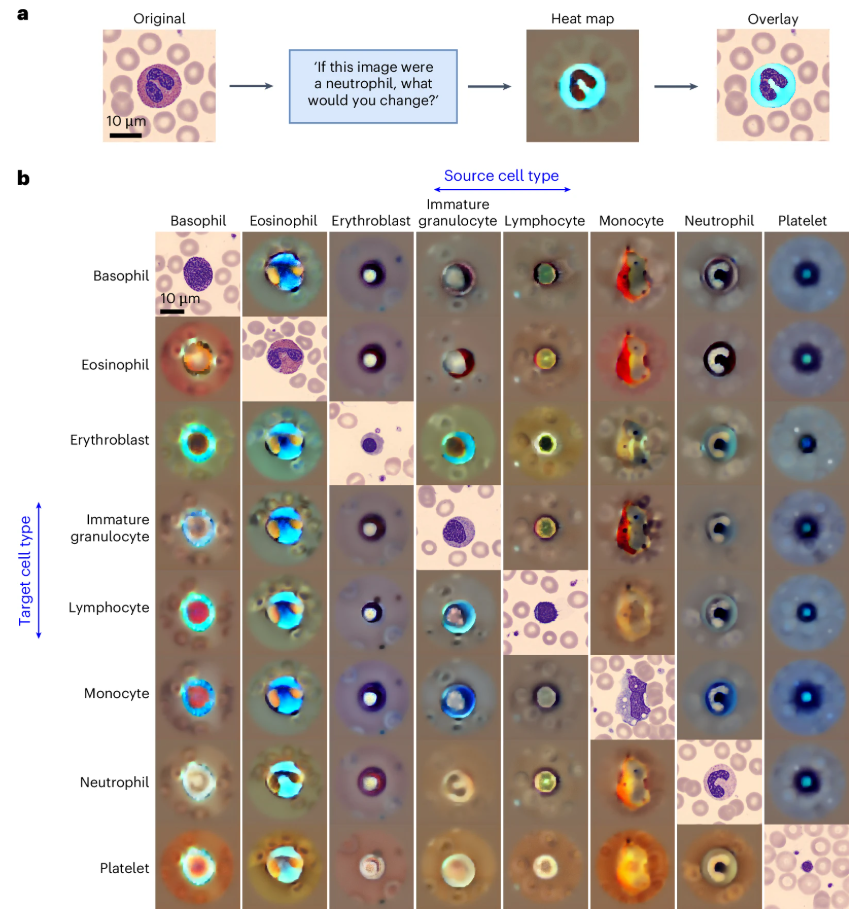
Diffusionsmodelle erweisen sich im biomedizinischen Bereich als bahnbrechend.
Die Forschung von CytoDiffusion demonstriert nicht nur das Potenzial von Diffusionsmodellen bei der Klassifizierung der Morphologie von Blutzellen, sondern spiegelt auch den rasanten Aufstieg diffusionsbasierter generativer Frameworks im gesamten biomedizinischen Bereich wider und zeigt bahnbrechenden Wert in vielfältigen Anwendungsszenarien auf.
Beispielsweise sind medizinische Daten oft begrenzt und mit erheblichen Datenschutzbedenken behaftet, was die Datenerfassung und -annotation zu einer großen Herausforderung macht. Diffusionsmodelle können dieses Problem durch die Generierung synthetischer medizinischer Bilder lösen.Dies hilft beim Training von Deep-Learning-Modellen und verbessert die Genauigkeit der medizinischen Bildanalyse;Neben der Erzeugung gewöhnlicher medizinischer Bilder wird die Erweiterung...Das Dispersionsmodell kann auch zur Erzeugung medizinischer Bilder für spezifische Krankheitsbilder (wie Tumore, Frakturen usw.) verwendet werden.Dies ist besonders wichtig beim Training medizinischer Diagnosemodelle, da es mehr Trainingsbeispiele für seltene Krankheiten oder schwer zu beschaffende Bilder liefert. Gleichzeitig können Diffusionsmodelle qualitativ hochwertige, klare und realistische Bilder erzeugen, was nicht nur die diagnostische Genauigkeit von Ärzten verbessert, sondern auch medizinischen KI-Systemen hilft, präzisere Vorhersagen zu treffen.
In vielen klinischen und Forschungsszenarien schränkt der Mangel an qualitativ hochwertigen medizinischen Bilddatensätzen das Potenzial künstlicher Intelligenz (KI) in klinischen Anwendungen ein. Im Dezember 2024 waren Professor Kang Zhang und Professor Jia Qu vom Augenkrankenhaus der Medizinischen Universität Wenzhou sowie Forscher Jinzhuo Wang von der Universität Peking die korrespondierenden Autoren, während Dr. Kai Wang von der Universität Peking und Dr. Yunfang Yu vom Sun-Yat-sen-Gedächtniskrankenhaus der Sun-Yat-sen-Universität als Erstautoren fungierten.Es wurde ein neuartiges Framework namens MINIM entwickelt, das auf einem Diffusionsmodell basiert. Dieses Modell kann anhand von Textbefehlen medizinische Bilder verschiedener Bildgebungsmodalitäten unterschiedlicher Organe generieren.Klinische Beurteilungen und strenge objektive Messungen bestätigten die hohe Qualität der von MINIM generierten Bilder. Angesichts neuartiger Datenbereiche demonstrierte MINIM verbesserte generative Fähigkeiten und stellte damit sein Potenzial als allgemeine medizinische KI (GMAI) unter Beweis.
Titel des Papiers:Selbstlernendes generatives Grundlagenmodell zur Erzeugung synthetischer medizinischer Bilder und für klinische Anwendungen
Papieradresse:https://www.nature.com/articles/s41591-024-03359-y
In der Zellbiologie sind lebende Zellen komplexe, dissipative Systeme, die weit vom chemischen Gleichgewicht entfernt sind. Wie sie kollektiv auf äußere Reize reagieren, ist seit jeher eine zentrale wissenschaftliche Frage, die Forscher zu entschlüsseln suchen. November 2025Forschungsteams der Columbia University, der Stanford University und anderer Institutionen haben das Squidiff-Rechenframework entwickelt.Dieses auf einem bedingt entrauschten Diffusionsmodell basierende Framework kann transkriptomische Reaktionen in verschiedenen Zelltypen unter Differenzierungsinduktion, Genperturbation und medikamentöser Behandlung vorhersagen. Sein Hauptvorteil liegt in der Integration expliziter Informationen aus Genomeditierungswerkzeugen und Wirkstoffen: Bei der Vorhersage der Stammzelldifferenzierung erfasst es nicht nur transiente Zellzustände präzise, sondern identifiziert auch nicht-additive Genperturbationseffekte und zellspezifische Reaktionscharakteristika. Das Forschungsteam wandte Squidiff zudem in der Forschung an vaskulären Organoiden an und sagte erfolgreich die Auswirkungen von Strahlenexposition auf verschiedene Zelltypen voraus sowie die Schutzwirkung strahlenschützender Medikamente.
Titel des Papiers:Squidiff: Vorhersage der zellulären Entwicklung und Reaktionen auf Störungen mithilfe eines Diffusionsmodells
Papieradresse:https://www.nature.com/articles/s41592-025-02877-y
Es ist absehbar, dass mit der Weiterentwicklung generativer Basismodelle im medizinischen Bereich Diffusionsmodelle in immer mehr realen klinischen Szenarien eingesetzt werden und somit eine wichtige Grundlage für die allgemeine medizinische Intelligenz bilden. Dies führt zu höherer Zuverlässigkeit, stärkerer Generalisierungsfähigkeit und einem breiteren Anwendungsbereich für die zukünftige medizinische Bilddiagnostik, Krankheitsvorhersage und intelligente Entscheidungsfindung.
Quellen:
1.https://www.nature.com/articles/s42256-025-01122-7
2.https://www.nature.com/articles/s41592-025-02877-y
3.https://mp.weixin.qq.com/s/9JEt-QwFxngv9XC0hSIcnw
4.https://bbs.huaweicloud.com/blogs/448218








