Command Palette
Search for a command to run...
Erfahren Sie in Einem Artikel Mehr Über Den PDB-Proteinstrukturdatensatz Hinter AlphaFold 2
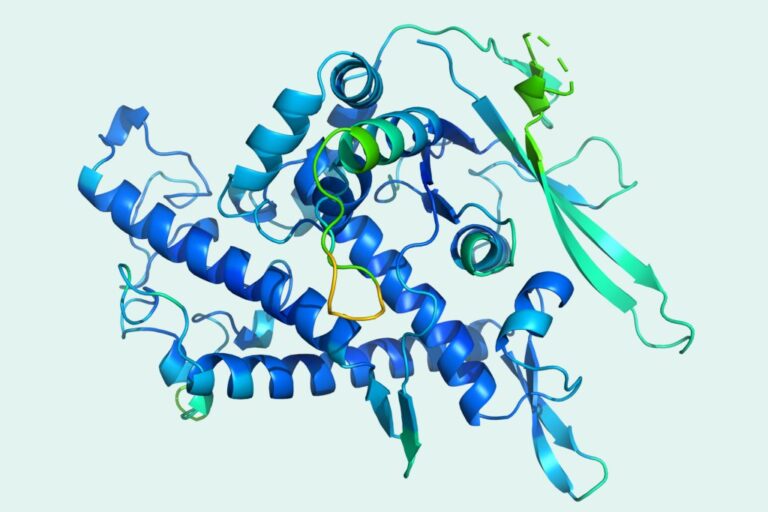
Der Algorithmus der neuesten Generation von DeepMind, AlphaFold 2, hat die Konkurrenz bei CASP, das kürzlich als „Olympiade der Proteine“ bezeichnet wurde, deutlich geschlagen und erstaunliche Durchbrüche erzielt, die die gesamte wissenschaftliche Forschungsszene schockierten. Nachdem wir von diesem wissenschaftlichen Forschungsergebnis überwältigt sind, werfen wir einen Blick auf den Datensatz hinter dem Algorithmus.
In den letzten zwei Tagen wurden wir mit Nachrichten über DeepMinds neuen Algorithmus für künstliche Intelligenz AlphaFold 2 bombardiert, insbesondere im biologischen Bereich, der einen Meilenstein in der Entwicklung darstellt.
Laut der offiziellen Ankündigung von DeepMind hat sein Deep-Learning-Algorithmus AlphaFold 2 in den letzten 50 Jahren große Probleme im biologischen Bereich erfolgreich gelöst.
Der Algorithmus kann die 3D-Struktur von Proteinen anhand ihrer Aminosäuresequenzen präzise vorhersagen, und zwar mit einer Genauigkeit, die mit 3D-Strukturen vergleichbar ist, die mithilfe experimenteller Techniken wie Kryo-Elektronenmikroskopie (Kryo-EM), Kernspinresonanz oder Röntgenkristallographie ermittelt werden.

Dieses bahnbrechende Ereignis begeisterte die Biologen, versetzte aber auch viele Leute in der Branche in Angst und Schrecken, und sie sagten, sie würden den Beruf wechseln, um Deep Learning zu erlernen.
Doch während alle diesem wissenschaftlichen Forschungsergebnis Aufmerksamkeit schenken, sollten wir den Helden dahinter nicht vergessen—— PDB-Proteinstrukturdatensatz, ein Datensatz zum Sammeln dreidimensionaler Strukturdaten von Proteinen und Nukleinsäuren.
Dieser Datensatz ist für einen bahnbrechenden Durchbruch unerlässlich
Laut DeepMind hat das Team das System anhand öffentlicher Daten trainiert.Diese Daten stammen aus dem Proteinstrukturdatensatz PDB und der großen Datenbank UniProt mit Proteinsequenzen unbekannter Struktur, die zusammen etwa 170.000 Proteinstrukturen umfassen.
In,PDB ist ein Datensatz, der sich der dreidimensionalen Struktur von Proteinen und Nukleinsäuren widmet. Es hat eine sehr lange Geschichte, die bis ins Jahr 1971 zurückreicht.
Walter Hamilton vom Brookhaven National Laboratory in den Vereinigten Staaten beschloss, diese Datenbank einzurichten. Im Oktober 1998 wurde das PDB an das Research Collaboratory for Structural Bioinformatics (RCSB) übertragen, das von Helen M. Berman von der Rutgers University geleitet wurde, die ebenfalls Mitglied des RCSB war.

Im Jahr 2003Aus der PDB entwickelte sich eine internationale Organisation, wwPDB (Worldwide Protein Database), die die PDB-Ressourcen überwacht. Andere Mitglieder von wwPDB, darunter PDBe (Europa), RCSB (USA) und PDBj (Japan), stellen ebenfalls Zentren für die Datensammlung, -verarbeitung und -veröffentlichung für PDB bereit.

Es ist erwähnenswert, dass die Daten in PDB zwar von Wissenschaftlern aus der ganzen Welt übermittelt werden, alle übermittelten Daten jedoch von wwPDB-Mitarbeitern überprüft und mit Anmerkungen versehen werden, um festzustellen, ob die Daten plausibel sind. Die PDB und die darin enthaltene Software stehen der Öffentlichkeit nun kostenlos zur Verfügung.
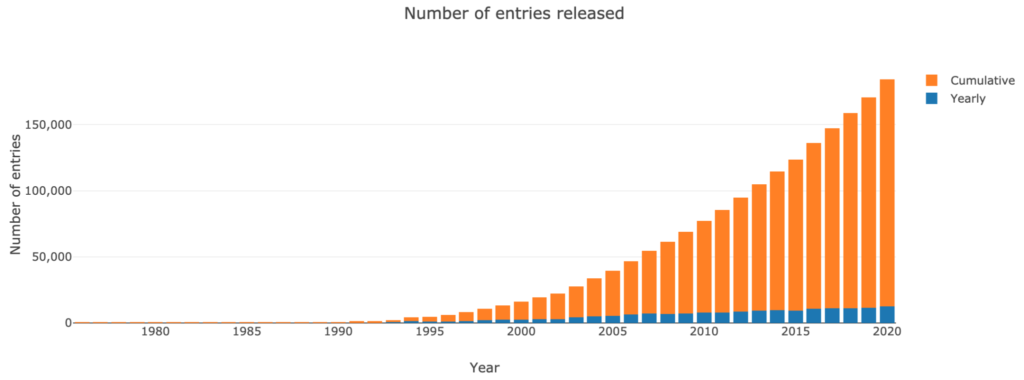
Mehr als 140.000 Strukturen, welche Informationen sind im PDB enthalten?
In den letzten Jahrzehnten ist die Anzahl der Strukturen im PDB nahezu exponentiell gewachsen:
- 100 im Jahr 1982;
- 1.000 im Jahr 1993;
- 10.000 im Jahr 1999;
- 100.000 im Jahr 2014.
Seit 2007 scheint sich die Rate, mit der neue Proteinstrukturen angesammelt werden, jedoch stabilisiert zu haben.
Strukturbiologen auf der ganzen Welt verwenden Methoden wie Röntgenkristallographie, NMR-Spektroskopie und Kryo-Elektronenmikroskopie, um die Position jedes Atoms in einem Molekül relativ zueinander zu bestimmen. Anschließend übermitteln sie diese Strukturinformationen, die von wwPDB kommentiert und öffentlich in der Datenbank veröffentlicht werden.
Sie können im PDB-Datensatz nach Strukturen von Ribosomen, Onkogenen, Arzneimittelzielen und sogar ganzen Viren suchen.Allerdings ist die Suche nach den benötigten Informationen aufgrund der schieren Anzahl der im PDB archivierten Strukturen eine gewaltige Aufgabe.
Die Informationen im PDB-Datensatz umfassen hauptsächlich:Die Quelle des Proteins/der Nukleinsäure, die Zusammensetzung der Protein-/Nukleinsäuremoleküle, Atomkoordinaten, zur Bestimmung der Struktur verwendete experimentelle Methoden sowie andere Daten und Informationen wie Temperaturfaktoren und Strukturdeterminatoren.
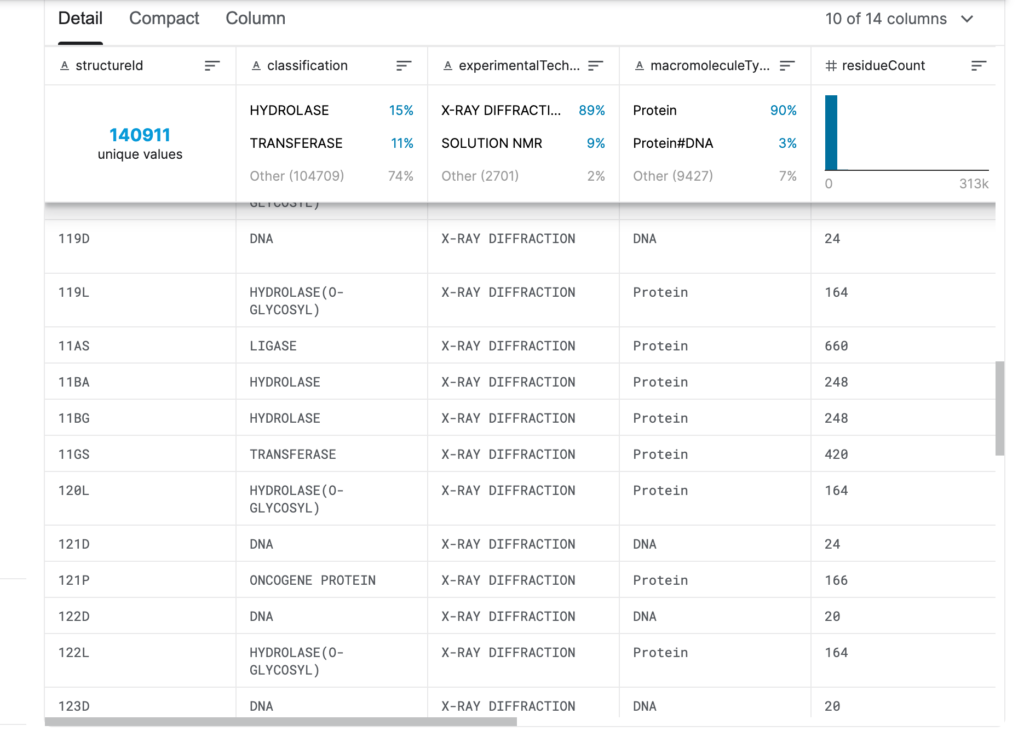
Wie lade ich das herunter?
Der Datensatz ist jetzt auf der offiziellen Website von Hyperneuron und openbayes.com verfügbar. Besuchen:https://orion.hyper.ai/datasets/13906 Oder klicken Sie auf „Originaltext lesen“, um den Datensatz mit einem Klick zu erhalten.
■ Details zum PDB-Proteinstrukturdatensatz
Veröffentlichungszeit:Gesammelt seit 1971
Verlag:wwPDB
Enthaltene Menge:Über 140.000 Protein-/Nukleinsäurestrukturen
Datenformat:CSV-Datei
Datengröße:27 MB (146 MB nach der Dekomprimierung)
Downloadadresse:https://orion.hyper.ai/datasets/13906
Derselbe Datensatz wie DeepMind, Sie verdienen ihn auch~
Wie benutzt man?
Unser Partner OpenBayes ist ein Cloud-Dienst, der Cloud-Rechenleistung für maschinelles Lernen bereitstellt. Sie verfügen über einen großen Supercomputing-Cluster und die GPU-Clusterarchitektur ist speziell für Matrix-Computing konzipiert. Es stellt Rechenleistungscontainer für KI-Anwendungen bereit, ist sehr einfach zu starten und sofort einsatzbereit.
Derzeit unterstützen die Containerprodukte von OpenBayes bereits TensorFlow, PyTorch, MXNet und andere CPU- und GPU-Umgebungen, verschiedene Versionen und Typen von Standard-Frameworks für maschinelles Lernen und verschiedene gemeinsame Abhängigkeiten.
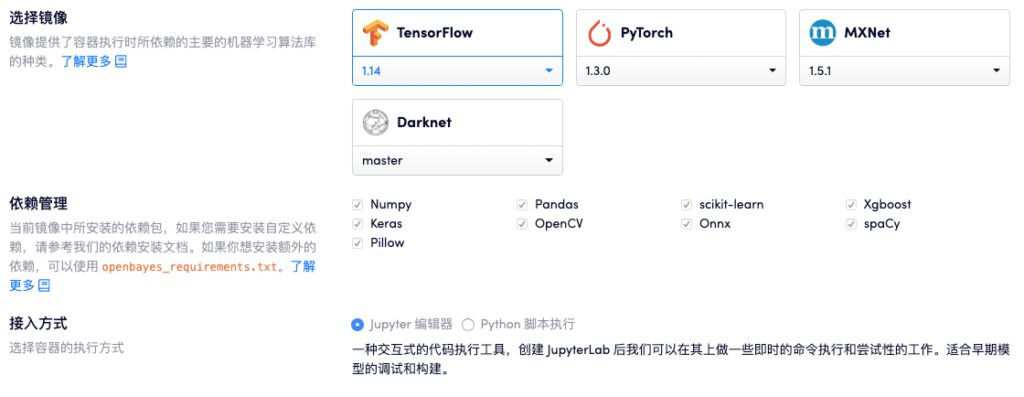
Derzeit unterstützt der OpenBayes-Computing-Container Standardbibliothekenund bieten CPU, NVIDIA T4, NVIDIA Tesla V100 und andere RechenressourcenUnabhängig davon, ob es sich um das zentrale Training großer Datenmengen oder den stromsparenden Betrieb von Modellen handelt, können die Benutzeranforderungen problemlos erfüllt werden.

Von CPU über T4 bis V100, eine breite Palette an Computing-Container-Konfigurationen OpenBayes-UnterstützungSkript-Upload und JupyterLab-EditorOnline-Programmierung und anschließend Modelltraining.
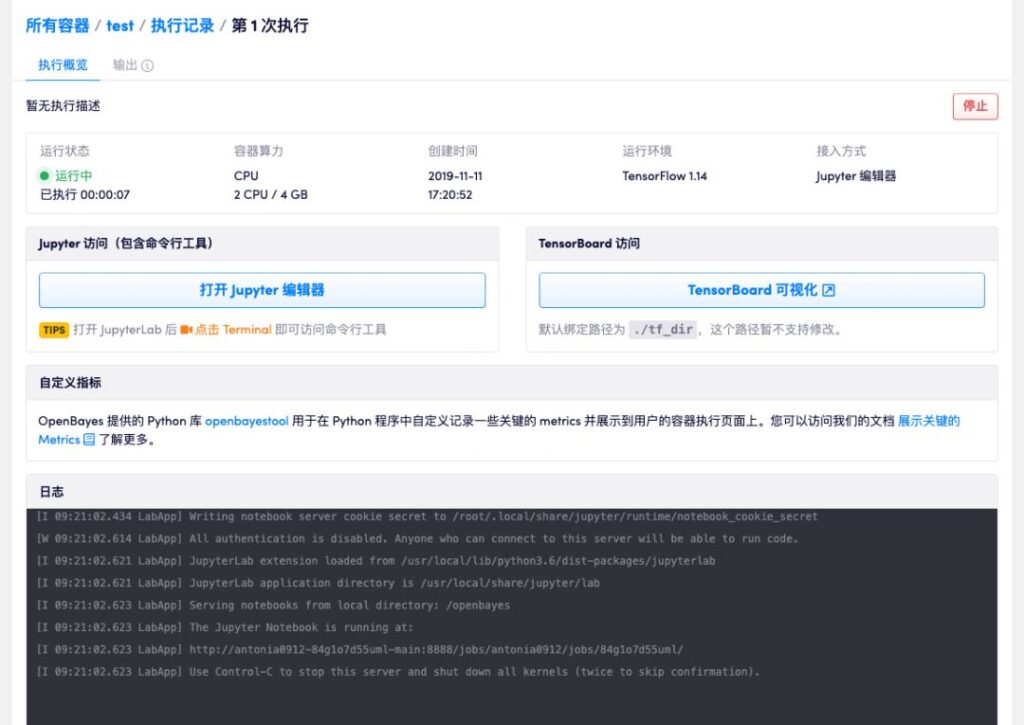
Klarer und prägnanter AusführungsprozessVollständiges Tutorial: https://openbayes.com/docs/quickstart/
Registrieren Sie sich als neuer Benutzer, um die GPU-Rechenleistung zu nutzen
openbayes.com besuchen, klicken Sie auf die offizielle Website, um sich sofort zu registrieren. Während der internen Testphase gibt es wöchentliche Geschenke, sodass Sie nicht mit Klassenkameraden und Kollegen um Rechenleistung konkurrieren müssen ~
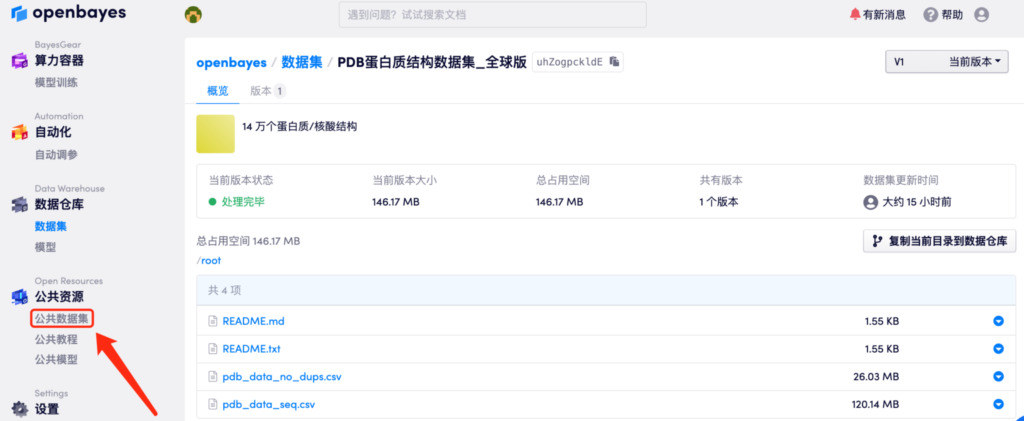
Veranstaltungsbeschreibung openbayes.com besuchen Registrieren Sie sich als neuer Benutzer mit dem Einladungscode [HyperAI]Sie können genießen
Freies CPU-Kontingent:300 Minuten/Woche
Freies vGPU-Kontingent:180 Minuten/Woche
Erfassung des vollständigen PDB-Datensatzes:
https://www.rcsb.org/#Category-download
Die Dateien im PDB-Datensatz können direkt mit einem Texteditor angezeigt werden. Besser ist jedoch die Verwendung eines Visualisierungstools. Das offiziell empfohlene Anzeigeprogramm ist der Swiss PDB Viewer:
https://spdbv.vital-it.ch/disclaim.html#
Weitere Referenzen:
https://www.novopro.cn/articles/201912021193.html
-- über--








