Command Palette
Search for a command to run...
تم اختياره لـ AAAI 2025! كانت جامعة تسينغهوا/جامعة كلية لندن رائدة في حل نموذج اللغة البروتينية-الحمض النووي الريبوزي، من خلال الجمع بين التنبؤ بالتقارب لتحديث SOTA
مرض الزهايمر، ومرض باركنسون، والصرع... هذه الأمراض العصبية التنكسية "المرعبة بشكل سيئ السمعة" هي قتلة غير مرئيين لصحة كبار السن، وغالباً ما يرتبط حدوث هذه الأمراض بالارتباط غير الطبيعي بين البروتين والحمض النووي الريبوزي.
في المجال الطبي الحيوي، تعتبر دراسة ارتباط البروتين بالحمض النووي الريبي ذات أهمية حيوية لأنها تلعب دورًا مركزيًا في العديد من العمليات البيولوجية مثل تنظيم التعبير الجيني ومعالجة الحمض النووي الريبي وربطه وتنظيم الترجمة واستجابة الخلايا للإجهاد.إن فهم آلية ارتباط البروتين بالحمض النووي الريبي هو المفتاح لكشف عمليات تنظيم الجينات المعقدة وتحليل الأساس الجيني للأمراض. وفي الوقت نفسه، تتمتع تفاعلات البروتين مع الحمض النووي الريبي أيضًا بتطبيقات مهمة في العلاج المستهدف للحمض النووي الريبي، مما يوفر اتجاهات جديدة لعلاج السرطان والأمراض الوراثية والأمراض الفيروسية.
ومن بين الإنجازات المختارة التي تم الإعلان عنها مؤخرًا في مؤتمر AAAI السنوي التاسع والثلاثين حول الذكاء الاصطناعي (AAAI 2025)، وهو المؤتمر الدولي الأبرز للذكاء الاصطناعي،فريق مشترك من جامعة تسينغهوا، وكلية لندن الجامعية، وجامعة موناش، وجامعة بكين للبريد والاتصالاتاقترحوقد حظي نموذج CoPRA باهتمام واسع النطاق في الصناعة وتم اختياره للمرحلة الشفوية.
هذه هي المحاولة الأولى لدمج نموذج لغة البروتين (PLM) ونموذج لغة الحمض النووي الريبي (RLM) من خلال بنية هيكلية معقدة للتنبؤ بتقارب ارتباط البروتين بالحمض النووي الريبي.لاختبار أداء CoPRA، قام الباحثون بتجميع أكبر مجموعة بيانات لتقارب ربط البروتين بالحمض النووي الريبي من مصادر بيانات متعددة وقاموا بتقييم أداء النموذج على 3 مجموعات بيانات. وأظهرت النتائج أن CoPRA حقق أداءً متطورًا على مجموعات بيانات متعددة.
النتائج ذات الصلة تحمل عنوان "CoPRA: ربط نماذج التسلسل المدربة مسبقًا عبر المجالات مع الهياكل المعقدة للتنبؤ بتقارب ربط البروتين-RNA" وقد تم نشرها كنسخة مسبقة على arXiv.

عنوان الورقة:
https://arxiv.org/abs/2409.03773
عنوان مستودع CoPRA:
https://github.com/hanrthu/CoPRA
يجمع مشروع المصدر المفتوح "awesome-ai4s" أكثر من 200 تفسير لورقة AI4S ويوفر مجموعات بيانات وأدوات ضخمة:
https://github.com/hyperai/awesome-ai4s
يواصل البحث الطبي الحيوي تطوير تفاعلات البروتين والحمض النووي الريبي
على مدى السنوات الماضية، لم يتوقف الباحثون في المجال الطبي الحيوي عن دراسة تفاعلات البروتين مع الحمض النووي الريبي، وحققوا تقدماً كبيراً.
تعتبر تقنية CLIP التجريبية واحدة من أهم التقنيات في أبحاث الحمض النووي الريبي. يمكنه تحليل خريطة ربط بروتين ربط الحمض النووي الريبي (RBP) على النسخ بأكمله وهو الأساس لفهم وظيفة بروتين ربط الحمض النووي الريبي (RBP) وآليته التنظيمية بشكل منهجي. ومع ذلك، فإن تجارب CLIP تستغرق وقتًا طويلاً وتتطلب جهدًا مكثفًا، ولا يمكنها توفير موقع ربط RNA لـ RBP معين في بيئة خلوية محددة في وقت واحد، ولديها متطلبات عالية للمواد التجريبية. ومع ذلك، فإن ارتباط البروتينات بالحمض النووي الريبوزي (RNA) قد يتغير بشكل كبير مع التغيرات في البيئة الخلوية، ولكن دراسة تنظيم البروتين على الحمض النووي الريبوزي (RNA) تتطلب معلومات الارتباط في نفس البيئة الخلوية.
من أجل حل مشكلة التغيرات الديناميكية في ارتباط RBP في بيئات خلوية مختلفة،في فبراير 2021، نشرت مجموعة أبحاث Zhang Qiangfeng في مركز الابتكار المتقدم في علم الأحياء البنيوي بجامعة Tsinghua نتيجة بحث بعنوان "التنبؤ بالتفاعلات الديناميكية بين البروتين الخلوي والحمض النووي الريبي من خلال التعلم العميق باستخدام هياكل الحمض النووي الريبي في الجسم الحي" في مجلة Cell Research. استخدم هذا العمل تجربة icSHAPE لتحليل خرائط البنية الثانوية للـ RNA لـ 7 أنواع شائعة من الخلايا، وطور خوارزمية ذكاء اصطناعي لدمج بنية RNA داخل الخلايا التي تم الحصول عليها من التجربة ومعلومات ربط RBP للبيئة الخلوية المقابلة، وأنشأ طريقة جديدة PrismNet للتنبؤ بالارتباط الديناميكي لـ RBP داخل الخلايا بناءً على معلومات بنية RNA داخل الخلايا.
من أجل التنبؤ بتقارب ارتباط البروتين بالحمض النووي الريبي، تم اقتراح العديد من الأساليب الحسابية في الصناعة.يتضمن طرقًا تعتمد على التسلسل وطرقًا تعتمد على البنية. تعمل الطرق القائمة على التسلسل على معالجة تسلسلات البروتين والحمض النووي الريبي بشكل منفصل باستخدام مشفرات تسلسل مختلفة ثم تقوم بعد ذلك بنمذجة التفاعلات بينهما. ومع ذلك، فإن أداء هذه الأساليب يكون محدودًا في كثير من الأحيان لأن تقارب الارتباط يتحدد في المقام الأول من خلال بنية واجهة الارتباط. وتركز طرق أخرى مقترحة مؤخرًا على استخراج السمات البنيوية لواجهة الترابط، مثل الطاقة ومسافة التلامس. وبناءً على هذه الميزات المستخرجة، قام الباحثون بتطوير نهج التعلم الآلي القائم على البنية للتنبؤ بالتقارب. ومع ذلك، بسبب محدودية حجم مجموعة البيانات، فإن هذه الأساليب لديها قدرة تعميم محدودة على العينات الجديدة وتعتمد بشكل كبير على هندسة الميزات.
مع ظهور تكنولوجيا الذكاء الاصطناعي، تم تطوير العديد من نماذج لغة البروتين (PLMs) ونماذج لغة الحمض النووي الريبي (RLMs)، والتي أظهرت أداءً ممتازًا وقدرات تعميم في مختلف المهام اللاحقة.وفي الوقت نفسه، وبما أن البنية ثلاثية الأبعاد للبروتينات/الحمض النووي الريبوزي ضرورية لفهم وظائفها، فقد أصبح دمج المعلومات البنيوية في نماذج اللغة أيضًا اتجاهًا جديدًا.
على سبيل المثال، استخدم فريق من جامعة ميسوري، وجامعة كنتاكي، وجامعة ألاباما تقنية التعلم المقارن متعددة المنظورات لدمج معلومات بنية البروتين الرئيسية في نموذج لغة البروتين. وبناءً على هذا المفهوم، قام الفريق بتطوير S-PLM: نموذج لغة البروتين الذي لديه القدرة على إدراك المعلومات البنيوية ثلاثية الأبعاد للبروتين. يظهر S-PLM أداءً ممتازًا في مهام التنبؤ بالبروتينات المتعددة. بعد التدريب باستخدام أداة ضبط خفيفة الوزن، يصل أداء S-PLM إلى أو يتجاوز الأساليب الحديثة في المهام مثل التنبؤ بوظيفة البروتين، والتنبؤ بفئة تفاعل الإنزيم، والتنبؤ بالبنية الثانوية. نُشر البحث ذو الصلة على bioRxiv تحت عنوان "S-PLM: نموذج لغة البروتين الواعي للبنية عبر التعلم التبايني بين التسلسل والبنية".
ومع ذلك، وعلى الرغم من أن أبحاث الصناعة الحالية قد أظهرت الإمكانات الكبيرة لنماذج اللغة البيولوجية المدفوعة بالمعلومات البنيوية في المهام التفاعلية، فإن العمل الذي يجمع بين النماذج المدربة مسبقًا من المجالات البيولوجية المختلفة لا يزال نادرًا.في مشروع CoPRA، الذي اقترحته جامعة تسينغهوا، وجامعة كلية لندن، وجامعة موناش، وجامعة بكين للبريد والاتصالات، جرت محاولة لأول مرة لدمج نماذج لغة البروتين والحمض النووي الريبي مع معلومات هيكلية معقدة للتنبؤ بتقارب ارتباط البروتين بالحمض النووي الريبي.
تصميم نموذج خفيف الوزن لـ Co-Former لبناء CoPRA
بشكل عام، تظهر عملية بناء نموذج CoPRA في الشكل التالي:
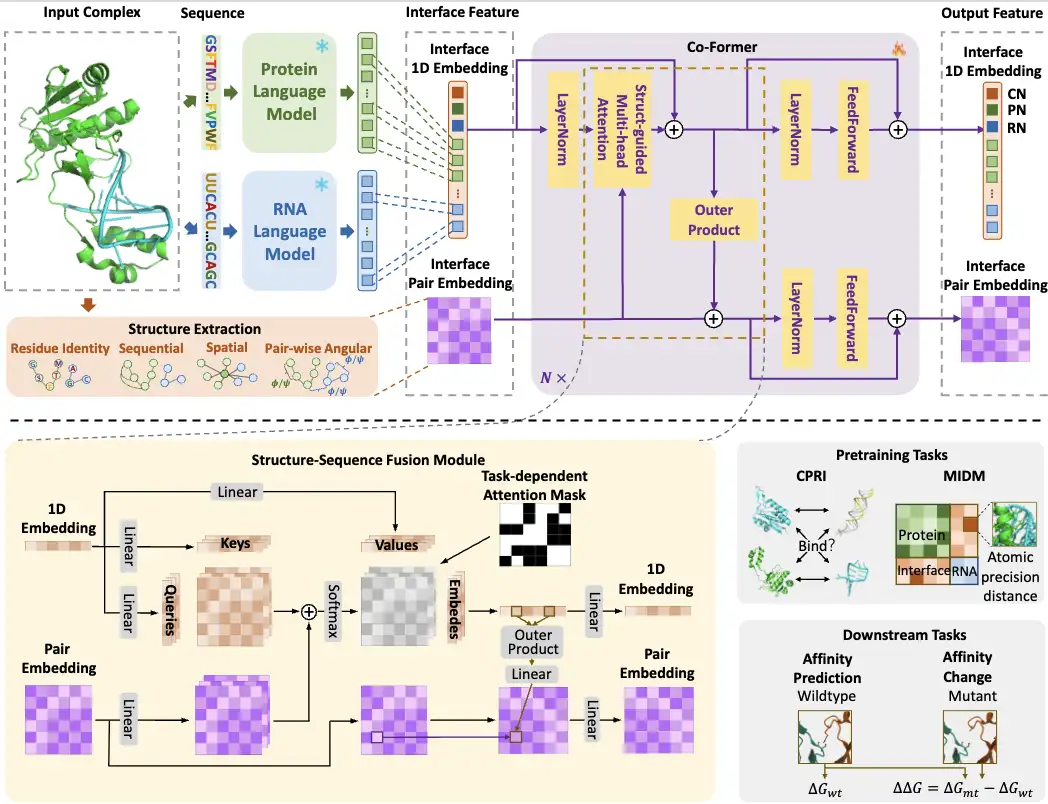
أولاً،قام الباحثون بإدخال تسلسلات البروتين والحمض النووي الريبي في PLM وRLM على التوالي، ثم قاموا باختيار التضمينات في واجهة التفاعل من مخرجات نموذجي اللغتين كتضمينات التسلسل للتعلم عبر الوسائط اللاحق. وفي الوقت نفسه، فإنه يستخرج أيضًا المعلومات البنيوية (ميزة الواجهة) من واجهة التفاعل باعتبارها تضمينًا مزدوجًا.
ثم،قام الباحثون بتصميم نموذج Co-Former خفيف الوزن يجمع بين تضمينات تسلسل الواجهة من نموذجين لغويين مع معلومات هيكلية معقدة لتشكيل وحدة دمج هيكلية-تسلسلية. على وجه التحديد، يقوم Co-Former بدمج التضمينات أحادية الأبعاد والتضمينات الزوجية عبر وحدات الانتباه الذاتي متعددة الرؤوس الموجهة بالهيكل ووحدات المنتج الخارجي، ويطبق أقنعة الانتباه المعتمدة على المهمة. يتم استخدام العقد الخاصة الناتجة والتضمينات المزدوجة لـ Co-Former وفقًا لمهام مختلفة، بما في ذلك مهمتان للتدريب المسبق ومهمتان للتقارب في اتجاه مجرى النهر.
واقترح الباحثون أيضًا استراتيجية تدريب مسبق ثنائية النطاق لـ Co-Former.لنمذجة تصنيف التفاعل التبايني الخشن (CPRI) والتنبؤ بمسافة الواجهة الدقيقة (MIDM)، والتي تم تعلمها بدقة على المستوى الذري.
لتقييم أداء CoPRA والنماذج الأخرى،يحتاج الباحثون إلى معالجة مشكلة عدم وجود مجموعات بيانات موحدة للمعايير التوضيحية. لذلك، قاموا بجمع عينات من ثلاث مجموعات بيانات عامة: PDBbind، وPRBABv2 وProNAB، وقاموا بتجميع أكبر مجموعة بيانات لتقارب ربط البروتين بالحمض النووي الريبي PRA310، وقاموا بتقييم قدرة نموذجهم على التنبؤ بتقارب ربط البروتين بالحمض النووي الريبي على مجموعات البيانات PRA310 وPRA201.
*مجموعة بيانات PRA201: مجموعة فرعية من PRA310، يحتوي كل مركب على سلسلة بروتين واحدة وسلسلة RNA واحدة فقط، ولديه قيود طول أكثر صرامة
يؤدي CoPRA أفضل أداء في التنبؤ بتقارب ارتباط البروتين بالحمض النووي الريبي
كما هو موضح في الجدول أدناه، فإن الإصدار المدرب حديثًا من CoPRA يحقق أفضل أداء على مجموعة البيانات PRA310. علاوة على ذلك، فإن معظم الطرق التي تستخدم تضمينات LM كمدخلات تتفوق على الطرق الأخرى، مما يشير إلى الإمكانات الكبيرة المتمثلة في الجمع بين LMs أحادية النمط المدربة مسبقًا للتنبؤ بالتقارب.
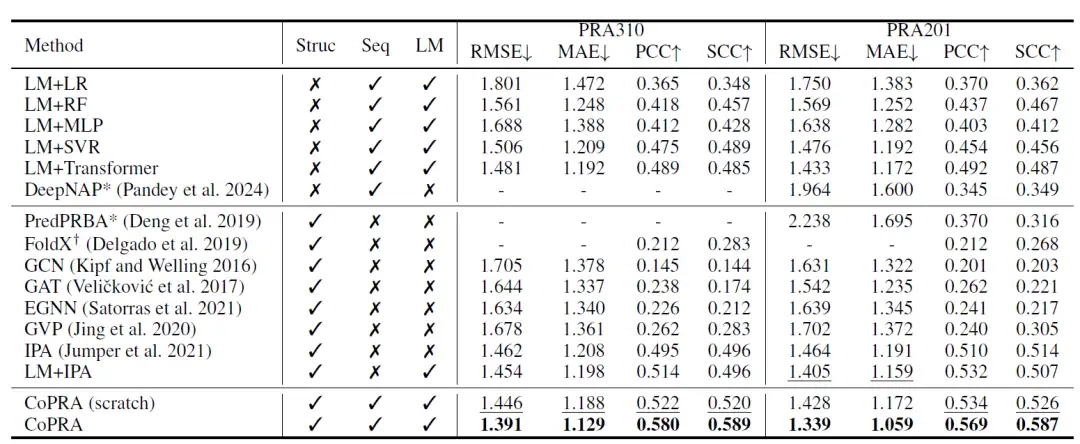
قام الباحثون بعد ذلك بتدريب النموذج مسبقًا باستخدام مجموعة البيانات غير الخاضعة للإشراف PRI30k، مما أدى إلى تحسين أدائه العام بشكل كبير على كلتا مجموعتي البيانات. في مجموعة بيانات PRA310، يحقق CoPRA RMSE يبلغ 1.391، وMAE يبلغ 1.129، وPCC يبلغ 0.580، وSCC يبلغ 0.589، وهو أفضل بكثير من ثاني أفضل نموذج CoPRA (تم تدريبه من الصفر). يدعم PredPRBA و DeepNAP التنبؤ بتقارب أزواج البروتين-RNA. قام الباحثون بمقارنة أداء هذه الطرق على مجموعة بيانات PRA201 وأظهروا أن أداءهم على PRA201 كان أقل بكثير من نتائجهم المبلغ عنها، على الرغم من ظهور ما لا يقل عن 100 عينة في PRA201 في مجموعات التدريب الخاصة بهم، مما يشير إلى أن هذه الطرق لديها قدرات تعميم ضعيفة.
إن CoPRA أقوى في التنبؤ بتأثير الطفرات على تقارب الارتباط ولديه قدرة تعميم ممتازة
لتقييم فهم النموذج الدقيق للتقارب بشكل أكبر، أعاد الباحثون توجيه النموذج للتنبؤ بتأثيرات الطفرات النقطية الفردية في البروتين على مجمع البروتين-RNA. في إشارة إلى الدراسات ذات الصلة حول التنبؤ بتأثير طفرة البروتين، قام الباحثون بمتوسط المقاييس في كل مستوى معقد وقاموا بتقييم أداء اللقطة الصفرية والضبط الدقيق لـ CoPRA بعد التدريب المسبق على PRI30k والضبط على PRA310.
كما هو موضح في الجدول أدناه، بعد الضبط الدقيق باستخدام مجموعة التحقق المتبادل لـ mCSM، تجاوز النموذج المقترح في هذه الدراسة النماذج الأخرى في جميع المؤشرات الأربعة، مع RMSE 0.957، وMAE 0.833، وPCC 0.550، وSCC 0.570.
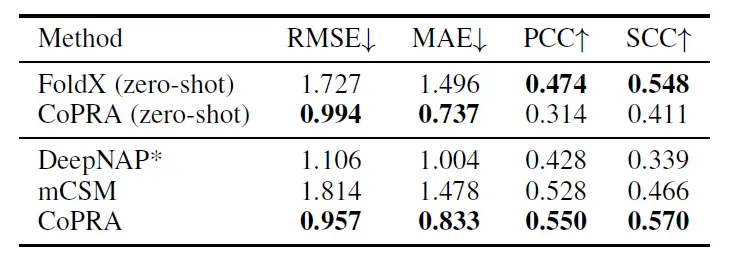
ينبع هذا الأداء المتفوق من هدف التدريب المسبق المزدوج على الرغم من عدم رؤية أي هياكل معقدة متحولة، مما يوضح قدرة CoPRA على التعميم في مهام مختلفة مرتبطة بالتقارب.
تقدم كبير في نماذج لغة البروتين متعددة الوسائط
جوهر فكرة البحث المقدمة أعلاه هو الجمع بين الوسائط البيولوجية المتعددة مثل البروتينات والحمض النووي الريبي مع المعلومات البنيوية المعقدة، وهو ما يسمى بالتعلم المتعدد الوسائط. ببساطة، التعلم المتعدد الوسائط هو عملية دمج أنواع مختلفة من البيانات في نموذج واحد في إطار التعلم العميق.
في السنوات القليلة الماضية، مع التطور السريع لنماذج اللغة الكبيرة، بدأ الباحثون في محاولة تطبيقها في مجال علم البروتينات لفهم وتوقع وظيفة وبنية وخصائص البروتينات بشكل دقيق. ومع ذلك، فإن نماذج اللغة الكبيرة الموجهة نحو البروتين السابقة تعالج بشكل أساسي تسلسلات الأحماض الأمينية كنص، مما يؤدي إلى فشلها في الاستفادة الكاملة من المعلومات البنيوية الغنية للبروتينات.واليوم، قدم التقدم في التعلم المتعدد الوسائط أفكارًا جديدة لمزيد من الأبحاث ذات الصلة.
على سبيل المثال، في مجال البحث والتطوير الدوائي، يعد التنبؤ الدقيق والفعال بتقارب الارتباط بين البروتينات والربيطات أمرا بالغ الأهمية لفحص الأدوية وتحسينها. ومع ذلك، لم تأخذ الدراسات السابقة في الاعتبار الدور المهم الذي تلعبه معلومات السطح الجزيئي في تفاعلات البروتين والربيط. وبناء على هذا،اقترح باحثون من جامعة شيامن إطار عمل جديد لاستخراج الميزات المتعددة الوسائط (MFE).يجمع هذا الإطار معلومات سطح البروتين والبنية ثلاثية الأبعاد والتسلسل لأول مرة، ويستخدم آلية الاهتمام المتبادل لمواءمة الميزات بين الوسائط المختلفة. وتظهر النتائج التجريبية أن هذه الطريقة حققت أداءً متطورًا في التنبؤ بتقارب ارتباط البروتين بالربيط. نُشر البحث ذو الصلة في مجلة Bioinformatics في يونيو 2024 تحت عنوان "التنبؤ بتقارب ارتباط البروتين بالربيط متعدد الوسائط القائم على السطح".
في ديسمبر 2024، اقترح فريق بحثي من جامعة شرق الصين العادية ومؤسسات أخرى حلاً مبتكرًا، وهو EvoLLama.هذا هو الإطار الذي يدمج مشفر بنية البروتين ومشفر التسلسل ونموذج اللغة الكبير للاندماج المتعدد الوسائط. في إعداد اللقطة الصفرية، أظهر EvoLLama قدرات تعميم قوية، مما أدى إلى تحسين أداء نماذج خط الأساس الأخرى المضبوطة بدقة بمقدار 1%-8%، وتجاوز الأداء المتوسط لنماذج الضبط الدقيق الخاضعة للإشراف الحديثة الحالية بمقدار 6%. وقد تم نشر نتائج البحث ذات الصلة كنسخة أولية على arXiv تحت عنوان "EvoLlama: تعزيز فهم طلاب الماجستير في القانون للبروتينات من خلال تمثيلات البنية والتسلسل المتعددة الوسائط".
وبطبيعة الحال، يعد التعلم المتعدد الوسائط مجرد أحد خيارات البحث المتاحة. وفي المستقبل، من خلال استخدام المزيد من أساليب التعلم الآلي لدراسة سطح البروتينات، يمكن لعلماء الأحياء الحصول على فهم أعمق لكيفية تفاعلها مع الجزيئات البيولوجية الأخرى، وبالتالي تقديم المساعدة لتطوير أدوية جديدة.
مراجع:
1.https://arxiv.org/abs/2409.03773
2.https://www.frcbs.tsinghua.edu.cn/index.php?c=show&id=873
3.https://www.sohu.com/a/846589543_121124715








