Command Palette
Search for a command to run...
まずはデモを体験してみませんか?基本ゲノムモデル Evo が Science の表紙に掲載され、分子スケールからゲノムスケールまでの予測と生成が可能になります

最近、生物学の分野でもAIが注目を集めています。米国のスタンフォード大学とアーク研究所の研究チームは、DNA、RNA、タンパク質のマルチモーダルタスクにおいてゼロサンプル予測と高精度生成を実現できるゲノムベースのモデルEvoを提案した。
関連する研究は「Evo を使用した分子からゲノムスケールまでの配列モデリングと設計」というタイトルで、Science 誌の表紙記事として掲載されました。
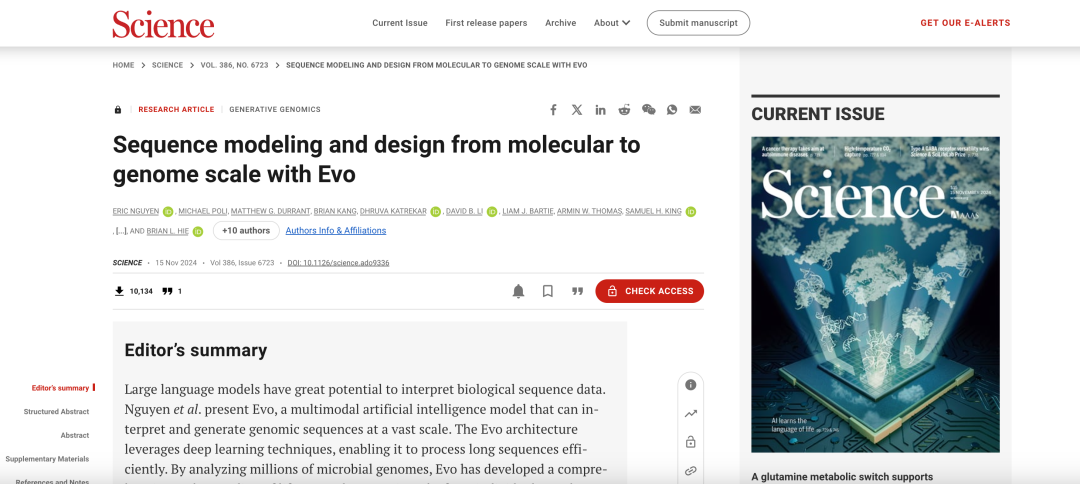
用紙のアドレス:
https://www.science.org/doi/10.1126/science.ado9336
公式アカウントをフォローし、バックグラウンドで「Evo」と返信すると、完全な PDF が表示されます
論文の筆頭著者として、Eric Nguyen は Evo の成果を紹介する更新情報をいくつか投稿し、チームの研究メンバーに何度も感謝の意を表し、「このような素晴らしいチームと仕事ができて本当に光栄です!」とさえ率直に言いました。

同紙によると、 Evo は、StripedHyena アーキテクチャを使用して、80,000 以上の細菌および古細菌のゲノムを含む大規模なゲノム データセットでトレーニングします。3,000億のヌクレオチドトークンをカバーする何百万もの予測ファージおよびプラスミド配列があり、長さ1メガベースを超える合理的なゲノム構造を持つDNA配列を生成できます。
また、Evo のパラメータ サイズは 70 億、最大コンテキスト長は 131,072 トークンで、コード配列と非コード配列の間の複雑な共進化を明らかにし、複雑な生物学的システムを設計できます。CRISPR-Cas複合体、IS200およびIS605トランスポゾンなど。
全体として、全ゲノム配列を予測、生成、設計する Evo の能力は、生命科学に新たな理論的裏付けを提供するだけでなく、遺伝子編集、創薬、疾患診断、農業およびその他の分野への応用も期待されており、研究を支援します。多くの分野で画期的な成果を上げています。
多くのネチズンは Evo のリリースに衝撃を受け、このモデルの具体的な用途への期待でいっぱいです。
Evoモデルの強力な機能を皆様に初めて体験していただくために、HyperAI ハイパーニューラル チュートリアル セクション「Evo: 分子からゲノム スケールまでの予測と生成」がオンラインになりました。コマンドを入力する必要はなく、ワンクリックでクローン作成をすぐに体験できます。
チュートリアルのリンク:
デモの実行
1. hyper.ai にログインし、「チュートリアル」ページで「Evo: 分子からゲノムスケールまでの予測と生成」を選択し、「このチュートリアルをオンラインで実行する」をクリックします。
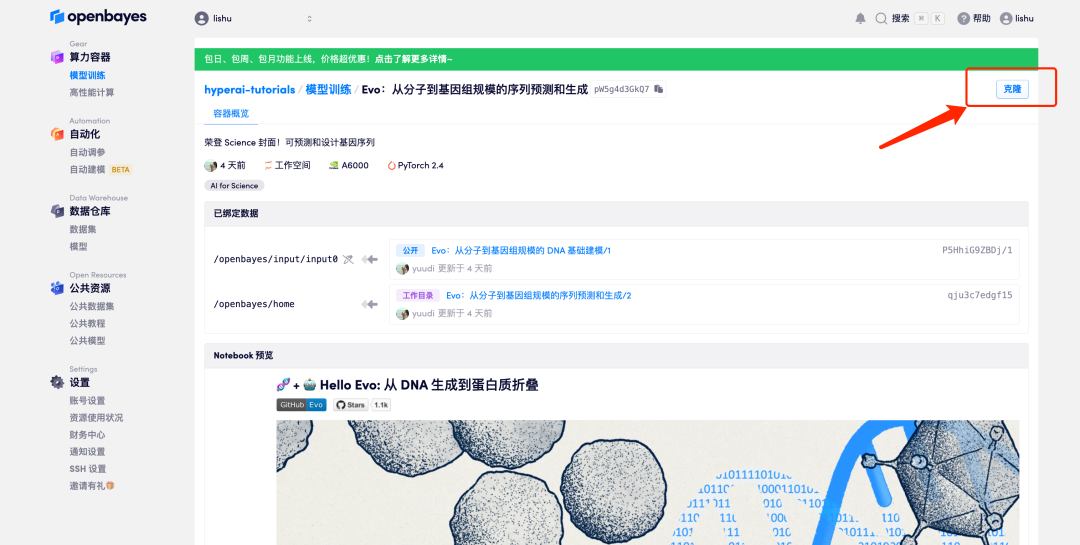
2. ページがジャンプしたら、右上隅の「クローン」をクリックしてチュートリアルを独自のコンテナにクローンします。
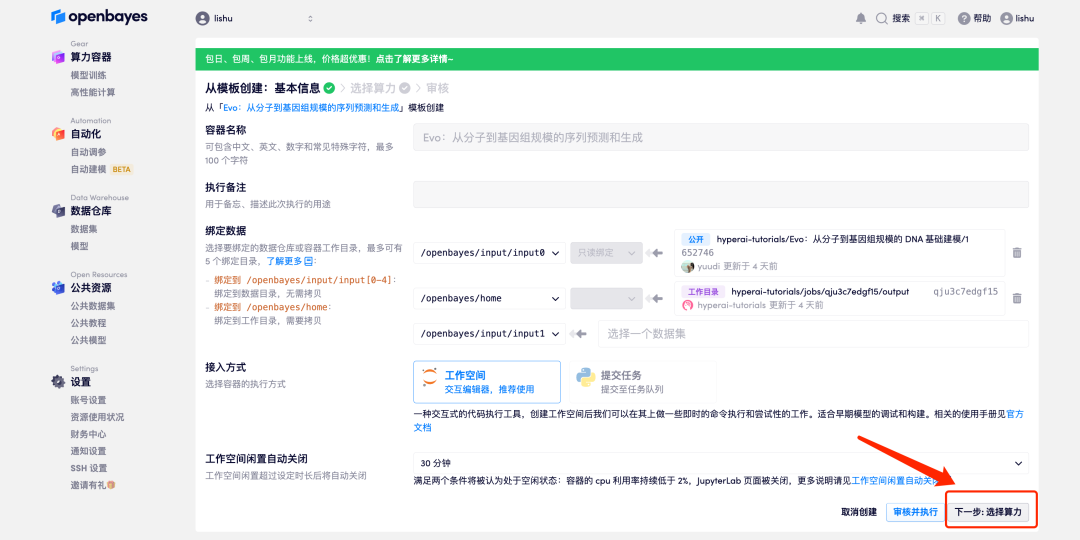
3. 右下隅の「次へ: コンピューティング能力の選択」をクリックします。
4. ページがジャンプしたら、「NVIDIA RTX A6000」を選択し、必要に応じて「Pay as you go」または「Daily/ Weekly/Monthly」を選択します。「PyTorch」イメージを選択した後、「Next: Review」をクリックします。以下の招待リンクを使用してサインアップした新規ユーザーは、4 時間の RTX 4090 + 5 時間の CPU を無料で入手できます。
HyperAI ハイパーニューラルの専用招待リンク (ブラウザに直接コピーして開きます):
https://openbayes.com/console/signup?r=Ada0322_QZy7
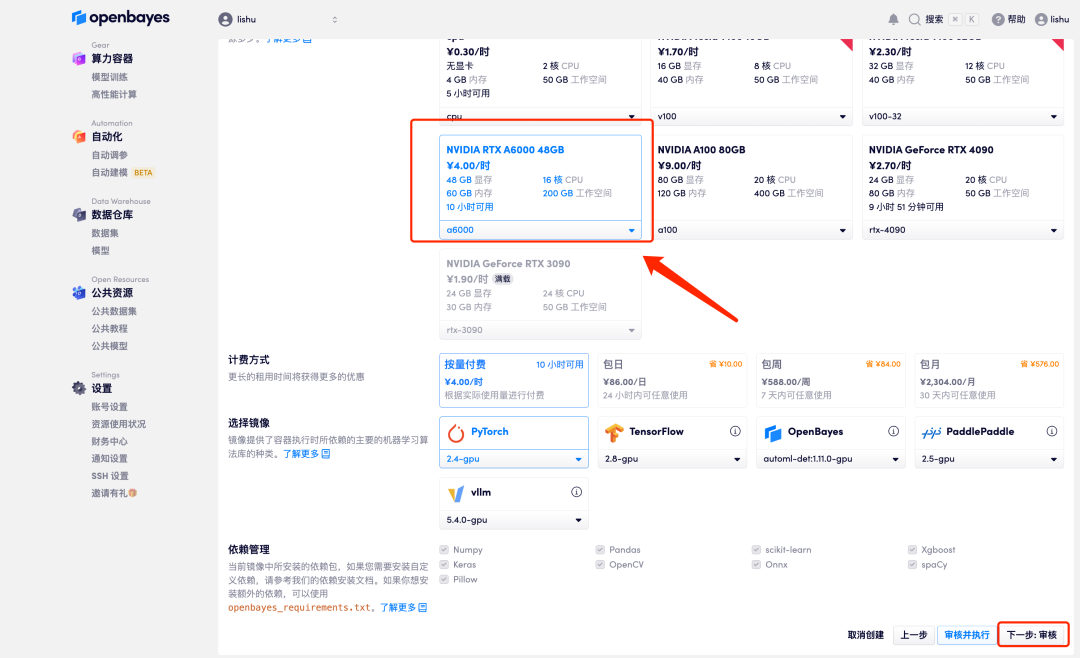
5. すべてが正しいことを確認したら、[続行] をクリックし、最初のクローンが割り当てられるまで待ちます。
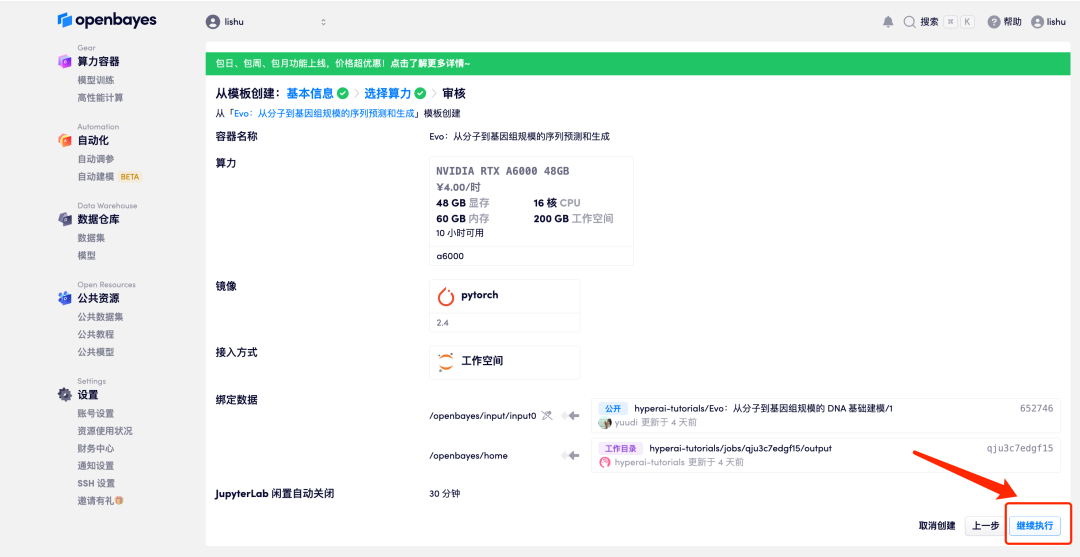
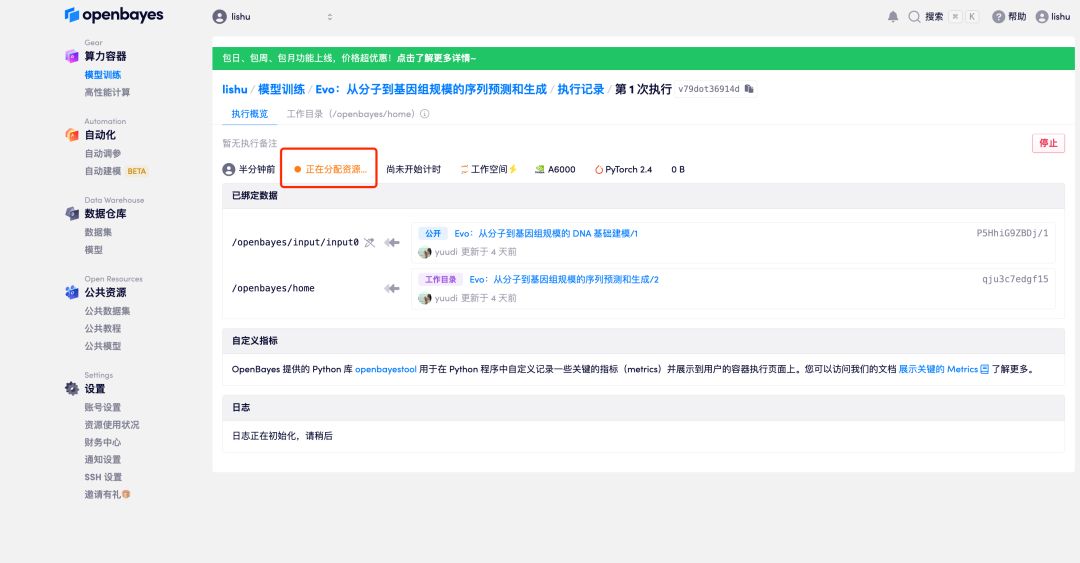
6. ステータスが「実行中」に変わったら、「ワークスペースを開く」オプションをクリックします。
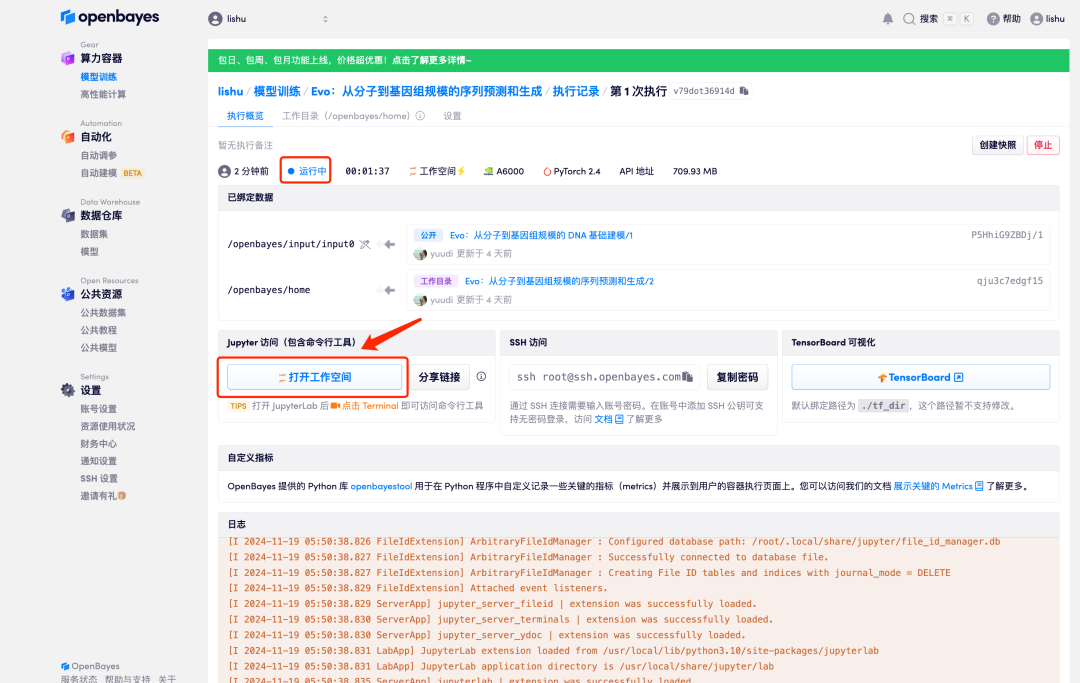
7. Jupyter ワークスペースに入った後、「README」ファイルをダブルクリックして、正式に Evo モデルの実行ページに入ります。
効果実証
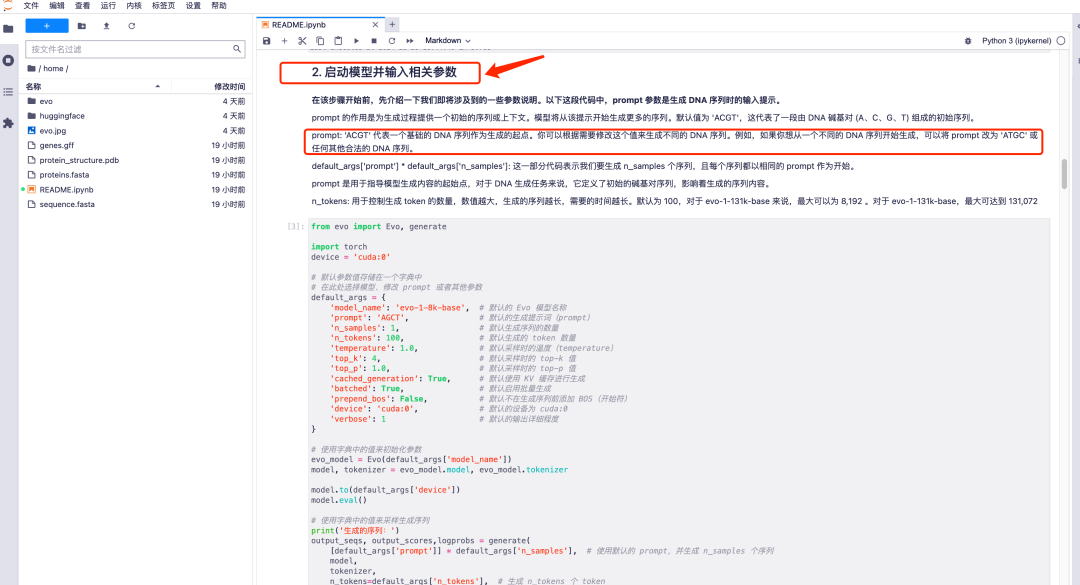
1. Evo モデルの実行ページに入ると、すべてのパラメーターがデフォルト モードになります。マウスを「2. モデルを開始して関連パラメータを入力」まで下にスライドさせ、必要に応じてプロンプトのパラメータ値を調整します。プロンプトのデフォルト値は、DNA 塩基対 (A、C、G、T) で構成される初期配列を表す「ACGT」であることに注意してください。必要に応じてこの値を変更して、異なる DNA シーケンスを生成できます。
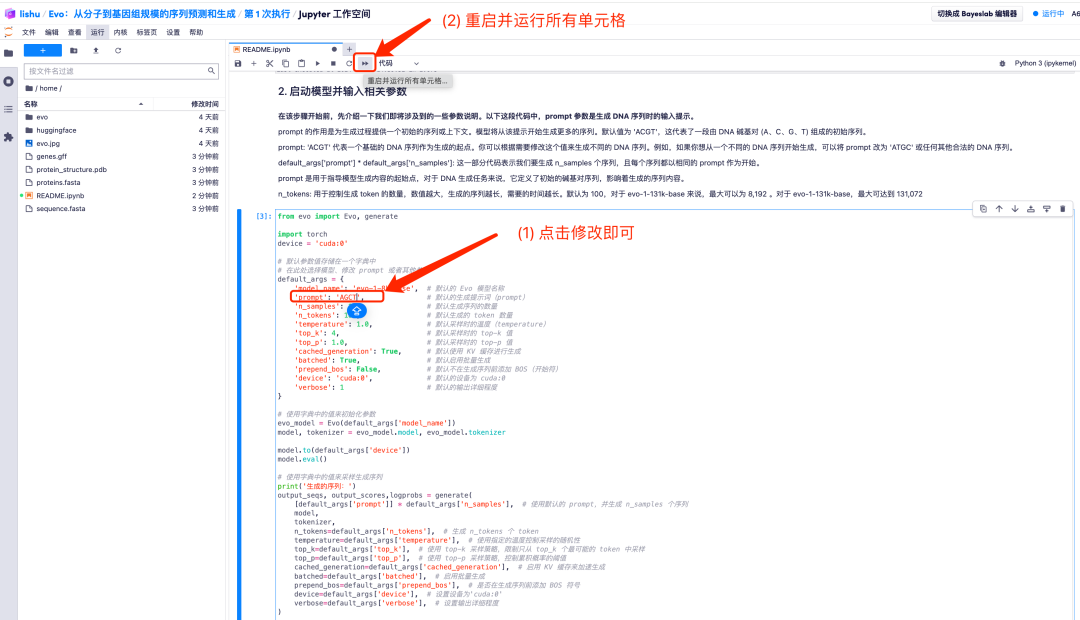
2. たとえば、プロンプトのデフォルト値を AGCT に変更します。デフォルトのパラメータ値を調整した後、[すべてのセルを再起動して実行する] オプションをクリックし、[再起動] を選択して実行します。
3. しばらく待ち、[*] 記号が数字に変わると、操作が完了したことを意味します。 「2. モデルを開始し、関連するパラメーターを入力します」の下部に、生成されたシーケンスが表示されます。
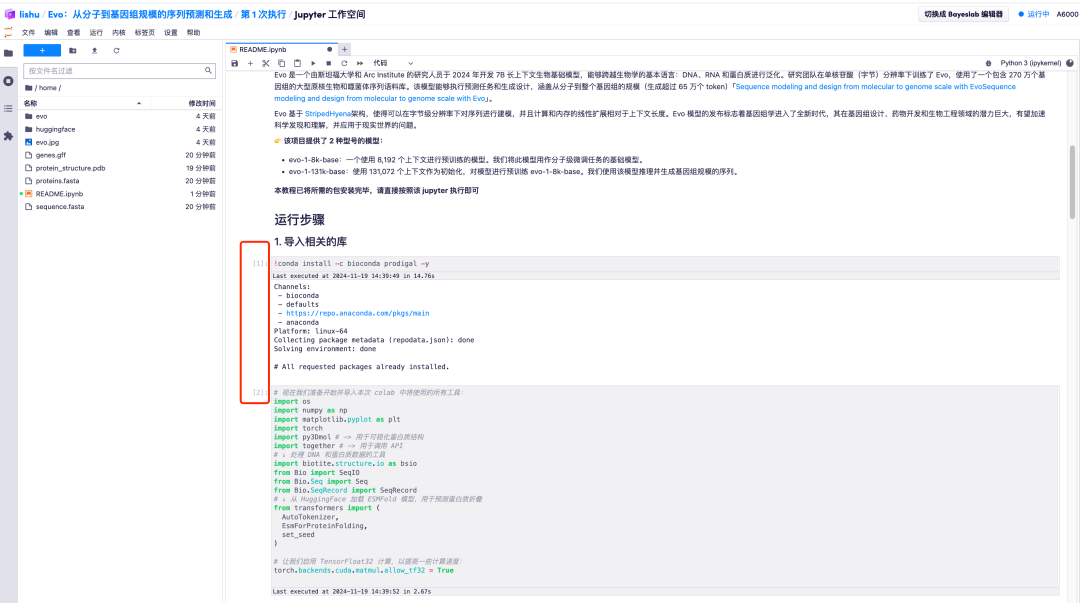
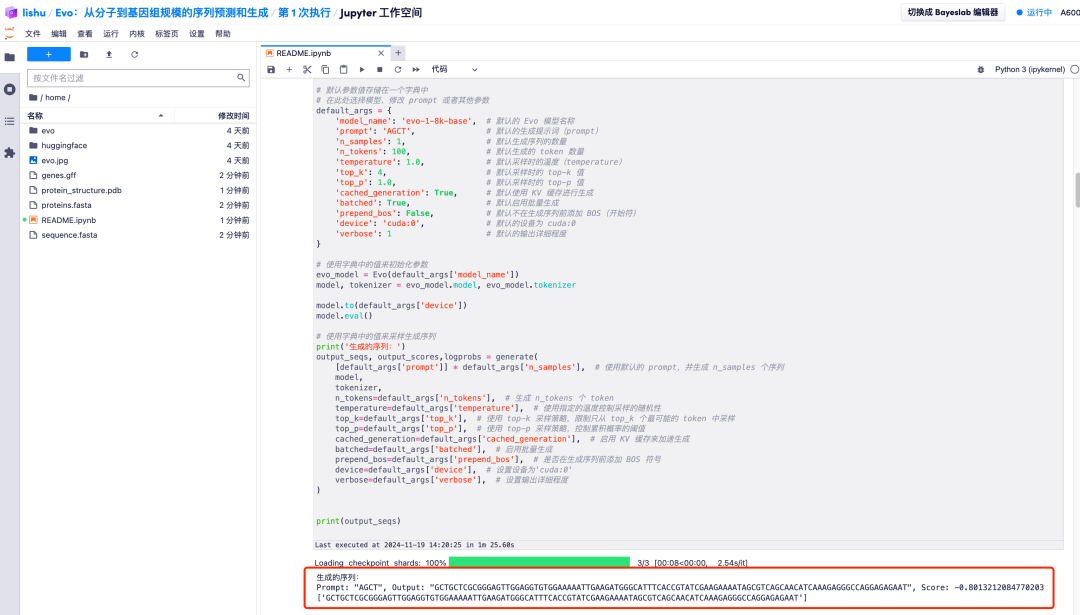
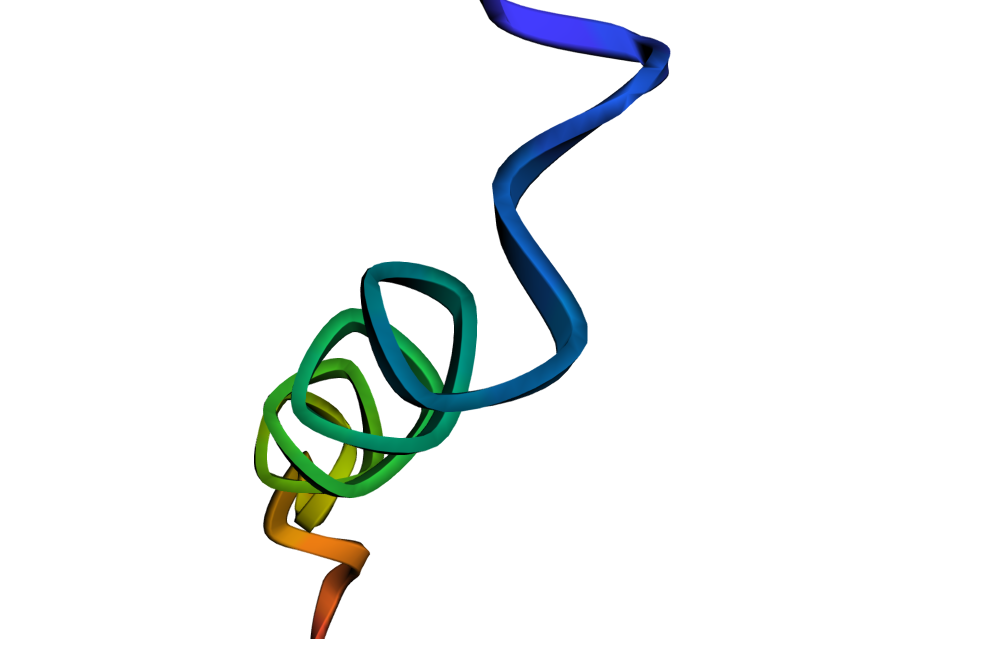
4. さらに、Evo モデルは、生成された DNA 配列を分析し、コーディング配列と非コーディング配列の間の共進化関係を学習することもできます。また、DNA 配列からタンパク質をコードする遺伝子を予測し、RNA システムをコード化して設計することで、生成されるタンパク質の折り畳み構造を予測し、最終的に画像の形で提示することもできます。
私たちは「安定拡散チュートリアル交換グループ」を設立し、さまざまな技術的問題について話し合ったり、アプリケーションの効果を共有したりするためにグループに参加する友人を歓迎します~
以下の QR コードをスキャンして Neurostar WeChat (WeChat ID: Hyperai01) を追加し、「SD チュートリアル交換グループ」とコメントしてグループ チャットに参加します。