Command Palette
Search for a command to run...
NeurIPS 2025に選ばれたトロント大学らは、特定の細胞における遺伝子発現の「標的制御」を実現するためのCtrl-DNAフレームワークを提案した。

特定の細胞における遺伝子発現の精密な制御は、遺伝子治療や合成生物学といった分野の進歩に不可欠です。このプロセスは、プロモーターやエンハンサーといった「シス調節エレメント(CRE)」と呼ばれるDNA配列群に依存しています。これらは遺伝子の「スイッチ」のような役割を果たし、標的細胞における遺伝子の「オン」と「オフ」を決定し、同時に他の正常細胞における異常な活性化を回避します。しかしながら、自然に発生する有効な CRE の数は限られており、多様な生物医学的応用シナリオに正確に一致させることは困難です。さらに重要なのは、DNA配列の可能性が飛躍的に拡大していることです。例えば、100塩基の配列には4¹⁰⁰通りの組み合わせがあり、それらを一つ一つ実験で検証するのは非常に困難です。時間と労力がかかるだけでなく、実用的なニーズを満たすこともできません。
現在のディープラーニングベースの手法により実験効率は大幅に向上しましたが、既存の手法には依然として複数の課題が残っています。例えば、既存のDNAの変異やランダム配列最適化に依存する手法もありますが、これらは容易に「局所最適性」の罠に陥り、生成される有効な配列の多様性が不十分になります。自己回帰言語モデルに基づくアプローチはDNA配列パターンを捉えることができますが、「既知の配列を模倣」することしかできず、細胞特異的な新しいCREを探索することはできません。強化学習(RL)に基づく手法は標的細胞における制御効果を向上させますが、他の細胞への「副作用」の制御を見落としています。さらに、これらの標準的な設計フレームワークは、生物学的妥当性に関する考慮を見落としがちです。生成された配列が主要な転写因子結合部位(TFBS)と一致しない場合があり、実際の制御機能が機能しない可能性があります。
細胞特異的 CRE の精密な設計におけるギャップを埋めるために、トロント大学のチームは Changping Laboratory や他の機関と共同で、Ctrl-DNA と呼ばれる制約強化学習フレームワークを開発しました。このフレームワークは、事前学習済みのDNA言語モデルをベースとし、強化学習アルゴリズムを用いて最適化プロセスにおいて2つの目的を同時に達成します。すなわち、標的細胞におけるCREの制御活性を最大化すると同時に、非標的細胞におけるCREの活性を厳密に制限することです。さらに、これら2つの要件のバランスをとるためにラグランジュ乗数という数学的ツールが用いられ、生成された配列の生物学的妥当性を保証するために、実際のDNAにおけるTFBSの分布が参照されます。
研究結果は次のことを示しています6 つのヒト細胞の設計タスクにおいて、Ctrl-DNA によって生成された CRE は、「標的細胞タイプにおける高い活性」と「非標的細胞タイプにおける制約」という 2 つの主要指標において既存の方法を大幅に上回りました。また、同社は多様性も大きく維持しており、「制御可能なシステムを作成する」合成生物学、「オフターゲットリスクを回避する」遺伝子治療、「細胞レベルのカスタマイズを実行する」精密医療に新たなソリューションを提供しています。
関連する研究成果は、「Ctrl-DNA: 細胞特異的シス調節要素設計のための制約付き強化学習」というタイトルでarXivプレプリントプラットフォームに掲載され、NeurIPS 2025に選出されました。
研究のハイライト:
* 正確な細胞タイプ特異的な遺伝子発現のための CRE を設計するためのツールを提供するため、新しい制約を考慮した強化学習フレームワークが提案されています。
* 最適化プロセスの簡素化、実験効率の向上、計算コストの削減
* 実験により、Ctrl-DNAは機能的有効性と生物学的妥当性の両方を備えていることが検証されました。

用紙のアドレス:
https://arxiv.org/abs/2505.20578
公式アカウントをフォローし、「Ctrl-DNA」と返信すると、完全なPDFを入手できます。
AIフロンティアに関するその他の論文:
データセット: 実際のヒトプロモーターおよびエンハンサーデータセットに基づく
この研究では、研究者らは実際のヒトプロモーターおよびエンハンサーデータセットを使用して、Ctrl-DNA を評価および検証しました。
で、ヒトプロモーターデータセットには、3 つの白血病由来細胞株からのプロモーター活性データが含まれています。3つの細胞株は、Jurkat、K562、THP1です。これら3つはいずれも中胚葉由来の造血細胞株であり、高い生物学的類似性を有しています。このデータセットの各配列は250塩基対です。以下の表をご覧ください。
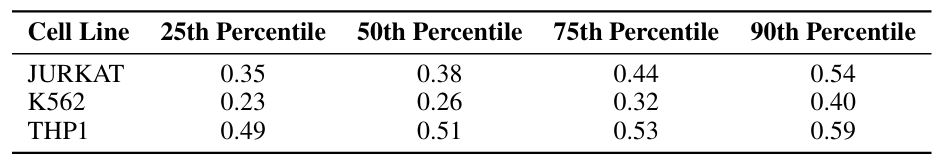
Human Enhancer Dataset には、超並列レポーターアッセイ (MPRA) によって測定された 3 つの細胞株の CRE 活性データが含まれています。3つの細胞株は、HepG2(肝細胞株)、K562(赤血球系細胞株)、SK-N-SH(神経芽腫細胞株)です。このデータセットの各配列は200塩基対の長さです。以下の表をご覧ください。
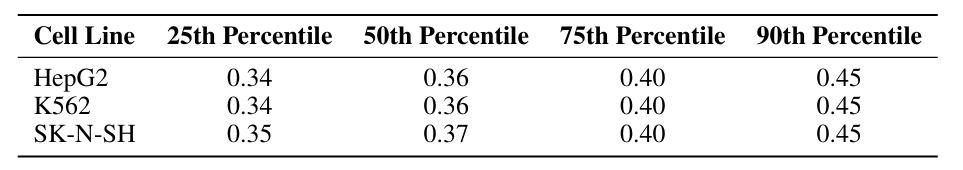
注目すべきは、THP1細胞株において25パーセンタイル活性が0.49に達し、右偏りの分布を示していることです。この分布の偏りは、THP1細胞株における活性の制限が困難になった一因である可能性があります。
モデルアーキテクチャ: 事前学習済みのDNA言語モデルに基づき、ラグランジュ緩和法と組み合わせる
Ctrl-DNA は、制約付き強化学習に基づく調節 DNA 配列設計フレームワークであり、その主な目標は制御可能な細胞タイプ特異性を持つ CRE を生成することです。機能実装の観点からは、標的細胞におけるCREの適応度を最大化(すなわち遺伝子発現を増強)すると同時に、オフターゲット細胞における適応度を予め設定された閾値内で厳密に制御する必要があります。同時に、生成された配列が実際の生物学的法則に適合していることを保証し、実験結果が要件を満たしているにもかかわらずアプリケーションが効果的でないという状況を回避する必要があります。
この目的のために、研究者はフレームワークの使いやすさ、合理性、その他の側面を考慮し、次の図に示すようにフレームワークの詳細な設計を実施しました。
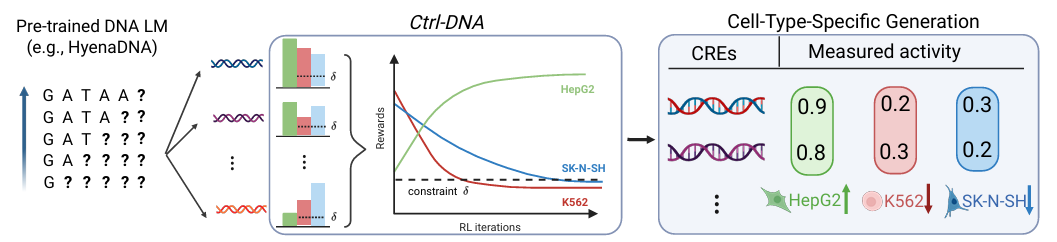
モデルと入力に関しては、Ctrl-DNA は、初期ポリシー モデルとしてヒトゲノムで事前トレーニングされた HyenaDNA 自己回帰ゲノム言語モデルを微調整し、Enformer アーキテクチャを使用して細胞タイプ固有の報酬モデルをトレーニングします。超並列レポート実験によって測定された「シーケンス適合性」データと組み合わせて、ターゲットセル報酬とオフターゲットセル報酬が個別に計算されます。
問題のモデリングレベルでは、研究者らは DNA 配列設計を制約付きマルコフ決定プロセス (CMDP) に変換しました。 Ctrl-DNAの中核となる最適化メカニズムは、制約付きバッチワイズ相対ポリシー最適化(CBROP)を利用しています。このメカニズムは、ラグランジュ緩和法を用いて、制約付き最適化問題を制約なしのプライマル・デュアル最適化問題に変換します。最適化プロセスは反復的で、ポリシーの更新はラグランジュ目的関数の勾配に従って学習率で行われます。オフターゲットセルの報酬は、ラグランジュ乗数を調整することで制約されます。ラグランジュ乗数を増やすと、閾値を超えるオフターゲットセルに対する制約が強化され、ラグランジュ乗数を減らすと、閾値を満たすオフターゲットセルに対する制約が弱まります。
トレーニングの複雑さを軽減するために、Ctrl-DNA は従来の強化学習における価値モデルへの依存を放棄します。正規化された利点は、バッチ データの統計に基づいて直接計算され、「高いターゲット報酬 + 低いオフターゲット報酬」を持つシーケンスを選択するための戦略の最適化をガイドします。
戦略更新目的関数の設計において、研究者らは「枝刈り置換目的関数」と「KL正則化」の組み合わせを採用しました。枝刈りによって戦略変異を制限し、現在の戦略と初期参照戦略の間にKLダイバージェンスを導入することで、生成された配列と天然DNAパターンの整合性を確保し、最終的に戦略更新目的関数を形成しました。
生物学的妥当性をさらに確保するため、Ctrl-DNAはTFBS頻度相関を追加制約として導入します。まず、FIMOツールを使用して、実際の高度に特異的なCRE配列からTFBSをスキャンし、真のTFBS頻度ベクトルを構築します。次に、生成された各配列に対応するTFBS頻度ベクトルを計算します。ピアソン相関係数を追加制約報酬として使用し、対応するラグランジュ乗数は[0, λmax](λmax ≤ 1)にクリップされます。これにより、生物学的妥当性と客観的な最適化のバランスが保たれ、モデルの探索能力を低下させる可能性のある過剰な制約を回避できます。
モデル学習の安定性を確保するため、研究者らは実験で使用したハイパーパラメータ設定を実証しました。すべてのモデルはAdamオプティマイザーを用いて学習され、ポリシー学習率は1e-4、バッチサイズは256、学習エポック数は100でした。実験は、下図に示すように、40GBのメモリを搭載した単一のNVIDIA A100 GPUで学習されました。
実験結果: 8種類のベースライン法と比較して、Ctrl-DNAには明らかな利点がある
Ctrl-DNAの性能評価実験は、ヒトエンハンサーとプロモーターという2つの主要な設計タスクを中心に、前述の6つの細胞株を対象としています。進化アルゴリズム(AdaLead、ベイズ最適化(BO)、CMA-ES、PEXなど)、生成モデル(RegLM)、強化学習法(TACO、PPO、PPO-Lagrangianなど)を含む8種類のベースライン手法と比較し、細胞種特異性、生物学的妥当性、配列多様性など、多角的な観点からその有効性と実用性を検証します。
Ctrl-DNA は、細胞タイプ特異的な閉じ込めに関して大きな利点を示します。下の図に示すように、横軸はオフターゲット細胞の適応度、縦軸はターゲット細胞の適応度を表しています。右上に示されている手法は、ターゲット細胞の適応度を最大化し、オフターゲット発現を最小化する最適なバランスを示しています。
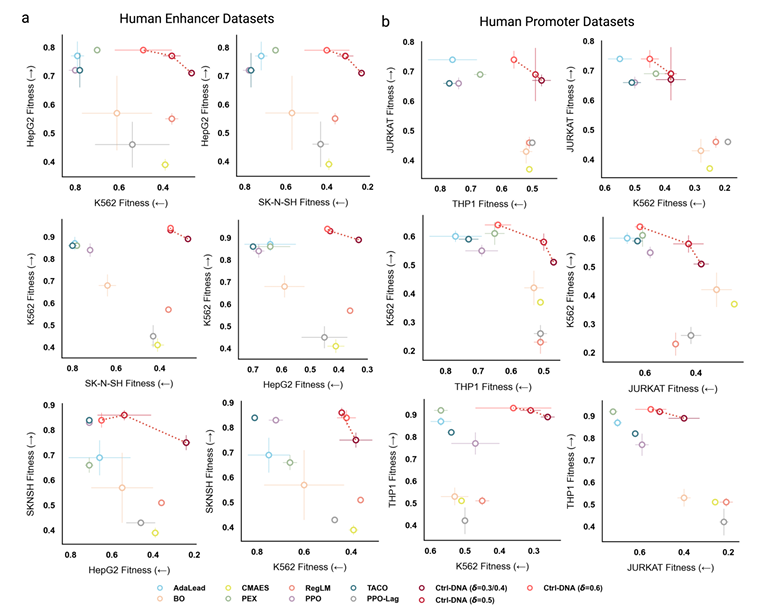
エンハンサー設計において、Ctrl-DNAは、様々な制約閾値(δ = 0.3、0.5、0.6)において、オフターゲット制約を満たしながら、一貫して最高の標的細胞適応度を達成しました。これは、オフターゲット制約を厳密に満たしながら、標的細胞適応度を最大化したことを意味します。さらに、TACOやCMAESなどの手法は標的細胞で高い発現を達成しましたが、オフターゲット細胞適応度を抑制することができず、細胞種特異性が低下しました。
プロモーター設計タスクでは、3 つのターゲット セル タイプすべてが中胚葉由来の造血細胞であるため、転写の類似性が高く、このタスクにとって大きな課題となりますが、Ctrl-DNA はそれでも優れたパフォーマンスを発揮します。この実験では、テスト用に 3 つの異なる制約しきい値 (δ = 0.4、0.5、および 0.6) を設定しました。Ctrl-DNA は、ターゲット セル タイプの適応度を最大化し、制約しきい値 δ = 0.5 および 0.6 を満たしたときに、すべてのベースラインを上回りました。また、アクティビティ分布が右に偏っている THP1 細胞などのケース (上記のデータセット セクションで述べたように、25 パーセンタイル アクティビティが 0.49 に達している) では、どの手法もオフターゲット アクティビティを厳密なしきい値 δ = 0.4 に抑制することはできませんが、Ctrl-DNA はすべての手法の中で制約要件に最も近い手法であることも注目に値します。
生物学的妥当性検証では、下図に示すように、Ctrl-DNAはヒトプロモーターおよびエンハンサーにおいて、すべての細胞種において最も高い報酬差(ΔR)を達成しました。これは、Ctrl-DNAがDNA配列の細胞特異的な適応度をより適切に最適化していることを示しています。モチーフ関連性に関しても、Ctrl-DNAはTHP1プロモーター設計を除き、ほとんどの細胞種において優れた性能を示しました。
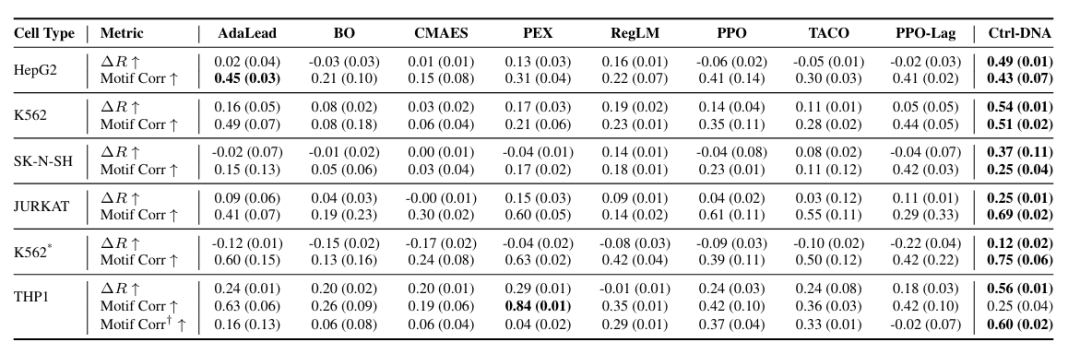
この矛盾をさらに調査するため、研究者らはTHP1の適応度が90パーセンタイルのプロモーター配列からモチーフを抽出しました。偽陽性を回避するためにq < 0.05の閾値を用いて、生成された配列と参照セットとの間のモチーフ相関を再評価しました(上図ではモチーフCorr†として示されています)。その結果、この厳格な設定下でもCtrl-DNAはすべてのベースラインを上回り、相関係数は0.60に上昇しました。一方、ほとんどのベースラインでは相関が低下し、機能的に重要な調節モチーフを優先的に捕捉できることが示されました。
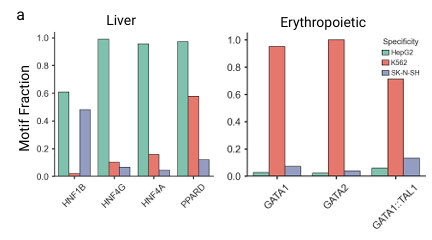
発見された特定の TFBS の頻度をさらに分析するために、研究者らは HepG2 肝細胞株と K562 赤血球細胞株に特有のモチーフに対して生成された配列を具体的に調べました。下図に示すように、Ctrl-DNAによって生成されたHepG2配列は、HNF4AやHNF4Gといった肝臓特異的モチーフの頻度が最も高いことがわかります。同様に、K562に対して生成された配列は、GATA1やGATA2といった赤血球特異的モチーフの頻度が最も高いことがわかります。これは、Ctrl-DNAが標的細胞の適応度を最適化するだけでなく、その根底にある細胞種特異性を反映した制御パターンを学習していることを示しています。
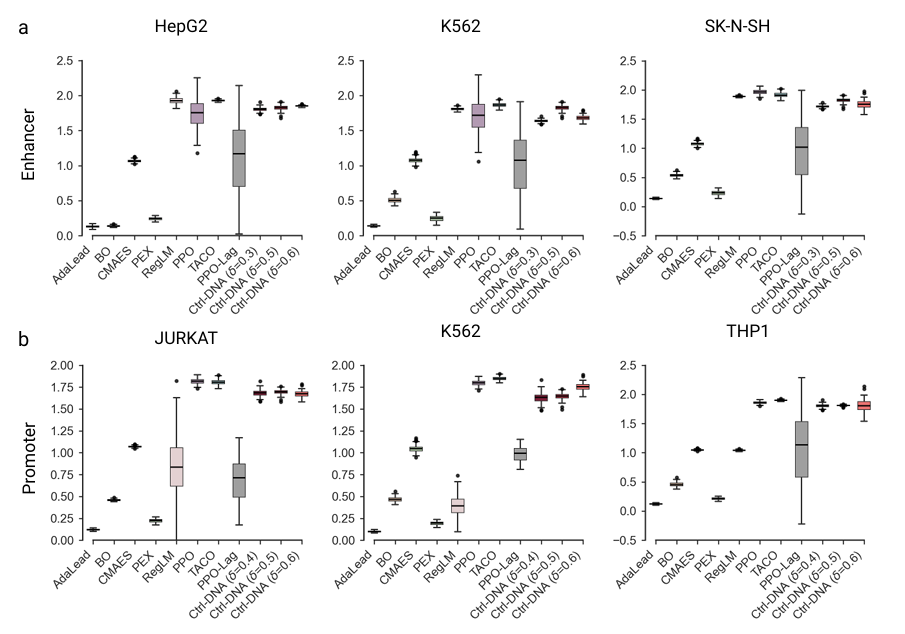
配列の多様性に関しては、Ctrl-DNA はほとんどのベースラインと同等かそれ以上の多様性を達成し、調節制御を犠牲にすることなく多様な配列を生成できることが確認されました。以下に示すように:
最後に、研究者らはアブレーション実験を通じてCtrl-DNAコアモジュールの有効性をさらに検証しました。TFBS正規化モジュールの役割も確認され、配列を生物学的に現実的なパターンへと効果的に誘導しました。
AI駆動型DNA「スイッチ」設計が新たな章を開く
これまで、調節 DNA 配列「スイッチ」の設計は、主に、多数の繰り返し手動スクリーニングによる「試行錯誤」に依存していました。現在では、AI技術と組み合わせることで、「どのDNA配列が標的調節タンパク質と最もよく一致するか」を予測するアルゴリズムを使用できるようになり、設計の効率と精度が大幅に向上しています。これは、AI による DNA スイッチ設計が新たな方向性となり、遺伝子治療や合成生物学などの分野を「広範囲」から「精密」へと直接推進する中核的な理由でもあります。
この論文は、「AI駆動型DNAスイッチ設計」という大きな木の大きな果実の一つに過ぎません。過去を振り返ると、多くの研究室がすでに関連研究を行ってきました。
たとえば、ジャクソン研究所、ブロード研究所、イェール大学のチームは、「細胞タイプをターゲットとするシス調節要素の機械誘導設計」と題する研究をネイチャー誌に発表しました。この研究では人工知能を利用して何千もの新しいDNAスイッチを設計した。これらのスイッチは、異なる細胞種における遺伝子発現を精密に制御することができます。具体的には、研究者らはCREの活性を正確に予測できる深層畳み込みニューラルネットワーク(マリノア)を構築し、特定の機能を持つCREを設計するためのモジュール式プラットフォーム(CODA)を開発しました。このプラットフォームは、レポーター遺伝子、CRISPR療法、遺伝子置換法などを開発するための強力なツールを提供します。
紙のアドレス:
https://www.nature.com/articles/s41586-024-08070-z
さらに、上記の記事で言及されているRegLMは、ジェネンテック社によるものです。「自己回帰言語モデルを用いた現実的な制御DNAの設計」というタイトルの研究では、我々は、特定の特性を持つ合成 CRE を設計するために、教師ありシーケンス関数モデルと組み合わせた自己回帰言語モデルに基づく RegLM と呼ばれるフレームワークを紹介します。同様に、RegLMもHyenaDNAフレームワークに基づいています。機能ラベルをヒントトークンとしてエンコードし、DNA配列プレフィックスに追加します。そして、モデルをトレーニングまたは微調整して次のトークン予測を実行し、目的の機能を持つDNA配列を生成します。同時に、教師あり配列アクティビティ回帰モデルと組み合わせることで、生成された配列をスクリーニングします。
用紙のアドレス:
https://genome.cshlp.org/content/34/9/1411.full#aff-1
まとめると、Ctrl-DNAの開発は、DNAスイッチ設計における新たな一歩であることは間違いありません。生成された配列の合理性と機能性をさらに向上させるために追加の生物学的制約を組み込むことや、ラグランジュ乗数の調整がまだ経験則であるなど、依然としていくつかの問題や緊急の改善が必要な領域はありますが、これらのツールの開発と改良は、DNAスイッチ設計に新たな章を開き、人工知能と生物学の学際科学の継続的な発展を促進したことは間違いありません。








