Command Palette
Search for a command to run...
20 Données Expérimentales Créent Une Étape Importante Dans La Protéine IA ! L'Université Jiao Tong De Shanghai Et Le Shanghai AI Lab Ont Publié Conjointement Le FSFP Pour Optimiser Efficacement Les Modèles De Pré-entraînement Des Protéines

Les protéines, ces minuscules mais puissantes molécules biologiques, sont à la base des activités de la vie et jouent de multiples rôles dans les organismes. Cependant, ajuster et optimiser précisément les fonctions des protéines pour répondre à des besoins industriels ou médicaux spécifiques est une tâche extrêmement difficile. Traditionnellement, les scientifiques ont eu recours à des méthodes de laboratoire humide pour explorer les mystères des protéines, mais cette approche est longue et coûteuse.
Heureusement, avec le développement rapide de l’intelligence artificielle, un nouvel outil – les modèles de langage protéique pré-entraînés (PLM) – nous aide à comprendre et à prédire le comportement des protéines de manière sans précédent. Les PLM apprennent les caractéristiques de distribution des séquences d'acides aminés dans des millions de protéines de manière non supervisée et présentent un grand potentiel pour révéler la relation implicite entre les séquences de protéines et leurs fonctions, contribuant ainsi à explorer efficacement un vaste espace de conception. maintenant,Les PLM pré-entraînés ont fait des progrès significatifs en l’absence de données expérimentales, mais leur précision et leur interprétabilité doivent encore être améliorées.De plus, les modèles d’apprentissage supervisé traditionnels nécessitent un grand nombre d’échantillons d’entraînement étiquetés, ce qui constitue également un obstacle difficile à surmonter dans les applications pratiques.
Afin de résoudre les problèmes ci-dessus,Le groupe de recherche du professeur Hong Liang de l'École des sciences naturelles/École de physique et d'astronomie/Institut d'études avancées de Zhangjiang/École de pharmacie de l'Université Jiao Tong de Shanghai, en collaboration avec Tan Pan, un jeune chercheur du Laboratoire d'intelligence artificielle de Shanghai,Utilisation complète de l'apprentissage par méta-transfert (MTL), de l'apprentissage du classement (LTR) et du réglage fin efficace des paramètres (PEFT),Nous avons développé une stratégie de formation, FSFP, qui peut optimiser efficacement les modèles de langage protéique lorsque les données sont extrêmement rares.Il peut être utilisé pour l’apprentissage sur de petits échantillons de l’adaptabilité des protéines. Il améliore considérablement l'effet des grands modèles traditionnels de pré-entraînement des protéines dans la prédiction des propriétés de mutation lorsqu'on utilise très peu de données expérimentales humides, et montre également un grand potentiel dans les applications pratiques.
La recherche connexe a été publiée dans Nature Communications, une filiale de Nature, sous le titre « Améliorer l'efficacité des modèles de langage protéique avec un minimum de données de laboratoire humide grâce à un apprentissage en quelques coups ».

Adresse du document :
https://doi.org/10.1038/s41467-024-49798-6
Adresse de téléchargement de l'ensemble de données sur les mutations protéiques ProteinGym :
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
La FSFP optimise le modèle de langage des protéines pour résoudre le problème de pénurie de données
L'approche FSFP comprend trois phases :Créez des tâches auxiliaires pour la méta-formation, méta-entraînez les PLM sur les tâches auxiliaires et transférez les PLM vers la tâche cible via LTR.
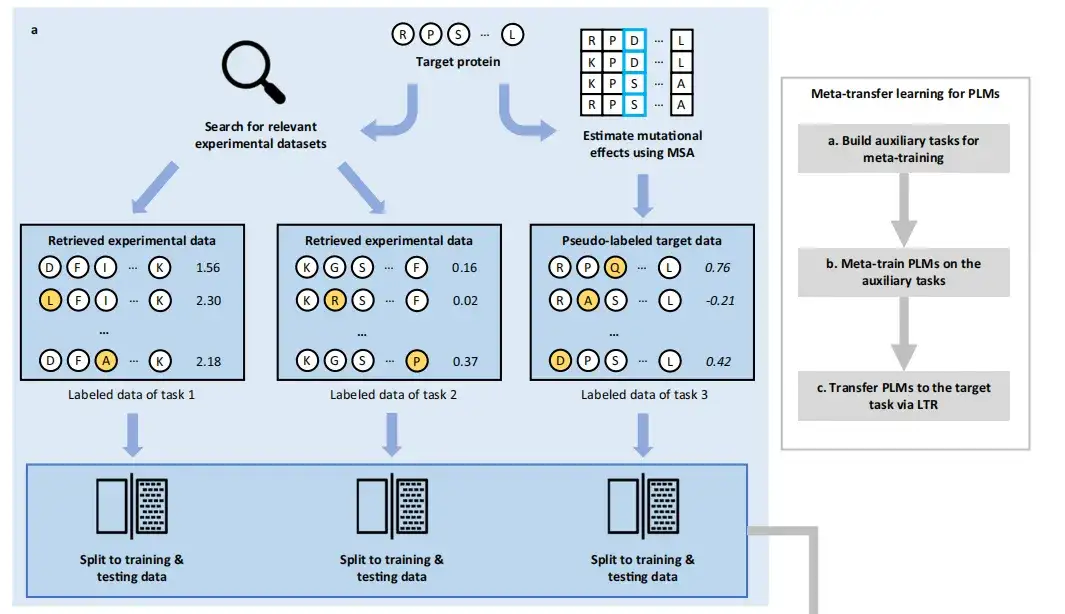
dans,Le méta-apprentissage vise à accumuler l’expérience de plusieurs tâches d’apprentissage pour former un modèle capable de s’adapter rapidement à de nouvelles tâches en utilisant seulement un petit nombre d’exemples et d’itérations de formation.. Par conséquent, cette étude a d’abord utilisé des PLM pour coder la séquence ou la structure de type sauvage de la protéine cible et la séquence ou la structure de la base de données dans un vecteur intégré.
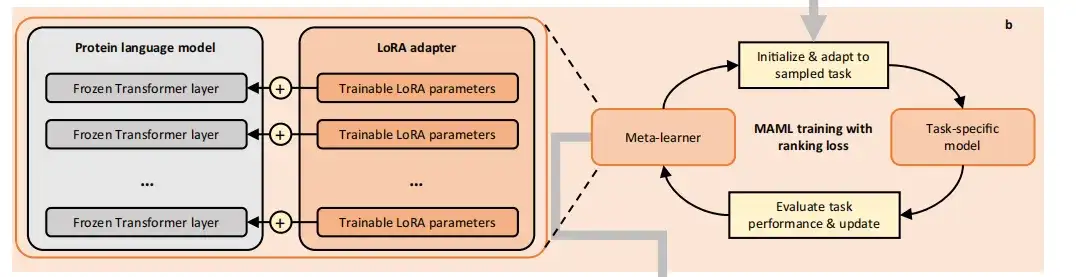
aussi,L’étude a utilisé une méthode de méta-apprentissage basée sur le gradient appelée Model-Agnostic Meta-Learning (MAML).Méta-trainer les PLM sur les tâches construites. MAML est capable de trouver des paramètres de modèle initiaux optimaux de telle sorte que même de petites modifications puissent conduire à des améliorations significatives de la tâche cible. À chaque itération, le processus de méta-formation se compose de deux niveaux d'optimisation et transforme finalement les PLM en méta-apprenants initialisés.
Dans l'optimisation interne, nous utilisons le méta-apprenant actuel pour initialiser un apprenant de base temporaire, qui est ensuite mis à jour vers un modèle spécifique à la tâche en échantillonnant les données de formation de la tâche. Dans l'optimisation externe, nous utilisons la perte de test d'un modèle spécifique à la tâche sur la tâche pour optimiser le méta-apprenant.
Afin d'éviter un surapprentissage catastrophique dû à un manque de données d'entraînement,FSFP utilise l'adaptation de rang faible (LoRA) pour injecter des matrices de factorisation de rang entraînables dans les PLM.Leurs paramètres pré-entraînés d'origine sont gelés et toutes les mises à jour du modèle sont limitées à un petit nombre de paramètres pouvant être formés.
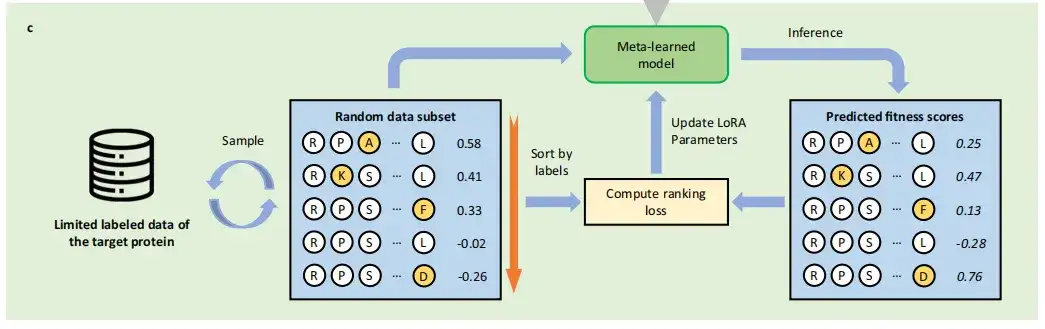
Après la méta-formation, l'étude peut obtenir l'initialisation basée sur les paramètres LoRA et enfin transférer les PLM méta-formés à la tâche d'apprentissage du petit échantillon cible, c'est-à-dire apprendre à prédire l'effet de mutation de la protéine cible avec des données étiquetées limitées. Différente des méthodes traditionnelles d’apprentissage supervisé pour la prédiction des mutations protéiques,FSFP le traite comme un problème de tri et utilise la technologie LTR.
Plus précisément, le FSFP apprend à classer la forme physique des mutations en calculant la perte ListMLE. À chaque itération, l’étude entraîne le modèle de sorte que ses prédictions sur un ou plusieurs sous-ensembles de données échantillonnées convergent vers la disposition de vérité fondamentale. Ces schémas de formation sont appliqués simultanément pour l'optimisation interne dans la phase d'apprentissage par transfert en utilisant les données de formation cibles et dans la phase de méta-formation en utilisant les données de formation des tâches auxiliaires.
Benchmark ProteinGym basé sur 87 ensembles de données de mutation à haut débit
Afin de construire les tâches de formation nécessaires au méta-apprentissage,Cette méthode récupère d'abord les ensembles de données mutantes étiquetées existantes, récupère les ensembles de données de mutation des deux premières protéines les plus proches de la protéine cible à partir de la plus grande collection publique d'ensembles de données DMS, ProteinGym, et utilise la méthode de pseudo-étiquetage GEMME basée sur MSA pour noter les informations de mutation de la protéine cible afin de construire l'ensemble de données pour la troisième tâche. Ces ensembles de données peuvent être utiles pour prédire les effets des variantes sur les protéines cibles. Les données étiquetées pour ces tâches sont divisées aléatoirement en données d’entraînement et données de test.
Pour évaluer les performances du modèle,Cette étude a sélectionné l’ensemble de données sur les mutations protéiques (ProteinGym) comme ensemble de données de référence. L'ensemble de données contient un total d'environ 1,5 million de variantes faux-sens provenant de 87 expériences de séquençage DMS. Étant donné que la longueur d’entrée maximale de l’ESM-1v est de 1 024, cette étude a tronqué les protéines avec plus de 1 024 acides aminés et a garanti que la plupart de leurs mutations dans les ensembles de données correspondants se sont produites dans l’intervalle généré.
Ensuite, l'étude a sélectionné au hasard 20 mutations ponctuelles comme ensemble d'entraînement initial, puis a ajouté 20 autres mutations ponctuelles pour étendre la taille de l'ensemble d'entraînement à 40, et a construit de manière similaire des ensembles d'entraînement de 60, 80 et 100. Après 5 processus de division aléatoire des données,Cette étude peut permettre de réaliser la moyenne des performances du modèle sur différentes partitions d’une certaine échelle de formation.
Le FSFP est appliqué avec succès à trois modèles de base et présente des avantages significatifs dans les tâches d'apprentissage sur de petits échantillons
Théoriquement, le FSFP peut être appliqué à n’importe quel modèle de langage protéique basé sur l’optimisation de la descente de gradient.Afin de vérifier son universalité,Cette étude a sélectionné trois PLM représentatifs - ESM-1v, ESM-2 et SaPro-t comme modèles de base pour la formation, et a sélectionné la version 650M pour l'évaluation.
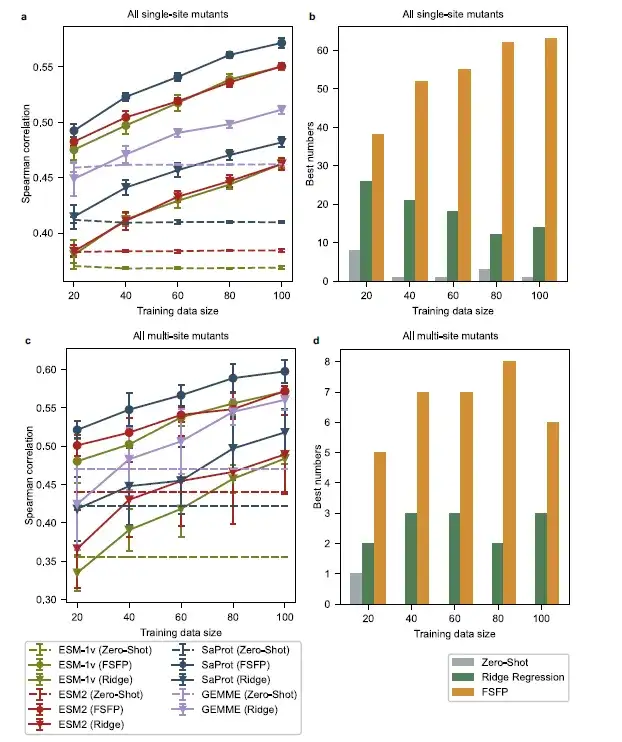
En termes de performance moyenne,Les PLM formés par FSFP surpassent systématiquement les autres lignes de base sur toutes les tailles de données de formation. Parmi eux, SaProt (FSFP) a obtenu les meilleurs résultats, tandis qu'ESM-1v (FSFP) et ESM-2 (FSFP) ont obtenu des résultats tout aussi bons. De plus, sur la plupart des ensembles de données de ProteinGym, les PLM entraînés par FSFP ont obtenu la meilleure corrélation de Spearman. Par rapport à la prédiction à zéro coup, le FSFP améliore les performances des PLM sur des mutants uniques de près de 0,1 en améliorant la corrélation de Spearman des mutants uniques en utilisant seulement 20 exemples d'entraînement, et cet écart devient encore plus grand lorsque plusieurs mutants sont impliqués. Ces améliorations augmentent à mesure que l’ensemble de données de formation augmente, ce qui est cohérent avec les résultats d’ablation de cette étude.
Le modèle utilisant FSFP réalise des améliorations significatives par rapport à GEMME et à sa version améliorée de régression de crête sur tous les échantillons d'entraînement. Cela indique que FSFP transfère non seulement les connaissances d'alignement de séquences multiples de GEMME vers PLM, mais les combine également avec succès avec les informations de supervision des données de formation cibles via l'apprentissage multitâche.Cela confirme une fois de plus l’avantage du FSFP dans les tâches d’apprentissage à quelques coups.
Évaluation des performances d'extrapolation, l'évaluation de la corrélation de Spearman des PLM formés FSFP est meilleure
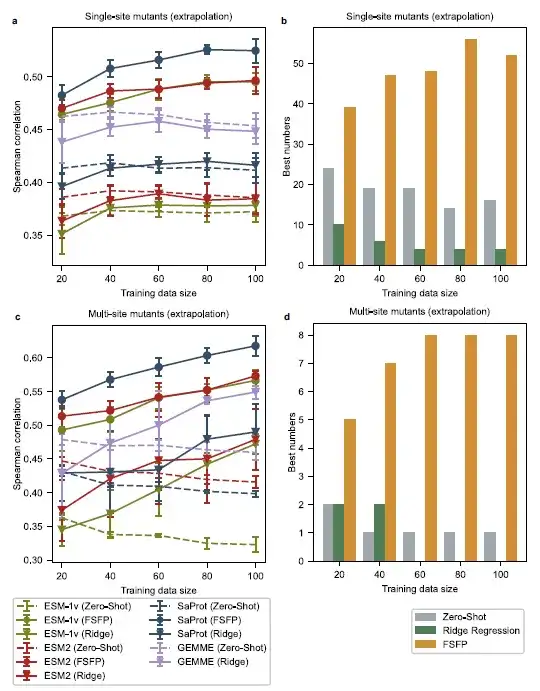
Les chercheurs ont sélectionné tous les mutants à point unique de chaque ensemble de tests d’origine dont les sites de mutation étaient différents des exemples d’entraînement, obtenant ainsi un ensemble de tests de mutants à point unique différents des exemples d’entraînement. Les chercheurs ont ensuite sélectionné des mutants multipoints dont les mutations individuelles ne chevauchaient pas celles des données d’entraînement, ce qui a donné lieu à un autre ensemble de tests difficiles. Dans ce contexte, nous constatons que les performances du modèle de base en mode zéro-shot évoluent considérablement avec la taille de l'ensemble d'entraînement.
Pour les mutations ponctuelles à différentes positions, le modèle amélioré par la régression de crête n'est pas plus performant que le modèle de base, même avec 100 exemples d'entraînement. Pour les mutations multipoints, lorsque la taille de la formation est inférieure à 60, la méthode de régression de crête ne peut pas améliorer efficacement les performances de GEMME et ESM-2. En revanche, les PLM formés à l’aide de FSFP avaient des scores de corrélation de Spearman plus élevés que tous les modèles de base à toutes les tailles de formation. aussi,Les modèles les plus performants sur la plupart des ensembles de données sont ceux formés avec FSFP.
Comparaison complète de 4 protéines, FSFP présente de plus grands avantages dans la formation avec de petits ensembles de données
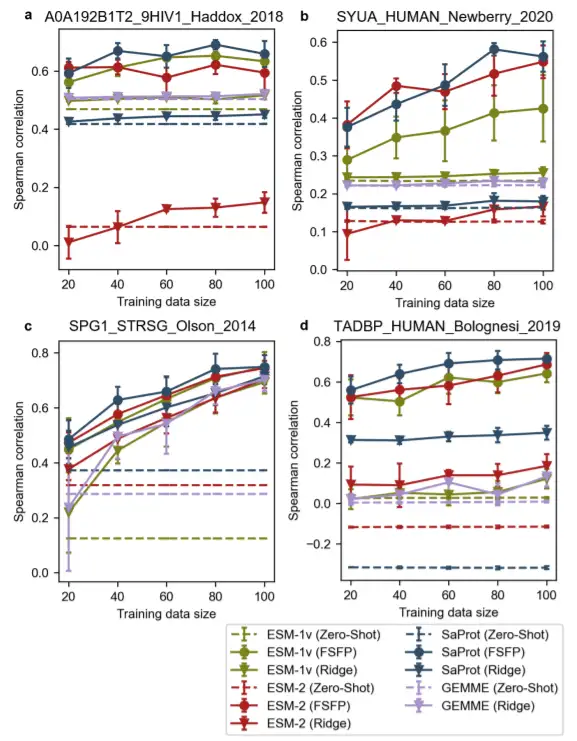
Pour démontrer davantage l’applicabilité et la généralisation du FSFP,L'étude a également montré les résultats de comparaison entre différentes méthodes pour quatre protéines : la protéine d'enveloppe Env du VIH, l'α-synucléine humaine, la protéine G (GB1), la protéine de liaison à l'ADN TAR humaine 43 (TDP-43). Dans plusieurs de ces cas, un ou plusieurs modèles non supervisés ont donné de mauvais résultats.
Notamment, pour TDP-43, toutes les corrélations de Spearman pour les prédictions à échantillon zéro sont proches de zéro. À l’exception de GB1, la plupart des modèles améliorés par la régression de crête n’obtiennent pas non plus d’améliorations significatives des performances sur des ensembles de données d’entraînement plus volumineux. En revanche, les modèles pré-entraînés peuvent réaliser des gains considérables lorsqu'ils sont formés sur de petits ensembles de données à l'aide de FSFP.
En utilisant FSFP pour concevoir l'ADN polymérase Phi29, le taux positif a augmenté de 25%
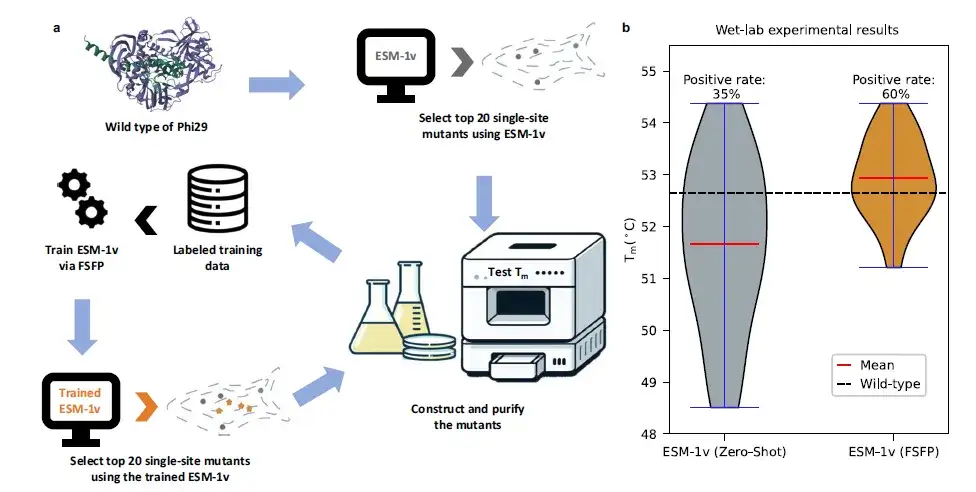
L’étude a également examiné un cas spécifique de modification de la protéine Phi29.Une vérification par test humide a été effectuée.Sur la base d'un ensemble limité de données expérimentales humides, cette étude a utilisé le FSFP pour entraîner ESM-1v, l'a utilisé pour trouver de nouveaux mutants à site unique et a effectué une vérification expérimentale. En comparant les 20 meilleurs résultats de prédiction de l'ESM-1v avant et après l'entraînement FSFP, la valeur moyenne de Tm a augmenté de plus de 1 ℃ et le taux positif a augmenté de 25%.
Plus précisément, les meilleurs mutants (c'est-à-dire les mutants avec les valeurs Tm les plus élevées) trouvés par ESM-1v (FSFP) ont également été recommandés par ESM-1v (zero-shot). Cependant, parmi les mutants positifs prédits par ESM-1v (FSFP), 9 n'apparaissent pas dans les données de formation, ce qui indique que FSFP peut permettre aux PLM d'identifier davantage de variantes de protéines.Ces résultats confirment le potentiel du FSFP pour accélérer les cycles itératifs de conception et de test de l’ingénierie des protéines.Cela pourrait aider à développer des protéines avec des caractéristiques fonctionnelles améliorées.
Représentant typique de l'IA pour la bio-ingénierie, une alliance forte se tient à l'avant-garde de l'époque
Aujourd’hui, alors que l’IA et la recherche scientifique sont étroitement intégrées, nous nous trouvons face à une opportunité historique. Le professeur Hong Liang estime que même si l'industrie biopharmaceutique chinoise est très forte, il existe encore une marge d'amélioration dans sa part de profit dans la chaîne industrielle internationale. Grâce à l’IA, nous avons la possibilité de « changer de voie et de dépasser » et d’utiliser directement la puissance de l’intelligence artificielle pour promouvoir le développement de l’industrie. C'est sur la base de ce concept que le professeur Hong Liang et le chercheur Tan Pan ont lancé des explorations sans fin dans le domaine de l'IA pour la bio-ingénierie.
Le Dr Tan Pan se concentre sur la biophysique moléculaire, la conception de protéines fonctionnelles par intelligence artificielle et la conception de molécules médicamenteuses.A publié 15 articles SCI dans des revues telles que Nature Communications, PRL, Journal of Cheminformatics, PCCP, etc. A développé une variété d'algorithmes de conception et de modification de protéines assistés par l'IA. En combinant l’expertise du professeur Hong Liang avec les algorithmes d’IA du Dr Tan Pan, la recherche collaborative entre les deux parties a donné lieu à des succès répétés.
Au fil des ans, les deux parties se sont concentrées sur la recherche innovante sur l'intelligence artificielle générale dans le domaine de l'ingénierie des protéines et ont développé avec succès la série pro d'intelligence artificielle générale pour l'ingénierie des protéines. De la même manière que ChatGPT comprend le langage humain, la série pro utilise de grands modèles pour comprendre la disposition des acides aminés des protéines dans la nature et conçoit des produits protéiques aux performances supérieures. Parmi eux, on compte deux produits phares dans l’application industrielle :
* Anticorps à domaine unique extrêmement résistant aux alcalis :Le premier produit protéique à grande échelle au monde, conçu conjointement avec Jinsai Pharmaceuticals, a atteint 5 000 litres de production industrielle, offrant une nouvelle solution pour la purification des macromolécules biologiques.
* Glycosyltransférase :En coopération avec Hanhai New Enzyme, nous avons développé l'enzyme permettant de produire l'EPS-G7, le matériau de base pour le dépistage de la pancréatite, brisant ainsi le monopole étranger à long terme et réduisant considérablement les coûts.
Ces deux cas marquent la première et la deuxième conception de modèles à grande échelle au monde et la réussite de la mise à l’échelle de la production vers l’étape d’industrialisation des produits protéiques. S'appuyant sur sa profonde expérience dans le domaine de la conception de protéines IA, le professeur Hong Liang a fondé Shanghai Tianwu Technology Co., Ltd. en 2021. En seulement trois ans, la société a non seulement mené à bien plusieurs projets de conception de protéines, mais a également obtenu des dizaines de millions de yuans lors d'un tour de financement pré-A. Les investisseurs comprennent des institutions bien connues telles que Glory Ventures et GSR Ventures.
À l'heure actuelle, les services de la société couvrent de nombreux domaines tels que les médicaments innovants, les diagnostics in vitro, la biologie synthétique, etc., et elle recherche activement une coopération avec davantage d'instituts de recherche scientifique et d'entreprises, et s'engage à établir une référence nationale et même mondiale dans le domaine de l'ingénierie des protéines.
Dans le domaine extrêmement compétitif de l’ingénierie des protéines, la vision du professeur Hong Liang est claire :Nous devons non seulement devenir un leader national, mais aussi un leader mondial.Dans le cadre de leur future recherche scientifique, le professeur Hong Liang et son équipe s'engagent à élargir leur coopération approfondie avec les institutions et entreprises de recherche scientifique mondiales, à explorer constamment les possibilités infinies de la conception des protéines, à s'efforcer de réaliser des percées technologiques et des innovations d'application dans ce domaine, à établir une référence au niveau national et à démontrer leur excellence à l'échelle internationale.
Enfin, je recommande une activité de partage académique en ligne. Les amis intéressés peuvent scanner le code QR pour participer !









