Command Palette
Search for a command to run...
L'université De Cambridge a Mis Au Point Un Classificateur d'images De Cellules Sanguines ; Son Modèle De Diffusion Facilite La Détection De La Leucémie, Surpassant Les Capacités Des Experts cliniques.

L'analyse d'images des cellules sanguines joue un rôle crucial dans le diagnostic clinique et la recherche scientifique. Les caractéristiques morphologiques des globules blancs, des globules rouges et des plaquettes reflètent non seulement la santé du système sanguin, mais peuvent également révéler des signes précoces de maladies telles que la leucémie et les syndromes myélodysplasiques. Cependant, l'analyse microscopique manuelle traditionnelle repose sur l'expertise de spécialistes pour la classification manuelle, ce qui est inefficace, chronophage et sujet à des biais subjectifs.
Ces dernières années, les technologies d'apprentissage profond ont été de plus en plus utilisées dans le domaine de l'analyse d'images médicales, et certaines études ont tenté d'appliquer des modèles discriminatifs, notamment les réseaux de neurones convolutifs (CNN), à l'évaluation de la morphologie des cellules sanguines. Bien que les modèles de classification ML discriminatifs les plus performants puissent se rapprocher des performances humaines dans la classification des cellules en catégories prédéfinies, ils apprennent principalement les frontières de décision à partir d'étiquettes d'experts. Par conséquent,Ils ne sont pas naturellement conçus pour saisir une distribution complète des données de morphologie cellulaire.Cette limitation réduit leurs capacités, notamment face à la complexité et à la variabilité inhérentes aux données d'hématologie clinique.
Dans ce contexte,Une équipe de recherche de l'Université de Cambridge, au Royaume-Uni, a proposé CytoDiffusion, une méthode de classification d'images de cellules sanguines basée sur un modèle de diffusion.Il peut modéliser fidèlement la distribution morphologique des cellules sanguines, réaliser une classification précise et possède de puissantes capacités de détection des anomalies, une résistance aux variations de distribution, une interprétabilité, une grande efficacité des données et des capacités de quantification de l'incertitude qui surpassent celles des experts cliniques.
Le modèle surpasse les modèles discriminants de pointe en matière de détection d'anomalies (AUC : 0,990 contre 0,916), de robustesse aux changements de distribution (précision : 0,854 contre 0,738) et de performances dans les scénarios de faible quantité de données (précision équilibrée : 0,962 contre 0,924).Le cadre d'évaluation complet développé dans cette étude établit une référence multidimensionnelle pour l'analyse d'images médicales hématologiques, ce qui devrait améliorer la précision du diagnostic en milieu clinique.
Les résultats de cette recherche, intitulée « Classification générative profonde de la morphologie des cellules sanguines », ont été publiés dans la revue Nature.
Points saillants de la recherche :
* Application d'un modèle de diffusion latente à la classification d'images de cellules sanguines.
* Nous proposons un cadre d'évaluation qui va au-delà des mesures standard telles que la précision, en intégrant la robustesse aux changements de distribution, les capacités de détection d'anomalies et les performances dans des scénarios de faible quantité de données.
* Construire un nouvel ensemble de données d'images de cellules sanguines incluant les artefacts d'imagerie et le niveau de confiance des annotateurs, en remédiant aux principales limitations des ensembles de données existants.

Adresse du document :
https://www.nature.com/articles/s42256-025-01122-7
Suivez le compte public et répondez à «CytodiffusionObtenez le PDF complet
Adresse du jeu de données :
Autres articles sur les frontières de l'IA :
https://hyper.ai/papers
Jeux de données : Combinaison de jeux de données accessibles au public et de jeux de données personnalisés
Les données constituent le fondement de l'analyse d'images de cellules sanguines et une garantie essentielle pour la performance et la capacité de généralisation des modèles d'IA. L'équipe CytoDiffusion a utilisé cinq ensembles de données, dont quatre sont accessibles au public et le cinquième est un ensemble de données auto-construit appelé CytoData.
L'ensemble de données CytoData est un jeu de données anonymisé contenant 2 904 frottis sanguins provenant de l'hôpital Addenbrooke de Cambridge, soit un total de 559 808 images de cellules uniques. Parmi celles-ci, 4 996 images sont annotées avec dix types de cellules sanguines, notamment les érythroblastes, les éosinophiles, les monocytes et les cellules immatures. Les images ont été acquises à l'aide d'un système CellaVision DM9600, et les annotations comprennent des scores de confiance attribués par chaque expert, fournissant ainsi une référence importante pour la quantification ultérieure de l'incertitude. CytoData inclut également des catégories d'artefacts afin de prendre en compte les interférences structurelles non cellulaires courantes dans les frottis sanguins, ce qui est d'une grande utilité pour les applications cliniques.
Raabin-WBC, PBC, Bodzas, LISC comme 4 ensembles de données publicsL'ensemble de données comprend des images de cellules sanguines obtenues à l'aide de différents microscopes, méthodes de coloration et équipements. Raabin-WBC propose deux partitions : Test-A et Test-B. Le Test-B utilise un équipement d'acquisition différent de celui de l'ensemble d'entraînement afin de simuler un changement de domaine. Du fait de ces différences d'équipement et de méthodes de coloration, l'ensemble de données LISC met davantage l'accent sur la capacité de généralisation du modèle.
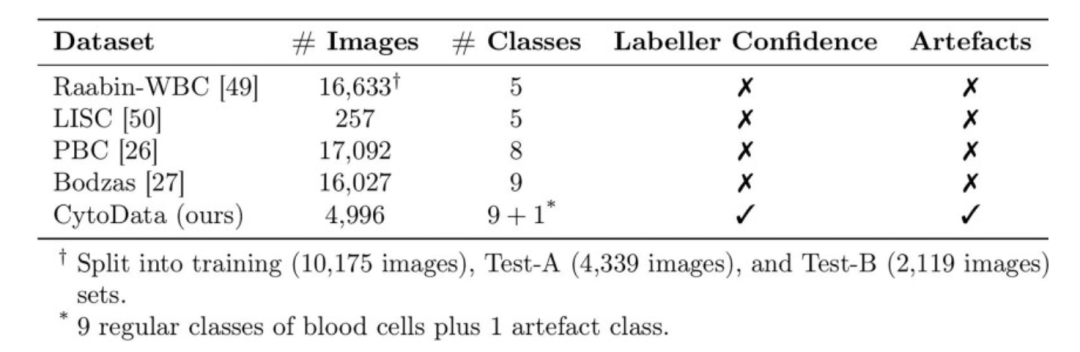
En combinant ces ensembles de données multi-sources, l'équipe a non seulement assuré la diversité de l'entraînement du modèle, mais a également fourni une base complète pour l'évaluation des performances inter-domaines, la détection des cellules anormales et le test du modèle dans des conditions de données rares.
Adresse du jeu de données :
Cadre conceptuel : Application d'un modèle de diffusion à la classification d'images de cellules sanguines
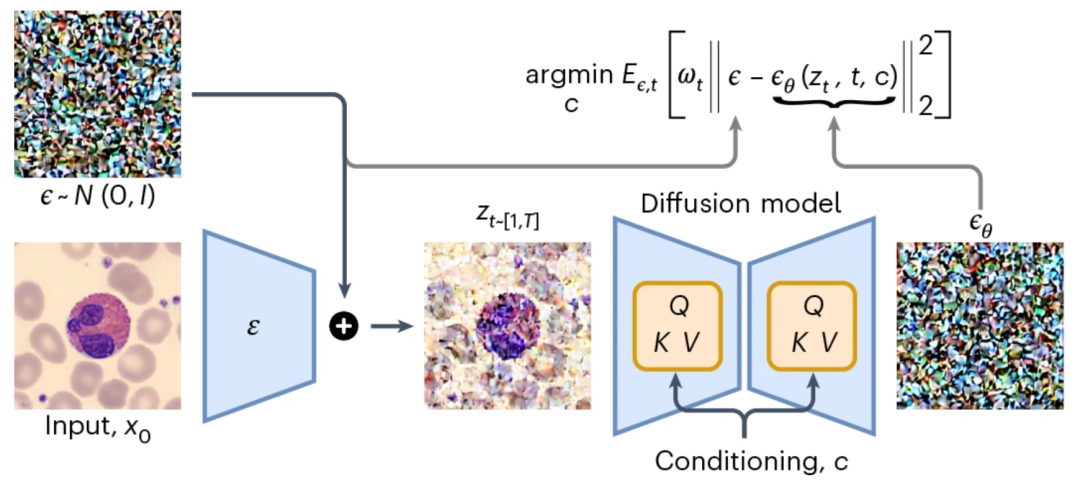
L’innovation fondamentale de CytoDiffusion réside dans l’application d’un modèle de diffusion à la classification d’images de cellules sanguines.Contrairement aux modèles discriminatifs traditionnels, les modèles de diffusion possèdent des propriétés génératives, leur permettant d'apprendre la distribution complète d'une image et de la classifier grâce à un mécanisme de prédiction du bruit.
Principes du modèle
Le principe fondamental du modèle de diffusion est de définir un processus de diffusion directe qui transforme les données en une distribution de type bruit en ajoutant progressivement du bruit. Le modèle apprend ensuite un processus inverse pour débruiter les données, reconstruisant ainsi efficacement la distribution des données originales.
Encodage de l'espace latent :L'image d'entrée est d'abord mappée sur l'espace latent par un encodeur, puis un bruit gaussien est ajouté pour former une représentation latente bruitée ;
Diffusion conditionnelle :Le modèle génère des prédictions de bruit pour chaque type de cellule et effectue la classification en minimisant l'erreur entre le bruit prédit et le bruit réel.
Éliminer progressivement :Un échantillonnage itératif est effectué sur toutes les catégories candidates, et les catégories impossibles sont éliminées étape par étape à l'aide du test t de Student apparié jusqu'à ce que la catégorie finale soit déterminée.
Cadres de formation générale
Les chercheurs ont utilisé le modèle de diffusion stable 1.5 comme modèle de base.Pour les conditions basées sur les catégories, l'algorithme contourne le tokenizer et l'encodeur de texte, fournissant directement des vecteurs encodés en one-hot pour chaque catégorie. Ces vecteurs sont dupliqués verticalement et complétés horizontalement pour correspondre à la matrice attendue de dimension 77 × 768. Un lot de 10, un taux d'apprentissage de 10⁻⁵ et une phase d'échauffement linéaire de 1 000 itérations ont été utilisés, l'entraînement étant réalisé sur un GPU A100-80GB.
Formation et raisonnement
Les chercheurs ont appliqué diverses méthodes d'augmentation de données pendant l'entraînement, notamment des retournements diagonaux aléatoires, des rotations aléatoires (échantillonnage uniforme entre 0 et 359 degrés), des variations de couleur (luminosité = 0,25, contraste = 0,25, saturation = 0,25, teinte = 0,125), Mixup (α = 0,3, appliqué aux entrées conditionnelles plutôt qu'à la cible) et RandAugment (en utilisant les paramètres par défaut).
L'entraînement a utilisé l'optimiseur AdamW (β1 = 0,9, β2 = 0,999, ϵ = 10⁻⁸, décroissance du poids de 0,01), un entraînement en précision mixte (fp16) et une moyenne mobile exponentielle (0,9999). Toutes les images ont été redimensionnées uniformément à 360 × 360 pixels.
Lors de la phase d'inférence, les mêmes méthodes d'augmentation de données que lors de la phase d'entraînement ont été appliquées, à l'exception de Mixup. Tirant parti du fait que les globules blancs sont généralement situés au centre de l'image, et afin de réduire les interférences dues à l'augmentation des données en périphérie, l'erreur d'inférence a été calculée uniquement dans un rayon de 20 pixels autour du centre de l'image dans l'espace latent. La dimension de cet espace latent était de 45 × 45 × 4.
Présentation des résultats : CytoDiffusion peut contribuer à résoudre les principaux défis liés au déploiement clinique.
Génération d'images et vérification d'authenticité
L'application clinique des systèmes d'intelligence artificielle exige non seulement des performances élevées, mais aussi des modèles dotés de capacités de représentation fiables. Afin de démontrer que CytoDiffusion apprend la distribution morphologique réelle des cellules sanguines, plutôt que de recourir à des « raccourcis » tels que des artefacts, les chercheurs ont mené un test de réalisme.
Sur la base de 32 619 images d'entraînement, les images de cellules sanguines générées par CytoDiffusion sont pratiquement indiscernables des images réelles. Dix hématologues ont réalisé des tests d'identification sur 2 880 images, obtenant une précision globale de 0,523 (niveau de réponse aléatoire), une sensibilité de 0,558 et une spécificité de 0,489. Ces performances, proches de celles obtenues par une réponse aléatoire, indiquent que même pour des professionnels expérimentés, les images de cellules sanguines générées par CytoDiffusion sont pratiquement indiscernables des images réelles.
La capacité à générer des images presque indiscernables des images réelles indique que CytoDiffusion a réussi à apprendre la véritable distribution de la morphologie des cellules sanguines, comme le montre la figure ci-dessous :
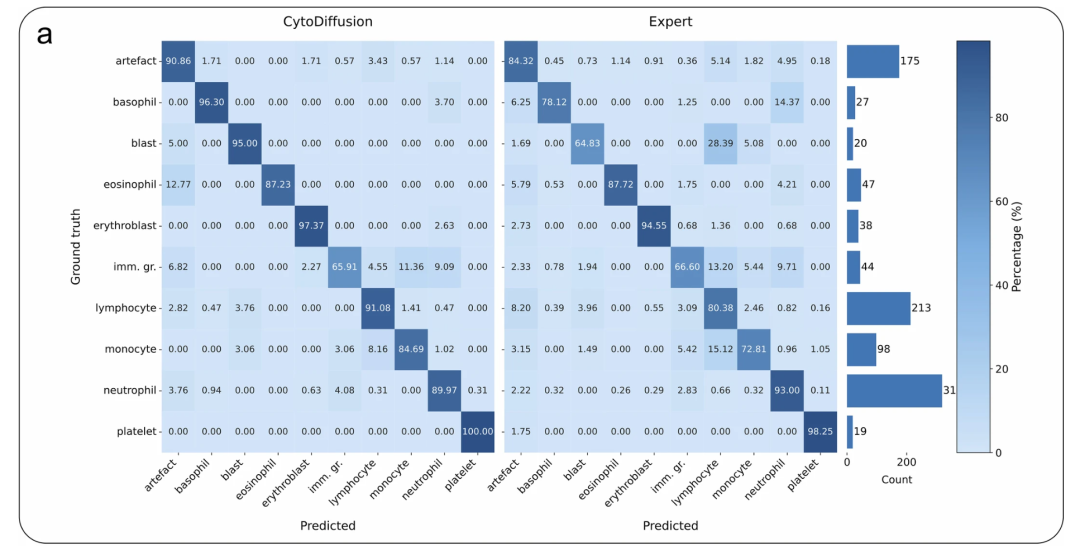
Comparaison des données CytoData : la matrice de gauche présente les résultats de CytoDiffusion, et la matrice de droite présente les performances moyennes des experts humains.
Performances de classification
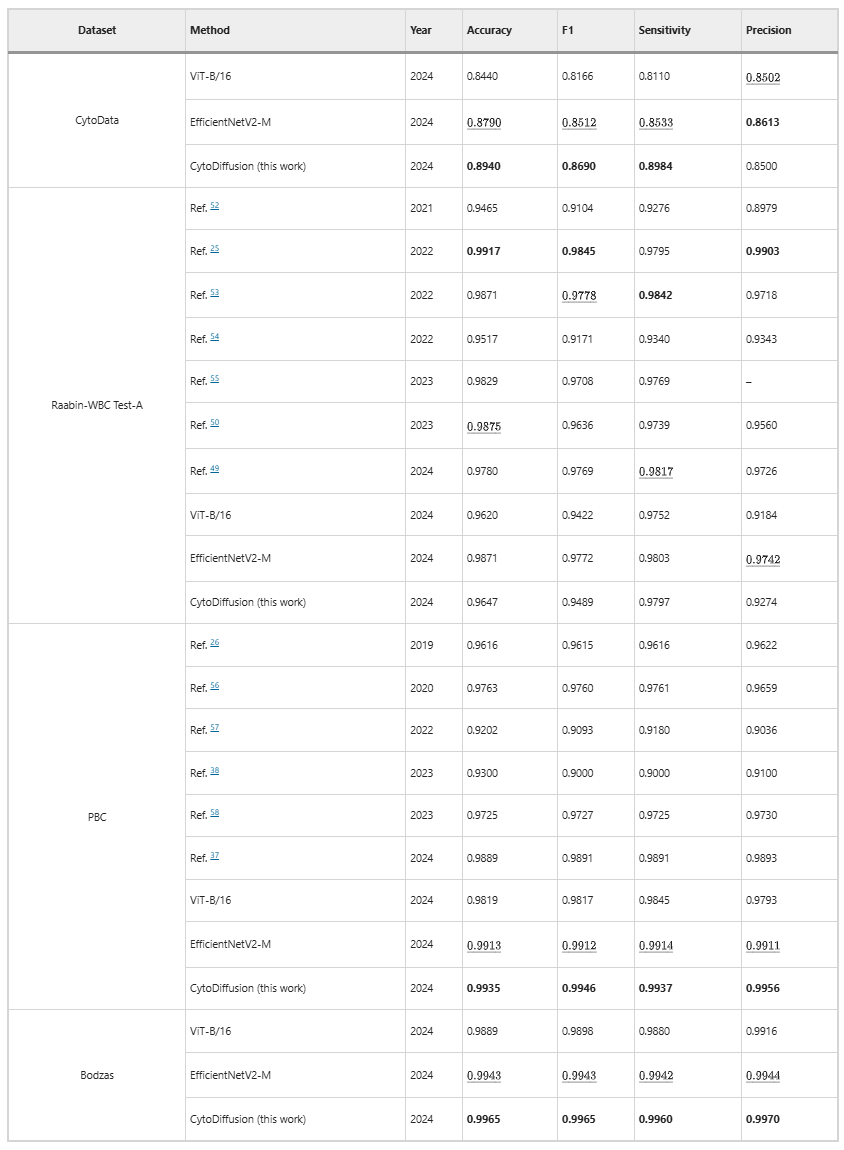
Sur quatre jeux de données (CytoData, Raabin-WBC, PBC et Bodzas), CytoDiffusion atteint, voire surpasse, les performances des modèles discriminatifs traditionnels. Plus particulièrement sur CytoData, PBC et Bodzas, le modèle affiche des performances de pointe, démontrant ainsi que les méthodes basées sur la diffusion peuvent égaler, voire surpasser, les modèles discriminatifs traditionnels, comme l'illustre le tableau ci-dessous :
La quantification de l'incertitude est supérieure à celle des experts humains
Les systèmes biologiques présentent par nature une incertitude irréductible. Dans toute tâche analytique, la mesure ne doit pas se limiter à la précision des prédictions, mais prendre également en compte l'incertitude de l'acteur (humain ou machine).
Les chercheurs ont utilisé des techniques de modélisation psychométrique bayésienne établies pour dériver la fonction psychométrique de CytoDiffusion, comme le montre la figure ci-dessous.Les résultats montrent que le modèle s'ajuste très bien et que les distributions a posteriori des principaux paramètres de seuil et de largeur sont très compactes (axes de coordonnées intégrés dans la figure ci-dessous).Bien qu'elle ne puisse être mesurée directement, ces résultats suggèrent que l'incertitude de la cytodiffusion est principalement due à une composante aléatoire, et que son comportement ressemble fortement à celui d'un observateur idéal.
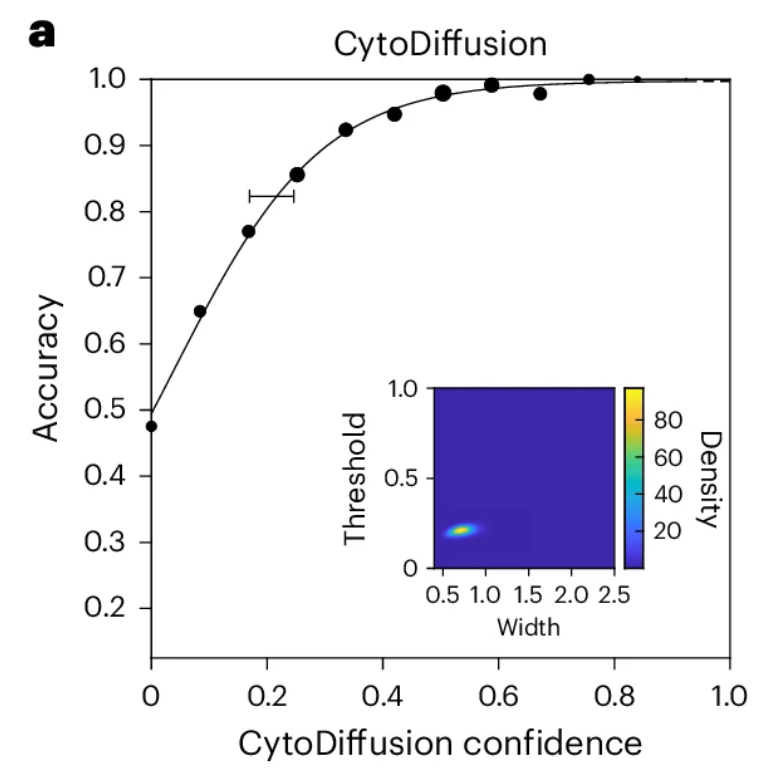
Détection et performance des cellules anormales dans des environnements à faible quantité de données
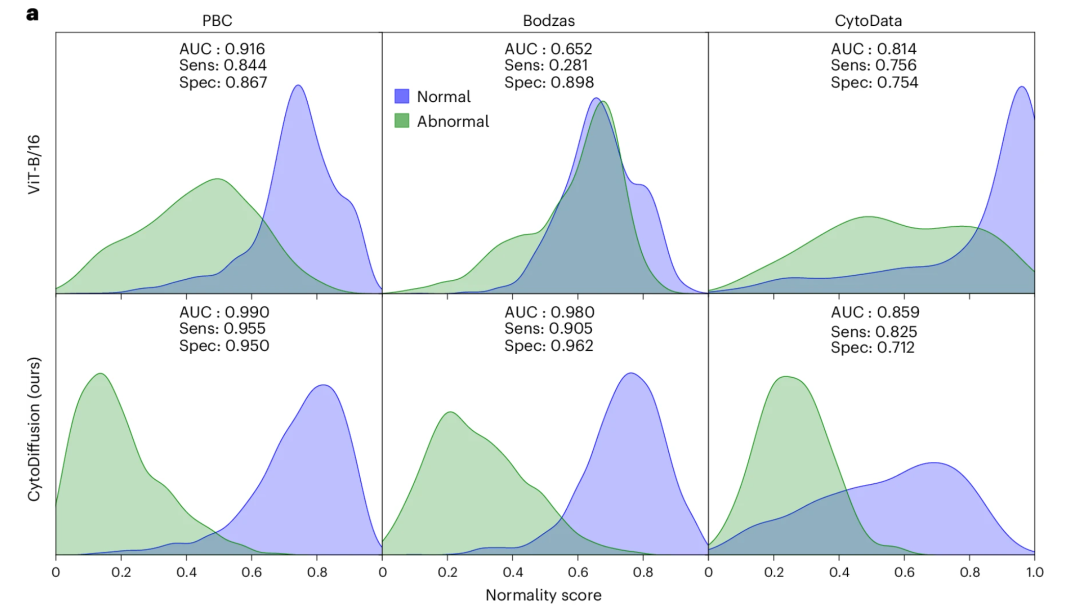
Pour la détection des cellules anormales, CytoDiffusion a obtenu une sensibilité élevée (0,905) et une spécificité élevée (0,962) en utilisant les cellules primitives comme catégorie d'anomalie dans l'ensemble de données Bodzas. En revanche, ViT a montré une sensibilité extrêmement faible (0,281), ne répondant clairement pas aux exigences des applications cliniques, comme le montre la figure ci-dessous :
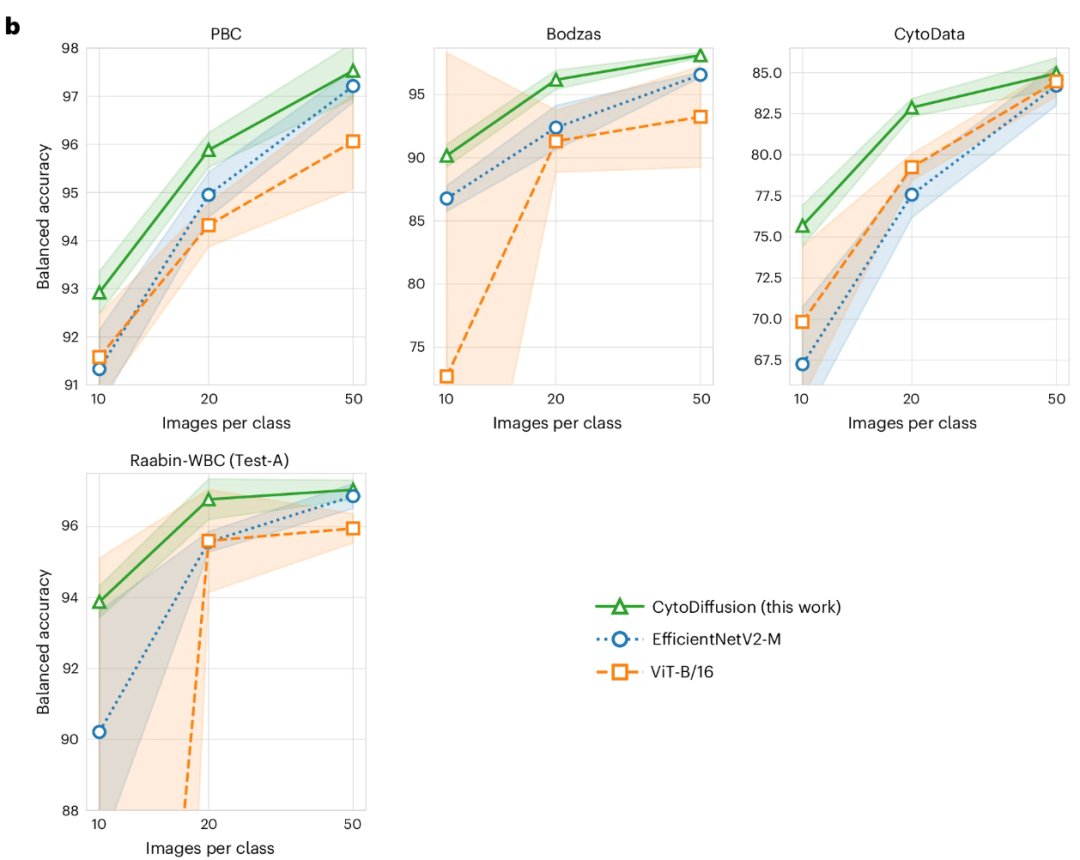
Dans les environnements à faible quantité de données, avec seulement 10 à 50 images d'entraînement par classe, CytoDiffusion surpasse significativement EfficientNetV2-M et ViT-B/16, démontrant ainsi sa capacité d'apprentissage efficace dans des conditions de données rares, comme le montre la figure ci-dessous :
Capacité de généralisation du modèle
Pour évaluer la capacité de généralisation du modèle, les chercheurs ont testé ses performances sur différents ensembles de données. Les modèles entraînés sur Raabin-WBC ont été testés sur les ensembles de données Test-B (avec différents microscopes et caméras) et LISC (avec différents microscopes, caméras et méthodes de coloration) ; les modèles entraînés sur CytoData ont été testés sur PBC et Bodzas. CytoDiffusion a atteint une précision optimale sur les quatre ensembles de données.Cet avantage en matière de cohérence, malgré différents degrés de dérive du domaine, démontre que CytoDiffusion est robuste aux variations des ensembles de données et possède une bonne capacité de généralisation dans des scénarios cliniques réels.
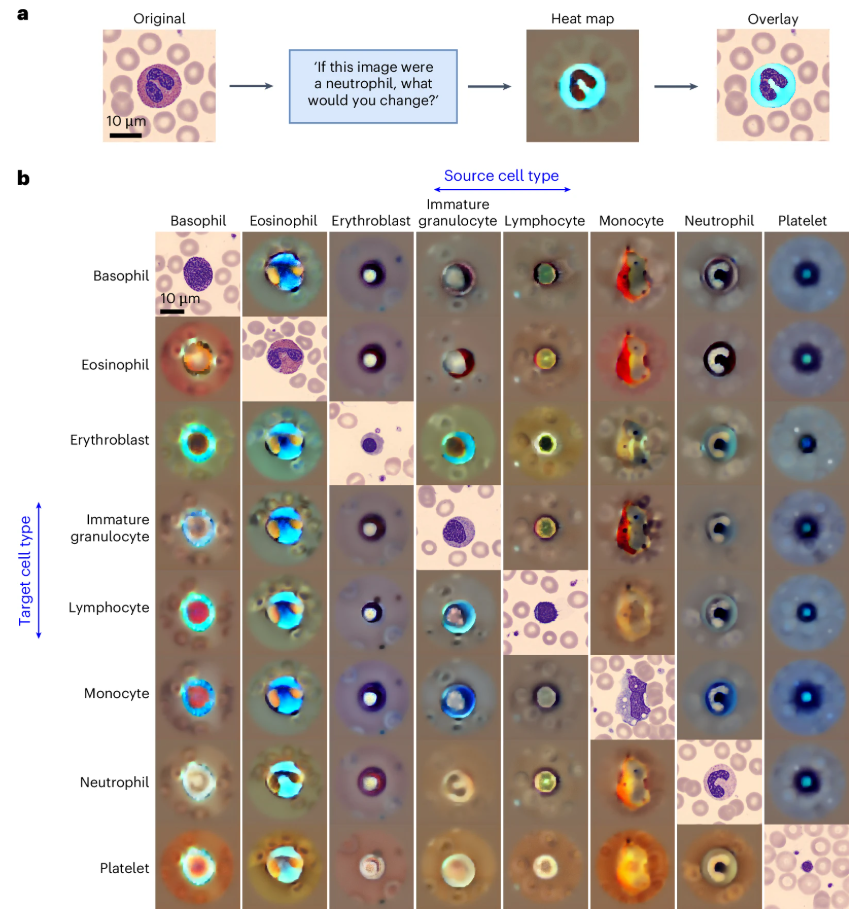
Vérification de l'explicabilité
Grâce à l'analyse de cartes thermiques contrefactuelles, le modèle identifie les principales caractéristiques morphologiques des cellules, comme illustré ci-dessous. Par exemple, lors de la transition des monocytes aux granulocytes immatures, le modèle met en évidence les différences d'acidité cytoplasmique et de remplissage vacuolaire. Cette visualisation valide non seulement la capacité d'apprentissage du modèle, mais permet également de détecter d'éventuels biais, garantissant ainsi la cohérence des critères de classification avec la logique scientifique clinique.
Les modèles de diffusion s'avèrent révolutionnaires dans le domaine biomédical.
Les recherches de CytoDiffusion démontrent non seulement le potentiel des modèles de diffusion dans la classification de la morphologie des cellules sanguines, mais reflètent également l'essor rapide des cadres génératifs basés sur la diffusion dans l'ensemble du domaine biomédical, démontrant une valeur révolutionnaire dans de multiples scénarios d'application.
Par exemple, les données médicales sont souvent limitées et soumises à d'importantes problématiques de confidentialité, ce qui rend leur acquisition et leur annotation particulièrement difficiles. Les modèles de diffusion peuvent pallier ce problème en générant des images médicales synthétiques.Cela permet de former des modèles d'apprentissage profond et d'améliorer la précision de l'analyse d'images médicales ;Outre la génération d'images médicales classiques, l'expansion...Le modèle de dispersion peut également être utilisé pour générer des images médicales pour des affections spécifiques (telles que des tumeurs, des fractures, etc.).Ceci est particulièrement important pour l'entraînement des modèles de diagnostic médical, car cela permet de fournir davantage d'exemples d'entraînement pour les maladies rares ou les images difficiles à obtenir. Parallèlement, les modèles de diffusion peuvent générer des images de haute qualité, nettes et réalistes, ce qui contribue non seulement à améliorer la précision des diagnostics médicaux, mais aussi à aider les systèmes d'IA médicale à effectuer des prédictions plus précises.
Dans de nombreux contextes cliniques et de recherche, la rareté des ensembles de données d'imagerie médicale de haute qualité limite le potentiel de l'intelligence artificielle (IA) dans les applications cliniques. En décembre 2024, les professeurs Kang Zhang et Jia Qu de l'hôpital ophtalmologique affilié à l'université de médecine de Wenzhou, ainsi que le chercheur Jinzhuo Wang de l'université de Pékin, étaient les auteurs correspondants, tandis que le docteur Kai Wang de l'université de Pékin et le docteur Yunfang Yu de l'hôpital commémoratif Sun Yat-sen de l'université Sun Yat-sen étaient co-premiers auteurs.Un nouveau cadre de travail, MINIM, basé sur un modèle de diffusion, a été développé. Ce modèle permet de générer des images médicales de différents organes selon diverses modalités d'imagerie, à partir de commandes textuelles.Des évaluations cliniques et des mesures objectives rigoureuses ont validé la haute qualité des images générées par MINIM. Face à des domaines de données inédits, MINIM a démontré des capacités de génération accrues, révélant son potentiel en tant qu'IA médicale générale (IAMG).
Titre de l'article :Modèle de base génératif auto-améliorant pour la génération d'images médicales synthétiques et les applications cliniques
Adresse du document :https://www.nature.com/articles/s41591-024-03359-y
En biologie cellulaire, les cellules vivantes sont des systèmes dissipatifs complexes, très éloignés de l'équilibre chimique. Comprendre leur réponse collective aux stimuli externes a toujours été une question scientifique fondamentale que les chercheurs s'efforcent de résoudre. Novembre 2025.Des équipes de recherche de l'Université Columbia, de l'Université Stanford et d'autres institutions ont développé le cadre de calcul Squidiff.Ce cadre, basé sur un modèle implicite de diffusion débruité conditionnellement, permet de prédire les réponses transcriptomiques de différents types cellulaires lors d'inductions de différenciation, de perturbations géniques et de traitements médicamenteux. Son principal atout réside dans sa capacité à intégrer des informations explicites issues d'outils d'édition génique et de composés médicamenteux : pour prédire la différenciation des cellules souches, il permet non seulement de saisir avec précision les états cellulaires transitoires, mais aussi d'identifier les effets non additifs des perturbations géniques et les caractéristiques de réponse spécifiques à chaque type cellulaire. L'équipe de recherche a ensuite appliqué Squidiff à la recherche sur les organoïdes vasculaires, prédisant avec succès les effets de l'exposition aux radiations sur différents types cellulaires et évaluant l'efficacité protectrice des médicaments radioprotecteurs.
Titre de l'article :Squidiff : prédiction du développement cellulaire et des réponses aux perturbations à l'aide d'un modèle de diffusion
Adresse du document :https://www.nature.com/articles/s41592-025-02877-y
Il est prévisible qu'à mesure que les modèles de base génératifs gagneront en maturité dans le domaine médical, les modèles de diffusion seront mis en œuvre dans des scénarios cliniques plus réels, devenant ainsi un fondement important de l'intelligence médicale générale, apportant une fiabilité accrue, une capacité de généralisation plus forte et un champ d'application plus large pour le futur diagnostic d'images médicales, la prédiction des maladies et la prise de décision intelligente.
Références :
1.https://www.nature.com/articles/s42256-025-01122-7
2.https://www.nature.com/articles/s41592-025-02877-y
3.https://mp.weixin.qq.com/s/9JEt-QwFxngv9XC0hSIcnw
4.https://bbs.huaweicloud.com/blogs/448218








