Command Palette
Search for a command to run...
Découvrez l'ensemble De Données De Structure Des Protéines PDB Derrière AlphaFold 2 Dans Un Seul Article
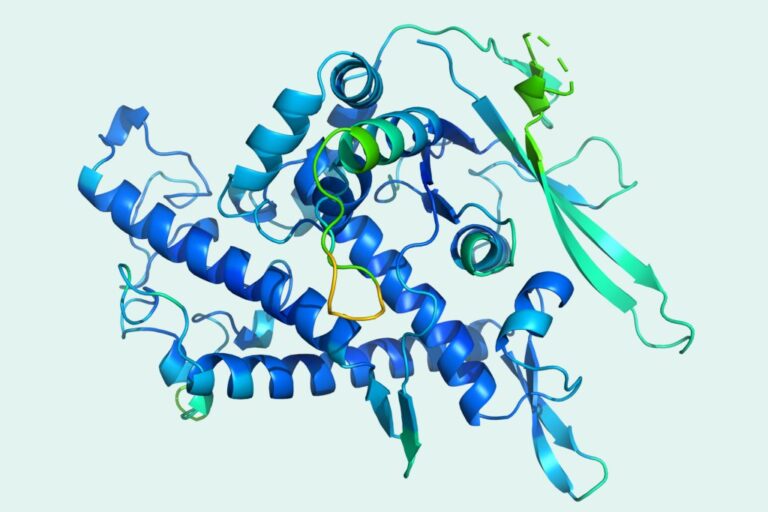
L'algorithme de dernière génération de DeepMind, AlphaFold 2, a complètement battu les autres concurrents du CASP, récemment surnommé les « Jeux olympiques des protéines », et a réalisé des percées étonnantes, choquant l'ensemble du cercle de recherche scientifique. Après avoir été submergé par ce résultat de recherche scientifique, examinons l’ensemble de données derrière l’algorithme.
Au cours des deux derniers jours, nous avons été bombardés de nouvelles concernant l'algorithme d'intelligence artificielle de nouvelle génération AlphaFold 2 de DeepMind, en particulier dans le monde biologique, qui peut être considéré comme ayant inauguré une avancée majeure.
Selon l'annonce officielle de DeepMind, son algorithme d'apprentissage profond AlphaFold 2 a résolu avec succès des problèmes majeurs dans le domaine biologique au cours des 50 dernières années.
L'algorithme peut prédire avec précision la structure 3D des protéines en fonction de leurs séquences d'acides aminés, avec une précision comparable aux structures 3D résolues à l'aide de techniques expérimentales telles que la microscopie cryoélectronique (CryoEM), la résonance magnétique nucléaire ou la cristallographie aux rayons X.

Cet événement marquant a enthousiasmé les biologistes, mais il a également fait trembler de peur de nombreuses personnes du secteur, qui ont déclaré qu'elles changeraient de carrière pour apprendre l'apprentissage profond.
Cependant, alors que tout le monde prête attention à ce résultat de recherche scientifique, n’oubliez pas le héros qui se cache derrière—— Ensemble de données sur la structure des protéines PDB, un ensemble de données dédié à la collecte de données sur la structure tridimensionnelle des protéines et des acides nucléiques.
Cet ensemble de données est essentiel pour une avancée révolutionnaire
Selon DeepMind, l’équipe a formé le système sur des données publiques.Ces données proviennent de l'ensemble de données sur la structure des protéines PDB et de la grande base de données UniProt contenant des séquences de protéines de structures inconnues, qui comprennent ensemble environ 170 000 structures de protéines.
dans,PDB est un ensemble de données dédié à la structure tridimensionnelle des protéines et des acides nucléiques. Son histoire est très longue, elle remonte à 1971.
Walter Hamilton du Brookhaven National Laboratory aux États-Unis a décidé de créer cette base de données. En octobre 1998, le PDB a été transféré au Research Collaboratory for Structural Bioinformatics (RCSB), dirigé par Helen M. Berman de l'Université Rutgers, également membre du RCSB.

En 2003,La PDB est devenue une organisation internationale, wwPDB (Worldwide Protein Database), pour superviser les ressources de la PDB. D'autres membres de wwPDB, notamment PDBe (Europe), RCSB (États-Unis) et PDBj (Japon), fournissent également des centres d'accumulation, de traitement et de publication de données pour PDB.

Il convient de mentionner que bien que les données du PDB soient soumises par des scientifiques du monde entier, chaque donnée soumise sera examinée et annotée par le personnel du wwPDB pour vérifier si les données sont raisonnables. La PDB et le logiciel qu’elle fournit sont désormais librement accessibles au public.
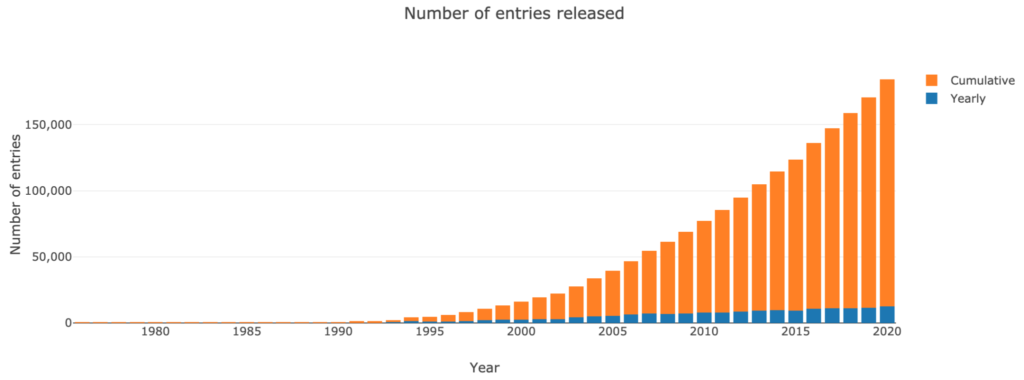
Plus de 140 000 structures, quelles informations contient la PDB ?
Au cours des dernières décennies, le nombre de structures dans la PDB a augmenté à un rythme quasi exponentiel :
- 100 en 1982;
- 1 000 en 1993;
- 10 000 en 1999;
- 100 000 en 2014.
Cependant, depuis 2007, le rythme auquel de nouvelles structures protéiques s’accumulent semble s’être stabilisé.
Les biologistes structurels du monde entier utilisent des méthodes telles que la cristallographie aux rayons X, la spectroscopie RMN et la microscopie cryoélectronique pour déterminer la position de chaque atome d’une molécule par rapport aux autres. Ils soumettent ensuite ces informations structurelles, qui sont annotées par wwPDB et publiées publiquement dans la base de données.
Vous pouvez rechercher des structures de ribosomes, d'oncogènes, de cibles médicamenteuses et même de virus entiers dans l'ensemble de données PDB.Cependant, le grand nombre de structures archivées dans la PDB rend la recherche des informations dont vous avez besoin une tâche ardue.
Les informations contenues dans l'ensemble de données PDB comprennent principalement :La source de protéines/acides nucléiques, la composition des molécules de protéines/acides nucléiques, les coordonnées atomiques, les méthodes expérimentales utilisées pour déterminer la structure, ainsi que d'autres données et informations telles que les facteurs de température et les déterminants de structure.
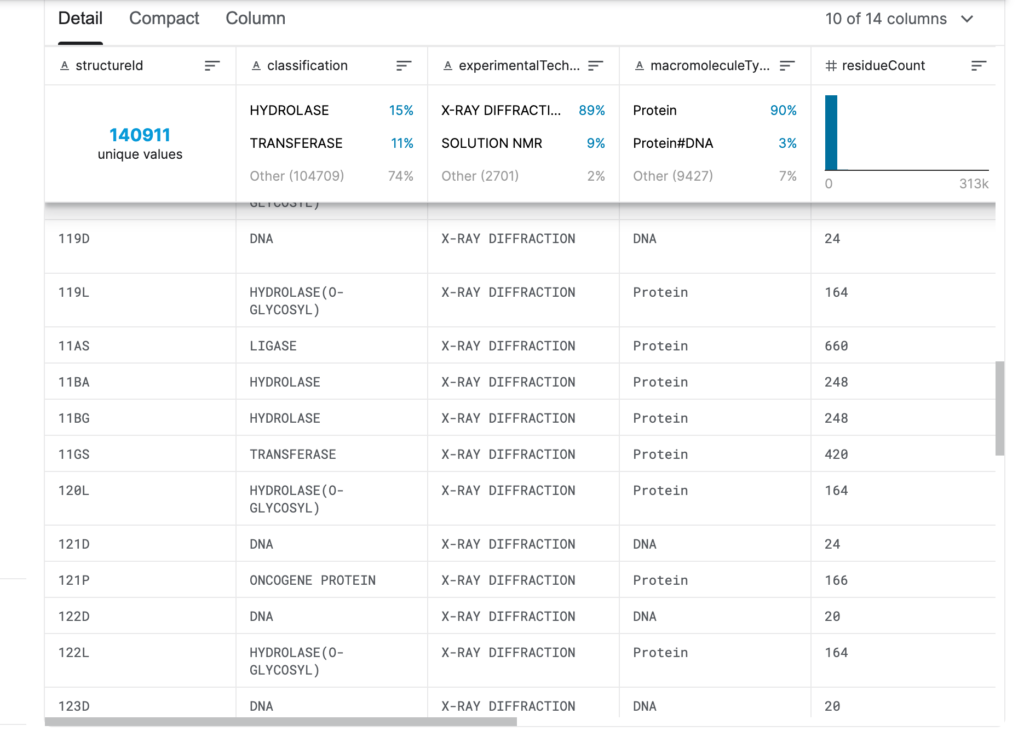
Comment télécharger ?
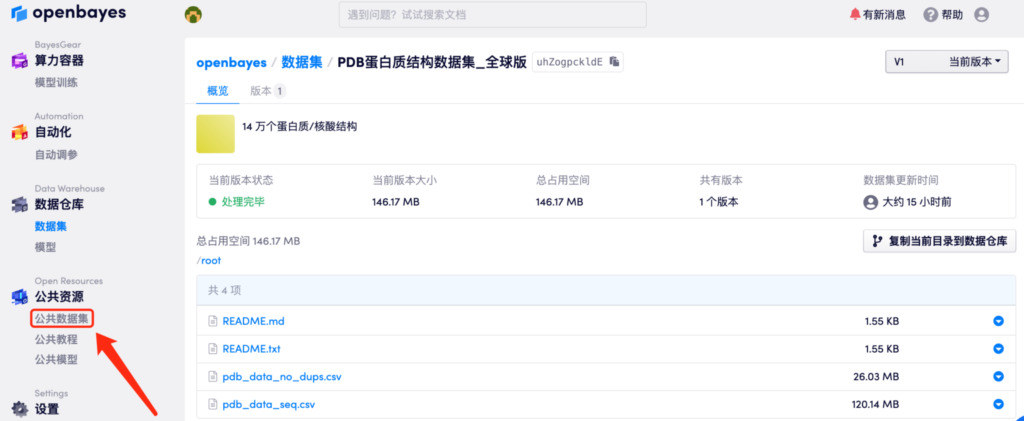
L'ensemble de données est désormais disponible sur le site officiel d'Hyperneuron et openbayes.com. Visite:https://orion.hyper.ai/datasets/13906 Ou cliquez sur « Lire le texte original » pour obtenir l'ensemble de données en un clic.
■ Détails de l'ensemble de données sur la structure des protéines PDB
Heure de sortie :Collectionné depuis 1971
Agence d'édition :wwPDB
Quantité incluse :Plus de 140 000 structures de protéines/acides nucléiques
Format des données :fichier csv
Taille des données :27 Mo (146 Mo après décompression)
Adresse de téléchargement :https://orion.hyper.ai/datasets/13906
Le même ensemble de données que DeepMind, vous le méritez aussi~
Comment l'utiliser ?
Notre partenaire OpenBayes est un service cloud qui fournit la puissance de calcul cloud pour l'apprentissage automatique. Ils disposent d'un cluster de supercalcul à grande échelle et l'architecture du cluster GPU est conçue spécifiquement pour le calcul matriciel. Il fournit des conteneurs de puissance de calcul pour les applications d'IA, et il est très facile à démarrer et peut être utilisé prêt à l'emploi.
Actuellement, les produits de conteneur de puissance de calcul d'OpenBayes prennent déjà en charge TensorFlow, PyTorch, MXNet et autres environnements CPU et GPU, différentes versions et types de frameworks d'apprentissage automatique standard et diverses dépendances courantes.
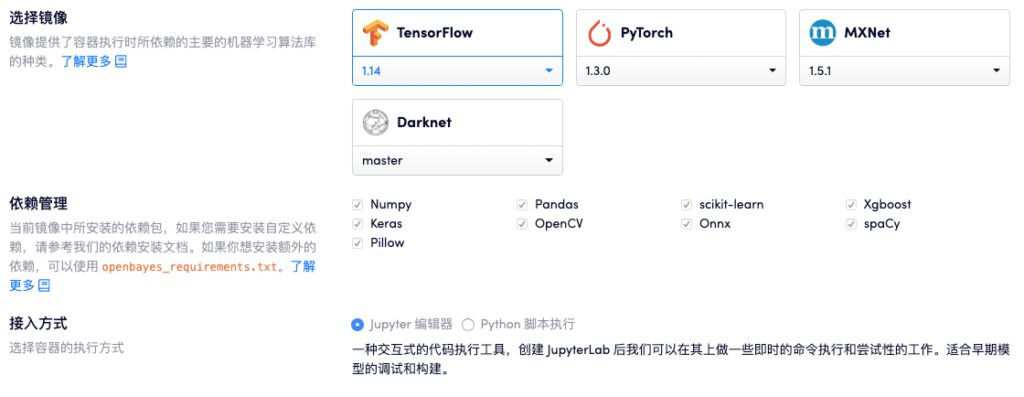
Actuellement, le conteneur informatique OpenBayes prend en charge les bibliothèques standardet fournir CPU, NVIDIA T4, NVIDIA Tesla V100 et autres ressources informatiquesQu'il s'agisse d'une formation centralisée de données massives ou d'un fonctionnement résident sur un modèle à faible consommation d'énergie, il peut facilement répondre aux besoins des utilisateurs.

Du CPU au T4 en passant par le V100, une large gamme de configurations de conteneurs informatiques Prise en charge d'OpenBayesTéléchargement de script et éditeur JupyterLabProgrammation en ligne puis formation sur modèle.
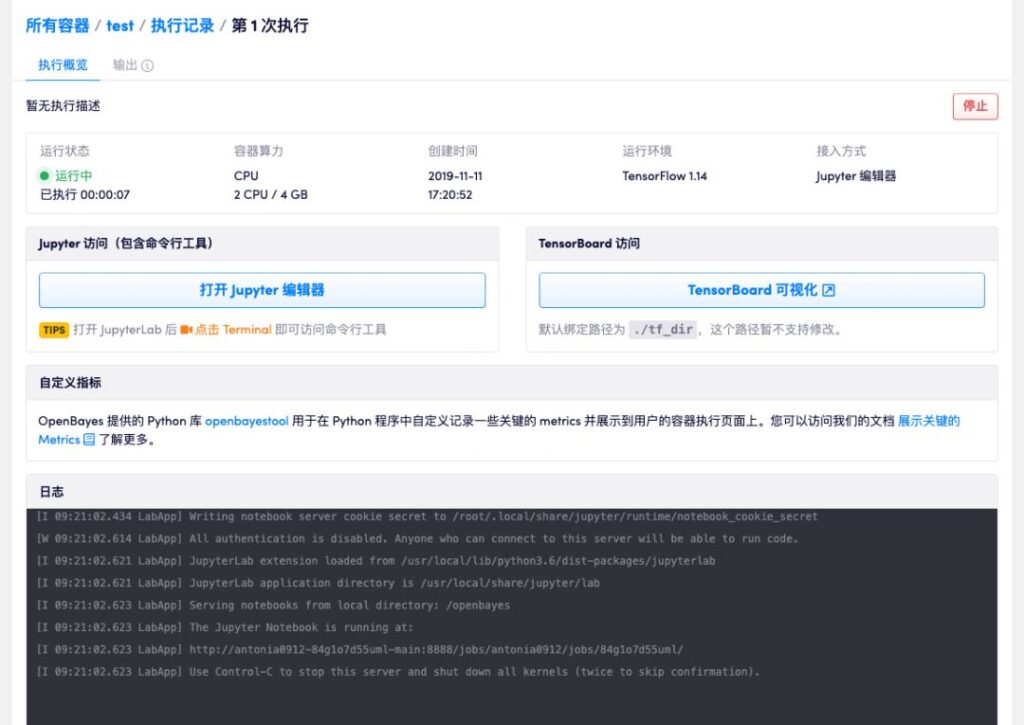
Processus d'exécution clair et concisTutoriel complet : https://openbayes.com/docs/quickstart/
Inscrivez-vous en tant que nouvel utilisateur pour profiter de la puissance de calcul du GPU
Visitez openbayes.com, cliquez sur le site officiel pour vous inscrire immédiatement, et il y aura des cadeaux hebdomadaires pendant la période de test interne, vous n'aurez donc pas à rivaliser avec vos camarades de classe et vos collègues pour la puissance de calcul~
Description de l'événement Visitez openbayes.com Inscrivez-vous en tant que nouvel utilisateur avec le code d'invitation [HyperAI]Vous pouvez profiter
Quota CPU gratuit :300 minutes/semaine
Quota vGPU gratuit :180 minutes/semaine
Acquisition de l'ensemble de données PDB complet :
https://www.rcsb.org/#Category-download
Les fichiers du jeu de données PDB peuvent être visualisés directement avec un éditeur de texte, mais il est préférable d'utiliser un outil de visualisation. Le programme de visualisation officiellement recommandé est Swiss PDB Viewer :
https://spdbv.vital-it.ch/disclaim.html#
Autres références :
https://www.novopro.cn/articles/201912021193.html
-- sur--








