Command Palette
Search for a command to run...
One-Stop-Protein-Zero-Shot-Mutationsvorhersage/Funktionsvorhersage, Protein-Engineering-Workbench VenusFactory Ermöglicht Full-Stack-Entwicklung
Die deutliche Verbesserung der Designeffizienz durch KI verändert das traditionelle Forschungsparadigma im Proteindesign. Im Vergleich zu herkömmlichen Proteindesign-Experimenten können große KI-Modelle nicht nur Proteinsequenzen, -strukturen und -funktionen in einem Bruchteil der Zeit vorhersagen und screenen, sondern auch neuartige, in der Natur nicht vorkommende Proteine auf der Grundlage physikalischer und chemischer Prinzipien und Datenmuster entwerfen. Durch Multi-Task-Learning und Deep-Learning-Modelle können sie sogar gleichzeitig Proteineigenschaften wie Stabilität, Bindungsaffinität und Kinetik vorhersagen.
Jedoch,Der komplexe Rechenrahmen des Modells und die riesige Proteindatenbank haben die Schwelle für den Einsatz von KI-Tools höher gelegt.Einerseits erfordert die Abhängigkeit des Proteindesigns von biologischen Daten, dass Forscher Daten aus mehreren Datenbanken abrufen, herunterladen, zusammenstellen und konvertieren müssen, was einen erheblichen Zeitaufwand bedeutet. Andererseits können Protein-KI-Modelle derzeit nur einzelne Aufgaben in Nischenbereichen lösen und es fehlt ein Bewertungssystem mit maßgeblichen Benchmark-Daten.
Darüber hinaus führte Dr. Tan Yang von der Forschungsgruppe von Professor Hong Liang an der Shanghai Jiao Tong University in Bezug auf die Herausforderungen des KI-Proteindesigns auch aus, dass die vorhandenen KI-Modelle im Bereich des Proteindesigns nicht nur Schwierigkeiten bei der Datenerfassung und Vereinheitlichung des Formats haben, sondern auch Schwierigkeiten bei der Anpassung von Parametern und eine langsame Trainingsgeschwindigkeit aufweisen.Die durch „Datenbarrieren, Modellbarrieren und Anwendungsbarrieren“ verursachten Hindernisse haben die Popularisierung und Anwendung von KI-Tools in einer breiteren wissenschaftlichen Forschungsgemeinschaft behindert.
Gleichzeitig sind Webserver im Vergleich zu bestehenden Lösungen zwar einfach und benutzerfreundlich, verfügen jedoch über eingeschränkte Funktionen, können nicht anhand individueller Daten trainiert werden und verfügen über eine begrenzte Intelligenz. Agenten reduzieren menschliche Eingriffe, können sich direkt auf die Ergebnisziele konzentrieren und eine oder mehrere Arbeitseinheiten autonom abschließen.
Um die breite Anwendung künstlicher Intelligenz im Bereich des Protein-Engineerings zu fördern,Die Forschungsgruppe von Professor Hong Liang an der Shanghai Jiao Tong University hat mit VenusFactory eine Open-Source-Workbench für die Proteinentwicklung aus einer Hand entwickelt.Die Plattform integriert die Abfrage biologischer Daten, standardisiertes Aufgaben-Benchmarking und vortrainierte Proteinsprachenmodelle (PLMs). Die Plattform kombiniert die Doppelfunktion eines Webservers und eines Agenten:
* Implementierung der 0-Code-Anpassung von KI-Modellen mithilfe privater Datensätze, Unterstützung der Befehlszeilenausführung und einer Gradio-basierten codelosen Schnittstelle.
* Bietet Open-Source-Downloads von über 30 großen Benchmark-Datensätzen zur Modellbewertung, integriert mehr als 40 proteinbezogene Datensätze und mehr als 40 beliebte PLMs und stellt einfache Links zu Proteindaten her.
* Es kann eine Nullstichproben-Mutationsvorhersage erreichen, KI-Modelle automatisch kombinieren, um Mutationen basierend auf der Nachfrage zu empfehlen, und überwachte Vorhersagemodule integrieren, um Eigenschaften durch KI-Modelle zur Zielintegration vorherzusagen.
derzeit,Die VenusFactory-Plattform für das Protein-Engineering-Design ist jetzt im Tutorial-Bereich der offiziellen Website von HyperAI (hyper.ai) verfügbar. Das Tutorial zur VenusFactory-Plattform umfasst 7 Funktionsmodule und Sie können es online mit der Ein-Klick-Bereitstellung erleben:
* Training: Zero-Code-Modelltraining, unterstützt über 40 große Modelle und verwendet private Datensätze, um Ihre eigenen Modelle zu trainieren.
* Bewertung: Ein benutzerfreundliches Tool zur umfassenden Leistungsbewertung von Proteinmodellen.
* Vorhersage: Verwenden Sie das trainierte Modell, um die Funktion neuer Proteinsequenzen vorherzusagen.
* Quick Tools: Benutzerfreundliche Version, die die Vorhersage von Nullstichprobenmutationen (gerichtete Evolution) und die überwachte Vorhersage (Funktions- oder Eigenschaftsvorhersage) unterstützt.
* Erweiterte Tools: Erweiterte angepasste Version, die die Vorhersage von Nullstichprobenmutationen (gerichtete Evolution) und die überwachte Vorhersage (Funktions- oder Eigenschaftsvorhersage) unterstützt.
* Download: Einfache Verknüpfung mit Proteindaten und Unterstützung des Multithread-Downloads wichtiger Mainstream-Datenbanken (RCSB, UniProt …).
* VenusAgent: Ein Protein-Engineering-Agent, der mit DeepSeek zusammenarbeitet, um die KI-Proteinberechnung zu ermöglichen.
Link zum Tutorial:
Darüber hinaus haben wir für neue Benutzer überraschende Vorteile in Bezug auf Rechenressourcen vorbereitet.Registrieren Sie sich mit dem Einladungscode „VenusFactory“, um 2 Stunden Dual-SIM-Nutzungszeit für A6000 zu erhalten (Gültigkeitsdauer der Ressource beträgt 1 Monat).Die Menge ist begrenzt, verpassen Sie es nicht!
Demolauf
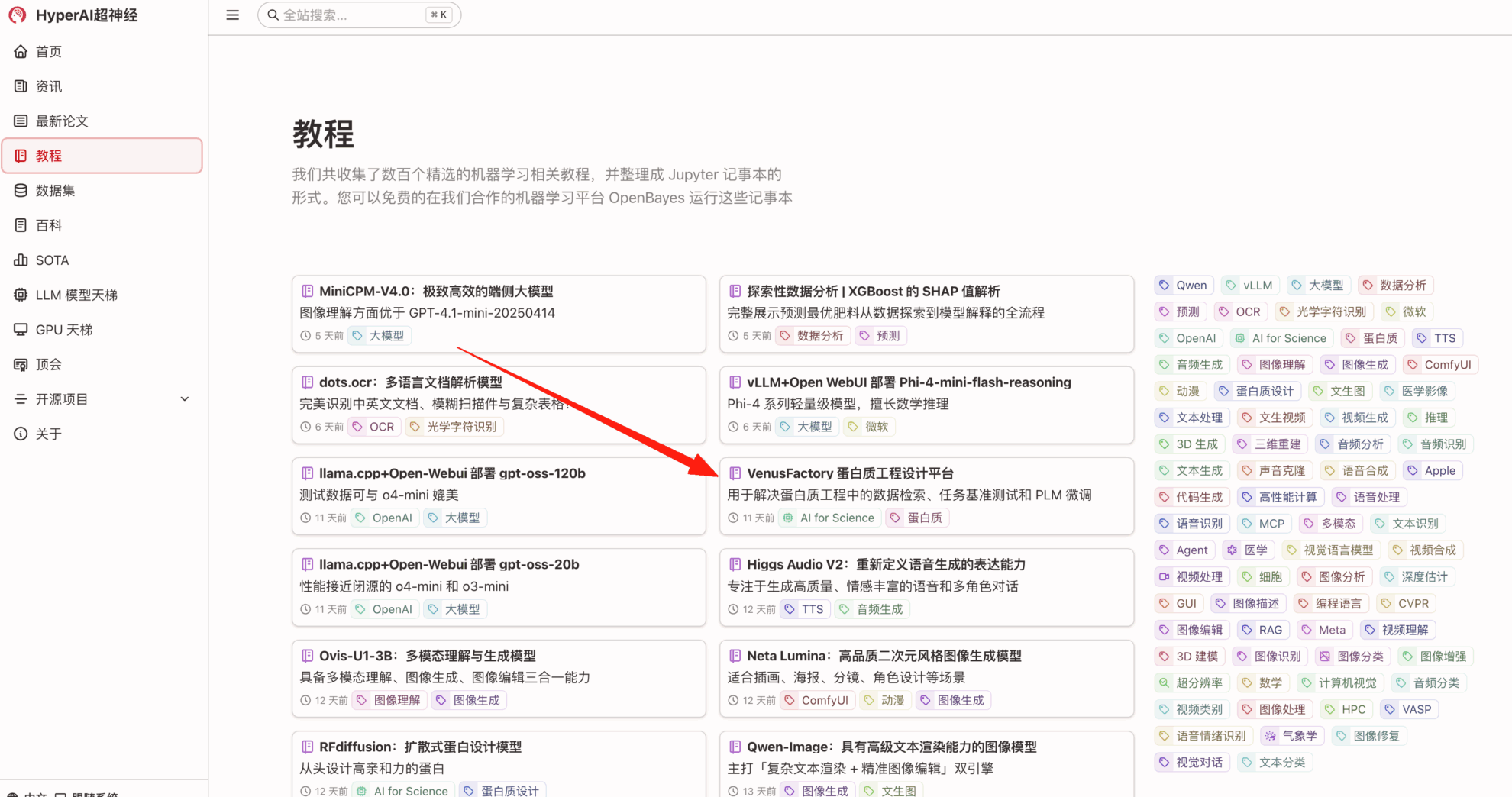
1. Geben Sie die URL hyper.ai in Ihren Browser ein. Klicken Sie auf der Startseite auf die Seite „Tutorials“, wählen Sie die VenusFactory Protein Engineering Platform aus und klicken Sie auf „Dieses Tutorial online ausführen“.
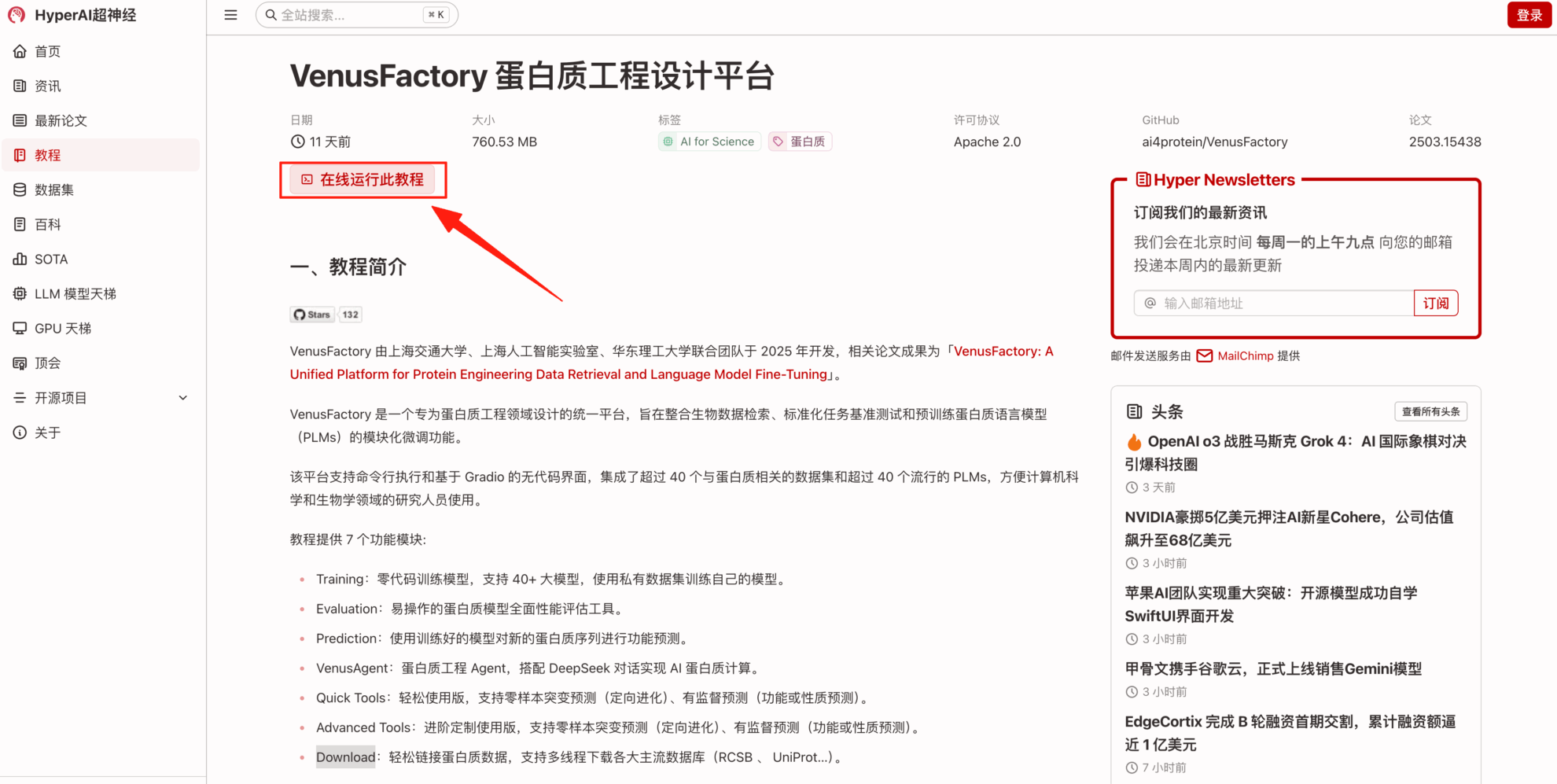
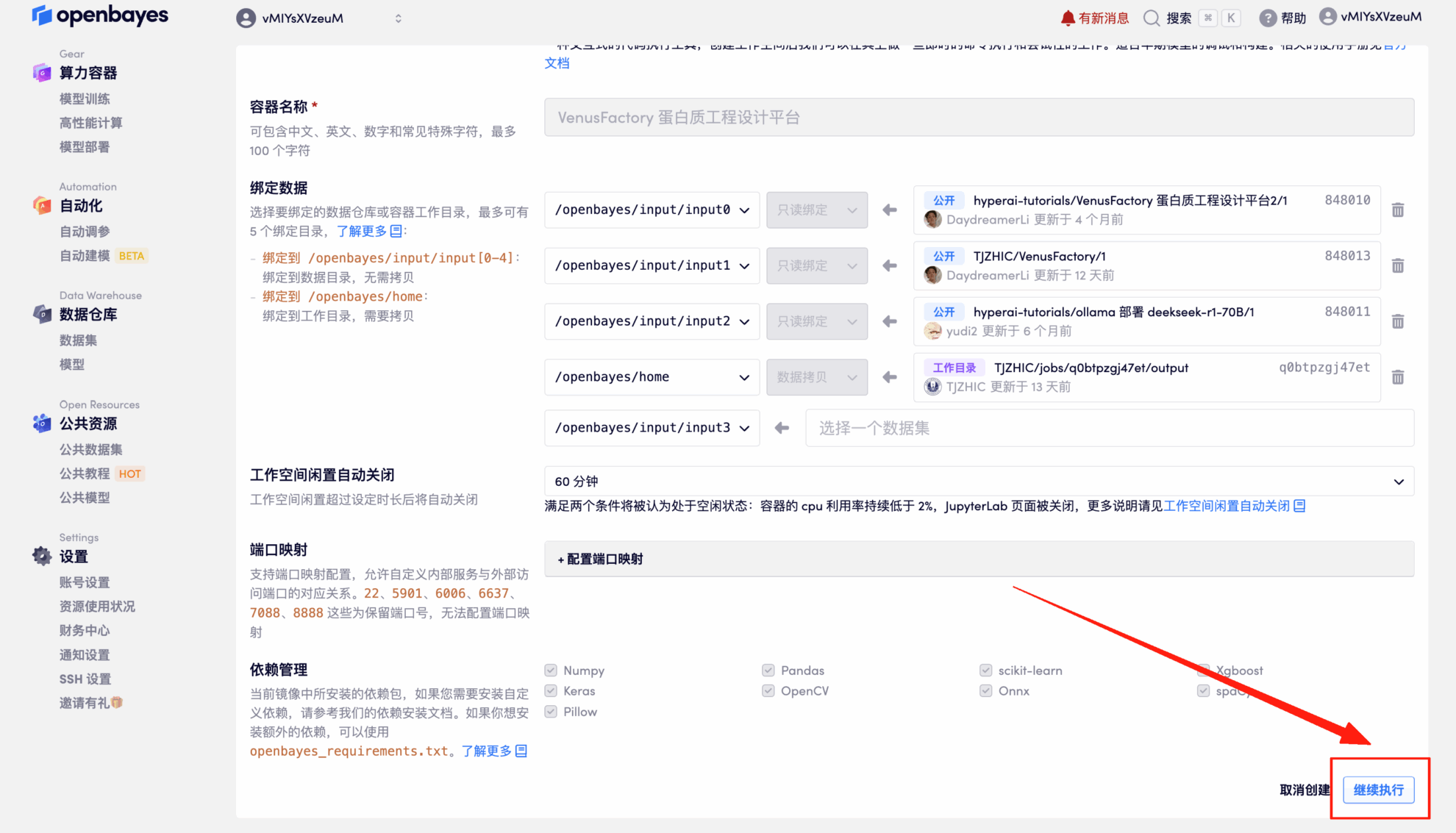
2. Klicken Sie nach dem Seitensprung oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
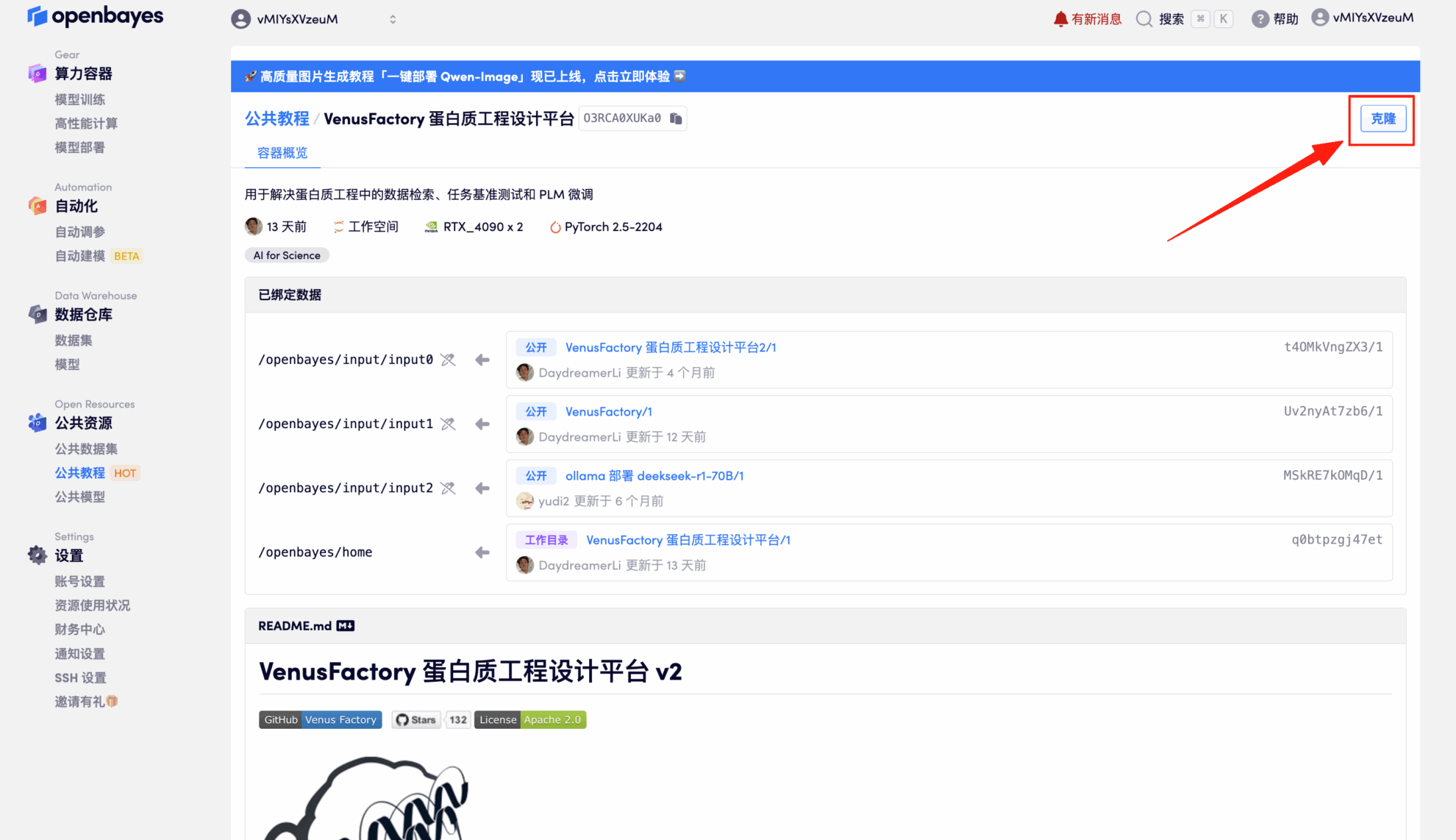
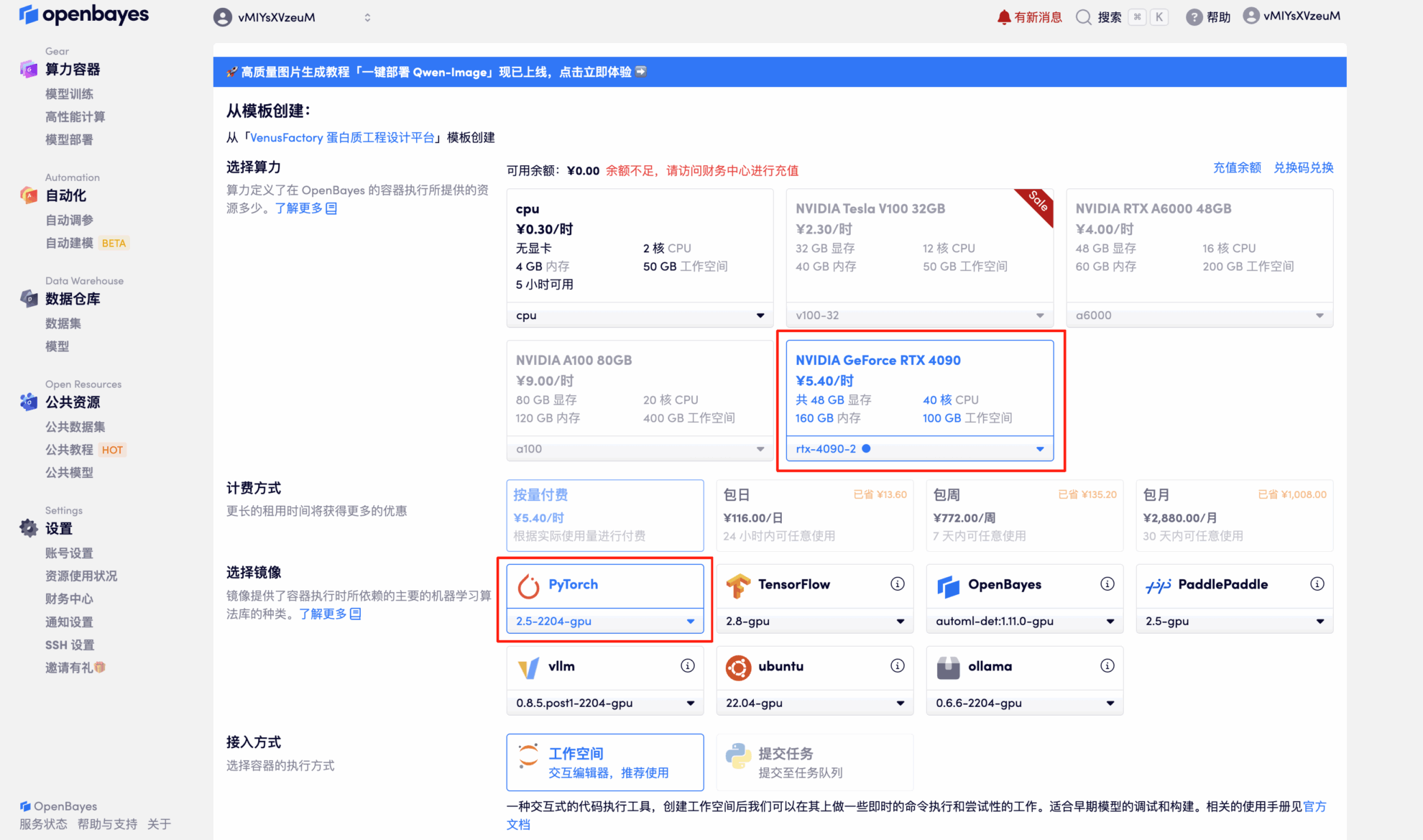
3. Wählen Sie die NVIDIA GeForce RTX 4090-2 und PyTorch-Images aus und klicken Sie auf „Weiter“. Die OpenBayes-Plattform bietet vier Abrechnungsoptionen: Pay-as-you-go oder Tages-/Wochen-/Monatstarife. Neue Benutzer können sich über den unten stehenden Einladungslink registrieren und erhalten 4 Stunden kostenlose RTX 4090 und 5 Stunden kostenlose CPU-Zeit!
Exklusiver Einladungslink von HyperAI (kopieren und im Browser öffnen):
https://openbayes.com/console/signup?r=Ada0322_NR0n
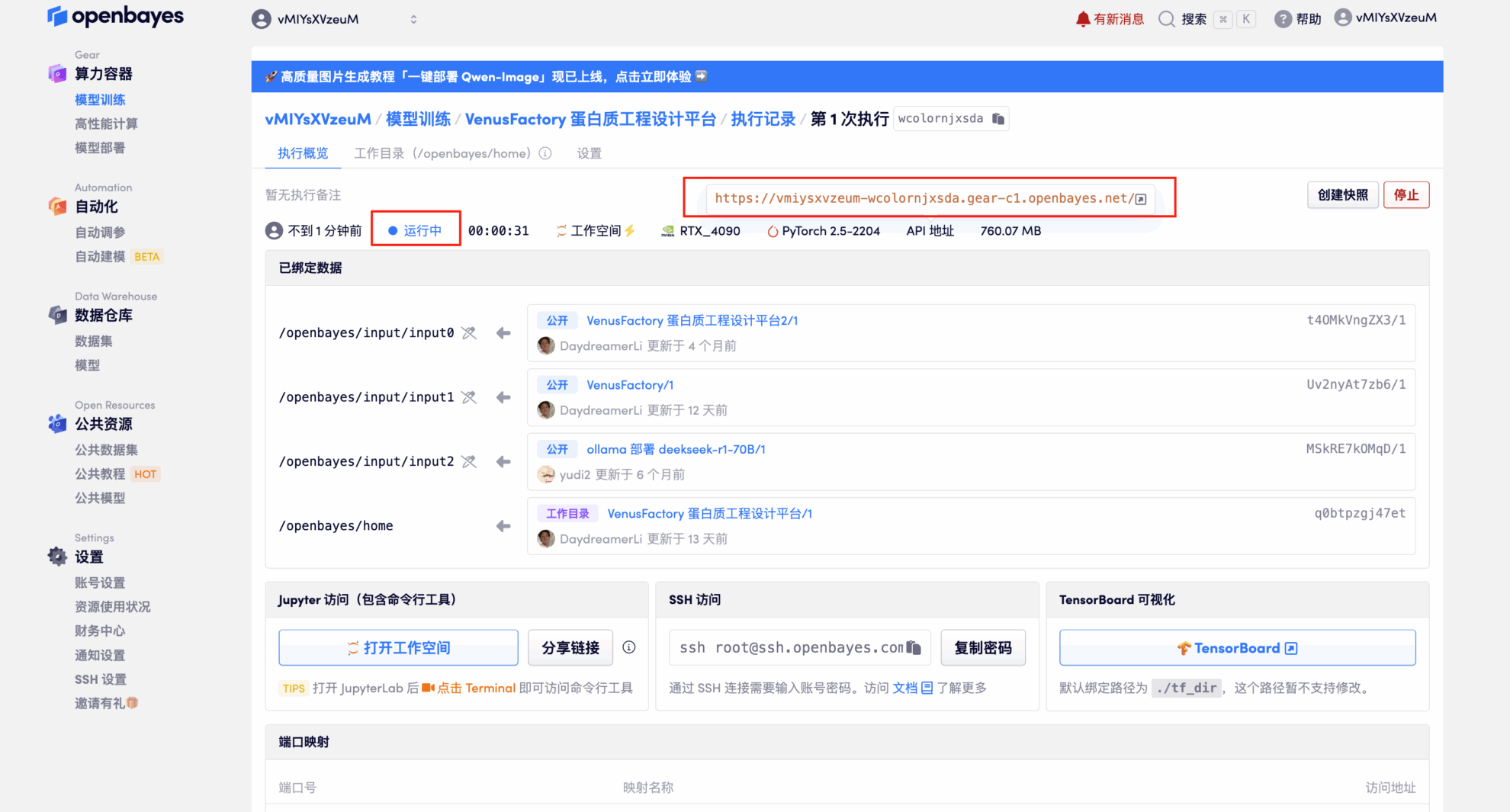
4. Warten Sie, bis die Ressourcen zugewiesen sind. Der erste Klonvorgang dauert etwa 2 Minuten. Wenn der Status auf „Läuft“ wechselt, klicken Sie auf den Pfeil neben „API-Adresse“, um zur Demoseite zu gelangen.Da das Modell groß ist, dauert es etwa 3 Minuten, bis die WebUI-Oberfläche angezeigt wird, andernfalls wird „Bad Gateway“ angezeigt.Bitte beachten Sie, dass Benutzer vor der Verwendung der API-Adresszugriffsfunktion eine Echtnamenauthentifizierung durchführen müssen.
Effektdemonstration
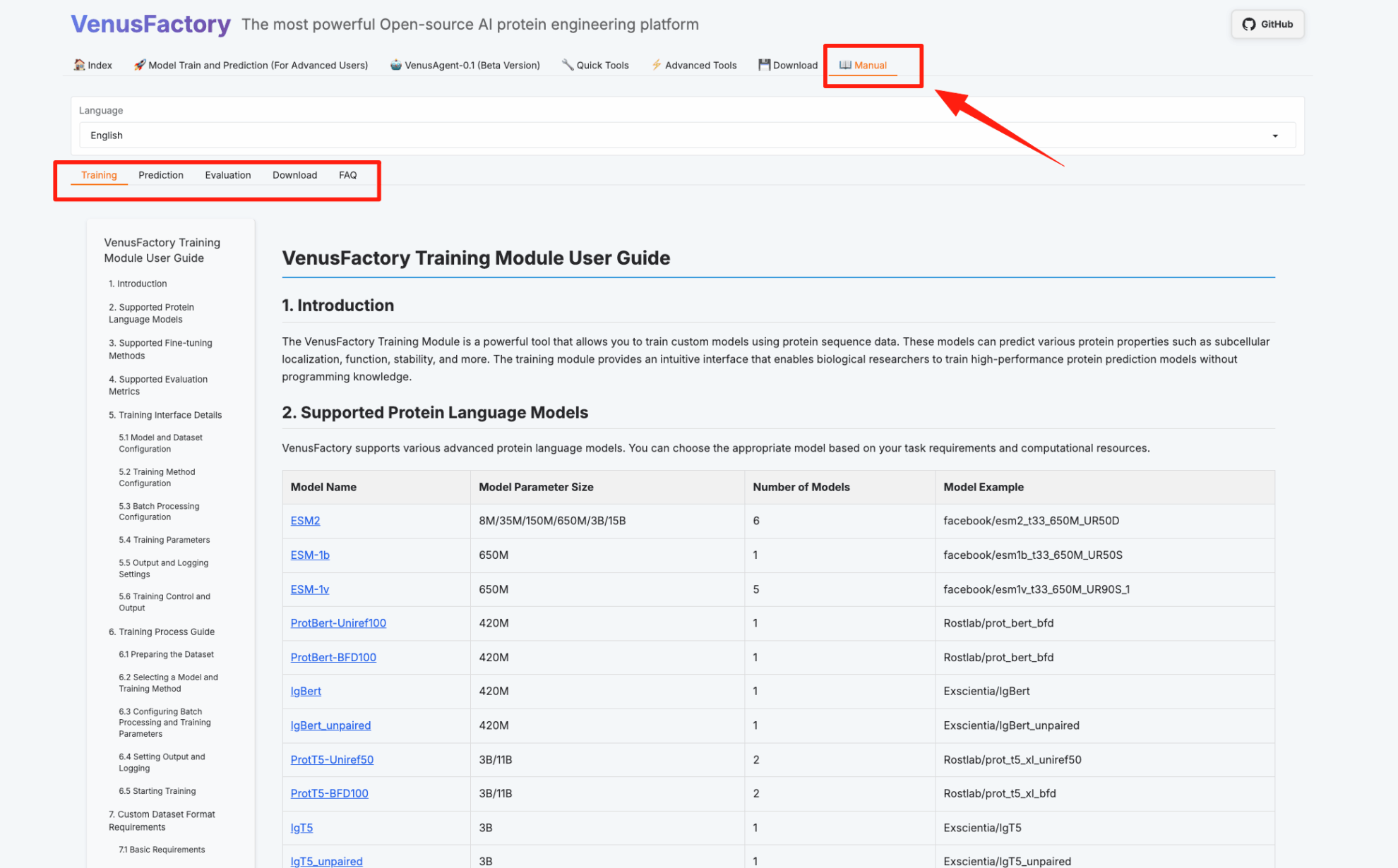
Nachfolgend finden Sie die VenusFactory-Nutzungsseite. Klicken Sie auf „Manuell“, um die Nutzungsanleitungen für das Trainingsmodul, das Vorhersagemodul, das Bewertungsmodul und das Downloadmodul direkt anzuzeigen:
Trainingsmodulanzeige
Klicken Sie im Modul „Model Train and Prediction Training“ auf das Modul „Training“:
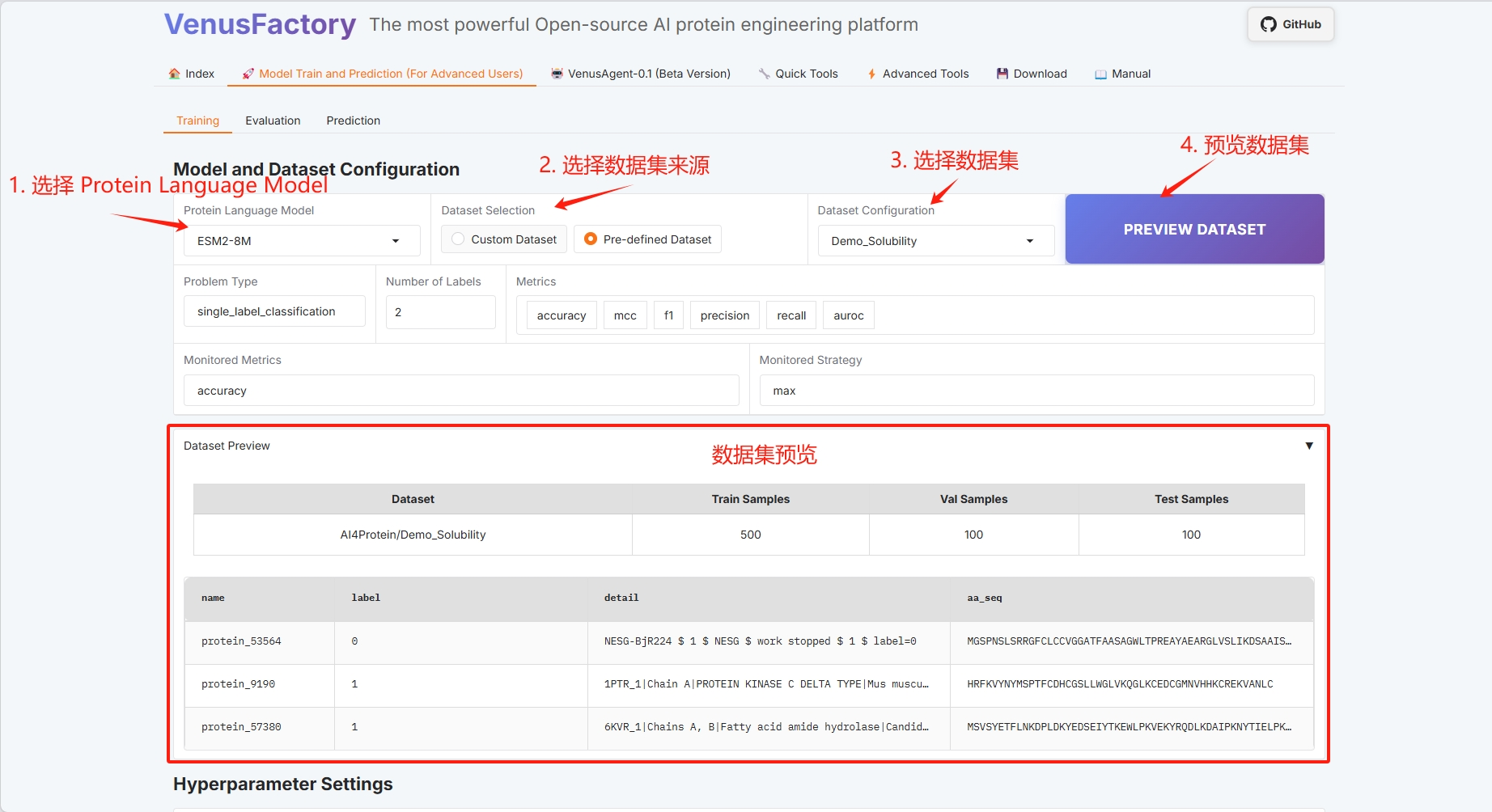
* Protein-Sprachmodell auswählen
* Datensatzauswahl
* Datensatzvorschau
* Konfiguration der Trainingsmethode (genaue Informationen finden Sie im Benutzerhandbuch)
* Batch-Konfiguration (Details siehe Benutzerhandbuch)
Legen Sie den Speicherpfad für das Trainingsmodell fest und klicken Sie auf „TRAINING STARTEN“, um mit dem Training zu beginnen.
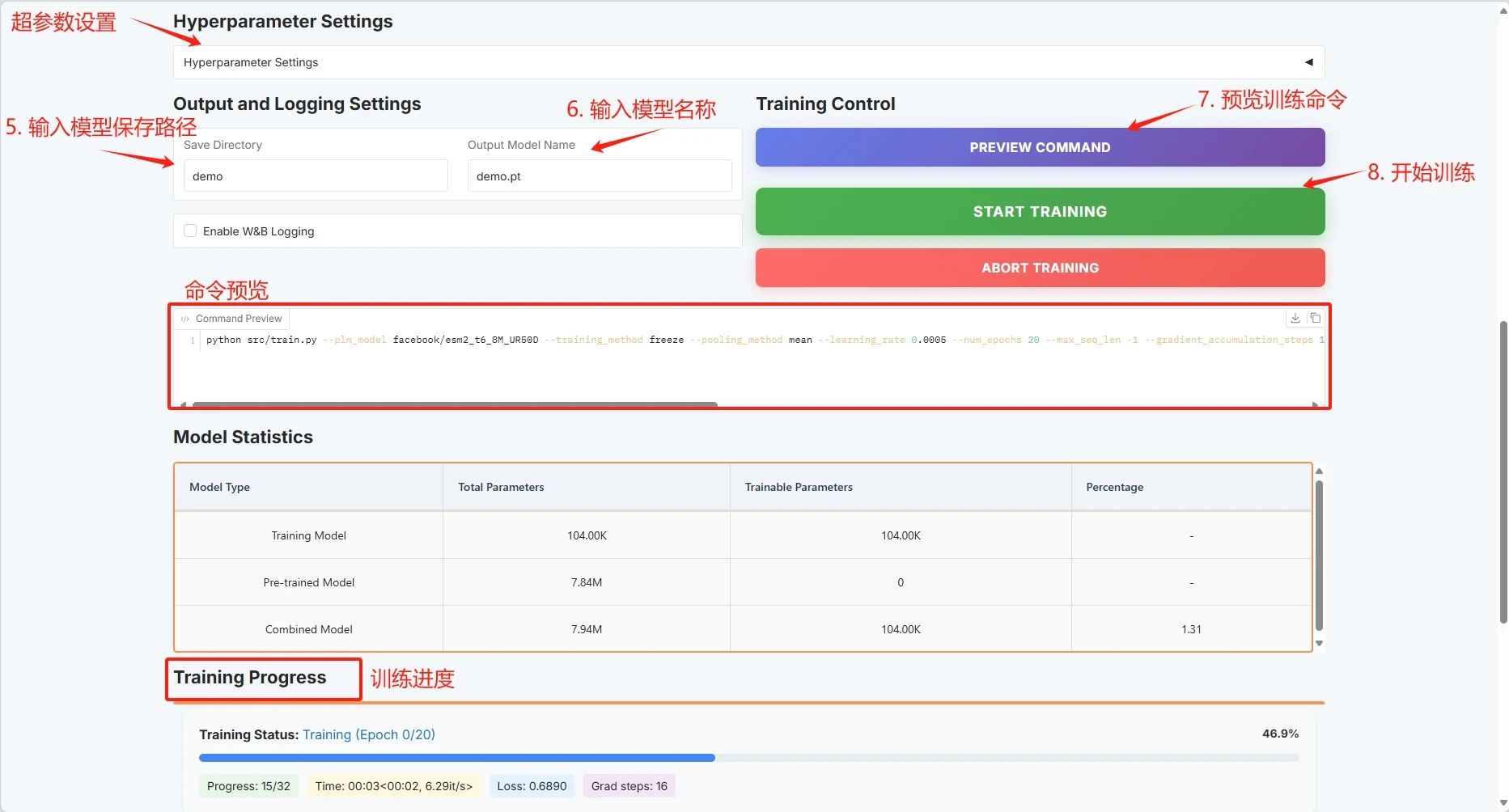
An dieser Stelle können Sie die Trainingsparameter und die Verlustkurve sehen:
Wenn Sie Ihren eigenen Datensatz verwenden möchten, können Sie die benutzerdefinierte Datensatzkonfiguration verwenden. Geben Sie einfach den Pfad Ihres Datensatzes ein (weitere Informationen finden Sie in der Handbuchdokumentation).
Anzeige des Auswertemoduls
Klicken Sie im Modul „Modelltraining und Vorhersagetraining“ auf das Modul „Auswertung“.
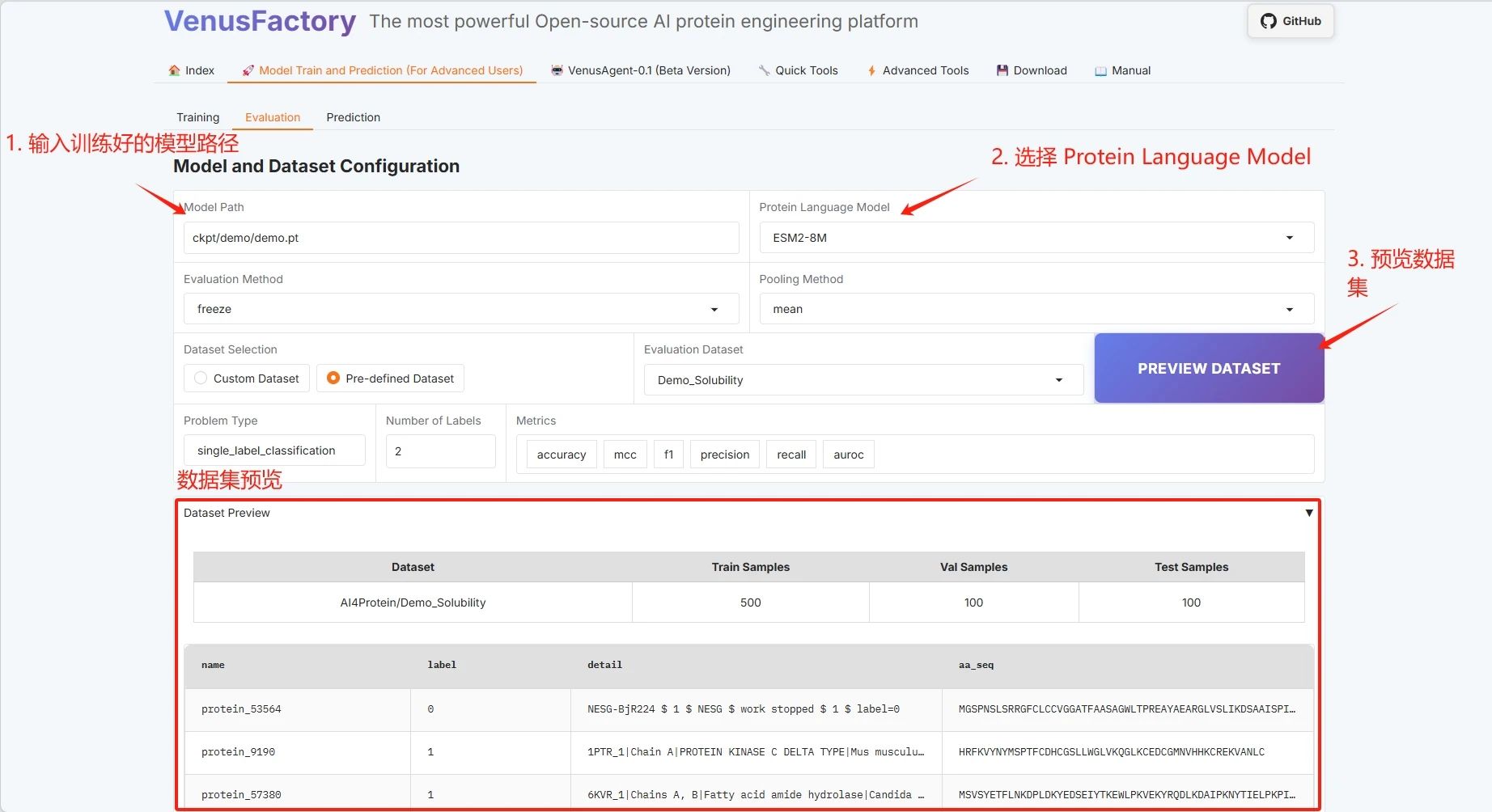
Batch-Konfiguration, klicken Sie auf „BEWERTUNG STARTEN“, um mit dem Training zu beginnen.
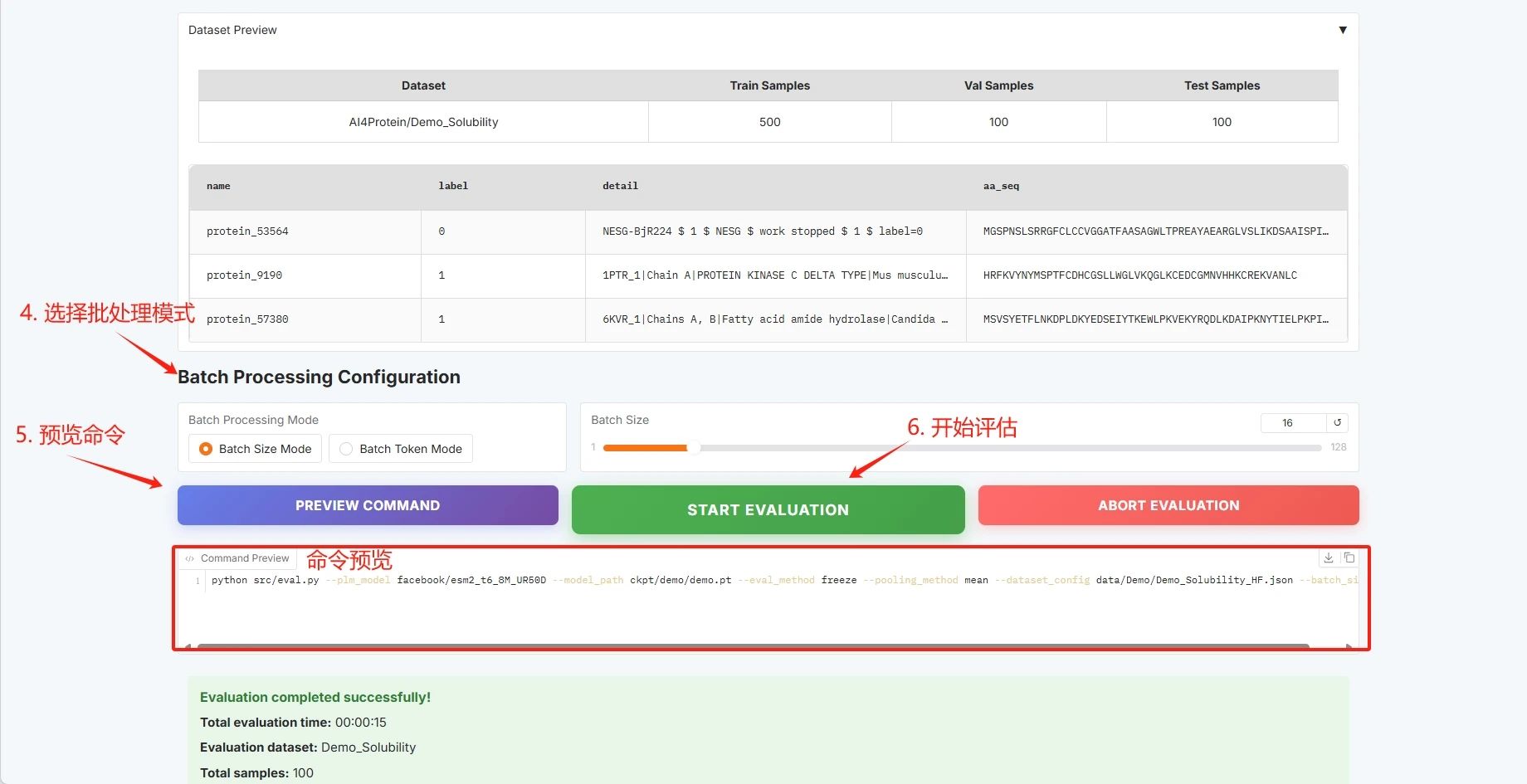
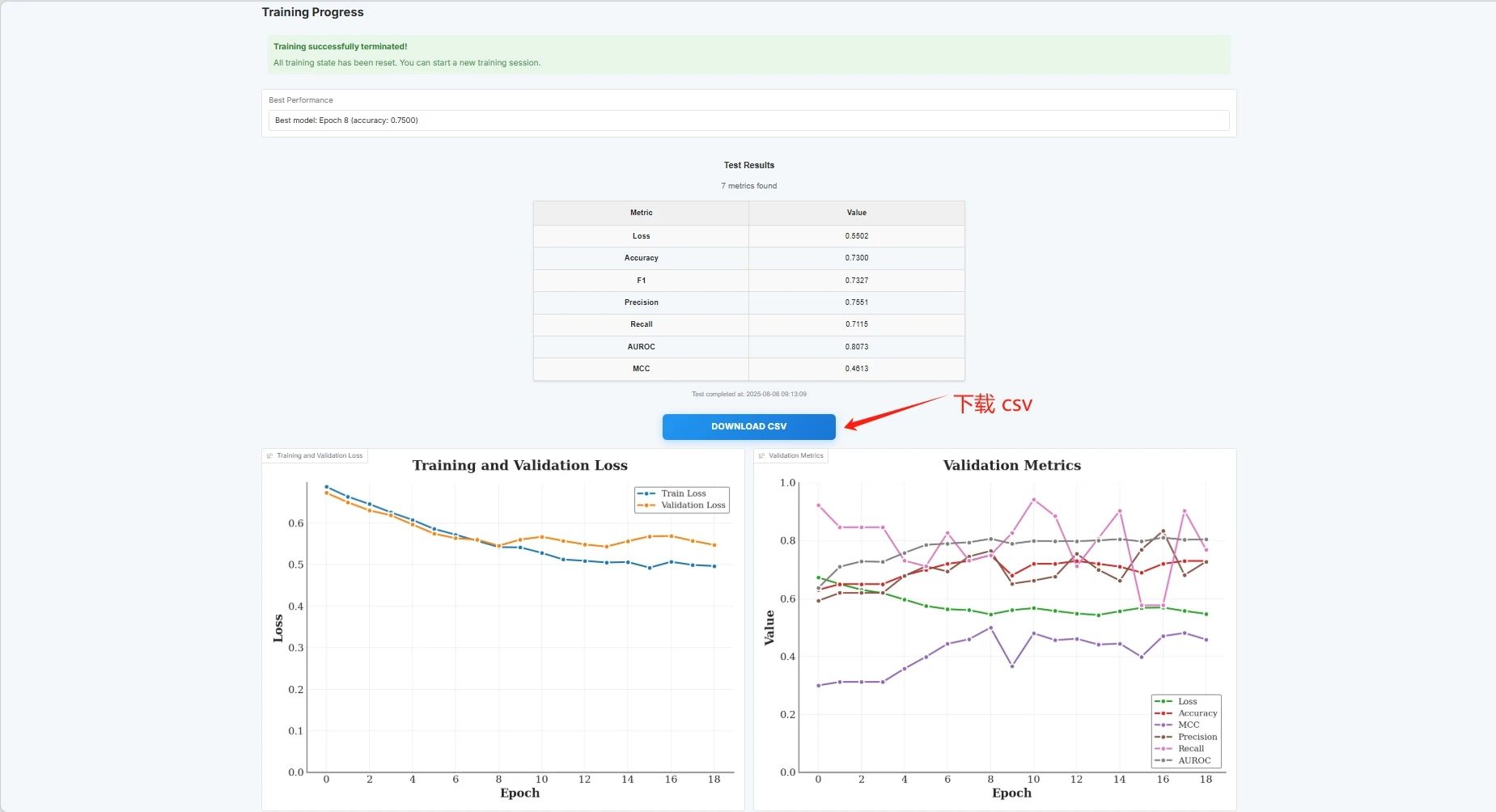
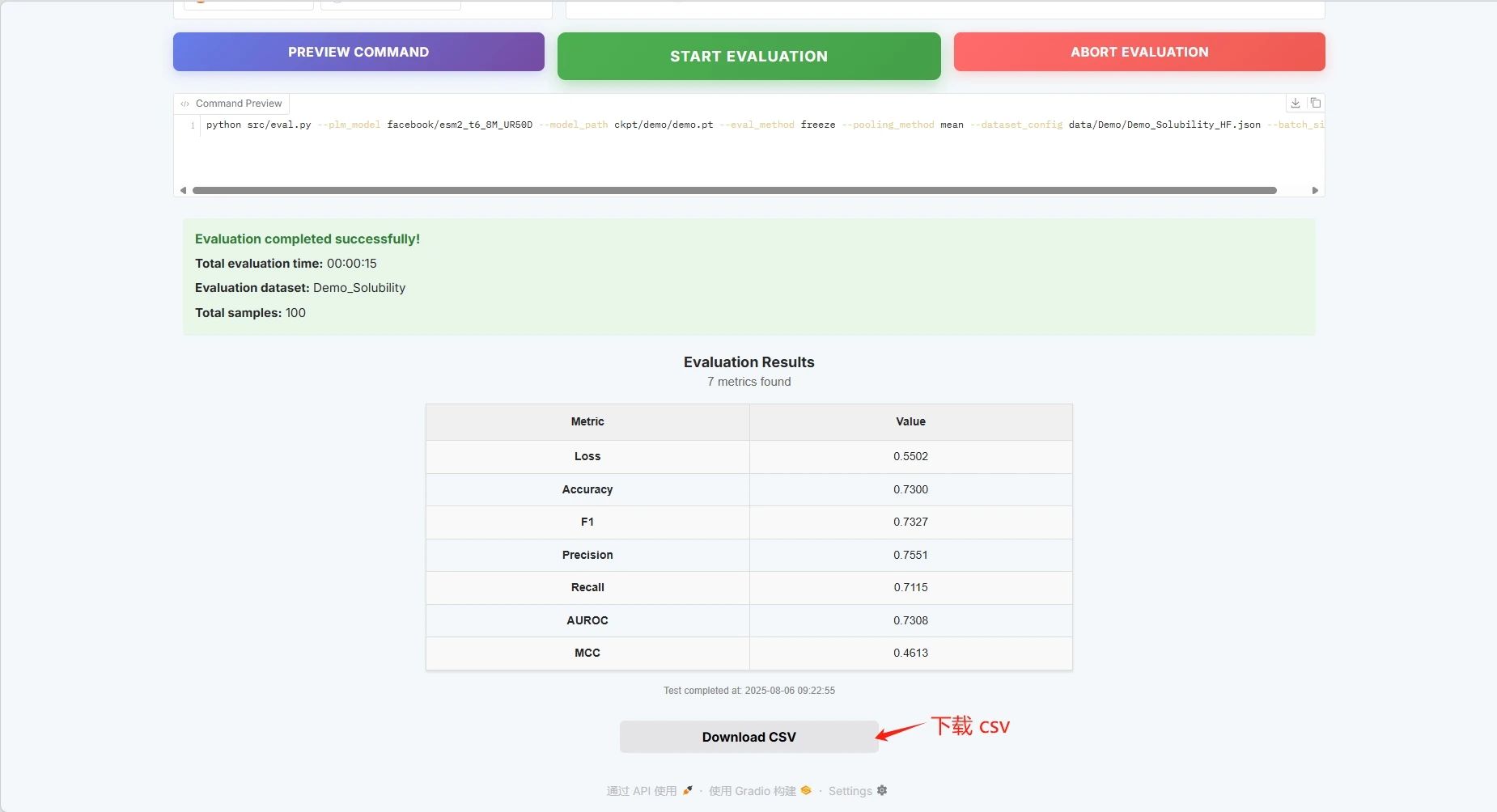
Die Auswertungsergebnisse sind wie folgt und Sie können die CSV-Datei herunterladen:
Wenn Sie Ihren eigenen Datensatz verwenden möchten, können Sie die benutzerdefinierte Datensatzkonfiguration verwenden. Geben Sie einfach den Pfad Ihres Datensatzes ein (weitere Informationen finden Sie in der Handbuchdokumentation).
Anzeige des Vorhersagemoduls
Klicken Sie im Modul „Modelltraining und Vorhersagetraining“ auf das Modul „Vorhersage“, legen Sie den Speicherpfad für das Trainingsmodell fest, wählen Sie das Proteinsprachenmodell aus und klicken Sie auf „VORHERSAGE STARTEN“, um mit dem Training zu beginnen.
Nehmen wir als Beispiel die Vorhersage einzelner Sequenzen:
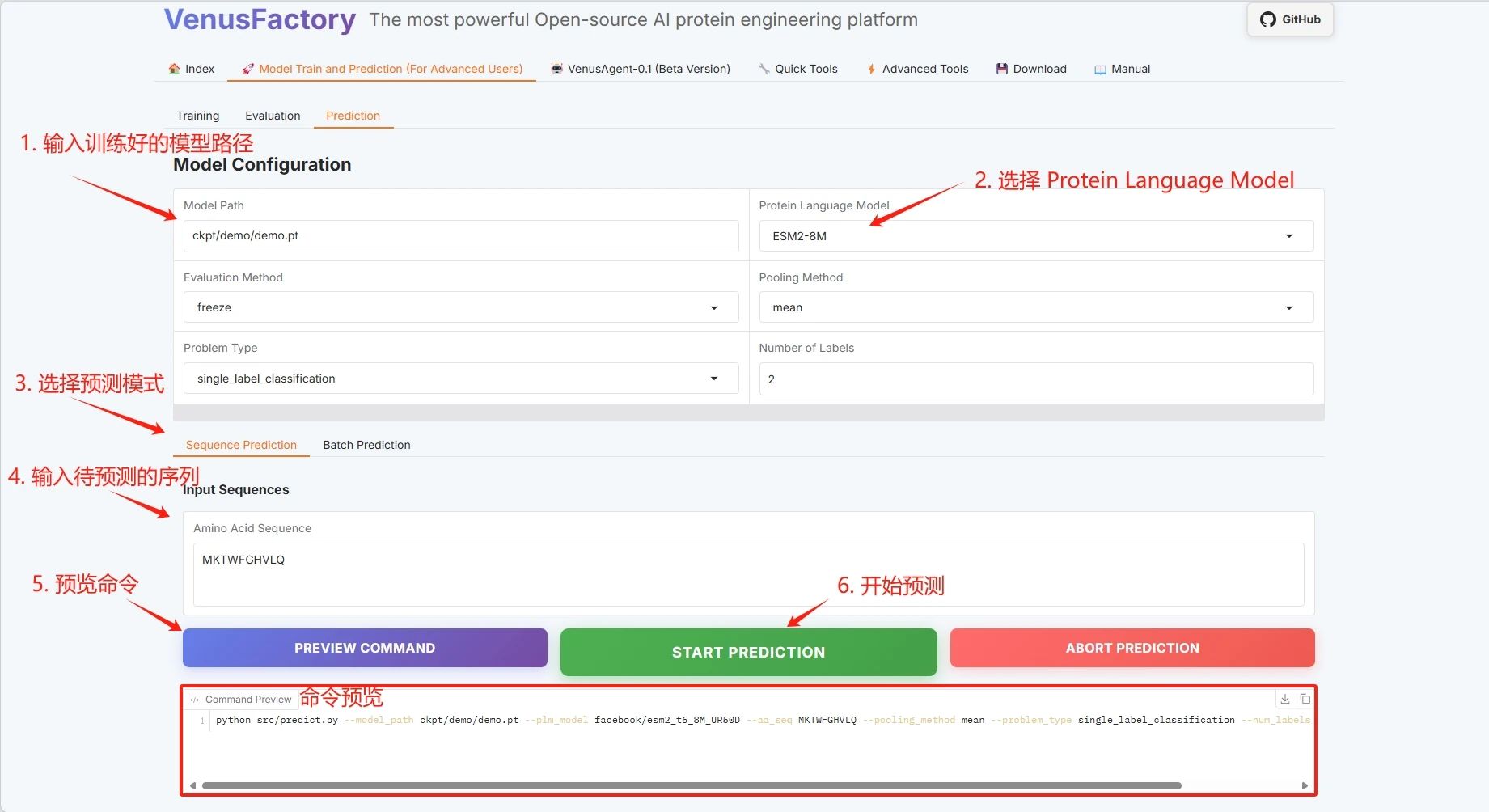
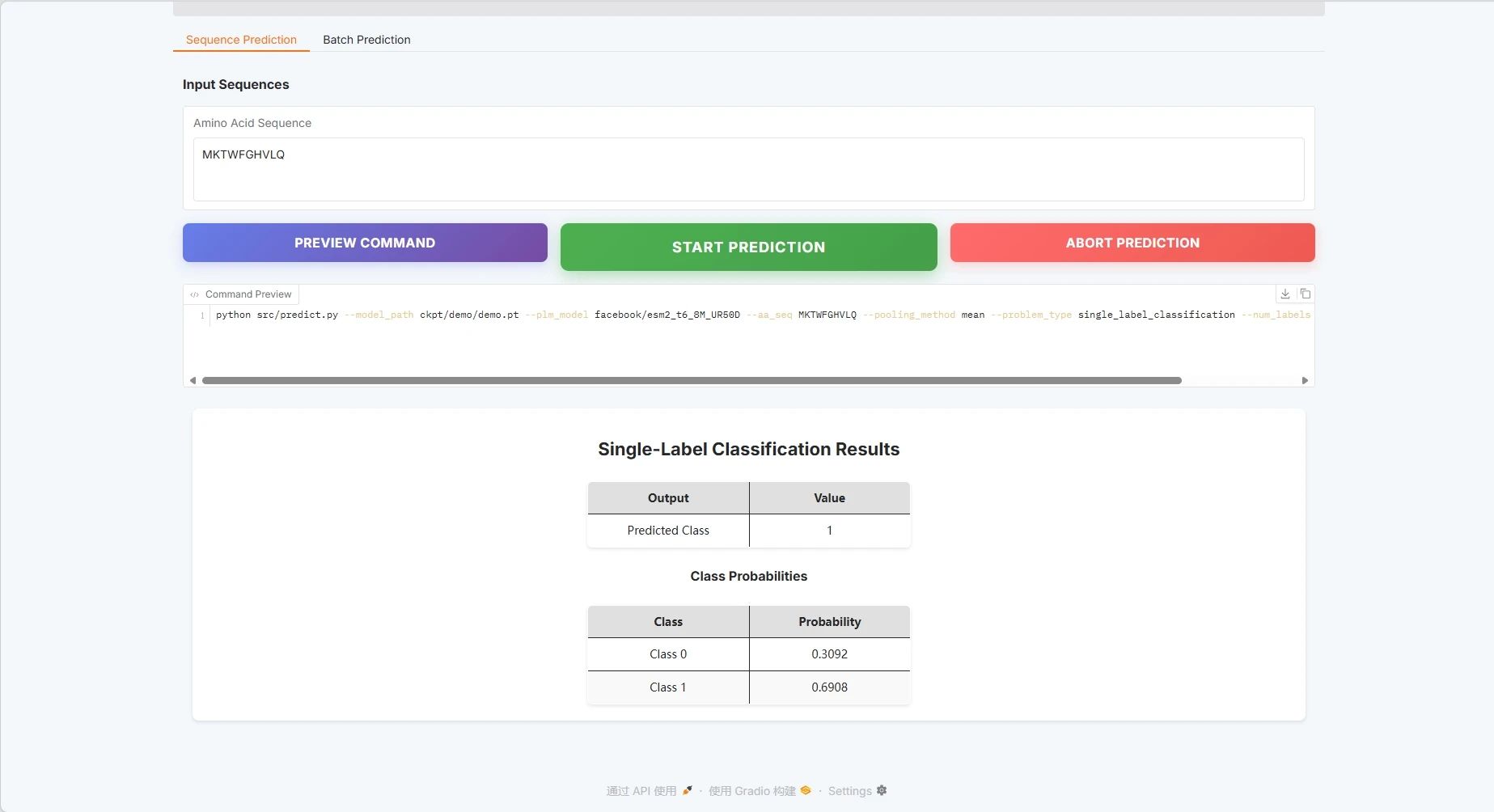
Beispiel einer Proteinsequenz: MKTWFGHVLQ
VenusAgent-Schaufenster
Klicken Sie auf das VenusAgent-Modul.
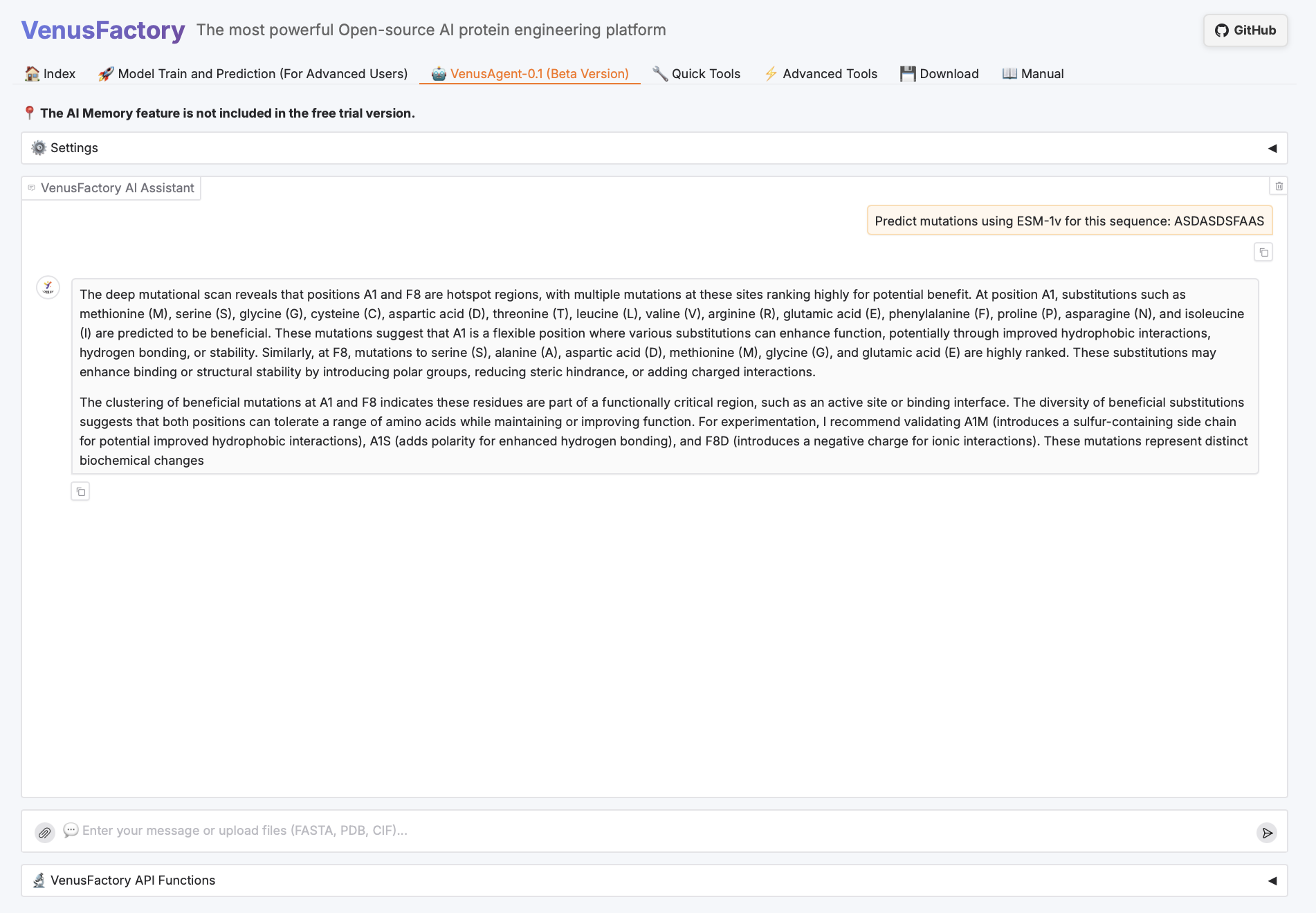
Da VenusAgent große DeepSeek-Modelle benötigt, bietet dieses Tutorial zwei Aufrufmethoden: die Eingabe des API-Schlüssels selbst oder die Verwendung des auf der Plattform bereitgestellten DeepSeek-R1-70B-Modells. Sie können je nach den benötigten Funktionen unterschiedliche Grafikkarten auswählen. Die Anweisungen zur Kartenauswahl lauten wie folgt:
* Bei Verwendung einer einzelnen RTX 4090-Grafikkarte unterstützt die VenusAgent-Funktion nicht die Verwendung lokal bereitgestellter großer Modelldienste (die Verwendung des DeepSeek-API-Schlüssels ist unbegrenzt).
* Wenn Sie zwei RTX 4090-Grafikkarten verwenden, können Sie nach der Verwendung der VenusAgent-Funktion nicht sofort (nach 1–2 Minuten) andere Funktionen verwenden (bei Verwendung des DeepSeek-API-Schlüssels gibt es keine Einschränkung).
* Bei Verwendung von zwei RTX A6000-Grafikkarten sind die VenusAgent-Funktionen unbegrenzt.
* Benutzer können den DeepSeek-API-Schlüssel eingeben. Andernfalls wird standardmäßig der im Tutorial lokal bereitgestellte Dienst für große Modelle verwendet. Bei Verwendung des lokalen Dienstes für große Modelle beträgt die Antwortzeit für die erste Konversation etwa 2–3 Minuten. Bitte haben Sie Geduld.
Oben finden Sie ein ausführliches Tutorial zur Verwendung der „VenusFactory Protein Engineering Design Platform“. Jeder ist herzlich eingeladen, vorbeizukommen und es zu erleben!
Link zum Tutorial:
Erhalten Sie mit einem Klick hochwertige Papiere und ausführliche Interpretationsartikel im Bereich AI4S von 2023 bis 2024 ⬇️









