Command Palette
Search for a command to run...
Greifen Sie Die Drei Wichtigsten Soliden Tumoren Direkt An! Team Der Shanghai Jiao Tong University Veröffentlicht Deep-Learning-System Zur Verbesserung Der Genauigkeit Der Krebsüberlebensvorhersage

Ein Bericht der Weltgesundheitsorganisation aus dem Jahr 2022 wies darauf hin, dassNichtübertragbare Krankheiten (NCDs) wie Krebs haben Infektionskrankheiten überholt und sind zur Todesursache Nummer eins weltweit geworden.Die neuesten vom Nationalen Krebszentrum Chinas veröffentlichten Daten zeigen, dass es im Jahr 2022 in China etwa 4,8247 Millionen neue Krebsfälle und 2,5742 Millionen neue Krebstodesfälle geben wird.
Lange Zeit hatten die Menschen Angst vor Krebs. Tatsächlich sind jedoch ein Drittel der Krebserkrankungen chronischer Natur und können durch Früherkennung, Frühdiagnose und Frühbehandlung verhindert werden. Ein Drittel der Krebserkrankungen ist unheilbar, kann aber durch die richtige Behandlung kontrolliert werden, um eine bessere Lebensqualität zu erreichen und das Überleben zu verlängern. Zur Prävention gehört vor allem, auf die persönliche Gesundheit zu achten, indem man sein Immunsystem stärkt, sich regelmäßig körperlich untersucht usw. Nach der Diagnose einer Krebserkrankung ist eine Prognoseanalyse sehr wichtig.
Unter Krebsprognose versteht man die Vorhersage des möglichen Verlaufs und Ausgangs bei Krebspatienten. Prognoseanalysen tragen dazu bei, die Überlebenschancen von Krebspatienten zu verbessern.In der Vergangenheit haben Forscher das Tumormikroumfeld (TME) aus der Perspektive der räumlichen Genexpression auf der Grundlage der räumlichen Transkriptomik (ST)-Technologie charakterisiert, um verschiedene prognostische Untergruppen von Krebspatienten zu unterscheiden. Die hohen Kosten und die lange Versuchsdauer von ST verhindern jedoch dessen Anwendung in großen Kohorten von Krebspatienten zur Überlebensvorhersage. Im Vergleich dazu sind histologische Bilder kostengünstig, im klinischen Umfeld leicht zugänglich und können umfassende Informationen zur Tumormorphologie liefern. Damit stellen sie eine bessere Alternative zur TME-Analyse auf molekularer Ebene dar und ermöglichen eine genauere Krebsprognose.
Kürzlich veröffentlichten die Forschungsgruppe von Yu Zhangsheng (Fakultät für Biowissenschaften und Technologie/Zentrum für klinische Forschung der Medizinischen Fakultät), die Forschungsgruppe von Wang Yuguang (Fakultät für Naturwissenschaften/Fakultät für Mathematische Wissenschaften) des Shanghai National Center for Applied Mathematics (Zweigstelle der Shanghai Jiao Tong University) und ihre Mitarbeiter in Cell Reports Medicine einen Artikel mit dem Titel „Nutzung von in histologischen Bildern dargestelltem TME zur Verbesserung der Krebsprognose durch ein Deep-Learning-System“.Im Rahmen dieser Studie wurde ein Deep-Learning-System entwickelt, das Informationen zur Tumormikroumgebung von Krebspatienten ohne räumliche Transkriptomdaten anhand histopathologischer Bilder vorhersagen und so eine genaue Krebsprognose ermöglichen kann.
Forschungshighlights:
- Vorhersage von TME-Informationen aus histopathologischen Bildern für Krebspatienten ohne ST-Daten
- Durch IGI-DL charakterisiertes TME verbessert die Genauigkeit der Krebsüberlebensvorhersage
- Erweitert die Nutzung von Informationen zur räumlichen Genexpression in großen öffentlichen Datenbanken mit Bildern aus der biomedizinischen Pathologie erheblich

Papieradresse:
https://www.cell.com/cell-reports-medicine/fulltext/S2666-3791(24)00205-2
Folgen Sie dem offiziellen Account und antworten Sie mit „Tumormikroumgebung“, um das vollständige PDF zu erhalten
Datensatz: Auswertung von Gewebeproben von 3 soliden Tumorarten
In dieser Studie wurden drei verschiedene Datensätze verwendet, um die Leistung des Modells anhand von Gewebeproben von drei verschiedenen soliden Tumorarten zu bewerten: Dickdarmkrebs (CRC), Brustkrebs und kutanes Plattenepithelkarzinom (cSCC).
Bei DickdarmkrebsDie Forscher verwendeten 41.492 Punkte aus 10 ST-Datensätzen von 10 CRC-Patienten des Ruijin-Krankenhauses der Shanghai Jiao Tong University School of Medicine, die mit 10× Visium als Leave-One-Patient-Out-Validierungssatz sequenziert wurden, wie in der folgenden Tabelle gezeigt.

Bei BrustkrebsDie Forscher verwendeten 34.678 Punkte aus 92 Gewebeproben von 27 Patienten, die mithilfe der herkömmlichen ST-Technologie sequenziert wurden, als Validierungssatz für einen Patienten, wie in der folgenden Tabelle dargestellt.

Bei Plattenepithelkarzinomen der HautDie Forscher verwendeten 4.353 Punkte aus 12 Gewebeproben von vier Patienten, die mithilfe der herkömmlichen ST-Technologie sequenziert wurden, als Validierungssatz für einen Patienten, wie in der folgenden Tabelle dargestellt.

Modellarchitektur: Neues Deep-Learning-System verbessert Krebsprognose
In dieser Studie entwickelten Forscher ein Deep-Learning-System, das die Krebsprognose mithilfe des in histologischen Bildern dargestellten TME verbessern kann.
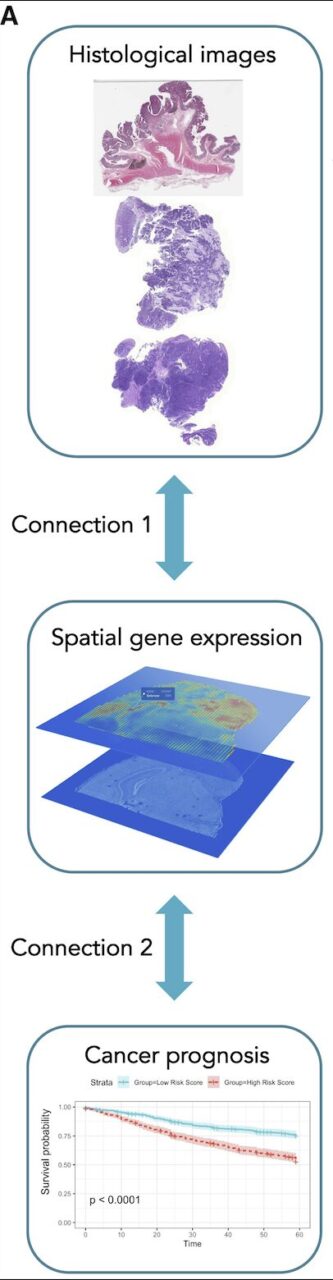
Das System besteht aus zwei Teilen:
Der erste Teil (Verbindung 1 in der Abbildung oben) ist ein Modell, das auf integriertem Graph- und Bild-Deep-Learning (IGI-DL) basiert und Convolutional Neural Networks und Graph Neural Networks verwendet, um H&E-gefärbte histologische Bilder in den Genexpressionsbereich zu projizieren.
Im zweiten Teil (Verbindung 2 in der Abbildung oben) verwendeten die Forscher den Super-Patch-Graph und die von IGI-DL vorhergesagte räumliche Genexpression als Knotenmerkmale in der Kohorte für Dickdarmkrebs und der Kohorte für Brustkrebs im Datensatz des Cancer Genome Atlas (TCGA), um die Prognose vorherzusagen, und überprüften diese dann im externen Testsatz MCO-CRC (Molecular and Cellular Oncology Colorectal Cancer).
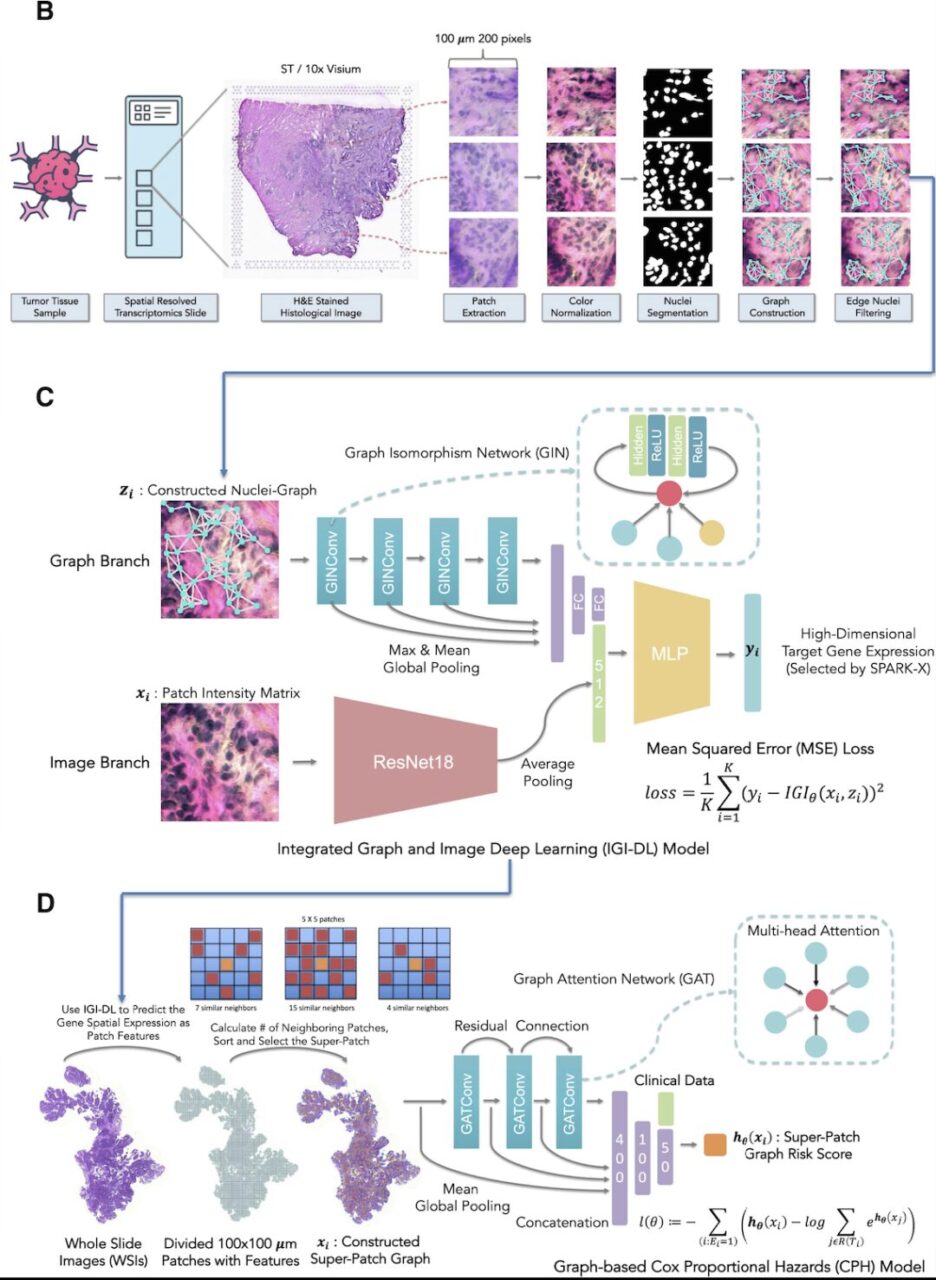
Im Einzelnen umfasst der Aufbau des Systems drei Schritte: Vorverarbeitung von H&E-gefärbten histologischen Bildern, Vorhersagemodell für räumliche Genexpression und Überlebensmodell für räumliche Genexpression im Super-Patch-Graph basierend auf der Vorhersage.
- Vorverarbeitung H&E-gefärbter histologischer Bilder:Zunächst wurde jedes H&E-gefärbte histologische Bild entsprechend den Koordinaten jedes Punkts in mehrere nicht überlappende Bereiche von 200 × 200 Pixeln mit einer Auflösung von 0,5 μm/Pixel segmentiert;
- Modell zur räumlichen Vorhersage der Genexpression:Für jeden Patch erstellten die Forscher einen Nuclei-Graph, in dem jeder von Hover-Net24 segmentierte Nukleus als Knoten dargestellt wurde und der Abstand zwischen jedem Nukleuspaar bestimmte, ob eine Kantenverbindung vorlag. Basierend auf der in Abbildung C oben dargestellten Architektur verwendeten die Forscher das IGI-DL-Modell, um die Expression von Zielgenen an jedem Punkt im Histologiebild vorherzusagen.
- Überlebensmodell des Super-Patch-Graphs basierend auf der vorhergesagten räumlichen Genexpression:Um die Prognose anhand der durch räumliche Genexpression abgegrenzten TME weiter vorherzusagen, erstellten die Forscher ein Super-Patch-Diagramm aus H&E-gefärbten Ganzglasbildern (WSI) jedes Krebspatienten und konstruierten dann ein diagrammbasiertes Überlebensvorhersagemodell unter Verwendung des erstellten Super-Patch-Diagramms und klinischer Merkmale als Eingabe.
Forschungsergebnisse: IGI-DL-Modell schneidet insgesamt gut ab
Insgesamt integriert das in dieser Studie erstellte IGI-DL-Modell die Vorteile von Convolutional Neural Networks und Graph Neural Networks und nutzt die Pixelintensität und Strukturmerkmale in histopathologischen Bildern voll aus, um eine genauere Vorhersage der räumlichen Expressionsniveaus von Genen zu erreichen.Das Modell zeigte bei drei Arten solider Tumore gute Ergebnisse: Dickdarmkrebs, Brustkrebs und Plattenepithelkarzinom der Haut, mit einer durchschnittlichen Verbesserung des Korrelationskoeffizienten von 0,171 im Vergleich zu fünf bestehenden Methoden.

Für Dickdarmkrebs verglichen die Forscher die Pearson-Korrelation von 179 von IGI-DL vorhergesagten Genen mit fünf SOTA-Modellen.IGI-DL erreichte bei den 10 verbleibenden Patienten eine durchschnittliche Pearson-Korrelation von 0,343 und übertraf damit die anderen Modelle mit einem durchschnittlichen Anstieg von 0,233 deutlich.Wie im Bild oben gezeigt.

Für Brustkrebs verglichen die Forscher die Pearson-Korrelationen von 187 Genen, die von IGI-DL vorhergesagt wurden, mit dem vorherigen Modell. IGI-DL erreichte bei den 27 verbleibenden Patienten eine durchschnittliche Korrelation von 0,231. Wie in der Abbildung oben gezeigt,Das IGI-DL-Modell übertrifft alle SOTA-Modelle mit einer durchschnittlichen Verbesserung von 0,142.

Für das kutane Plattenepithelkarzinom verglichen die Forscher die von IGI-DL vorhergesagte Pearson-Korrelation von 487 Genen mit früheren Modellen. IGI-DL erreichte bei den vier beibehaltenen Patienten eine durchschnittliche Korrelation von 0,198, was die beste Leistung aller Modelle darstellte.Die durchschnittliche Leistung anderer SOTA-Modelle wird um 0,131 verbessert.Wie im Bild oben gezeigt.
In Bezug auf die plattform- und krebsübergreifende Leistung ist das beste SOTA-Modell, wie in den obigen Experimenten, nicht auf die interne Validierung und externe Testsätze verschiedener Krebsarten festgelegt.Die Leistung des IGI-DL-Modells ist jedoch immer besser als bei anderen Modellen, mit einer durchschnittlichen Verbesserung von 0,171, was auf eine gute plattformübergreifende Generalisierungsfähigkeit hinweist.
Darüber hinaus untersuchten die Forscher die Leistung der krebsübergreifenden Vorhersage von IGI-DL. Das auf Dickdarmkrebs trainierte Modell zeigte bei der internen Validierung und den externen Testsätzen für kutanes Plattenepithelkarzinom gute Ergebnisse mit durchschnittlichen Korrelationen von 0,204 bzw. 0,143. Allerdings war die Leistung der Vorhersagen für verschiedene Krebsarten meist geringer als beim Training und Testen für eine einzelne Krebsart.Dieses Ergebnis lässt darauf schließen, dass die räumliche Genexpression in Tumorregionen eine gewisse Krebsspezifität aufweist und dass es inhärente Schwierigkeiten bei der krebsübergreifenden Vorhersage gibt.

Was die Leistung der Prognosevorhersage betrifft, kann das Super-Patch-Graph-Überlebensmodell, das auf räumlicher Genexpression als Knotenmerkmalen basiert, in der Brustkrebs-Kohorte des Cancer Genome Atlas (TCGA-BRCA) bei 5-facher Kreuzvalidierung einen durchschnittlichen Konsistenzindex (C-Index) von 0,747 erreichen; In der Kohorte des Cancer Genome Atlas Colorectal Cancer (TCGA-CRC) weist das Überlebensmodell bei der 5-fachen Kreuzvalidierung einen C-Index von 0,725 auf, der besser ist als bei anderen Prognosemodellen, wie in der obigen Abbildung gezeigt.
Dieses Überlebensprognosemodell behält auch seinen Genauigkeitsvorteil bei der Vorhersage der Prognose von Patienten im Frühstadium (Stadium I und II) und der vorhergesagte Risikowert kann als unabhängiger Prognoseindikator für Patienten aller Stadien und Patienten im Frühstadium verwendet werden. Im externen Testsatz MCO-CRC, der Daten von mehr als tausend Patienten enthält, behielt das Überlebensprognosemodell einen stabilen Vorteil und verfügte über Generalisierungsfähigkeit.
Brustkrebs und Bauchspeicheldrüsenkrebs zuerst: KI zur Verbesserung der Prognose
Bei der Diagnose und Behandlung von Krebs kann eine Krebsprognoseanalyse Überbehandlung und Verschwendung medizinischer Ressourcen wirksam vermeiden und dem medizinischen Personal und seinen Familien eine wissenschaftliche Grundlage für medizinische Entscheidungen bieten. In den letzten Jahren ist es zu einem heißen Thema in der Krebsforschung geworden.
Um die Behandlungsergebnisse bei Brustkrebs zu verbessern, arbeiteten Salesforce-Forscher im Jahr 2020 mit Klinikern des Lawrence J. Ellison Institute der University of Southern California zusammen, umEinführung des maschinellen Lernsystems ReceptorNet,Sein Algorithmus kann den Hormonrezeptorstatus anhand kostengünstiger und leicht zugänglicher Gewebebilder vorhersagen – ein wichtiger Biomarker für Kliniker bei der Entscheidung über den geeigneten Behandlungsweg für Brustkrebspatientinnen. Die Genauigkeit des Systems erreichte 92%.

Im Februar 2024 verwendeten Forscher der University of Kentucky, der Macau University of Science and Technology, der University of Macau und des First Affiliated Hospital der Guangzhou Medical University ein neuronales Netzwerkmodell, umEs wurde ein präzises Prognosebewertungssystem, MIRS (Metastasen- und Immunogenomrisiko-Score), entwickelt.Die Bewertung des Tumormetastasierungs- und immungenomischen Risikos stellt ein Vorhersageinstrument dar, das bei Brustkrebspatientinnen nahezu universell anwendbar ist und neue Behandlungsmöglichkeiten für Brustkrebspatientinnen bietet. (Klicken Sie hier für einen ausführlichen Bericht: Chinesische Wissenschaftler haben das Brustkrebs-Prognosesystem MIRS entwickelt, um die weltweit am häufigsten auftretende Krebsart zu bekämpfen.)
Darüber hinaus ist Bauchspeicheldrüsenkrebs einer der häufigsten bösartigen Tumoren des Verdauungstrakts und die Fünfjahresüberlebensrate nach der Diagnose beträgt nicht mehr als 10%. Ein entscheidender Schritt zur Verbesserung der Überlebensraten von Patienten besteht darin, das prognostische Risiko des Patienten genau vorherzusagen, um gezielte Behandlungspläne zu entwickeln. Die Histopathologie ist eine Routineuntersuchung in der Onkologie, mit der Tumoreigenschaften auf mikroskopischer Ebene analysiert werden können und die eine wichtige Methode zur Beurteilung des Risikos einer Tumorprogression darstellt. Aufgrund der Größe der Schnitte und der komplexen Gewebezusammensetzung können die Auswertungsergebnisse jedoch leicht durch subjektive Faktoren beeinflusst werden.

Im Jahr 2023 wird ein Forschungsteam der Nanjing University of Information Science and Technology und des Institute of Smart Healthcare, School of Artificial Intelligence,Veröffentlichung eines Forschungspapiers mit dem Titel „Multi-Gewebesegmentierungsmodell für Ganzschichtbilder von Bauchspeicheldrüsenkrebs basierend auf Multitasking und Aufmerksamkeit“.Die Gewebesegmentierung von 8 Kategorien pathologischer Abschnitte des Bauchspeicheldrüsenkrebses wurde untersucht. Durch die Einführung eines Aufmerksamkeitsmechanismus und die Gestaltung einer hierarchischen und gemeinsam genutzten Multitasking-Struktur konnte die Modellleistung durch die Verwendung verwandter Hilfsaufgaben erheblich verbessert werden.
Das in dieser Studie vorgeschlagene Modell wurde anhand des Datensatzes des Shanghai Changhai Hospital trainiert und getestet und extern anhand des öffentlichen TCGA-Datensatzes validiert. Die F1-Werte im internen Testsatz waren höher als 0,97 und die F1-Werte im externen Validierungssatz waren höher als 0,92. Die Generalisierungsleistung war deutlich besser als bei der Basismethode.
Es muss betont werden, dass KI Pathologieexperten nicht ersetzen kann, sondern vielmehr als unterstützende Diagnosetechnologie dient, um die pathologische Diagnose zu vereinfachen und die Arbeitseffizienz von Pathologen weiter zu verbessern. Langfristig gesehen besteht bei der KI noch viel Entwicklungspotenzial in der digitalen Biomarkererkennung, der medizinischen Bildanalyse, der Krankheitsverlaufsvorhersage und anderen Bereichen.
Quellen:
1.https://news.un.org/zh/story/2018/09/1017602
2.https://mp.weixin.qq.com/s/VE68FKL6kwpO1IFsbR-LVA
3.https://ins.sjtu.edu.cn/articles/286
4.https://www.cdstm.cn/theme/khsj/khzx/khcb/202012/t20201214_1039028.html








