Command Palette
Search for a command to run...
NVIDIA と他の企業は、100 万種の数十億の遺伝子に基づいて EDEN シリーズのモデルを構築し、最先端 (SOTA) のゲノムおよびタンパク質予測機能を実現しました。

プログラム可能な生物学の根本的な目標は、生体システムの合理的な設計と精密な制御を実現し、複雑な疾患に革新的な治療法をもたらすことです。しかしながら、このプロセスは、生物系に固有の複雑さによって長い間制約されてきました。スケールを越えた制御ネットワーク、隠れた長い配列の依存性、環境の変化に対する生物の多様な適応性により、従来の「試行錯誤」による研究開発は、カスタマイズ、低スループット、高コストのジレンマに陥っています。
結局のところ、現在の計算モデルが依存する訓練データは、規模と多様性の両面において、生命が数十億年にわたる進化の過程で発展させてきた広大な設計空間を網羅するには程遠い。そのため、これらのモデルは普遍的な設計原則を捉えるのに苦労している。マルチモーダルかつクロススケールな革新的な治療設計に直面すると、一般化する能力が著しく欠如します。
この根本的な限界を克服するために、Basecamp Research、NVIDIA、およびいくつかの主要な学術機関が共同で、メタゲノム基本モデルの EDEN シリーズを開発しました。種をまたぎ、環境情報と関連する膨大な自然進化データを学習することで、生物設計の深遠な「文法」と普遍的な原理を初めて体系的に抽出しました。このモデルは280億のパラメータを有し、複数のベンチマークテストで最先端の結果を達成しました。その核心的なブレークスルーは、種をまたぐ配列を理解し、生成する卓越した能力にあり、バイオエンジニアリングを「スクリーニング」から「予測可能なプログラミング」という新たな段階へと前進させます。
EDENの統合バイオデザインエンジンとしての能力を検証するため、研究チームは複数の治療法を対象とした体系的な試験を実施しました。遺伝子治療において、EDENは標的部位のわずか30塩基のヒントを用いて、大きな断片をヒトゲノムに正確に組み込むことができる活性リコンビナーゼをde novo設計することができます。抗菌ペプチドの設計に関しては…同じモデルによって生成されたペプチドライブラリは、多剤耐性病原体に対して最大 97% の活性を示しました。また、マイクロモルレベルの効力も有しています。生態系レベルでは、EDENは数万の人工ゲノム、正確な代謝経路、そして合理的な種間関係を含む合成マイクロバイオームの構築に成功しました。
「EDENファミリーの基礎モデルを使用したAIプログラム可能な治療法の設計」と題された関連研究成果が、bioRxivでプレプリントとして公開されています。
研究のハイライト:
* 進化の歴史から普遍的な設計原理を直接学習する新しいパラダイムを開拓し、地球規模の生物多様性を網羅するメタゲノムデータベースであるBaseDataを使用したトレーニングを通じて、優れた種間配列理解および生成機能を実現しました。
* この研究は、マルチスケール、マルチモーダルな治療設計を推進する上で単一の基本モデルの強力な汎用性を検証し、単一のモデルが分子から生態系に至るまでの複雑な設計課題に均一に対処できることを実証しています。
* EDEN は、DNA の手がかりのみを使用して、複数の疾患関連部位の機能的なリコンビナーゼを設計し、訓練されていないターゲットで 63.21 TP3T の機能ヒット率を達成できます。

用紙のアドレス:
https://doi.org/10.64898/2026.01.12.699009
弊社の公式 WeChat アカウントをフォローし、バックグラウンドで「EDEN」と返信すると、完全な PDF を入手できます。
AIフロンティアに関するその他の論文:
https://hyper.ai/papers
BaseData データセット: 高品質の長いシーケンスを使用して生物学的 AI データ ベンチマークを再構築します。
本研究で使用したBaseDataデータセットは、従来の生物学データベースの限界を根本的に打破するものです。従来のデータベースは、限られた参照ゲノムと断片化された短い配列に依存しているのに対し、BaseDataは完全な進化シグナルを体系的に捉え、地球規模の生物多様性を網羅する進化ゲノムデータサプライチェーンを構築することを目指しています。
BaseDataのコアバリューは、主にその規模と戦略的構成に反映されています。下の図に示すように、トレーニング用の 9.7 兆ヌクレオチド マーカーが含まれており、100 万を超える新しい種と 1,000 億の新しい遺伝子をカバーしています。さらに重要なのは、データがランダムに収集されるのではなく、環境メタゲノミクス、バクテリオファージ、可動性遺伝要素といった高情報密度配列が意図的に追加されていることです。これらのデータは、バクテリオファージと宿主の相互作用や遺伝子水平伝播といった重要な進化ダイナミクスを自然に記録しており、種を超えた普遍的な機能ルールをモデルが学習するための中核的な材料となります。
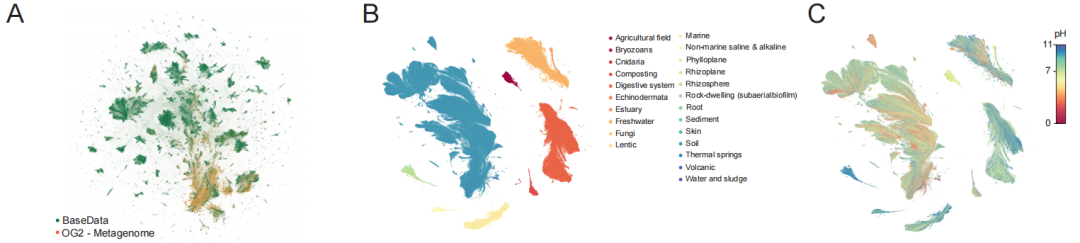
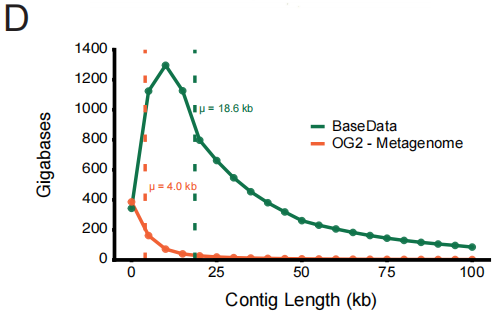
データ品質の面では、BaseDataは、主に配列コンテキストの完全性において質的な向上を達成しています。広く使用されているOpenGenome-2(OG2)と比較すると、連続した配列断片(オーバーラップ)の長さの中央値は18.6 kbp(OG2は4.0 kbp)に達し、各アセンブリには大幅に多くの遺伝子が含まれています。このより長い連続した背景は、モデルが遺伝子間制御と代謝経路を理解する上で非常に重要です。
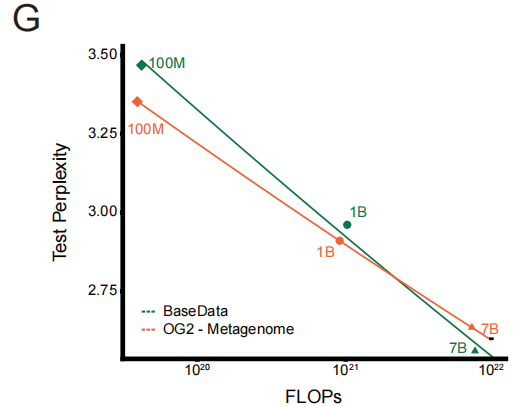
この品質優位性を定量化するために、研究チームは制御実験を実施しました。これは、BaseDataとOG2の同サイズのデータセットを用いて一連のモデルを学習させるというものです。その結果、「品質を考慮したスケーリング則」が明確に検証されました。同じ計算オーバーヘッドにおいて、BaseDataで学習したモデルはテストパープレキシティの減少が速かったことが分かりました。重要な発見は、大規模モデル(例えば70億パラメータ)がBaseDataの長シーケンス情報を最大限に活用し、最終的にOG2で学習した同様のモデルを上回る性能を発揮できることです。これは、長期的なコンテキストがモデルのパフォーマンスに決定的な影響を与えることを直接示しています。
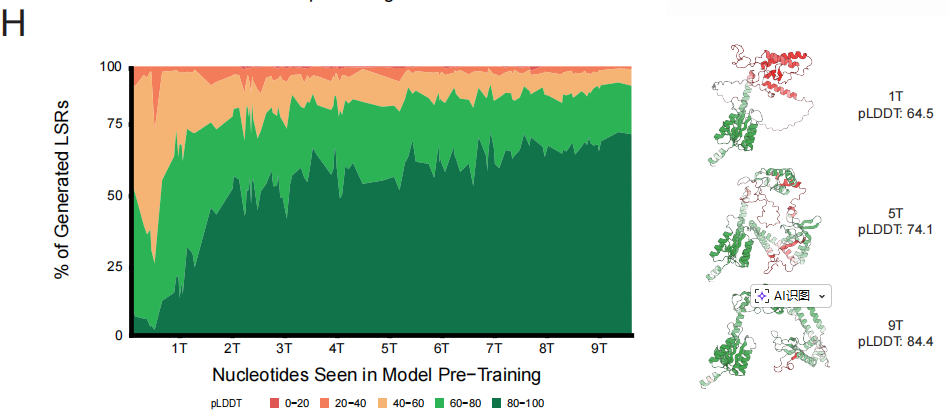
このパターンに基づいて研究チームは、完全な BaseData を使用して 280 億のパラメータを持つ EDEN-28B モデルをトレーニングしました。このモデルは、テストパープレキシティが最も低かっただけでなく、そのパフォーマンス向上の軌跡は、小規模モデルから得られたスケーリング予測と完全に一致しています。下流タスクモニタリングにおいて、事前学習中にモデルによって生成されたタンパク質の構造信頼度指数は、学習プロセスとともに継続的な単調増加を示しました。これは、高品質なデータが、実用的な治療のためのデータ生成能力を直接的かつ着実に向上させることを示しています。
さらに、すべてのデータは、28か国と208のライセンスを網羅する標準化された法的合意を通じて取得され、情報源から使用までの追跡可能性と利益分配の枠組みを確立し、大規模な生物学的AI研究に必要な倫理およびガバナンス基準を設定しました。
汎用生物設計エンジン「EDEN」
EDEN モデル ファミリは、「スケーラビリティ、普遍性、拡張性」を中核設計原則として設計されており、モデル パラメータは 1 億から 280 億に及びます。その中でも、中核となる作業モデルである EDEN-28B は、メタゲノム データの固有の特性に合わせて深く適応したアーキテクチャとトレーニング戦略を備えています。
モデルアーキテクチャの観点から言えば、EDEN は、特に Llama 3.1 の設計スタイルに基づいて、大規模言語モデルによって検証されたデコーダーのみの Transformer アーキテクチャを採用しています。この選択は、Transformerの優れた長距離依存性モデル化能力によって可能になりました。EDEN-28Bは、6,144層の隠れ層と48個のアテンションヘッドを備えた48層ネットワークで構成され、SwiGLU活性化関数とRoPE位置エンコーディングを採用しています。このモデルは、512語彙の単一ヌクレオチド解像度トークン化手法を採用しており、最も基本的な「文字」レベルでDNA配列を理解し、生成することができます。
重要な技術的ハイライトは、長いシーケンスを生成できる能力にあります。モデルのコンテキストウィンドウは8,192ラベルに設定されていますが、実用的には正しい遺伝子順序、読み取りフレーム、および調節要素構造を維持しながら、13,000 塩基対を超える一貫したゲノム配列を安定して生成し、正確にアセンブルできます。これは、モデルが局所的なパターンマッチングをはるかに超える学習を行っていることを示しています。つまり、物理的なウィンドウ長を超えた、ゲノム構造のより深い「文法」を推論し、適用できるということです。トレーニング全体は1,008基のH100 GPUで完了し、大規模な分散コンピューティングを通じて膨大な量の進化データから効率的な学習を実現しました。
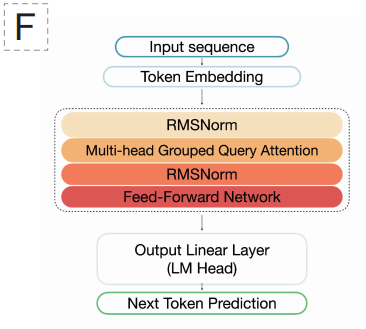
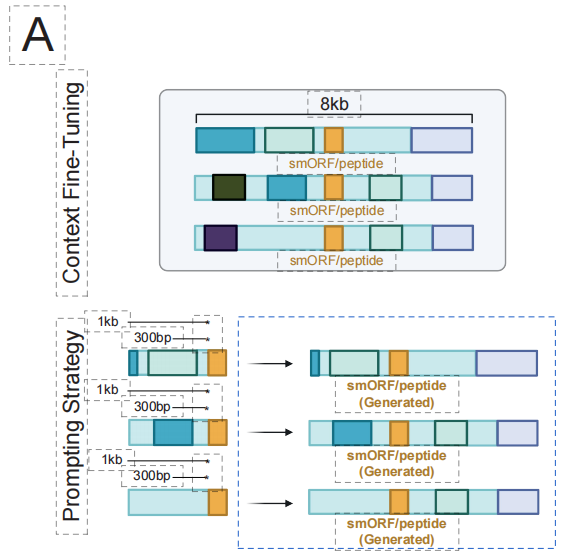
EDENの中核となる設計哲学は、「事前学習と微調整」というパラダイムに基づいています。第一段階では、様々な種の進化史を網羅するBaseDataを用いて、モデルを大規模に事前学習します。これにより、タンパク質の折り畳みや代謝経路の構築といった生物学的設計の一般原則が内部化されます。
この強固な基盤を基にして、特定の DNA サイトをターゲットとするリコンビナーゼの設計や、新しい抗菌ペプチドの生成など、特定の治療設計タスクに取り組むことができます。軽量な微調整のための少量の高品質タスクペアデータのみを使用して、モデルはタスクの「方言」をすぐに習得できます。この設計により、単一の EDEN モデルが汎用的な「生物学的シーケンス エンジン」として機能することが可能になり、遺伝子挿入やペプチド設計からマイクロバイオーム工学に至るまで、多様な治療法に柔軟に適応して駆動できるようになり、「1 つのモデルで複数の機能」というプログラム可能な生物学のビジョンを真に実現します。
分子レベル、細胞レベルから生態系レベルまで、治療のイノベーションを推進
実際の治療設計における EDEN モデルの普遍性と有効性を体系的に検証するために、研究チームは、規模、パターン、生物学的複雑性が大きく異なる 4 つの主要な方向性を選択して実験検証を行いました。
AIプログラマブル遺伝子挿入(aiPGI)の分野では、チームは長年のボトルネックであった「大きなDNA断片の正確な統合」の克服に注力してきました。従来のCRISPR技術は二本鎖切断を誘発する手法に依存しており、天然の大型セリンリコンビナーゼはヒトゲノム配列を認識できません。下図に示すように、EDENのソリューションは、モデルに含まれる数百万ものLSR結合部位のペアを微調整することで、「標的DNA配列 → 対応するリコンビナーゼ」のマッピング関係を理解できるEDEN-LSRモデルを構築することです。
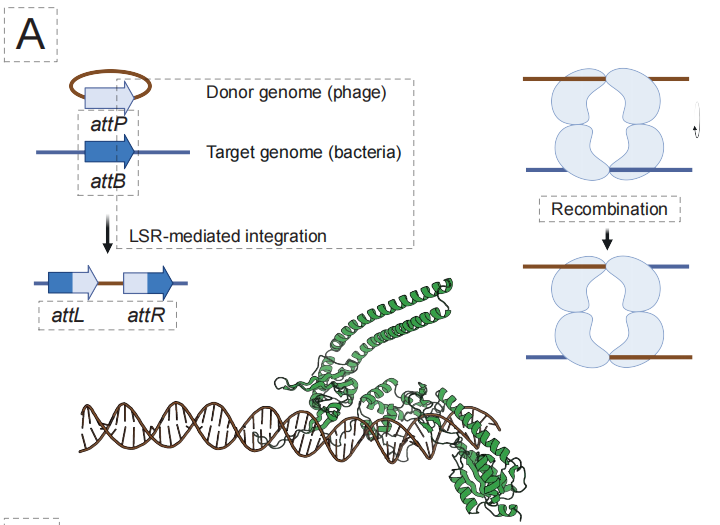
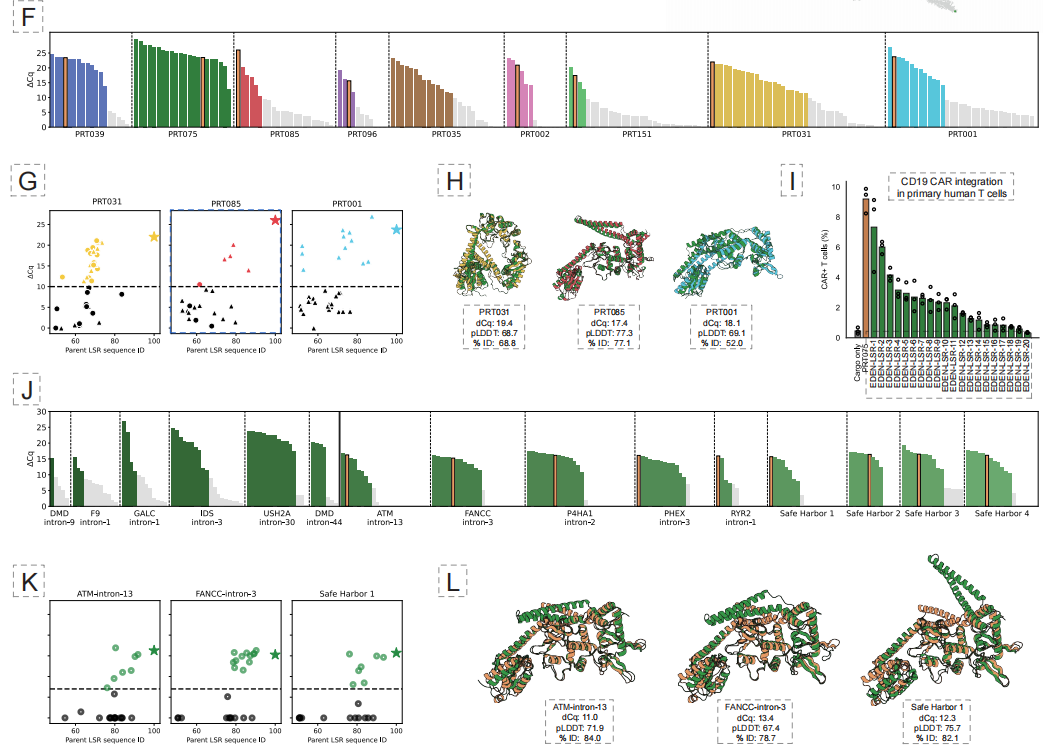
実験結果によると、この手法は10の異なる疾患関連遺伝子座と4つの潜在的な「セーフハーバー」座に対して活性LSRを生成することに成功し、全体的な機能的ヒット率は53.6%でした。さらに重要なのは、50% 設計酵素は、初代ヒト T 細胞での治療関連の CAR 遺伝子挿入を実現でき、いくつかの変異体は細胞株で最大 40% の統合効率を実現しています。これは臨床応用の可能性を実証しています。
新規ブリッジングリコンビナーゼ(BR)の分野では、EDEN モデルの機能は、よりプログラム可能な遺伝子編集システムであるブリッジングリコンビナーゼにまで拡張されました。下の図に示すように、設計を最適化するために、チームは数百万の BR を含むゲノム領域でモデルを微調整して、EDEN-BR 固有のモデルを構築しました。
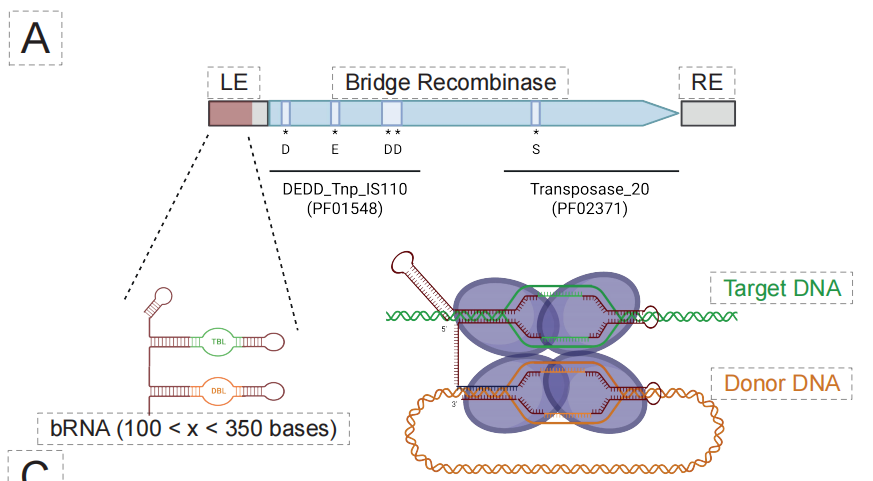
主要な生化学実験により、この設計プロセスの実現可能性が検証されました。下図に示すように、予備的な無細胞試験において、EDEN-BRによって生成された49個の候補配列のうち2個が明確なリコンビナーゼ活性を持つことが確認されました。DF3843とDF3881と名付けられたこれらの2つの人工的に設計されたタンパク質は、既知の天然BR配列との類似性は最大でそれぞれ851 TP3Tと65.81 TP3Tしかありません。よく研究されている参照タンパク質であるISCro4との配列類似性は351 TP3Tよりもさらに低いものの、3次元構造は非常に類似しています。これは、EDEN が単に配列を模倣するのではなく、タンパク質の機能と折り畳みを決定するコア構造ロジックを習得していることを証明しています。
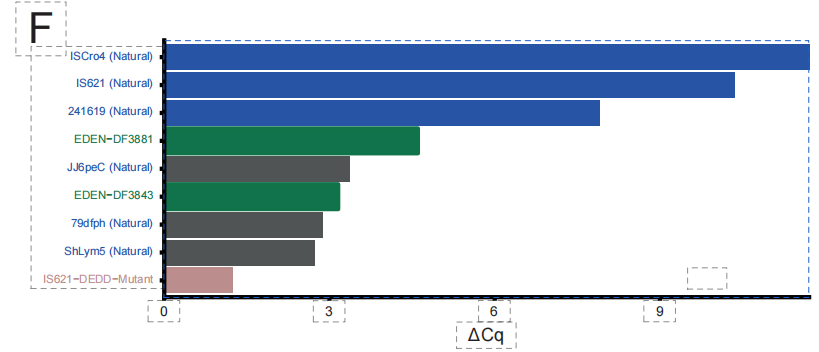
新規抗菌ペプチド(AMP)の分野において、研究チームはEDENの新規抗菌ペプチド設計能力を検証しました。下図に示すように、ゲノムコンテキスト情報を組み込んだ微調整戦略を採用することで、モデルは新しい抗菌ペプチド配列を生成することができます。
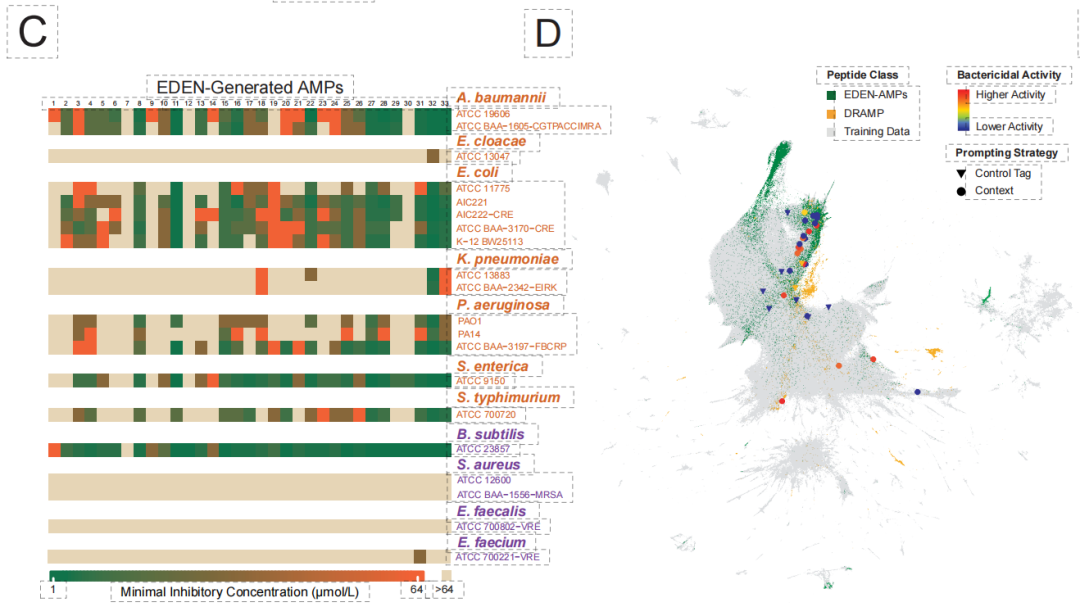
実験検証により画期的な結果が得られました。下の図に示すように、33 個の生成ペプチドからなる AMP ライブラリでは、最大 971 個の TP3T 配列が抗菌活性を示しました。その中で、最も優れた設計の候補は、多剤耐性グラム陰性細菌(アシネトバクター・バウマニなど)に対してマイクロモルレベルの阻害濃度を達成し、強力な外膜透過能を示しました。生成された配列は、一般的に既存のデータベースとの類似性が低く、このモデルが従来の相同性制限を克服し、真の「de novo設計」を実現できることが確認されました。
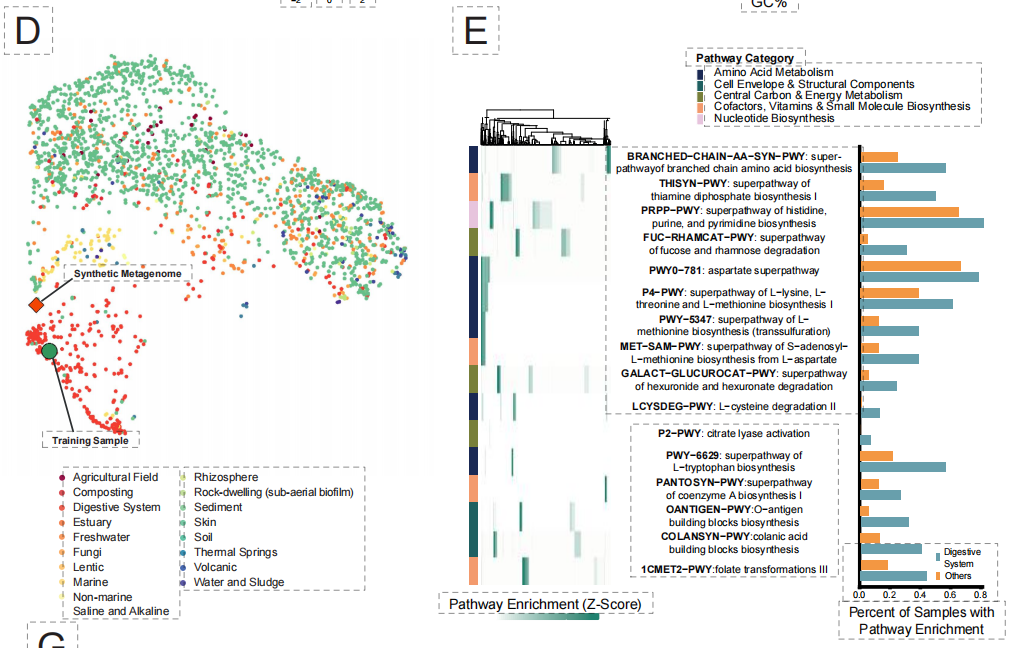
最後に、最も複雑な生態系レベルにおいて、この研究は「合成マイクロバイオーム」の設計に挑戦しました。従来の方法では、複数の種間の代謝相互作用と生態系のバランスを調整することが困難でした。下図に示すように、EDENは消化器系のマイクロバイオームデータを用いて微調整を行い、機能遺伝子またはニッチの手がかりのみに基づいて、90,000 種以上を含みギガベースに及ぶ合成メタゲノミクスが正常に生成されました。
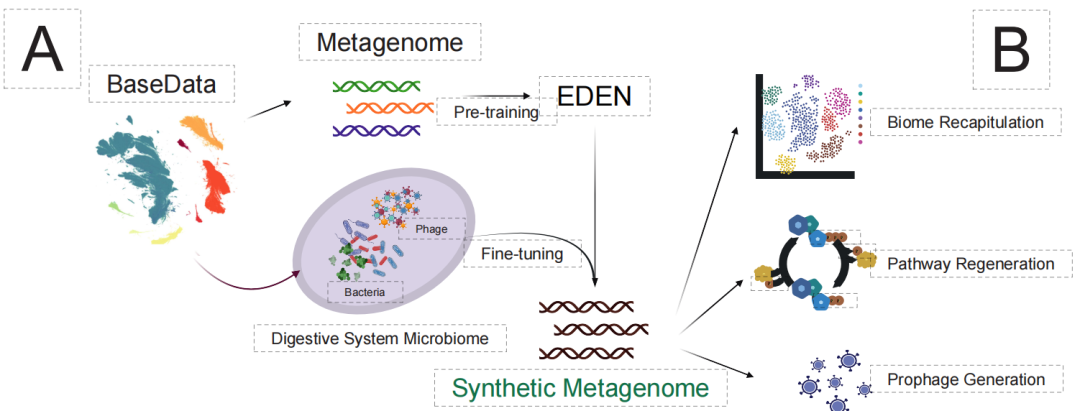
生成された結果は、高度な生態学的リアリズムを示しています。種 99% は消化器系関連の生物群に正しく分類され、種間の代謝経路は完全に保存されていました。さらに、このモデルは宿主ゲノムに統合されたプロファージ構造を正確に生成することができ、宿主とウイルス間の複雑な相互作用ロジックを捉えていることを実証しています。
これら4つのクロススケール実験は、統一された進化データで事前学習されたEDENモデルが、汎用的な生物学的設計エンジンとして機能できることを総合的に実証しています。このモデルは、最小限のタスク固有のデータガイダンスで、分子レベル、細胞レベル、そして生態系レベルから迅速かつ確実に治療革新を推進することができ、プログラム可能な生物学のための確固たる実用的な基盤を築きます。
AIと合成生物学の統合によるイノベーション
近年、プログラム可能な生物学の分野における学界と産業界の統合と革新が大幅に加速しており、一連の大きな進歩によりバイオデザインの境界が再定義されています。
世界中の一流学術機関が、進化の知恵を、かつてない規模と精度で計算可能なモデルへと変換しています。例えば、2024年初頭には、DeepMind、Isomorphic Labs、そして複数の大学からなる共同チームが、タンパク質の構造と相互作用を同時に予測し、特定の機能を持つ新規タンパク質を生成するAlphaFold 3モデルを発表しました。このモデルは、生体分子の複雑な相互作用を高精度シミュレーションのための統一されたフレームワークに組み込んだ初めてのモデルです。ネイチャー誌はこれを「生命の分子機械の内部の仕組みを解明する上での飛躍的進歩」と称賛した。
業界では、これらのブレークスルーをプラットフォームや治療法へと転換する動きが加速しています。AIを活用した医薬品開発の分野では、NVIDIAとRecursion Pharmaceuticalsが共同で、創薬を「干し草の山の中の針を探す」ような作業から「誘導型アプローチ」へと転換することを目指した生化学AIモデルライブラリ「BioNeMo」をリリースしました。合成生物学企業のGinkgo Bioworksは、自動化プラットフォームを活用し、炭素回収と化学物質生産のための微生物群集を体系的に設計することで、「合成エコシステム」のエンジニアリングを推進しています。
データとアルゴリズムによって推進されるこの新たな波は、生物学を観察と記述に基づく科学から、プログラム可能でデバッグ可能、そして予測可能な工学分野へと押し進めています。これは、生命をより正確にコード化して病気を克服できるようになるだけでなく、資源、環境、そして健康といった分野における地球規模の課題に対処するために、生物システムを体系的に設計する能力を予兆するものでもあります。
参考リンク:
1.https://nvidianews.nvidia.com/news/nvidia-announces-broad-expansion-of-its-biomedicine-platform
2.https://www.ginkgobioworks.com/2024/01/04/ginkgo-bioworks-and-pfizer-expand-collaboration-to-advance-rna-based-therapeutics/








