Command Palette
Search for a command to run...
香港中文大学などの研究者らは、遺伝子発現データと細胞形態画像をリンクさせることで、表現型薬物の開発を加速するトランスクリプトーム誘導拡散モデルを開発した。
細胞形態学は、単一細胞生物学における中核研究分野です。その価値は、ハイスループット画像解析を用いて、遺伝子や薬剤の摂動下における細胞形態の動的な変化を体系的に解析することにあります。この研究は、化合物の作用機序(MOA)予測の精度を大幅に向上させるだけでなく、化合物の生物活性評価の精度も向上させます。最終的には、候補化合物のスクリーニングや作用機序の検証といった、表現型創薬における重要なステップをデータで裏付け、研究開発プロセスを効果的に加速させます。
しかし、遺伝子や薬剤による撹乱後の細胞形態変化を観察・分析することは容易ではありません。スクリーニング可能な化合物の数は数百万を超え、編集可能な遺伝子の数も数万に及びます。従来の実験手法を用いて一つ一つ検証することは、極めて非効率的であるだけでなく、多大な時間と費用がかかります。細胞形態予測には様々な計算手法が提案され、適用されてきましたが、その精度と忠実度は依然として実用的な研究のニーズを満たすには至っていません。
具体的には、既存の方法の限界は、主に次の 2 つの点に反映されます。まず、IMPA(IMage Perturbation Autoencoder)などの高度なモデルの性能は、既知の生物学的知識や特定のデータセットに大きく依存するため、一般化能力が弱く、幅広い適用性に欠けます。次に、細胞形態データは、バッチ効果やウェル位置効果といった実験干渉要因の影響を受けやすく、ノイズレベルが高いため、真の細胞形態特性を効果的に捉えることが困難です。これは、データの安定性と信頼性に直接影響を与え、その後の解析結果の精度を制限します。
上記の課題に対処するため、香港中文大学、モハメド・ビン・ザイード人工知能大学などの研究者は、スケーラブルなトランスクリプトーム誘導拡散モデルである MorphDiff を提案しました。このモデルは、摂動に対する細胞形態応答の高忠実度シミュレーション用に特別に設計されています。潜在拡散モデル(LDM)アーキテクチャをベースとし、L1000遺伝子発現プロファイルをノイズ除去学習の条件付き入力として使用します。
研究の結果は、MorphDiff の主な利点は、「目に見えない摂動条件」下で細胞形態を正確に生成できることです。この機能は、2つの重要なメリットをもたらします。第一に、研究者が広大な表現型変動スクリーニング領域を効率的に探索するのを支援し、大規模なフィールド実験への依存を大幅に低減することで、実験コストを削減し、スクリーニング効率を向上させます。第二に、構造的に多様な薬物分子の作用機序の解明を支援し、化合物の作用機序検証に重要なサポートを提供します。したがって、MorphDiffは、表現型医薬品の開発を加速させる高性能ツールとして機能します。
研究結果は「トランスクリプトーム誘導拡散モデルによる摂動下での細胞形態変化の予測」というタイトルでNature Communicationsに掲載されました。
研究のハイライト:
* この研究では、拡散モデルを革新的に適用して初めて細胞形態を予測し、新たな道を切り開き、表現型薬物開発のための新しいツールを提供します。
* 広範なベンチマーク テストにより、MorphDiff の有効性が実証されています。特に MOA 検索においては、グラウンド トゥルース形態学と同等の精度を達成でき、ベースライン メソッドをそれぞれ 16.9% と 8% 上回っています。

用紙のアドレス:
https://www.nature.com/articles/s41467-025-63478-z
公式アカウントをフォローし、「トランスクリプトーム誘導拡散」と返信すると、完全なPDFが入手できます。
AIフロンティアに関するその他の論文:
https://hyper.ai/papers
データセット: 有効性を検証するための大規模マルチオミクスデータセット
摂動条件下での細胞形態の予測におけるMorphDiffモデルの有効性と一般性を体系的に検証するために、この研究では、「遺伝子変動と薬物変動」という二重の次元に基づいて、マルチセルラインおよびマルチソースデータセットシステムを構築しました。実験では、各サンプルについて、L1000遺伝子発現プロファイルと細胞形態画像の2種類のデータが収集され、ペアデータを形成しました。前者は「分子特性入力」、後者は「表現型特性入力」として機能しました。この手法により、遺伝子レベルの摂動シグナルと形態レベルの表現型応答との相関が確保され、標的摂動のみによって駆動されます。これにより、細胞株の違いや実験バッチなどの無関係な変数による干渉が排除されます。
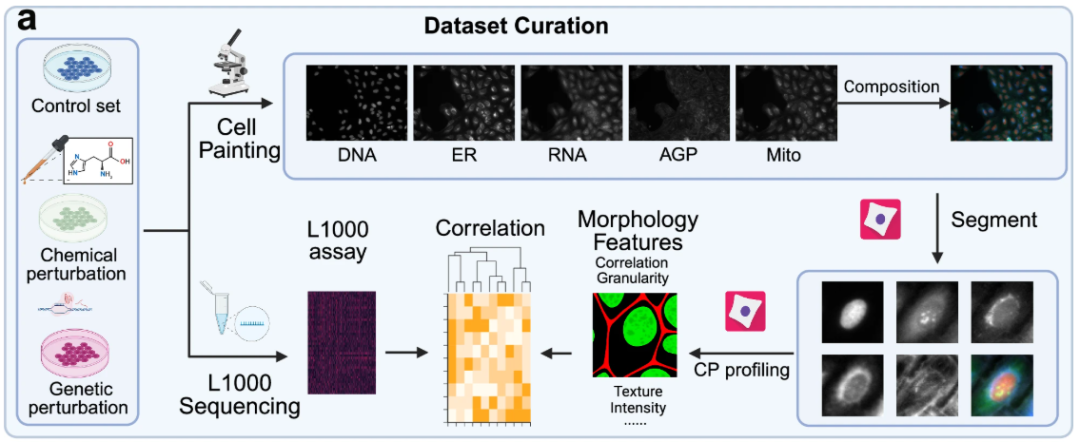
細胞形態画像データセットに関しては、本研究では、モデルの学習、評価、および解析のために、3つの大規模細胞形態画像データセットを使用しました。1つは遺伝子変動データセット、2つは薬物変動データセットです。遺伝子変動データセットは、U2OS細胞株に基づくJUMPデータセットから派生したもので、130個の遺伝子変動を含んでいます。薬物変動データセットは、U2OS細胞株に基づくCDRPデータセットとA549細胞株に基づくLINCSデータセットから派生したもので、それぞれ1,028個の薬物変動と61個の薬物変動を含んでいます。
3つの細胞形態画像データセットはすべて前処理され、セグメント化されました。より詳細な分析を行うために、CellProfiler 4.2.5を使用して、バルク細胞プレート画像を単一細胞画像にセグメント化しました。セルペインティング技術によって取得される細胞形態画像には、5 つのコア チャネルが含まれます。つまり、DNA(核)、RNA(核小体と細胞質)、ER(小胞体)、AGP(ゴルジ体/細胞膜/アクチン骨格)、Mito(ミトコンドリア)です。
その上、この実験ではさらに、「対応する形態学的画像がない」L1000 データセットを計画しました。このデータセットは主に、「遺伝子発現データのみを取得する」というシナリオでのモデルの適用をさらに探求するために使用され、その後のメカニズム検証、薬物スクリーニングなどの基礎を築き、「データ駆動型」の仮説を提供することができます。
モデル構造と手法:遺伝子発現データと細胞形態画像のリンク
MorphDiff の主な目標は、トランスクリプトーム誘導潜在拡散モデル フレームワークを通じて、L1000 遺伝子発現プロファイルから細胞形態画像へのエンドツーエンドの正確なマッピングを実現することです。簡単に言えば、ある「擾乱」に対応する L1000 遺伝子発現データを入力し、この擾乱を受けた細胞の形態画像または擾乱形態を出力するという、「橋」のようなモデルを設計し、トレーニングすることです。
MorphDiff モデルの中核は、次の 2 つの主要モジュールで構成されています。下の図 b に示すように、形態学変分オートエンコーダ (MVAE) と潜在拡散モデル (LDM) を使用します。
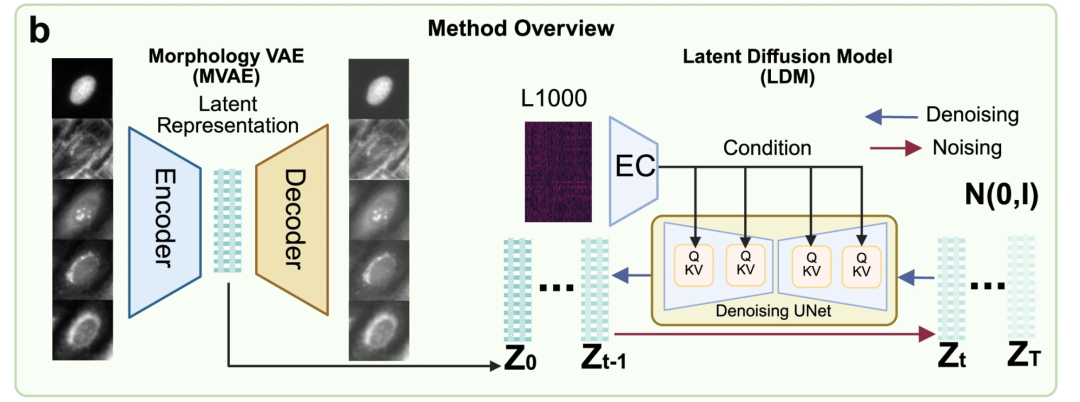
その中で、MVAE はモデルの「画像圧縮エンジン」であり、マルチチャネルの高解像度の細胞形態画像を低次元の解釈可能な潜在的な表現に変換する役割を担っています。このアプローチは、高次元画像で拡散モデルを直接学習させる際に生じる高い計算コストと不安定な学習に対処します。MVAEは構造的に、エンコーダとデコーダの2つの部分で構成されます。エンコーダは複雑な5チャンネルの形態学的画像をより単純な低次元表現に圧縮し、デコーダはこの低次元表現から元の形態学的画像を復元します。
LDM は主に「遺伝子発現」と「圧縮された形態学的特徴」を結合する役割を担い、モデルが「遺伝子から形態学的特徴まで」の関係を完成させるのに役立ちます。 LDMは、ノイズ除去プロセスとノイズ除去プロセスから構成されます。ノイズ除去プロセスでは、圧縮された形態学的特徴に徐々にガウスノイズを追加し、完全にランダムになるまで続けます。このノイズ除去プロセスにより、モデルはL1000遺伝子の既知の発現を前提として、ランダムノイズを徐々に元の形態学的特徴に復元することができます。このモデルはU-Netネットワークアーキテクチャを採用し、アテンションメカニズムを組み込むことで、主要な遺伝情報と形態学的情報をより正確に結び付けます。
下の図cは、事前学習済みMorphDiffモデルの2つの応用例、G2IとI2Iを示しています。前者は、L1000遺伝子の発現を条件として、対応する細胞形態画像からランダムノイズ分布を除去し、対応する細胞形態画像を生成します。後者は、特定の摂動下でのL1000遺伝子の発現を条件として、形態画像を対照細胞形態から予測された摂動後の形態画像に変換し、「正常形態から摂動後の形態」への予測機能を実現します。
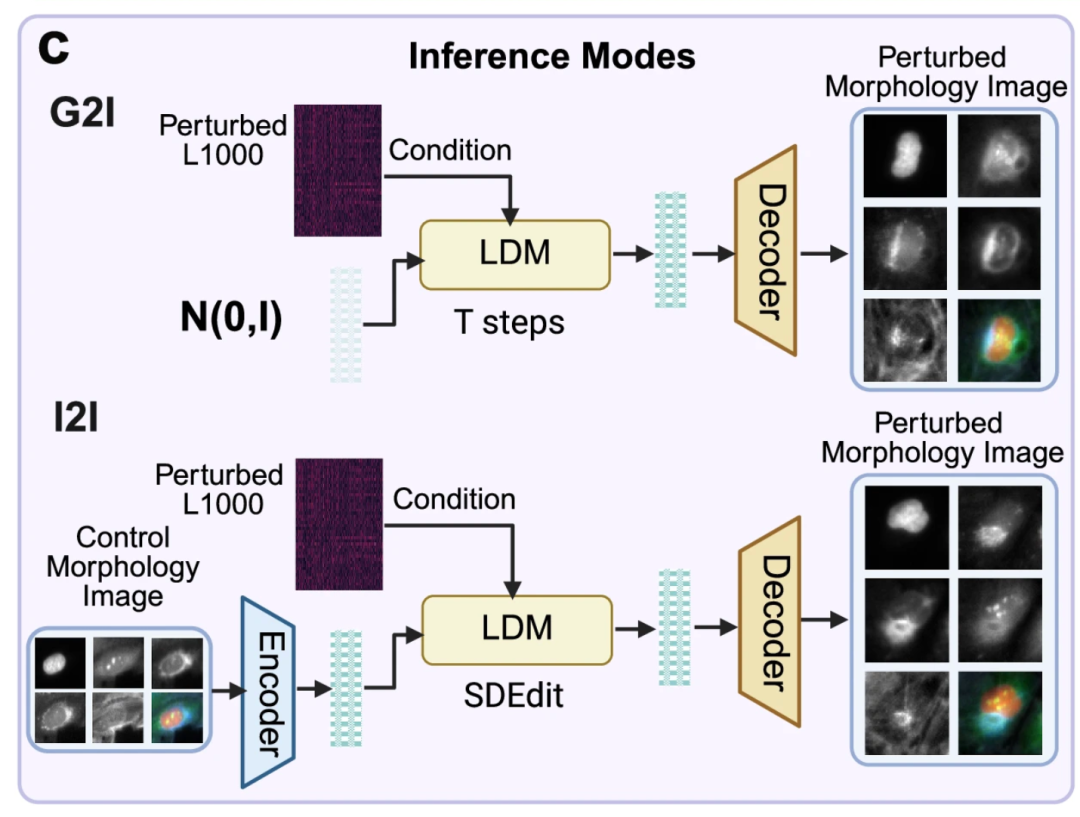
下の図 d は、実際のアプリケーションにおける MorphDiff モデルの価値を示しています。まず、このモデルは、トレーニング中に遭遇しなかった「目に見えない摂動」によって引き起こされる細胞の形態変化を予測することができます。これにより、研究者は物理的な実験を行うことなく、コンピュータで新薬の妨害による細胞の変化の状態をシミュレートすることができ、より多くの可能性を迅速かつ低コストで探索できるようになります。モデル フレームワークは、CellProfiler や DeepProfiler などのツールを組み合わせたものです。薬物のMOAを特定し、表現型薬物の開発を促進するのに役立ちます。
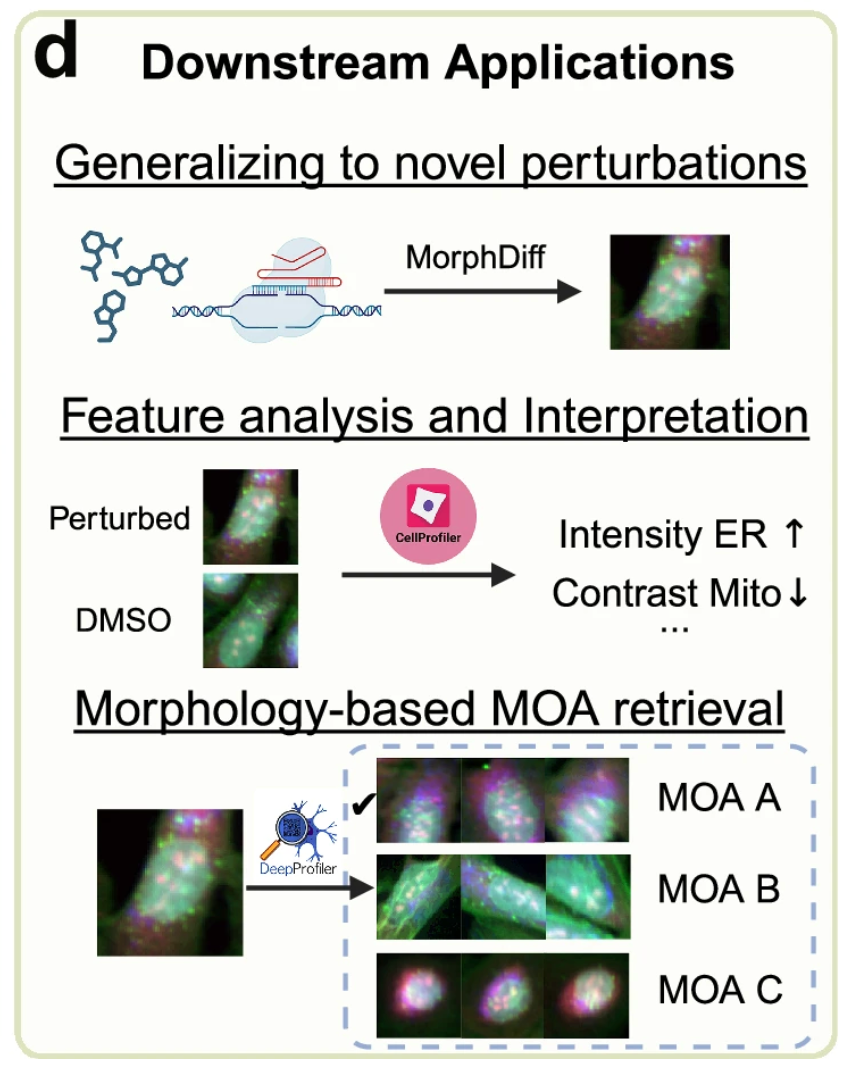
研究者らによると、MorphDiff は現在、遺伝子発現からの形態学的画像の生成と、非撹乱形態から撹乱形態への変換をサポートする唯一のツールだという。
実験結果:IMPAを上回る性能で、表現型薬物の開発を加速
MorphDiff モデルの有効性を検証するために、研究者はさまざまな目的で一連の実験を設計しました。高度なツールとの実験的な比較を通じて、MorphDiff の有効性と実用性が体系的に検証されます。
まず、実験により遺伝子変動の予測が検証されました。研究者らは、JUMP OODデータセットでベンチマークテストを実施し、MorphNet、DMIT(Disentanglement for Multi-mapping Image-to-Image Translation)、DRIT++(Disentangled Representation for Image-to-Image Translation)、StarGANv1、IMPA、VQGAN(Vector Quantized Generative Adversarial Network)、MDTv2(Masked Diffusion Transformers)などの複数のベースライン手法と比較しました。
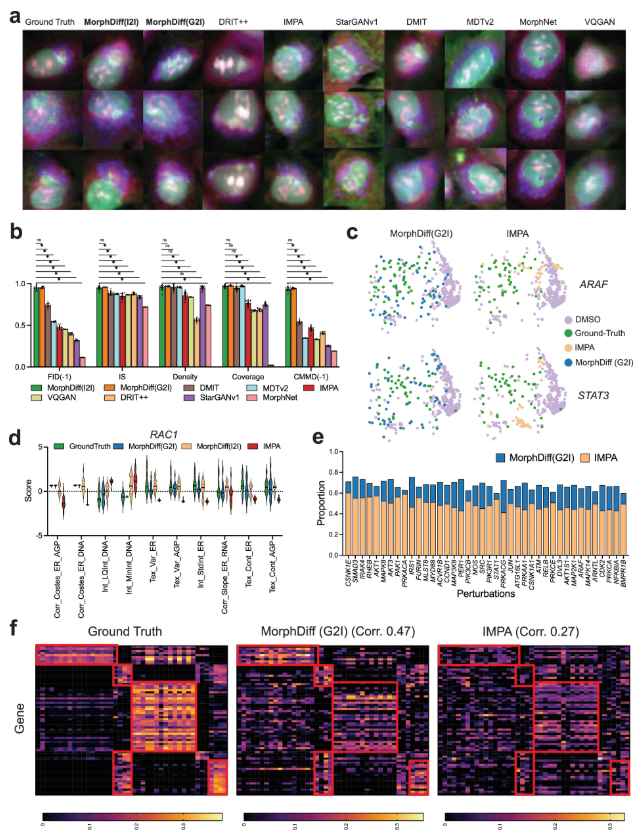
結果は次のようになります。MorphDiff の 2 つのモードによって生成された結果は、視覚的な品質と構造形態の点で実際のベースラインに近くなります。FID、IS(インセプションスコア)、CMMD、密度、カバレッジなどの定量的指標において、MorphDiffの両モードは、一般化、忠実度、多様性においてベースライン手法を上回り、より高い出力品質を達成しました。細胞形態変化の予測において、MorphDiff (G2I) の出力はより多様性が高く、真のベースラインに近い値を示しました。一方、MorphDiff (I2I) によって生成された特徴は、真のベースライン特徴との重なりが大きく、その予測精度が真の摂動形態と有意に一致していることを示しています。
その後、薬物摂動予測の検証実験が行われました。研究者らはまず、CDRP OODデータセットを用いて全ての手法をベンチマークしました。MorphDiff は、ほとんどのメトリックにおいて他のベースライン メソッドと比較して優れたパフォーマンスを実現し、より強力で安定した包括的な生成機能を発揮します。その後、研究者らはLINCSデータセットでモデルのより厳密な評価を実施し、生成された結果をCellProfilerの機能と比較することで、小分子化合物に対する細胞形態の反応をさらに調査しました。
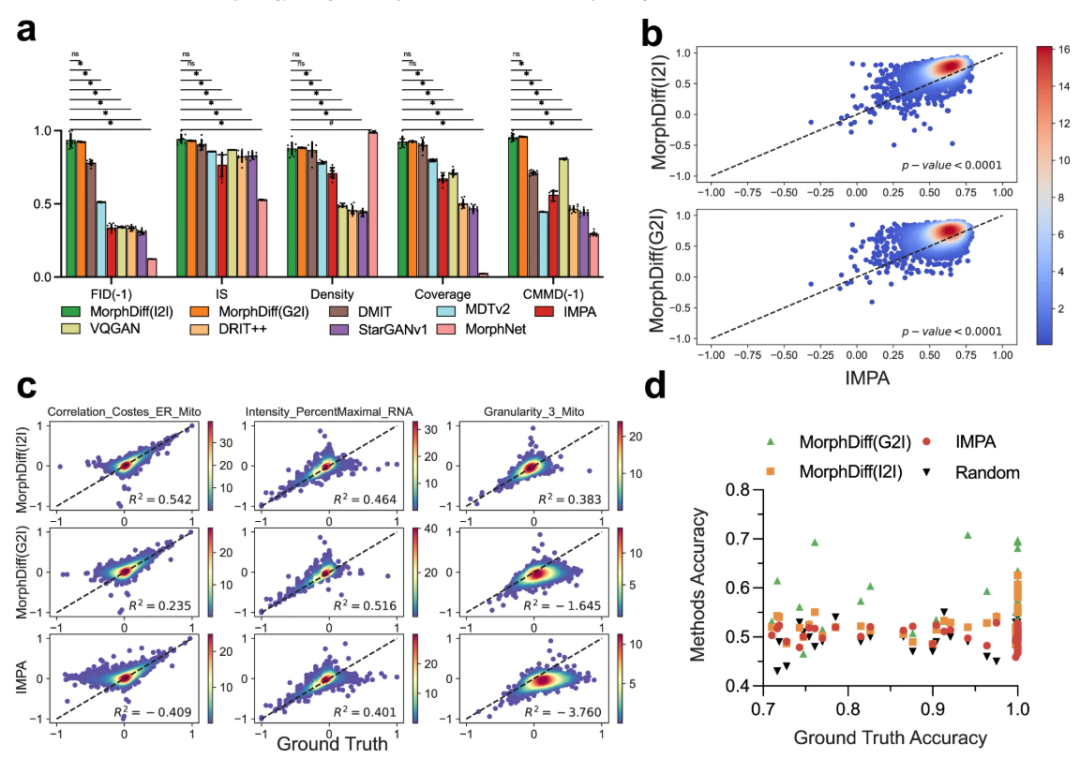
結果は、MorphDiff の両方のモードがベースラインを大幅に上回っていることを示しています。G2Iでは、TP3Tサンプルのうち87.61個が0.5以上のスコアを記録し、16.21個が0.8を超えました。I2Iでは、TP3Tサンプルのうち891個が0.5以上のスコアを記録し、27.21個が0.8を超えました。一方、IMPAでは、TP3Tサンプルのうち78.31個が0.5以上のスコアを記録しましたが、0.8を超えるものはありませんでした。CDRP OODデータセットとLINCS Leave-one-outデータセットで同様の分析を行った結果、どちらのMorphDiffモデルもベースライン手法を上回り、p値が0.0001未満であることから、この手法の一般化可能性が示されました。
DeepProfiler埋め込み分析では、MorphDiff (G2I) は、摂動特有の細胞形態パターンを最も確実かつ正確に捉えることができます。製薬業界では、G2Iパターンによって生成される出力は、I2Iよりも高い摂動特異性を示します。これらのデモンストレーションは、MorphDiffが薬剤スクリーニングに持つ可能性をさらに示しています。
最後に、この実験では医薬品開発におけるMorphDiffの能力も検証しました。研究者らは、CDRP Target_MOAデータセットを選択し、MorphDiffの2つのアプリケーションモードとIMPAをベンチマークしました。
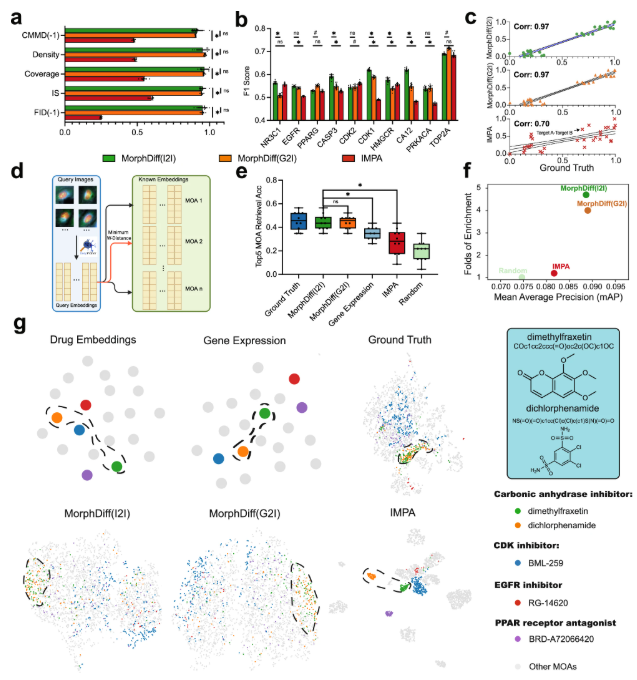
結果は、MorphDiff によって生成された摂動形態が真のベースラインと非常に一致していることを示しています。これは、標的関連の多様性を捉え、薬物の摂動と形態の複雑な関係を効果的に学習する能力を実証しました。MOA検索タスクにおいて、MorphDiffによって生成された結果の平均精度は、IMPAベースの検索よりも16.9%、遺伝子発現ベースの検索よりも8%高くなりました。さらに、実験により、MorphDiffは細胞形態に補完的な情報が含まれていることを発見し、MOAは同じだが構造が異なる薬物を識別できることが実証されました。これは、表現型医薬品の開発を加速させるのに役立ちます。
シリコンベースのシミュレーションが主流となり、下流の医薬品開発を加速
フェノタイプ創薬は、標的に基づく創薬とは異なり、生物システム全体または細胞の表現型への影響を観察することで薬剤の探索・開発を行います。このアプローチは、新たな作用機序や標的の発見、そして複雑な疾患の治療において大きな利点をもたらします。多くの研究室や研究機関がこのテーマを積極的に研究しており、計算技術と生物医学を融合させることで、フェノタイプ創薬の新たな時代を切り開いています。
論文でも述べられているように、IMPA モデルはドイツのミュンヘン工科大学とイギリスのオックスフォード大学のチームによって提案された深層生成モデルです。スタイル転送法を使用すると、細胞画像は「スタイル」(摂動/バッチ表現)と「コンテンツ」(細胞表現)に分解され、摂動に対する細胞の反応を予測し、バッチ効果を除去できます。「生成モデリングを用いた摂動に対する細胞形態学的反応の予測」と題されたこの論文は、Nature Communications にも掲載されました。
さらに、ミシガン大学のチームが発表した「MorphNetが単一細胞遺伝子発現から細胞形態を予測する」という論文では、遺伝子発現プロファイルに基づいて細胞の形態画像を描画できるMorphNetと呼ばれる計算手法を提案した。この方法では、形態学的データと分子データをペアにしてニューラル ネットワークをトレーニングし、遺伝子発現に基づいて核または細胞全体の形態を予測します。
まとめると、遺伝子や薬剤による摂動下における細胞状態の変化を観察・解析することで、表現型創薬や生物学研究を促進することが重要な課題となっています。MorphDiffに関しては、学習データ外の多数の新たな摂動に対して未知の摂動を予測することが難しいなど、依然として多くの欠点はあるものの、MorphDiffは先行技術を基盤として着実に改善を続けており、実用性、汎用性、使いやすさ、拡張性において先行技術を凌駕していることは明らかです。
2023年から2024年にかけてのAI4S分野の高品質な論文と詳細な解釈記事をワンクリックで入手⬇️









