Command Palette
Search for a command to run...
8k長の配列モデリング、タンパク質言語モデルProt42は、標的タンパク質配列のみを使用して高親和性バインダーを生成できます。
タンパク質結合剤(抗体や阻害ペプチドなど)は、疾患診断、画像分析、標的薬物送達などの重要なシナリオにおいてかけがえのない役割を果たします。従来、高度に特異的なタンパク質結合剤の開発は、ファージディスプレイや指向性進化などの実験技術に大きく依存してきました。しかし、このような方法は一般に、膨大なリソースの消費と長期にわたる研究開発サイクルという課題に直面しており、タンパク質配列の組み合わせの複雑さという固有のボトルネックによって制限されています。
人工知能の発展に伴い、タンパク質言語モデル(PLM)は、タンパク質配列と機能の関係を理解するための重要なツールとなっています。タンパク質バインダーの設計において、PLMは言語モデルの生成能力に基づき、標的タンパク質配列に基づいて、高い結合親和性を持つリガンドタンパク質や抗体フラグメントを直接設計することができます。しかし、ロングコンテキストモデリング能力と真の生成能力を両立するPLMが不足しているなどの課題にも直面しており、特に複雑な結合インターフェースや長いタンパク質バインダーの設計においては、大きな技術的ギャップが存在しています。
これを基に、アラブ首長国連邦アブダビのインセプションAI研究所と米国シリコンバレーのセレブラスシステムズの共同研究チームが、最初の PLM ファミリーである Prot42 は、タンパク質配列情報のみに依存し、3 次元構造の入力を必要としません。このモデルは、自己回帰とデコーダーのみのアーキテクチャの生成力を活用します。構造情報がなくても、高親和性タンパク質結合剤と配列特異的 DNA 結合タンパク質の生成を可能にします。Prot42 は、PEER ベンチマーク、タンパク質バインダー生成、および DNA 配列特異的バインダー生成実験で優れたパフォーマンスを発揮しました。
関連研究は「Prot42: ターゲット認識型タンパク質バインダー生成のための新しいタンパク質言語モデルファミリー」と題され、arXiv でプレプリントとして公開されています。
研究のハイライト* Prot42 は、初期の 1,024 個のアミノ酸から 8,192 個のアミノ酸まで段階的に拡張する漸進的コンテキスト拡張トレーニング戦略を採用しています。 * PEER ベンチマークテストでは、Prot42 はタンパク質機能予測、細胞内局在、相互作用モデリングなど 14 個のタスクで優れたパフォーマンスを発揮しました。 * 3D 構造に依存する AlphaProteo とは異なり、Prot42 はバインダーを生成するためにターゲットタンパク質配列のみを必要とします。

用紙のアドレス:
AIフロンティアに関するその他の論文:
https://go.hyper.ai/UuE1o
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 3つの大規模なデータセットがモデル開発とトレーニングをサポート
本研究では、いくつかの主要なデータセットを用いてモデルの学習と性能評価を行いました。これらのデータセットは、幅広いタンパク質配列情報をカバーするだけでなく、タンパク質-DNA相互作用データも含み、Prot42の豊富な学習教材となっています。
タンパク質-DNAインターフェースデータベース(PDIdb)2010
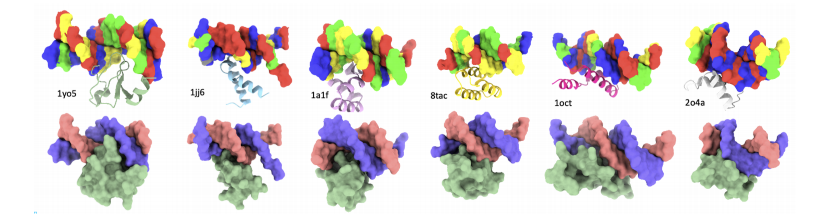
標的の DNA 配列に結合できるタンパク質を設計するために、研究者らは PDIdb 2010 データセットを使用しました。このデータセットには 922 個の固有の DNA-タンパク質ペアが含まれており、特定の DNA 配列に結合するタンパク質を生成する Prot42 の能力をトレーニングおよび評価するために使用されました。4 つの DNA タンパク質モデルを評価するために、研究者らは 1TUP、1BC8、1YO5、1L3L、2O4A、1OCT、1A1F、1JJ6 などのさまざまな PDB 構造から DNA 断片を抽出しました。
UniRef50データセット
Prot42 モデルの事前トレーニング データセットは、主に UniRef50 データベースから取得されます。データベースには 6,320 万個のアミノ酸配列が含まれており、広範囲の生物種とタンパク質機能をカバーしています。これらの配列はクラスター化され、類似度が 50% を超える配列がグループ化されるため、データの冗長性が削減され、トレーニング効率が向上します。
Prot42 をトレーニングする前に、研究チームは UniRef50 データセットを前処理しました。これらは、20 種類の標準アミノ酸の語彙を使用してラベル付けされています。アミノ酸残基を表すには Xtoken を使用します (X は、一般的でない、またはあいまいなアミノ酸残基をマークするために使用されます)。
データ前処理段階では、研究チームは、最大コンテキスト長が 1,024 トークンのシーケンスを処理し、これより長いシーケンスを除外して、最終的に 5,710 万シーケンスのフィルタリングされたデータセットを取得しました。初期の充填密度は27%です。データ利用率と計算効率を向上させるため、研究チームは可変配列長(VSL)充填戦略を採用しました。固定されたコンテキスト長内でのトークンの占有率を最大化し、最終的にデータセットを 1,620 万の埋め込みシーケンスに削減しました。充填効率は96%に達します。
STRINGデータベース
STRING データベースは、包括的なタンパク質間相互作用データベースです。実験データ、計算予測、テキストマイニングの結果を統合し、タンパク質相互作用の信頼度スコアを提供します。Prot42にタンパク質結合体を生成するよう訓練するため、研究チームはSTRINGデータベースから信頼度スコアが90%以上のタンパク質相互作用ペアを選別し、訓練データの高い信頼性を確保しました。さらに、扱いやすい単一ドメイン結合タンパク質に焦点を合わせるために、配列の長さは 250 アミノ酸に制限されました。スクリーニング後、最終データセットには 74,066 個のタンパク質間相互作用ペア、59,252 個のサンプルを含むトレーニング セット D(train)(pb)、および 14,814 個のサンプルを含む検証セット D(val)(pb) が含まれます。
モデルアーキテクチャ: 自己回帰デコーダアーキテクチャから派生した2つの主要なバリエーション
本論文で言及されているProt42は、自己回帰デコーダーアーキテクチャに基づくPLMであり、アミノ酸配列を一つずつ生成し、以前に生成されたアミノ酸を用いて次のアミノ酸を予測します。このアーキテクチャにより、モデルは配列内の長距離依存性を捉えることができます。大規模なラベルなしタンパク質配列データベースから直接豊富な表現を学習できるため、既知のタンパク質配列の膨大な数と比較的少ない割合のタンパク質配列 (<0.3%) の間のギャップを効果的に埋めることができます。同時に、モデルには複数の Transformer レイヤーが含まれており、各レイヤーには、シーケンス内の複雑なパターンをキャプチャするためのマルチヘッド自己注意メカニズムとフィードフォワード ニューラル ネットワークが含まれています。
その設計は、自然言語処理、特に LLaMA モデルにおける画期的な進歩にインスピレーションを得ています。 Prot42 は、大規模なラベルなしタンパク質配列を事前トレーニングすることで、タンパク質の進化、構造、機能に関する情報を取得し、高親和性タンパク質バインダーの生成を可能にします。
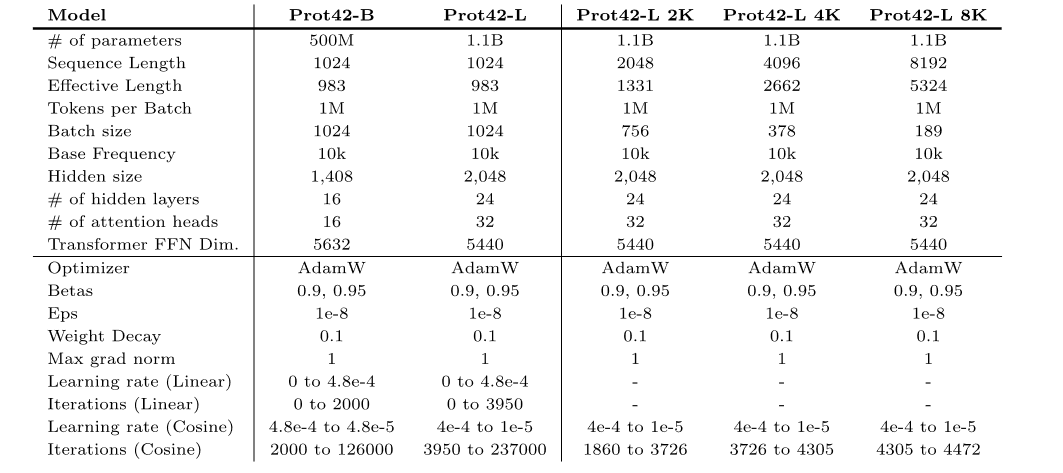
これを踏まえて、研究者らは2つのモデルバリアントを事前に訓練した。つまり、Prot42-B と Prot42-L です。
* プロト42-B:基本バージョンでは、モデルには 5 億のパラメータがあり、最大 1,024 アミノ酸のシーケンス長をサポートします。
* プロト42-L:大規模バージョンには 11 億のモデル パラメータがあり、最大 1,024 アミノ酸のシーケンス長もサポートされます。Prot42-L のコンテキスト長は、1,024 アミノ酸から 8,192 アミノ酸まで徐々に拡張されました。このプロセスでは、徐々に増加するコンテキストの長さと一定のバッチ サイズ (100 万の未充填トークン) を使用することで、長いシーケンスを処理する際のモデルの安定性と効率性が確保され、長いシーケンスと複雑なタンパク質構造を処理するモデルの能力が大幅に向上しました。Prot42-L には 24 個の隠し層も含まれており、各隠し層には 32 個のアテンション ヘッドがあります。隠れ層の次元は 2,048 です。
実験的結論:6つのタスクすべてにおいて大きな可能性が示された
下流タスクでの検証前に Prot42 モデルのパフォーマンスを評価するために、研究者は、自己回帰言語モデルを評価するためのパラメトリック複雑度 (PPL) の標準測定基準、つまり異なるコンテキスト長での Prot42 モデルのパフォーマンスを使用しました。すべてのモデルは 1,024 トークンでは比較的高い困惑度を示しますが、2,048 トークンでは約 6.5 に大幅に改善されます。結果は、ベースモデルと、より短いコンテキスト向けに微調整されたモデルが、それぞれの最大コンテキスト長にわたって同様のパフォーマンスパターンを示していることを示しています。8kコンテキストモデルのパフォーマンスは特に顕著で、中程度の長さのシーケンス(2,048~4,096トークン)ではパープレキシティがわずかに高くなりますが、最大8,192トークンのシーケンスを処理でき、最大長でも最小のパープレキシティ5.1を達成しています。4,096 トークンを超えると、困惑度曲線は下降傾向を示します。以下に示すように。
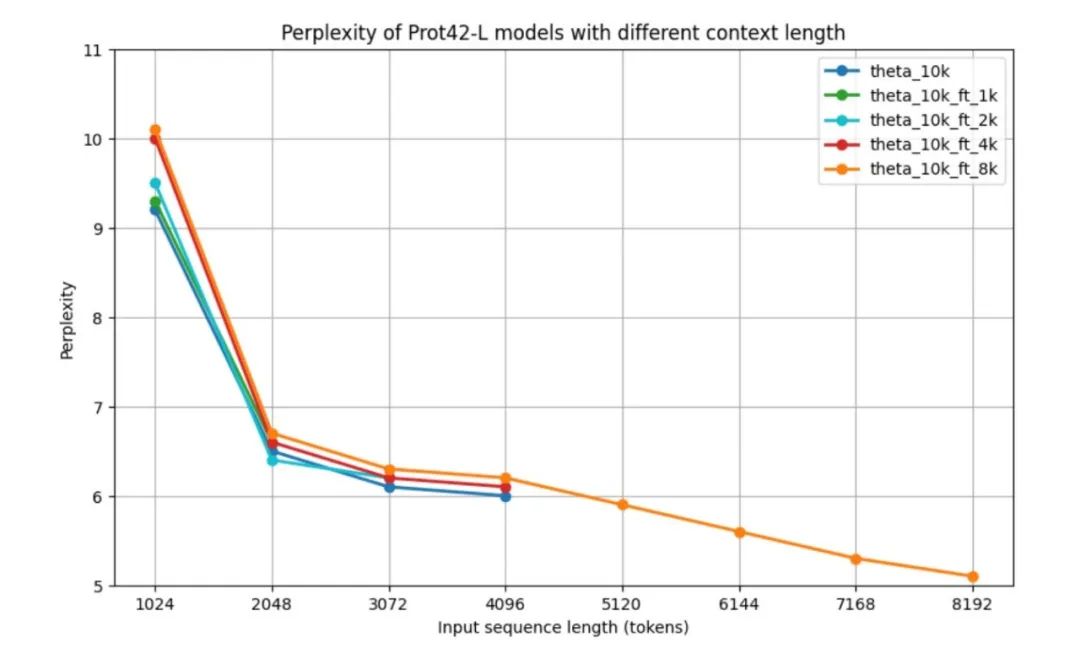
コンテキストの長さが長くなるにつれて、モデルの PPL は徐々に減少し、長いシーケンスを処理するモデルの能力が大幅に向上したことを示しています。特に、8K コンテキスト モデルは最も低い PPL を実現しており、拡張コンテキスト ウィンドウを効果的に利用して、タンパク質配列内の長距離依存性を捉えることができることを示しています。拡張されたコンテキスト ウィンドウは、タンパク質配列モデリングの分野における大きな進歩であり、複雑なタンパク質とタンパク質間相互作用をより正確に表現することを可能にし、効果的なタンパク質結合剤を生成するために重要です。
一連の厳格な実験評価を通じて、Prot42 は複数の主要タスクで優れたパフォーマンスを発揮しました。タンパク質結合剤の生成や特定の DNA 配列に結合するタンパク質の設計に効果があることが実証されています。
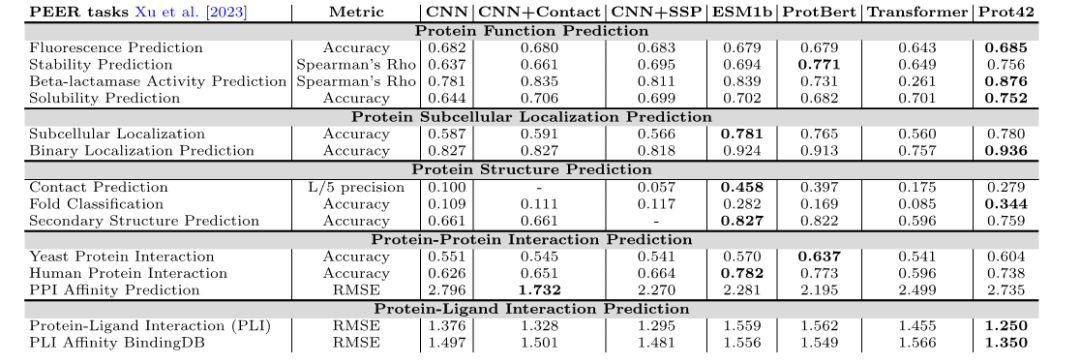
タンパク質機能予測
PEERベンチマークテストにおいて、Prot42モデルは蛍光予測、安定性予測、β-ラクタマーゼ活性予測、溶解度予測など、複数のタンパク質機能予測タスクにおいて優れた性能を示しました。既存のモデルと比較して、Prot42 は、安定性予測、溶解度予測、β-ラクタマーゼ活性予測において大きな利点を実現しました。これは、高解像度のタンパク質工学タスクにおける大きな可能性を示しています。
タンパク質の細胞内局在予測
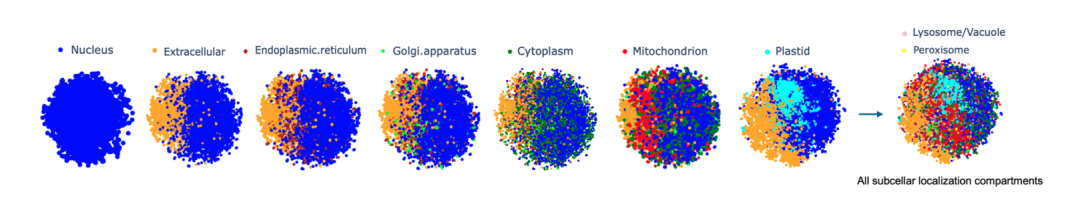
研究者らは、各タンパク質配列を32×2048の高次元ベクトルとして表現し、Prot42-Lモデルをタンパク質配列全体に埋め込み計算を行った。埋め込みとコンパートメントにおける品質の差異を直感的に評価するため、t分布確率的近傍埋め込み(t-SNE)を適用して次元数を削減し、タンパク質グループの可視化を明確にした。Prot42 はタンパク質の細胞内局在予測のタスクにおいて優れたパフォーマンスを発揮することが検証されており、その精度は既存の高度なモデルに匹敵します。研究チームは視覚的な分析を通じて、タンパク質の細胞内局在特性を捉える Prot42 モデルの有効性をさらに検証しました。
タンパク質構造予測
タンパク質構造予測タスクでは、Prot42 モデルは、接触予測、折り畳み分類、二次構造予測において優れた結果を達成しました。これらの結果は、Prot42 モデルがタンパク質構造の微妙な違いを捉えることができ、複雑な生物学的相互作用モデリングと医薬品アプリケーションを強力にサポートできることを示しています。
タンパク質間相互作用予測
タンパク質間相互作用およびタンパク質-リガンド相互作用の予測タスクでは、Prot42 モデルは高い精度と信頼性を示しました。研究者らは Chem42 を使用して化学物質埋め込みベクトルを生成し、それを ChemBert と比較しました。は、別の化学表現モデルとして知られていますが、それでもそのパフォーマンス指標は既存の手法よりも優れており、Chem42を用いた結果に近いものとなっています。特に、Chem42を用いて化学埋め込みを生成する場合、その予測結果は専門的な化学モデルの予測結果に近いものとなっています。これは、Prot42 が化学情報を組み合わせる際に優れた拡張性を持っていることを示しています。医薬品の設計を強力にサポートします。
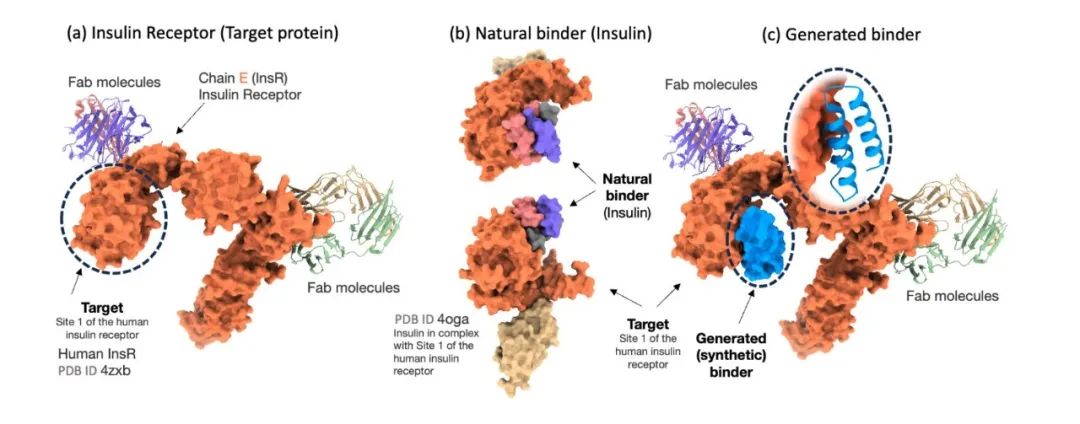
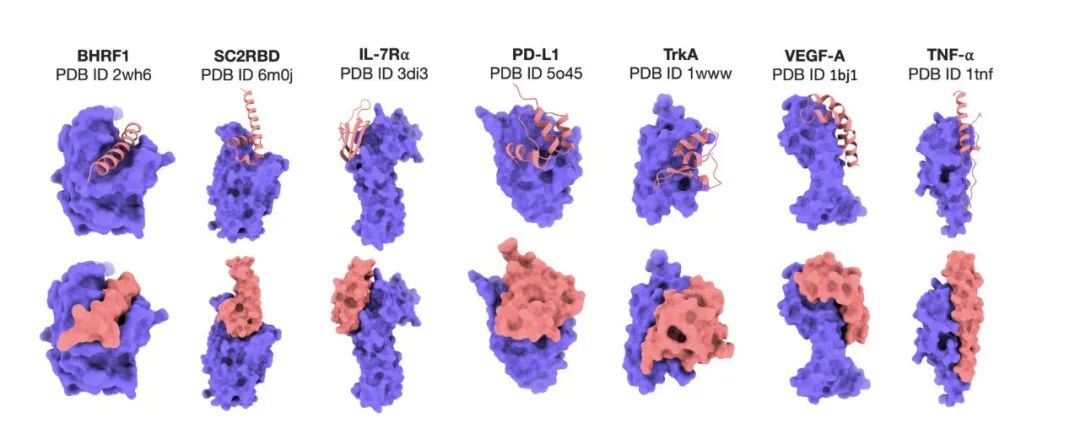
タンパク質結合剤の生成
タンパク質結合体生成におけるProt42モデルの有効性を厳密に評価するため、研究者らは、タンパク質結合体予測のために特別に設計された高度なモデルであるAlphaProteoと比較した。実験結果から、Prot42 モデルは、複数の治療関連ターゲットに対して強い予測親和性を持つ結合剤を生成しました。特にIL-7Rα、PD-L1、TrkA、VEGF-Aなどの標的に対して、Prot42 モデルは AlphaProteo モデルよりも大幅に優れたパフォーマンスを発揮しました。これらの結果は、下の図に示すように、Prot42 モデルがタンパク質結合剤の生成に大きな利点があることを示しています。
DNA配列特異的バインダー生成
DNA配列特異的な結合剤生成実験においても、Prot42は顕著な結果を達成した。実験結果から、Prot42 モデルは、遺伝子埋め込みとタンパク質埋め込みのマルチモーダル戦略を組み合わせることで、標的の DNA 配列に特異的に結合し、高い親和性を示すタンパク質を生成することができます。DeepPBSモデルによって評価された結合特異性は高かった。これらの結果は、Prot42モデルがDNA配列特異的な結合剤の生成においても大きな可能性を秘めており、遺伝子制御およびゲノム編集アプリケーションのための新たなツールとなることを示唆している。
タンパク質設計における人工知能のブレークスルーとイノベーション
バイオテクノロジーと人工知能の深い融合により、タンパク質設計のフロンティア分野は革命的な変化を遂げています。生命活動の中核を担うタンパク質の構造と機能の解析は、科学研究における難題であり続けてきましたが、AI技術の介入により、この複雑なパズルの解決が加速され、新薬開発や酵素工学の転換といった新たなシナリオへの道が開かれています。
近年、AI技術は再び飛躍的な進歩を遂げ、生成AIを中心とした新たな技術がタンパク質設計を「創世」の段階へと押し進めています。
ミズーリ大学の Xu Dong 教授のチームは、マルチビューコントラスト学習を導入することでタンパク質配列と 3D 構造情報を統一された潜在空間に配置する構造認識型タンパク質言語認識型モデル (S-PLM) を提案しました。Swin Transformer を使用して、AlphaFold によって予測された構造情報を処理し、それを ESM2 ベースのシーケンス埋め込みと融合して、構造を認識した PLM を作成します。論文「S-PLM:配列と構造の対照学習による構造を考慮したタンパク質言語モデル」がAdvanced Science誌に掲載されました。S-PLMは、タンパク質配列とその3次元構造を統合された潜在空間に整列させることで、構造情報を配列表現に巧みに組み込みます。また、効率的な微調整戦略も探求しており、様々なタンパク質予測タスクにおいて優れた性能を発揮することを可能にしています。これは、タンパク質の構造と機能の予測分野における重要な進歩です。
用紙のアドレス:
https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202404212
さらに、清華大学の研究チームをはじめとする共同研究チームは、1000億パラメータまで拡張可能な統合事前学習フレームワークおよび基本モデルである統合タンパク質言語モデルxTrimoPGLMを提案しました。このモデルは、双方向アテンションと自己回帰目標のバックボーンとして一般言語モデル(GLM)を活用しており、従来のエンコーダのみ、あるいは因果デコードのみのPLMとは異なります。本研究は、超大規模PLMの統合理解および生成事前学習を探求し、タンパク質配列設計の新たな可能性をさらに明らかにし、より幅広いタンパク質関連アプリケーションのさらなる発展を促進しました。この研究は、「xTrimoPGLM:タンパク質言語の解読のための統合1000億パラメータ事前学習済みトランスフォーマー」というタイトルでNature誌のサブジャーナルに掲載されました。
用紙のアドレス:
https://www.nature.com/articles/s41592-025-02636-z
Prot42の画期的な進歩は、単なる技術進歩にとどまらず、「データ駆動型+AI設計」モデルがライフサイエンス分野において着実に成熟していることを反映しています。今後、研究チームはProt42によって生成されたバインダーを実験的に検証し、計算による評価を実際の機能試験で補完する予定です。これにより、モデルの実用性を強化し、予測精度を向上させ、AI駆動型配列生成と実験バイオテクノロジーのギャップを埋めることができるでしょう。
参考文献:
1.https://arxiv.org/abs/2504.04453
2.https://mp.weixin.qq.com/s/SDUsXpAc8mONsQPkUx4cvA
3.https://mp.weixin.qq.com/s/x7_Wnws35Qzf3J0kBapBGQ
4.https://mp.weixin.qq.com/s/SDUsXpAc8mONsQPkUx4cvA








