Command Palette
Search for a command to run...
AAAI 2025に選出されました!清華大学/UCLは、親和性予測とSOTAの刷新を組み合わせたタンパク質-RNA言語モデル融合ソリューションの先駆者です。
アルツハイマー病、パーキンソン病、てんかん...これらの「悪名高いほど恐ろしい」神経変性疾患は、高齢者の健康を目に見えない形で殺すものであり、これらの疾患の発生は、多くの場合、タンパク質とRNAの異常な結合に関連しています。
生物医学分野では、タンパク質と RNA の結合を研究することは、遺伝子発現の調節、RNA の処理とスプライシング、翻訳の調節、細胞のストレス応答など、複数の生物学的プロセスにおいて中心的な役割を果たすため、極めて重要です。タンパク質とRNAの結合メカニズムを理解することは、複雑な遺伝子制御プロセスを明らかにし、疾患の遺伝的基礎を分析するための鍵となります。同時に、タンパク質とRNAの相互作用はRNA標的療法にも重要な応用があり、がん、遺伝性疾患、ウイルス性疾患の治療に新たな方向性をもたらします。
最近、人工知能に関する国際会議のトップである第39回AAAI人工知能会議(AAAI 2025)で発表された選ばれた成果の中で、清華大学、ロンドン大学ユニバーシティ・カレッジ、モナシュ大学、北京郵電大学の合同チーム提案するCoPRA モデルは業界で広く注目を集めており、経口セグメントに選択されました。
これは、タンパク質-RNA 結合親和性を予測するための複雑な構造アーキテクチャを通じて、タンパク質言語モデル (PLM) と RNA 言語モデル (RLM) を組み合わせる初の試みです。CoPRA のパフォーマンスをテストするために、研究者らは複数のデータ ソースから最大のタンパク質-RNA 結合親和性データセットをコンパイルし、3 つのデータセットでモデルのパフォーマンスを評価しました。結果は、CoPRA が複数のデータセットで最先端のパフォーマンスを達成したことを示しました。
関連する結果は「CoPRA: タンパク質-RNA 結合親和性予測のための複雑な構造を持つクロスドメイン事前トレーニング済みシーケンス モデルのブリッジング」と題され、arXiv でプレプリントとして公開されています。

用紙のアドレス:
https://arxiv.org/abs/2409.03773
CoPRA倉庫住所:
https://github.com/hanrthu/CoPRA
オープンソース プロジェクト「awesome-ai4s」は、200 を超える AI4S 論文の解釈をまとめ、膨大なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
生物医学研究はタンパク質とRNAの相互作用を進歩させ続けている
過去数年にわたり、生物医学分野の研究者はタンパク質とRNAの相互作用の研究を止めず、大きな進歩を遂げてきました。
CLIP 実験技術は、RNA 研究において最も重要な技術の 1 つです。トランスクリプトーム全体における RNA 結合タンパク質 (RBP) の結合マップを解析することができ、RBP の機能とその制御メカニズムを体系的に理解するための基礎となります。しかし、CLIP 実験は時間がかかり、労力もかかる上、一度に特定の細胞環境内の特定の RBP の RNA 結合部位しか提供できず、実験材料に対する要件も高くなります。しかし、タンパク質とRNAの結合は細胞環境の変化によって大きく変化する可能性がありますが、RNA上のタンパク質の調節を研究するには、同じ細胞環境における結合情報が必要です。
異なる細胞環境におけるRBP結合の動的変化の問題を解決するために、2021年2月、清華大学構造生物学先端イノベーションセンターの張強鋒氏の研究グループは、「生体内RNA構造を用いたディープラーニングによる動的な細胞内タンパク質-RNA相互作用の予測」と題する研究結果をCell Research誌に発表した。本研究では、icSHAPE実験を用いて7種類の一般的な細胞のRNA二次構造マップを分析し、実験から得られた細胞内RNA構造と対応する細胞環境のRBP結合情報を統合する人工知能アルゴリズムを開発し、細胞内RNA構造情報に基づいて細胞内RBPの動的結合を予測する新しい方法PrismNetを確立しました。
タンパク質と RNA の結合親和性を予測するために、業界ではいくつかの計算方法が提案されてきました。シーケンスベースおよび構造ベースの方法が含まれます。シーケンスベースの方法では、異なるシーケンス エンコーダーを使用してタンパク質シーケンスと RNA シーケンスを個別に処理し、その後それらの間の相互作用をモデル化します。しかし、結合親和性は主に結合インターフェースの構造によって決まるため、これらのアプローチのパフォーマンスは制限されることがよくあります。最近提案された他の方法は、エネルギーや接触距離など、結合界面の構造的特徴を抽出することに重点を置いています。研究者らは、抽出されたこれらの特徴に基づいて、親和性予測のための構造ベースの機械学習アプローチを開発しました。ただし、データセットのサイズが制限されているため、これらの方法では新しいサンプルに対する一般化能力が制限されており、特徴エンジニアリングに大きく依存します。
人工知能技術の台頭により、多くのタンパク質言語モデル (PLM) と RNA 言語モデル (RLM) が開発され、さまざまな下流タスクで優れたパフォーマンスと一般化機能が実証されています。同時に、タンパク質/RNAの3次元構造はそれらの機能を理解する上で重要であるため、構造情報を言語モデルに組み込むことも新たなトレンドとなっています。
たとえば、ミズーリ大学、ケンタッキー大学、アラバマ大学のチームは、多視点の比較学習技術を使用して、主要なタンパク質構造情報をタンパク質言語モデルに組み込みました。この概念に基づいて、チームはタンパク質の 3D 構造情報を認識できるタンパク質言語モデルである S-PLM を開発しました。 S-PLM は、複数のタンパク質予測タスクで優れたパフォーマンスを発揮します。軽量チューニング ツールを使用してトレーニングした後、タンパク質機能予測、酵素反応クラス予測、二次構造予測などのタスクにおける S-PLM のパフォーマンスは、現在の最先端の方法に達するか、それを上回ります。関連研究は、「S-PLM: 配列と構造の対照学習による構造認識型タンパク質言語モデル」というタイトルでbioRxivに掲載されました。
しかし、現在の業界の研究では、インタラクティブなタスクにおいて構造情報によって駆動される生物学的言語モデルの大きな可能性が実証されているものの、異なる生物学分野の事前トレーニング済みモデルを組み合わせる研究はまだまれです。清華大学、ロンドン大学ユニバーシティ・カレッジ、モナッシュ大学、北京郵電大学が共同で提案したCoPRAでは、タンパク質とRNAの言語モデルを複雑な構造情報と組み合わせて、タンパク質とRNAの結合親和性を予測する試みが初めて行われました。
CoPRAを構築するための軽量Co-Formerモデルの設計
全体として、CoPRA モデルの構築プロセスを次の図に示します。
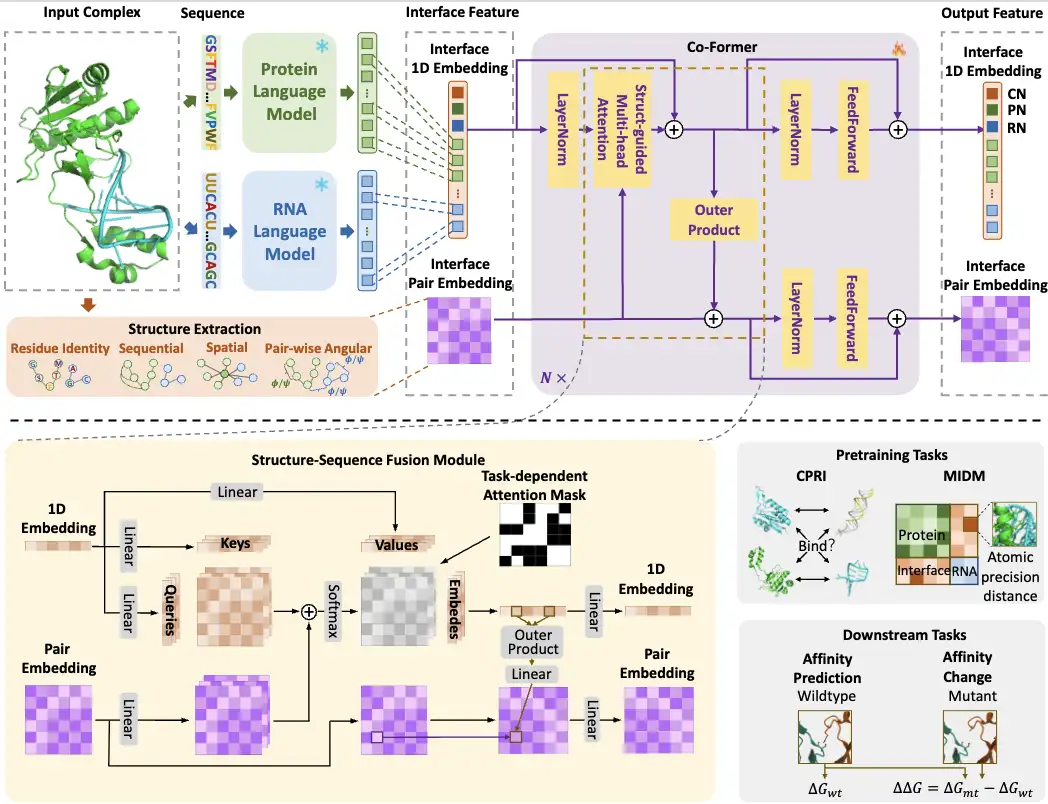
初め、研究者らは、タンパク質配列とRNA配列をそれぞれPLMとRLMに入力し、その後、2つの言語モデルの出力から相互作用インターフェースでの埋め込みを、その後のクロスモーダル学習のための配列埋め込みとして選択しました。同時に、インタラクションインターフェースから構造情報(インターフェース特徴)をペア埋め込みとして抽出します。
それから、研究者らは、2 つの言語モデルからのインターフェース シーケンス埋め込みと複雑な構造情報を組み合わせて構造シーケンス融合モジュールを形成する軽量の Co-Former モデルを設計しました。具体的には、Co-Former は、構造ガイド付きマルチヘッド自己注意および外積モジュールを介して 1D およびペアワイズ埋め込みを融合し、タスク依存の注意マスクを適用します。 Co-Former の出力特殊ノードとペアになった埋め込みは、2 つの事前トレーニング タスクと 2 つの下流のアフィニティ タスクを含むさまざまなタスクに応じて使用されます。
研究者らは、Co-Former のデュアルレンジ事前トレーニング戦略も提案しました。原子レベルの精度で学習した、粗粒度コントラスト相互作用分類 (CPRI) と細粒度インターフェース距離予測 (MIDM) をモデル化します。
CoPRAと他のモデルのパフォーマンスを評価するために、研究者は、統一された注釈標準データセットの不足に対処する必要があります。そこで、研究者らは、PDBbind、PRBABv2、ProNAB という 3 つの公開データセットからサンプルを収集し、最大のタンパク質-RNA 結合親和性データセット PRA310 をコンパイルして、PRA310 および PRA201 データセットでタンパク質-RNA 結合親和性を予測するモデルの能力を評価しました。
*PRA201データセット:PRA310のサブセット。各複合体には1つのタンパク質鎖と1つのRNA鎖のみが含まれており、より厳しい長さの制約があります。
CoPRAはタンパク質-RNA結合親和性の予測に最も優れている
下の表に示すように、CoPRA の de novo トレーニング済みバージョンは、PRA310 データセットで最高のパフォーマンスを実現します。さらに、LM 埋め込みを入力として使用する方法のほとんどは他の方法よりも優れており、親和性予測のために事前トレーニング済みの単峰性 LM を組み合わせる大きな可能性を示しています。
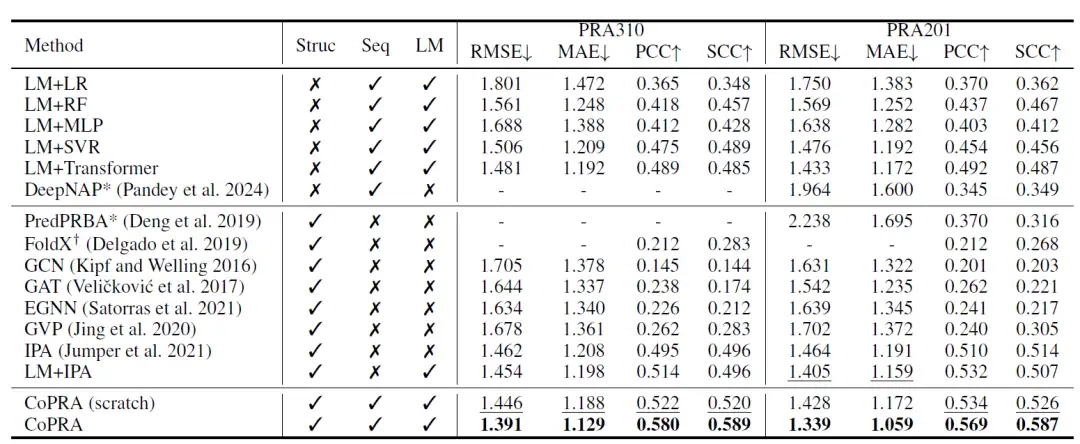
その後、研究者らはコンパイルされた教師なしデータセット PRI30k を使用してモデルを事前トレーニングし、両方のデータセットでの全体的なパフォーマンスを大幅に向上させました。 PRA310 データセットでは、CoPRA は RMSE 1.391、MAE 1.129、PCC 0.580、SCC 0.589 を達成しており、これは 2 番目に優れたモデルである CoPRA (最初からトレーニング) よりもはるかに優れています。 PredPRBA と DeepNAP は、タンパク質と RNA のペアの親和性の予測をサポートします。研究者らは、PRA201 データセットでこれらの方法のパフォーマンスを比較し、PRA201 の少なくとも 100 個のサンプルがトレーニング セットに含まれていたにもかかわらず、PRA201 でのパフォーマンスは報告された結果よりも大幅に低いことを示しました。これは、これらの方法の一般化能力が低いことを示しています。
CoPRAは、変異が結合親和性に与える影響を予測する能力に優れており、一般化能力に優れている。
モデルの親和性に関する詳細な理解をさらに評価するために、研究者らはモデルを再設計し、タンパク質の単一点変異がタンパク質-RNA複合体に与える影響を予測するようにした。研究者らは、タンパク質変異効果予測に関する関連研究を参考に、各複合体レベルでの指標を平均化し、PRI30kで事前トレーニングし、PRA310でチューニングした後、CoPRAのゼロショットおよび微調整のパフォーマンスを評価しました。
下の表に示すように、mCSM のクロス検証セットを使用して微調整した後、本研究で提案されたモデルは、RMSE 0.957、MAE 0.833、PCC 0.550、SCC 0.570 となり、4 つの指標すべてで他のモデルを上回りました。
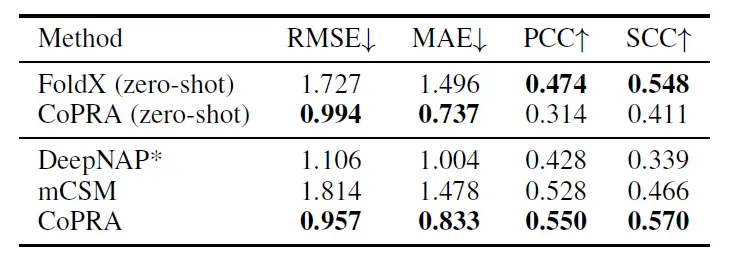
この優れたパフォーマンスは、変異した複雑な構造が見られなかったにもかかわらず、二重の事前トレーニング目標から生まれたものであり、さまざまな親和性関連のタスクにおける CoPRA の一般化能力を示しています。
マルチモーダルタンパク質言語モデルにおける画期的な進歩
上記で紹介した研究アイデアの本質は、タンパク質やRNAなどの複数の生物学的モダリティと複雑な構造情報を組み合わせること、いわゆるマルチモーダル学習です。簡単に言えば、マルチモーダル学習とは、ディープラーニングのフレームワークの下でさまざまな種類のデータを 1 つのモデルに統合するプロセスです。
過去数年間、大規模言語モデルの急速な発展に伴い、研究者はそれをタンパク質科学の分野に適用し、タンパク質の機能、構造、特性を正確に理解し予測しようとし始めました。しかし、これまでのタンパク質指向の大規模言語モデルは、主にアミノ酸配列をテキストとして処理しており、タンパク質の豊富な構造情報を十分に活用できていませんでした。今日、マルチモーダル学習の進歩により、より多くの関連研究に新たなアイデアがもたらされています。
例えば、医薬品の研究開発の分野では、タンパク質とリガンド間の結合親和性を正確かつ効果的に予測することが、医薬品のスクリーニングと最適化に不可欠です。しかし、これまでの研究では、タンパク質-リガンド相互作用における分子表面情報の重要な役割は考慮されていませんでした。これを踏まえて、厦門大学の研究者らは、新しいマルチモーダル特徴抽出(MFE)フレームワークを提案した。このフレームワークは、タンパク質表面、3D 構造、配列の情報を初めて組み合わせ、クロスアテンション メカニズムを使用して異なるモダリティ間の特徴を調整します。実験結果によると、この方法はタンパク質-リガンド結合親和性の予測において最先端の性能を達成しています。関連研究は、2024年6月に「表面ベースのマルチモーダルタンパク質-リガンド結合親和性予測」というタイトルでバイオインフォマティクスに掲載されました。
2024年12月、華東師範大学などの研究チームが革新的なソリューション「EvoLLama」を提案しました。これは、タンパク質構造エンコーダー、シーケンスエンコーダー、大規模言語モデルをマルチモーダルに統合するフレームワークです。ゼロショット設定では、EvoLLama は強力な一般化機能を発揮し、他の微調整されたベースライン モデルのパフォーマンスを 1% ~ 8% 向上させ、現在の最先端の教師あり微調整モデルの平均パフォーマンスを 6% 上回ります。関連する研究結果は、「EvoLlama: マルチモーダル構造および配列表現による LLM のタンパク質理解の向上」というタイトルで arXiv にプレプリントとして公開されています。
もちろん、マルチモーダル学習は利用可能な研究オプションの 1 つにすぎません。将来的には、より多くの機械学習手法を使用してタンパク質の表面を研究することで、生物学者はタンパク質が他の生物学的分子とどのように相互作用するかをより深く理解し、新薬の開発に役立てることができます。
参考文献:
1.https://arxiv.org/abs/2409.03773
2.https://www.frcbs.tsinghua.edu.cn/index.php?c=show&id=873
3.https://www.sohu.com/a/846589543_121124715








