Command Palette
Search for a command to run...
Tutoriel En Ligne : RFdiffusion2 Permet La Génération De Protéines Au Niveau Atomique À Partir De Réactions Chimiques, Atteignant Un Taux De Réussite De 100% Dans 41 Cas

Auparavant, le modèle de conception générative de protéines par diffusion RF générait principalement des structures protéiques avec des sites fixes précis en décrivant des sites actifs idéaux. Cependant, cette approche présentait deux limitations majeures difficiles à surmonter :
* La géométrie du site actif ne peut être spécifiée qu'au niveau du résidu, et les chercheurs doivent déterminer les positions possibles du squelette des résidus en énumérant les rotamères de la chaîne latérale ;
* Les positions des résidus catalytiques doivent être prédéterminées dans la séquence, ce qui limite considérablement l'espace des solutions échantillonnées.
En réponse aux défis connexes, en avril 2025, l'Institute for Protein Design de l'Université de Washington a publié un nouveau modèle génératif, Rosetta Fold diffusion 2 (RFdiffusion2).Cela a permis d'obtenir un chemin optimisé pour générer des squelettes de protéines avec des sites actifs personnalisés basés sur des descriptions de réactions chimiques simples, brisant ainsi le goulot d'étranglement technique de longue date dans la conception des catalyseurs.Il fournit un support technique solide pour une variété d’applications, y compris la dégradation du plastique.
RFdiffusion2 utilise une nouvelle approche générative profonde qui permet la conception de protéines à partir de descriptions indépendantes de la séquence des positions des groupes fonctionnels, sans nécessiter de génération d'isomères par rotation inverse. Comparé aux modèles précédents, l'optimisation de RFdiffusion2 se concentre sur trois axes clés :
* Élimination de la dépendance à l'égard du dénombrement des résidus et de l'indexation des séquences, et introduction de techniques de correspondance de flux et de centrage stochastique, permettant au modèle de générer des squelettes protéiques directement au niveau atomique ;
* Prend en charge la génération de sites actifs au niveau atomique sans index, peut traiter les informations sur les ligands et échantillonner automatiquement les conformations des ligands pendant le processus de génération, améliorant ainsi la flexibilité de conception ;
* Utilise une nouvelle référence AME, une référence de conception de protéines informatiques couvrant 41 sites actifs différents (la génération précédente n'en couvrait que 16).
Lors d'expériences ultérieures, l'équipe de recherche a conçu des protéines autour de trois sites catalytiques différents. Les résultats expérimentaux ont montré queRFdiffusion2 montre des améliorations de performances significatives par rapport aux méthodes précédentes, générant avec succès des squelettes protéiques qui répondent aux contraintes dans 41 cas, avec un taux de réussite de 100% ;Dans les mêmes conditions, le taux de réussite de RFdiffusion1 n'était que d'environ 39%. On peut dire que le lancement de RFdiffusion2 marque une avancée majeure dans la conception de protéines pilotée par l'IA.
RFdiffusion2 : un outil de conception de protéines est désormais disponible sur le site web d'HyperAI (hyper.ai) dans la section Tutoriels. Découvrez le tout dernier outil de génération de protéines en un seul clic.
Lien du tutoriel :
https://hyper.ai/cn/tutorials/44096
Essai de démonstration
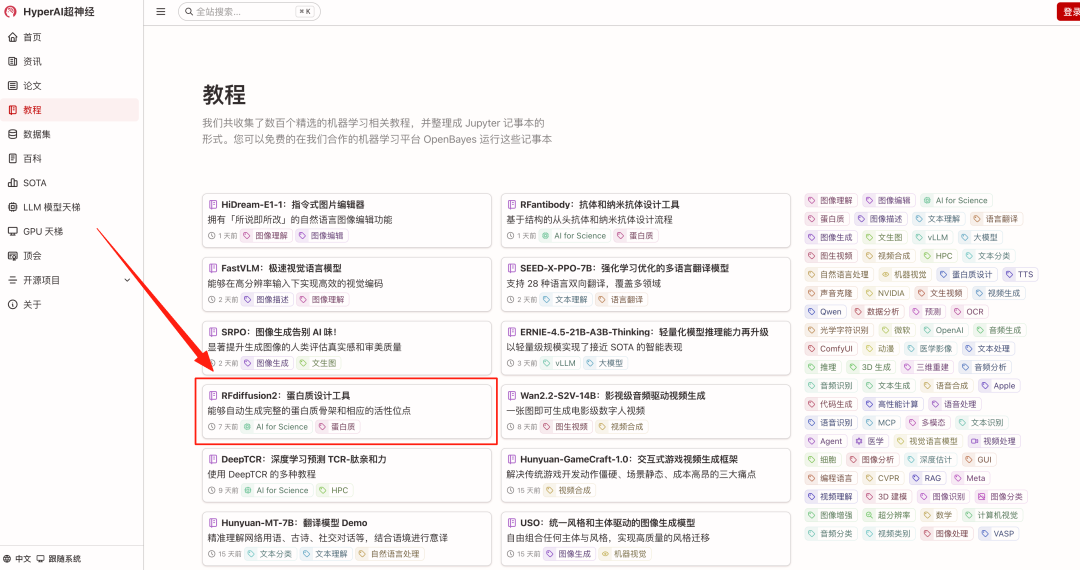
1. Saisissez l'URL hyper.ai dans votre navigateur. Une fois sur la page d'accueil, cliquez sur la page « Tutoriels », sélectionnez RFdiffusion2 : Outil de conception de protéines, puis cliquez sur « Exécuter ce tutoriel en ligne ».
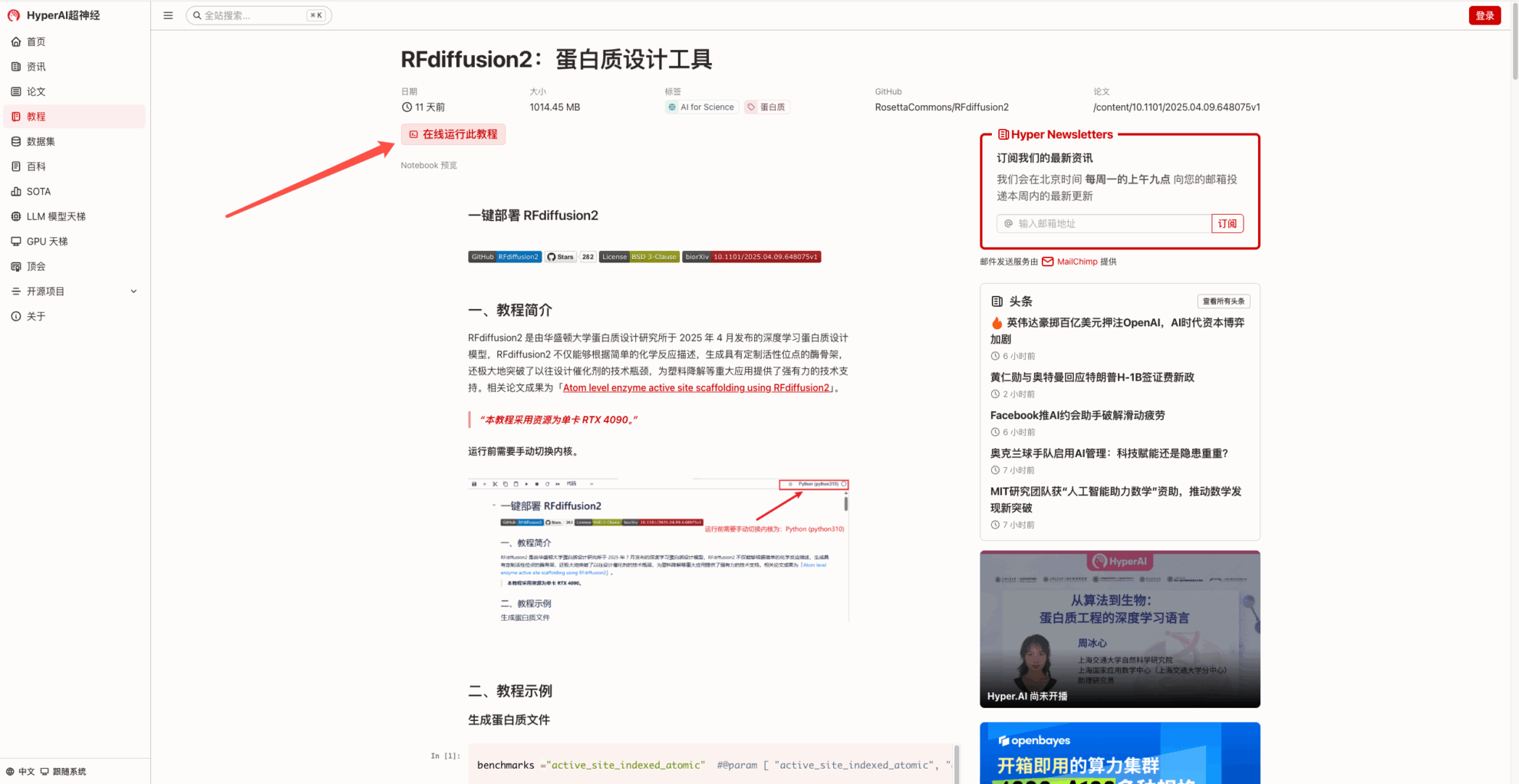
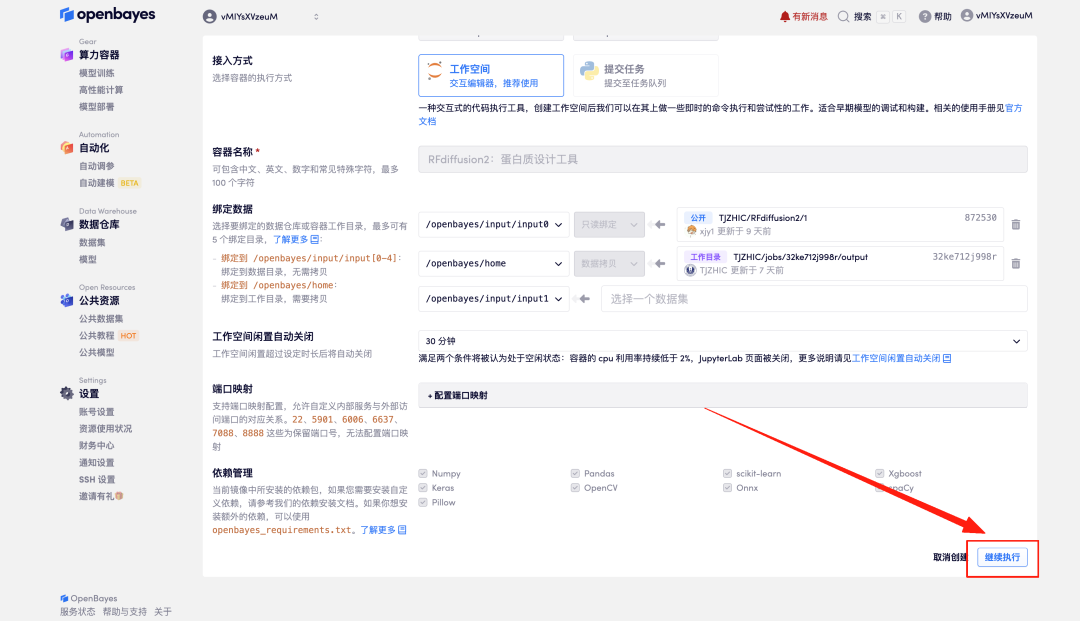
2. Une fois la page affichée, cliquez sur « Cloner » dans le coin supérieur droit pour cloner le didacticiel dans votre propre conteneur.
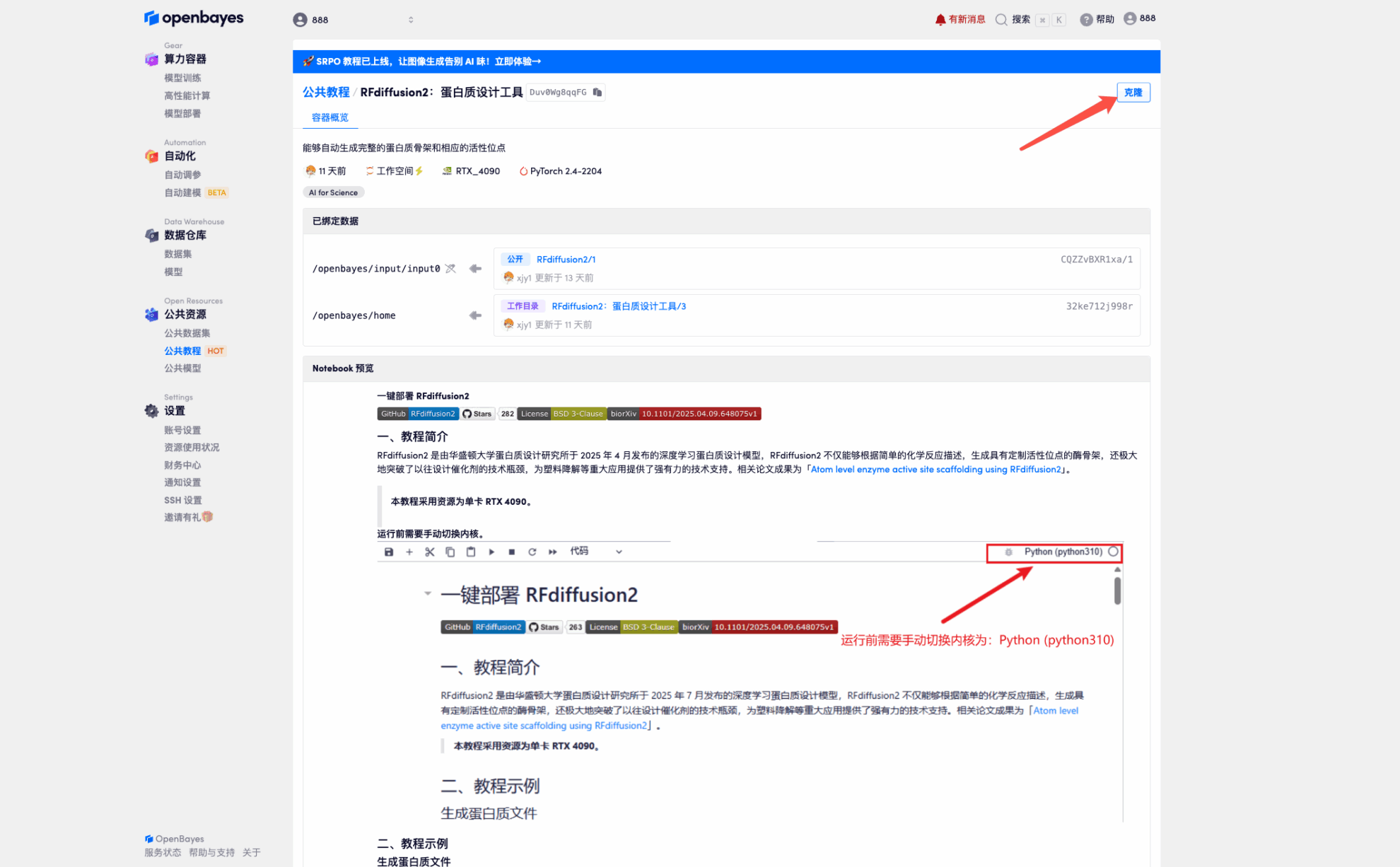
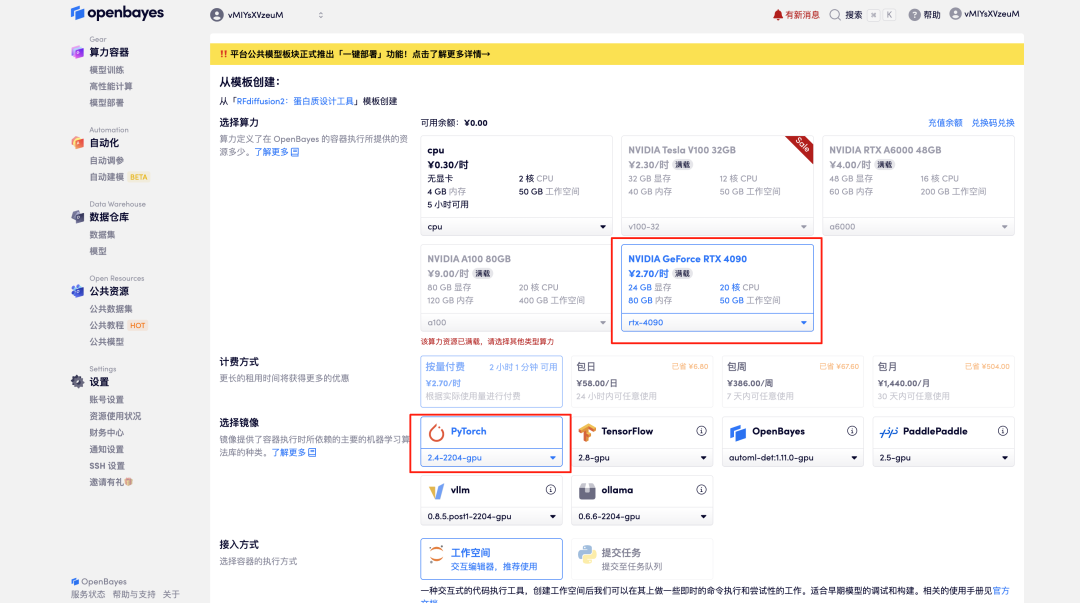
3. Sélectionnez les images NVIDIA GeForce RTX 4090 et PyTorch, puis cliquez sur Continuer. La plateforme OpenBayes propose quatre méthodes de facturation. Vous pouvez choisir « payer à l'utilisation » ou « quotidien/hebdomadaire/mensuel » selon vos besoins. Les nouveaux utilisateurs peuvent s'inscrire en utilisant le lien d'invitation ci-dessous pour obtenir 4 heures de RTX 4090 + 5 heures de temps CPU gratuit !
Lien d'invitation exclusif HyperAI (copier et ouvrir dans le navigateur) :
https://openbayes.com/console/signup?r=Ada0322_NR0n
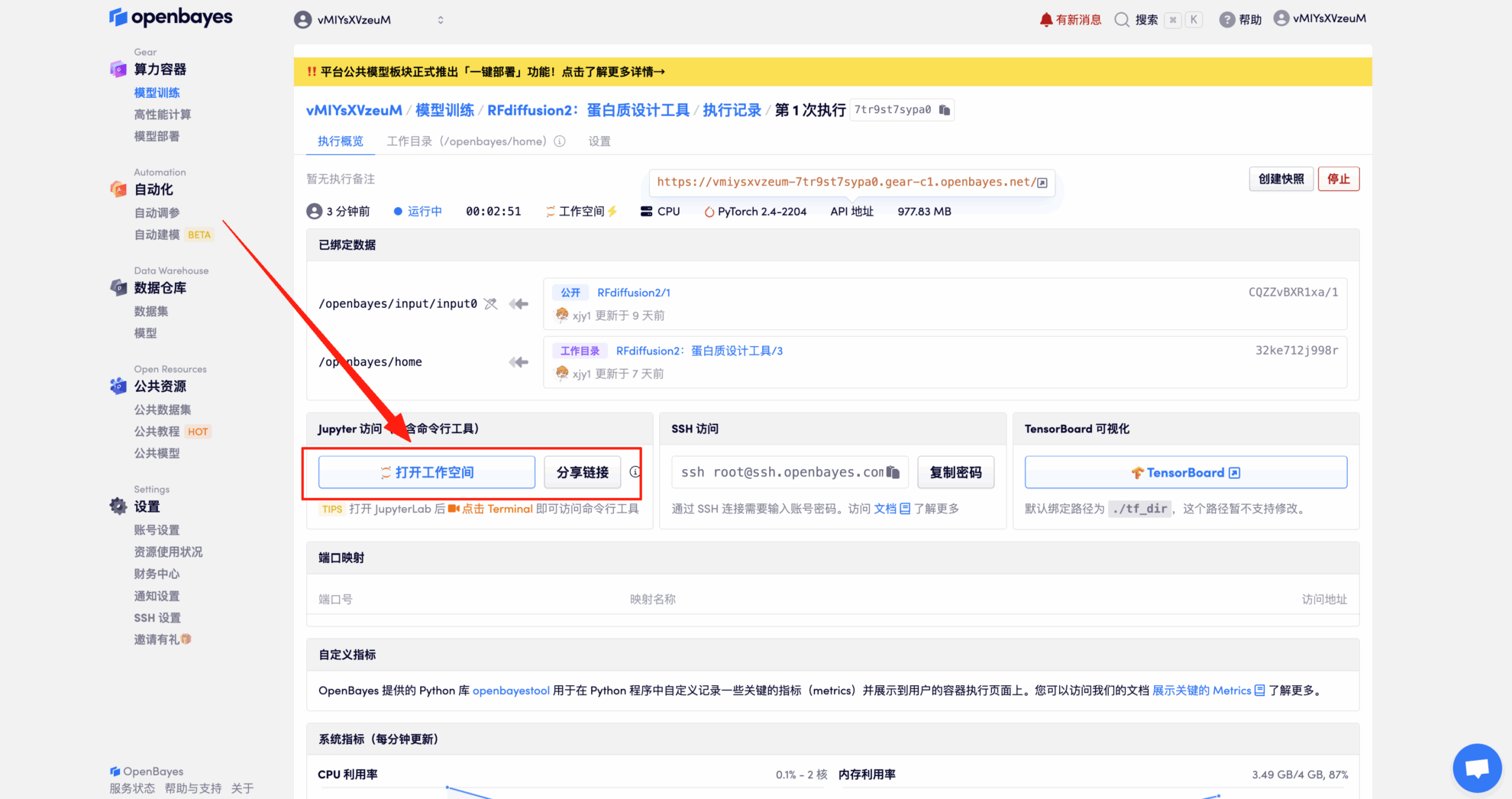
4. Attendez que les ressources soient allouées. Le premier clonage prendra environ 2 minutes. Lorsque le statut passe à « En cours d'exécution », cliquez sur la flèche de raccourci à côté de « Ouvrir l'espace de travail » pour accéder à la page de démonstration.
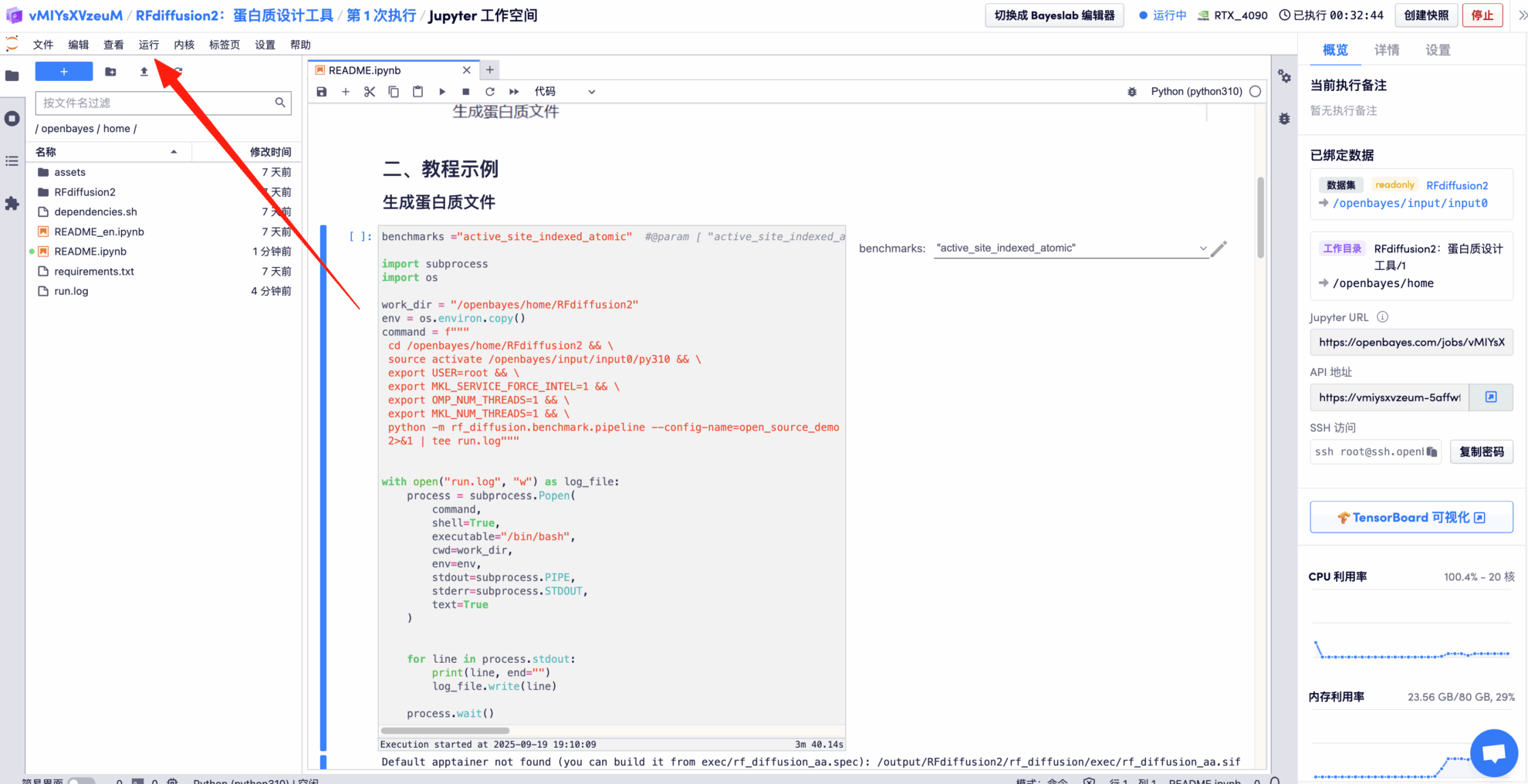
Démonstration d'effet
Après avoir accédé à la page d'exécution de démonstration, entrez les repères et cliquez sur Exécuter pour obtenir les résultats de sortie.
Ici, nous prenons « active_site_indexed_atomic » comme exemple pour montrer
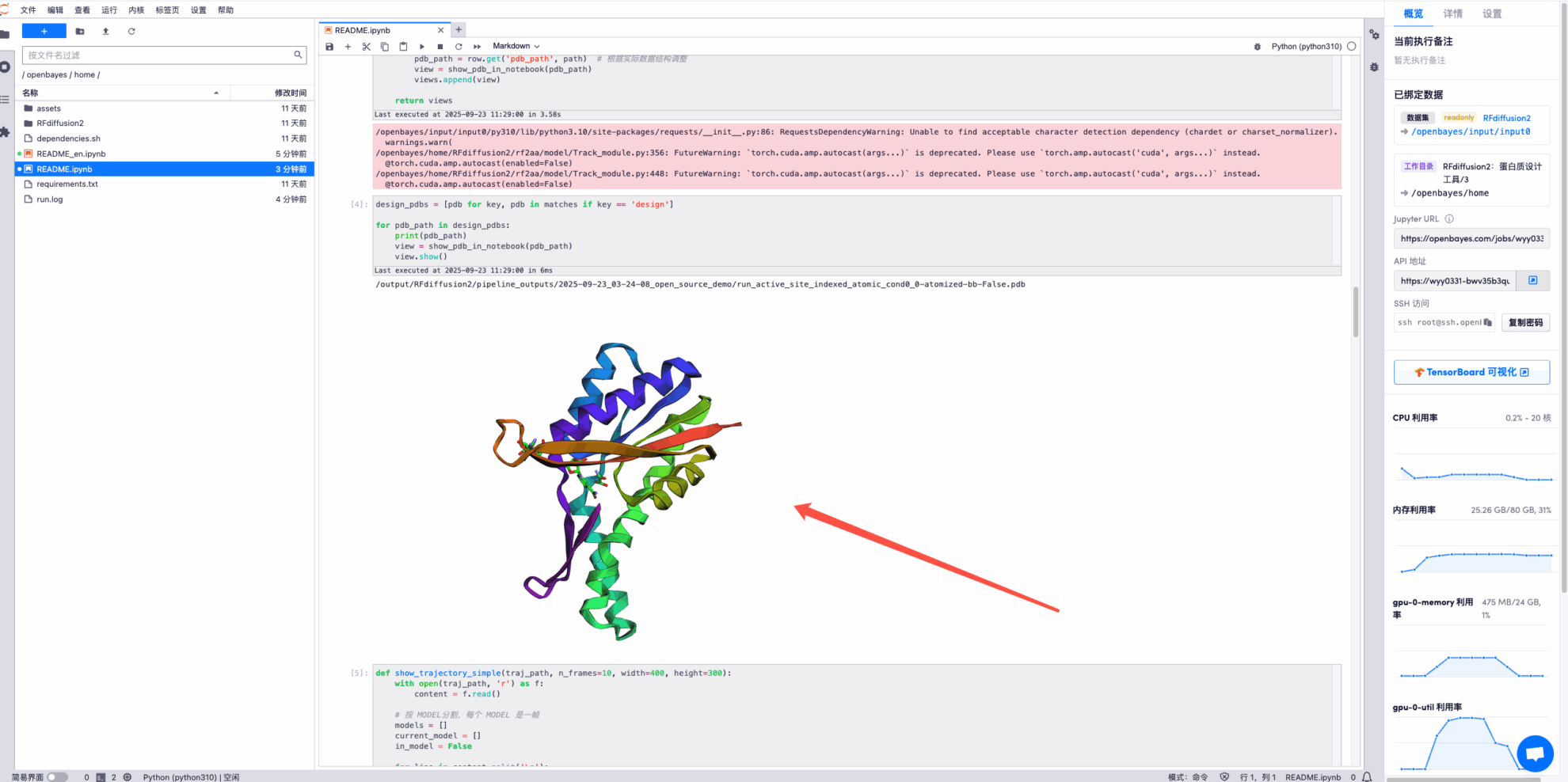
* Afficher le fichier final de conception de la protéine
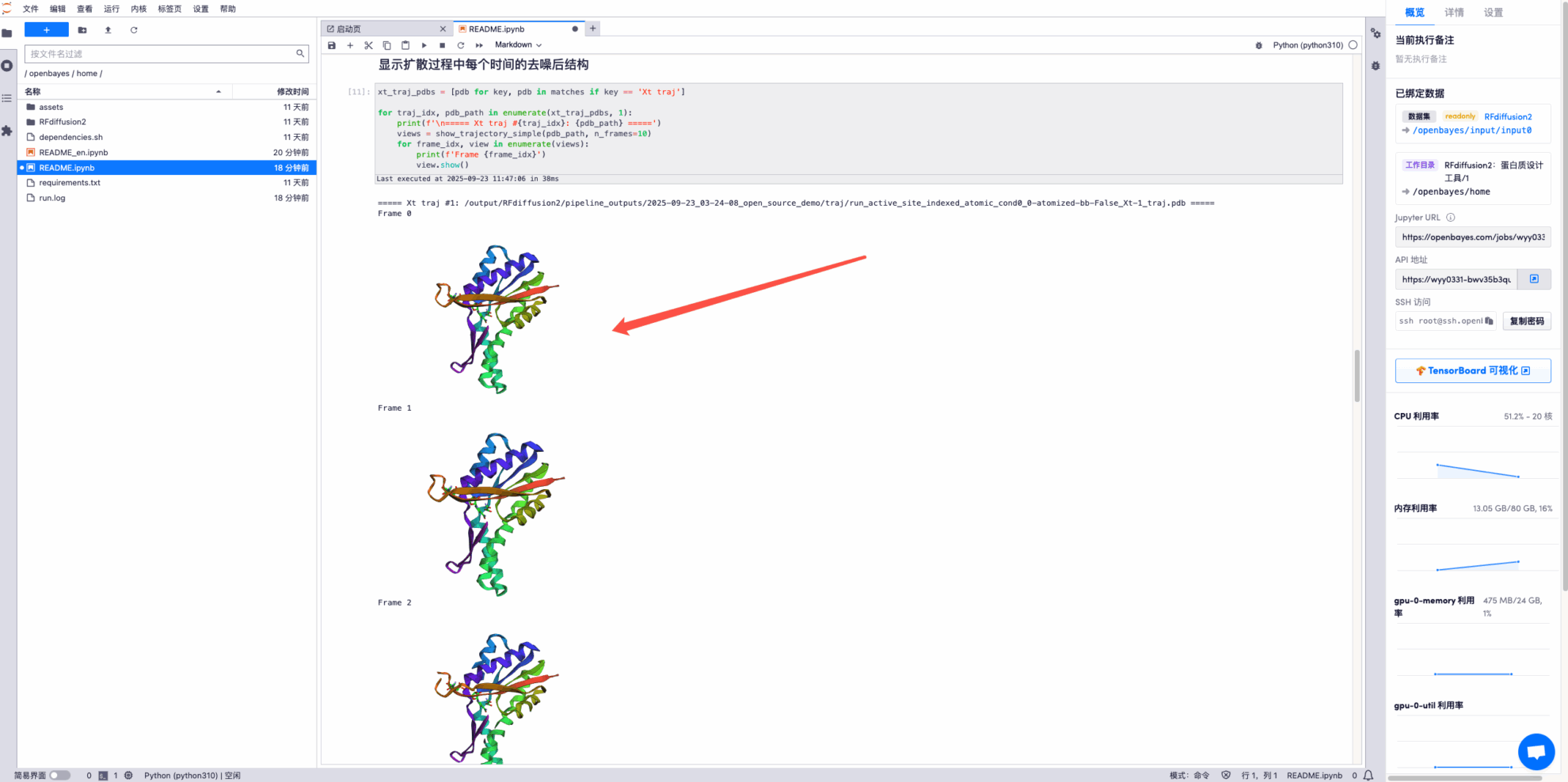
* Afficher la structure débruitée à chaque instant du processus de diffusion. Voici quelques exemples :
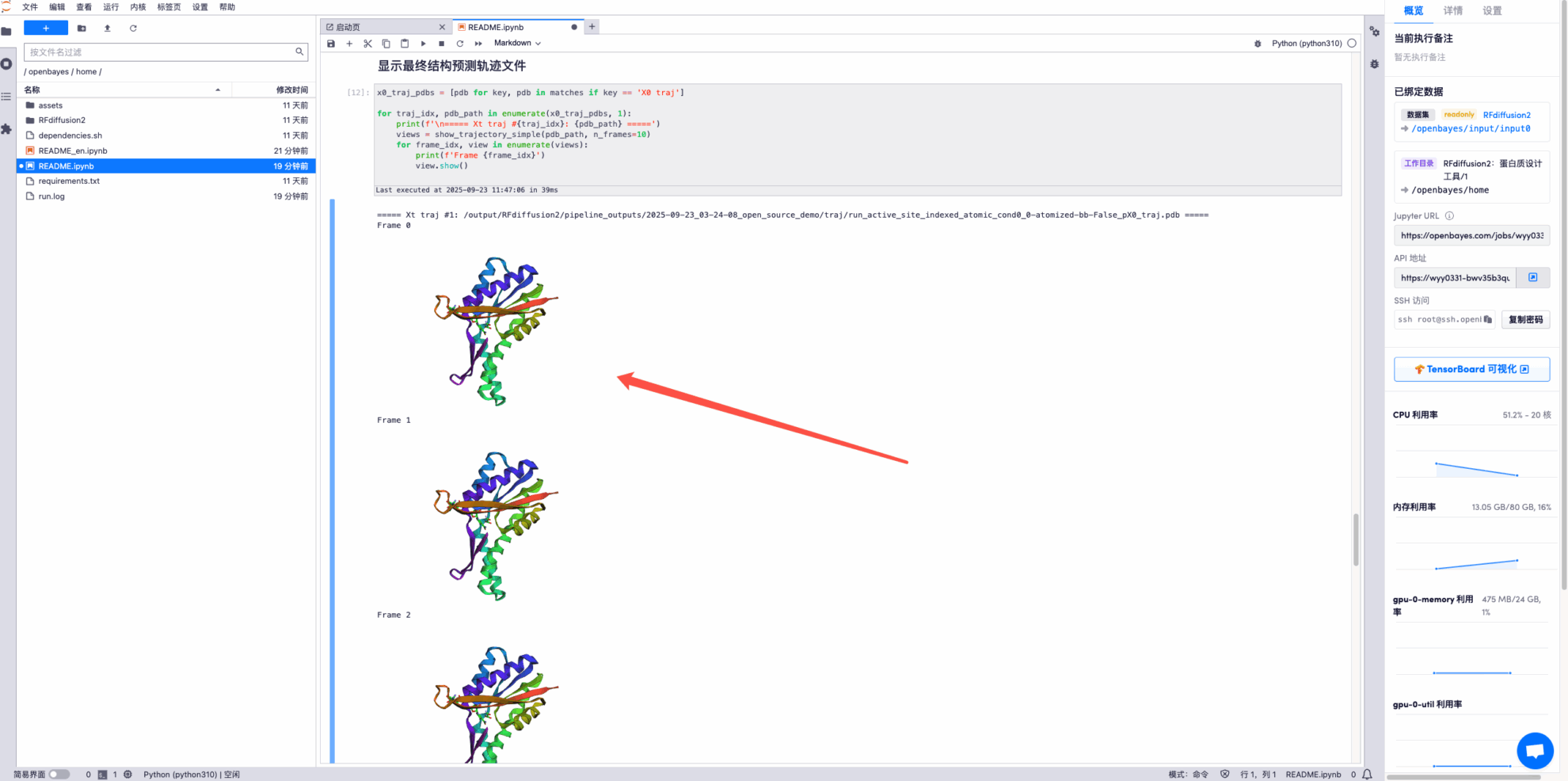
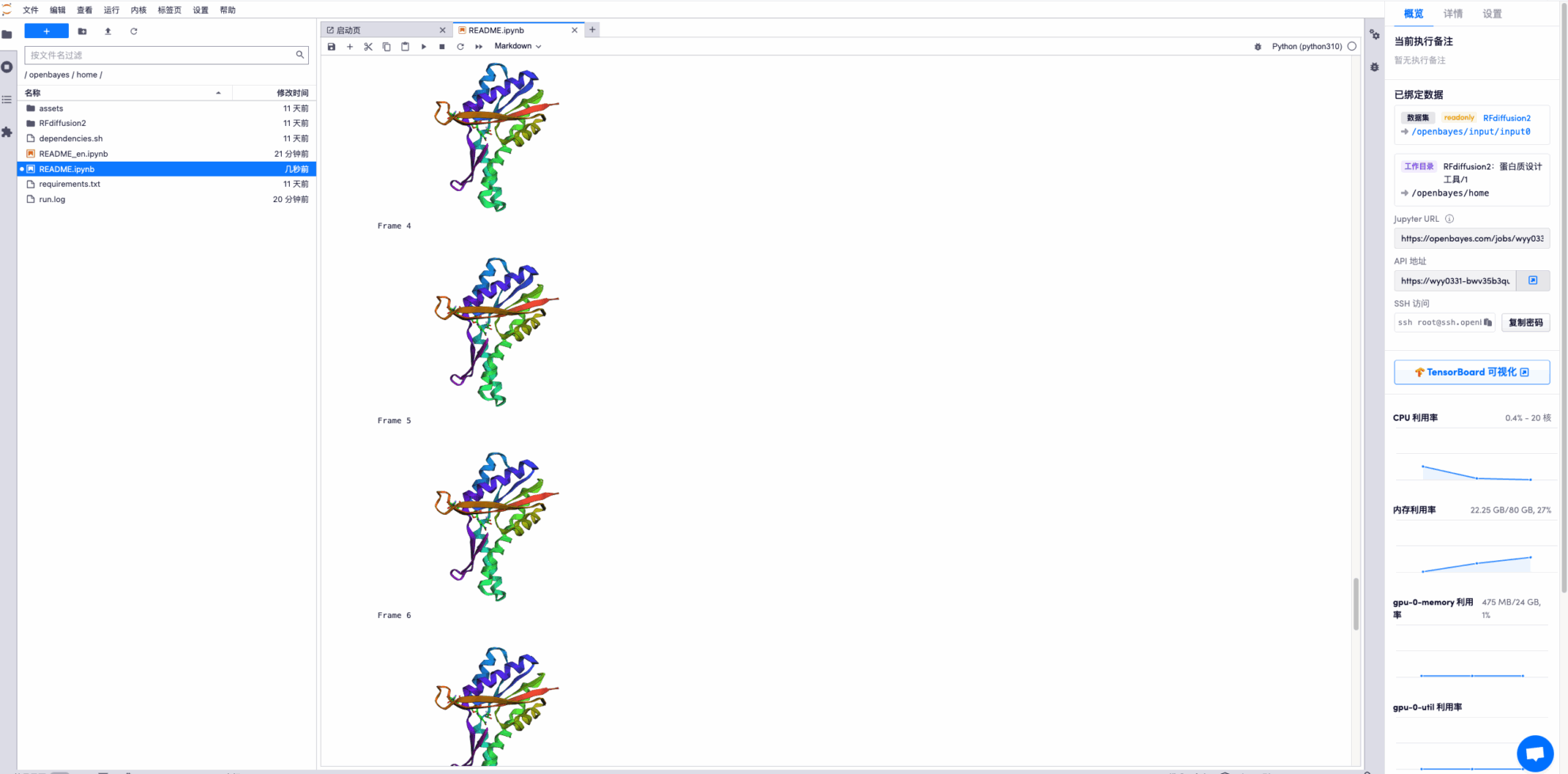
* Afficher le fichier de trajectoire de prédiction de la structure finale, voici quelques exemples :
Le tutoriel ci-dessus est celui recommandé par HyperAI cette fois-ci. Bienvenue à tous pour le découvrir !
Lien du tutoriel :