Command Palette
Search for a command to run...
Tutoriel En Ligne | L'équipe De David Baker Rend RFdiffusion3 Open Source, Réalisant Une Percée Générative Dans La Conception De Protéines À l'échelle atomique.

Ces dernières années, des progrès significatifs ont été réalisés dans la conception de nouvelles protéines fonctionnelles grâce aux méthodes d'apprentissage profond génératif. Actuellement, la plupart des méthodes, dont RFdiffusion (RFD1) et BindCraft, utilisent une représentation des protéines au niveau des résidus d'acides aminés et ont permis de concevoir avec succès des monomères, des assemblages et des systèmes d'interaction protéine-protéine. Cependant, leur résolution reste insuffisante pour concevoir avec précision des structures interagissant spécifiquement avec des composants non protéiques (tels que des ligands de petites molécules et des acides nucléiques) par le biais d'interactions de chaînes latérales.
Bien que RFdiffusion2 (RFD2) ait surmonté cette limitation dans une certaine mesure, son processus de diffusion reste limité au niveau des résidus et il est difficile de l'étendre davantage pour former des interactions supplémentaires entre chaînes latérales et composants non protéiques.Les recherches existantes suggèrent que les processus de diffusion à l'échelle atomique peuvent être utilisés pour générer des squelettes protéiques et peuvent être étendus à la modélisation des chaînes latérales, mais ces tentatives n'ont pas encore permis de modéliser efficacement les interactions entre les composants non protéiques.
Sur cette base,L'équipe du lauréat du prix Nobel David Baker a développé RFdiffusion3 (RFD3), qui peut générer des conformations tridimensionnelles de protéines dans des structures composées de ligands, d'acides nucléiques et d'autres atomes non protéiques.Ce modèle, qui modélise explicitement tous les atomes du polymère, gère plus facilement et efficacement les contraintes complexes à l'échelle atomique, notamment pour la conception d'enzymes. Son architecture native tout atome simplifie également considérablement la spécification de ces contraintes, offrant un contrôle précis des liaisons hydrogène, des contacts ligand-protéine et des interactions avec les acides nucléiques.
Contrairement à AlphaFold3 (AF3), qui s'appuie sur un module Pairformer gourmand en ressources de calcul pour extraire des informations telles que la distance à partir de la séquence d'entrée, l'équipe de recherche a conçu un module d'extraction d'informations plus léger.Cela permet à RFD3 de réduire drastiquement le nombre de couches du Pairformer de 48 à seulement 2, diminuant ainsi considérablement la charge de calcul. Le modèle final ne contient que 168 millions de paramètres entraînables.L'équipe de recherche a démontré la large applicabilité de RFD3 en concevant et en caractérisant expérimentalement des protéines de liaison à l'ADN et des cystéine hydrolases. RFD3 peut générer rapidement des structures protéiques guidées par des contraintes complexes à l'échelle atomique, à partir de n'importe quel environnement atomique non protéique, ce qui élargira encore le champ des fonctionnalités accessibles à la conception de protéines.
Le modèle de conception de protéines « RFdiffusion3 » est désormais disponible sur le site web d'HyperAI (hyper.ai) dans la section « Tutoriels ». Téléchargez-le et découvrez-le en un clic !
Pour célébrer le Nouvel An, HyperAI offre des avantages en matière de puissance de calcul à tous.Les nouveaux utilisateurs peuvent obtenir 2 heures de crédit NVIDIA en utilisant le code de réduction « 2026 Bonne Année » après leur inscription. GeForce RTX 5090 Durée d'utilisation,Quantités limitées, profitez dès maintenant de vos avantages pour les fêtes !
Lien du tutoriel :
Essai de démonstration
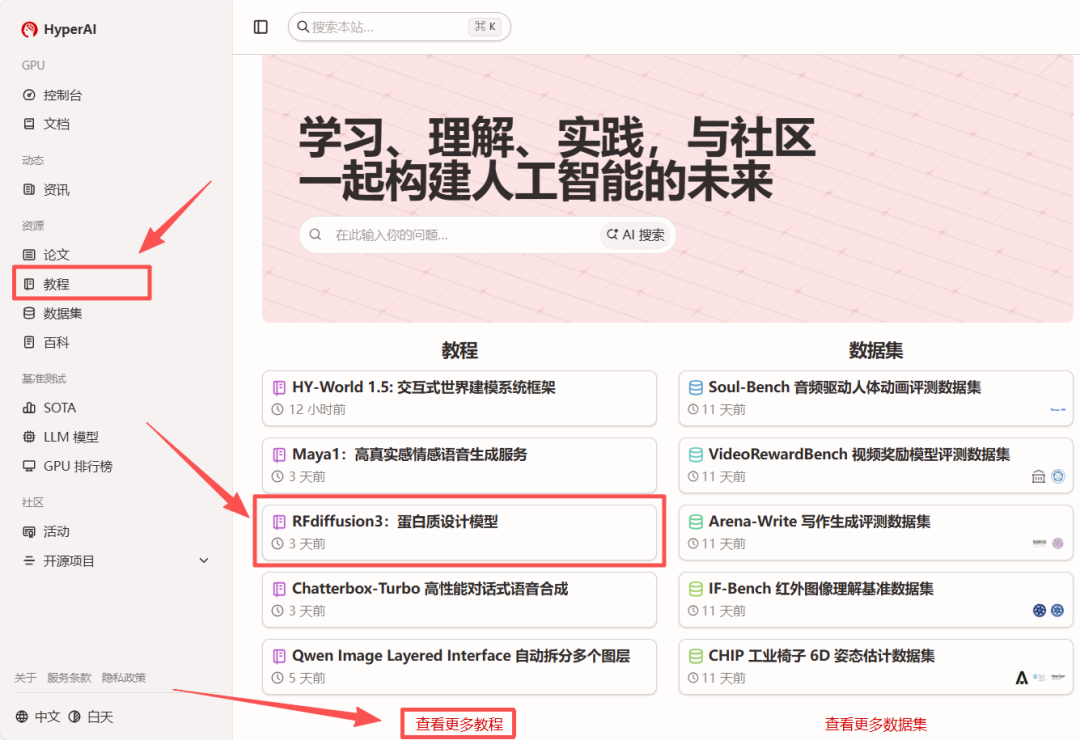
1. Après avoir accédé à la page d'accueil d'hyper.ai, sélectionnez « RFdiffusion3 : Modèle de conception de protéines » ou rendez-vous sur la page « Tutoriels » pour le sélectionner. Cliquez ensuite sur « Exécuter ce tutoriel en ligne ».
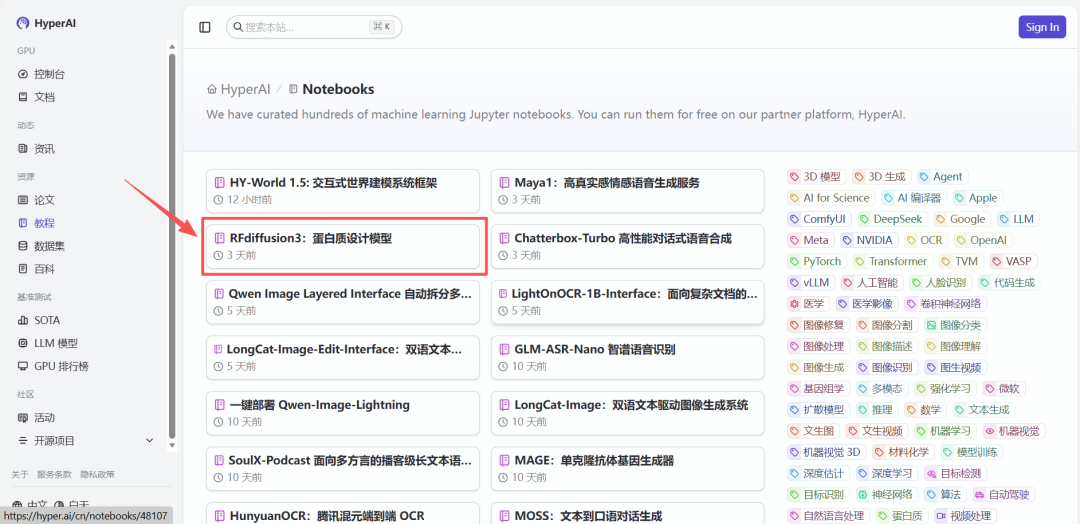
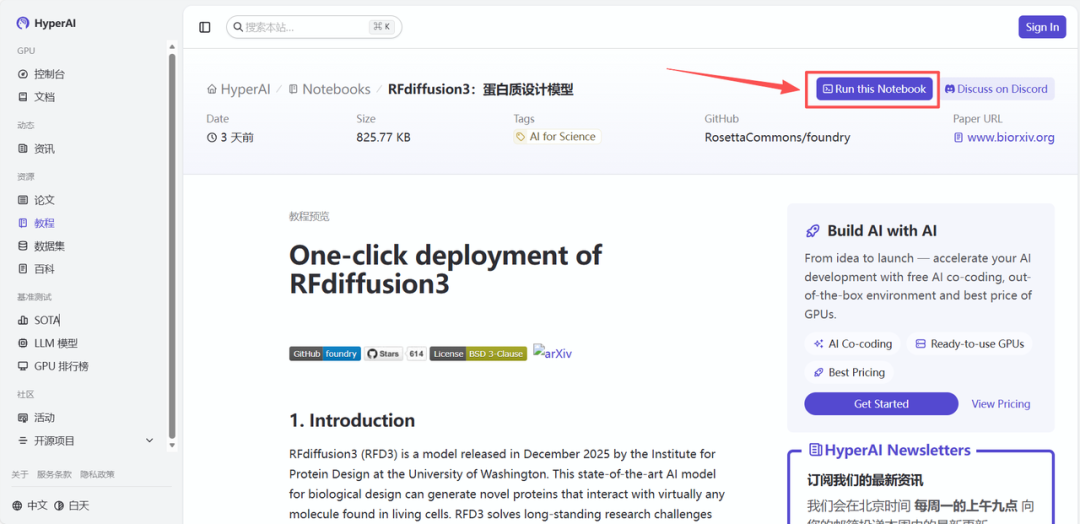
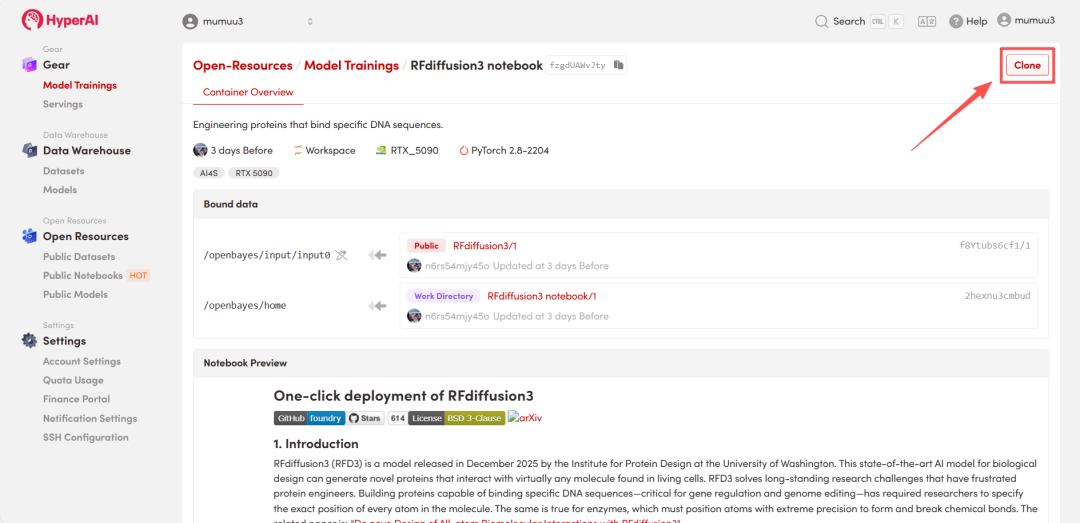
2. Une fois la page redirigée, cliquez sur « Cloner » en haut à droite pour cloner le tutoriel dans votre propre conteneur.
Remarque : Vous pouvez changer de langue en haut à droite de la page. Actuellement, le chinois et l’anglais sont disponibles. Ce tutoriel présente les étapes en anglais.
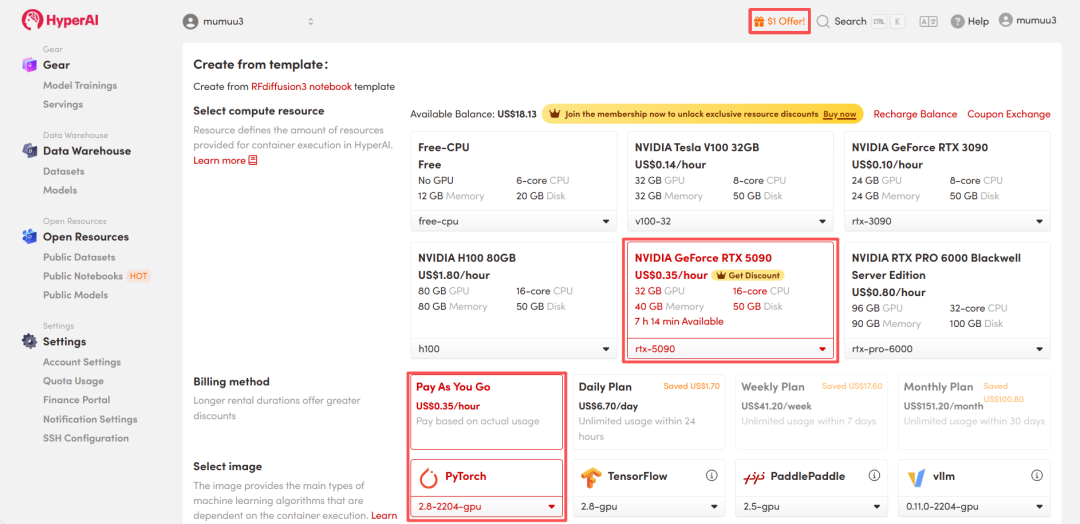
3. Sélectionnez les images « NVIDIA GeForce RTX 5090 » et « PyTorch », puis choisissez « Pay As You Go » ou « Daily Plan/Weekly Plan/Monthly Plan » selon vos besoins, puis cliquez sur « Continuer l’exécution de la tâche ».
HyperAI propose un bonus d'inscription pour les nouveaux utilisateurs : pour seulement $1, vous pouvez obtenir 20 heures de puissance de calcul RTX 5090 (au lieu de $7), et les ressources sont valables indéfiniment.
4. Attendez que les ressources soient allouées. Une fois que le statut passe à « En cours d'exécution », cliquez sur « Ouvrir l'espace de travail » pour accéder à l'espace de travail Jupyter.
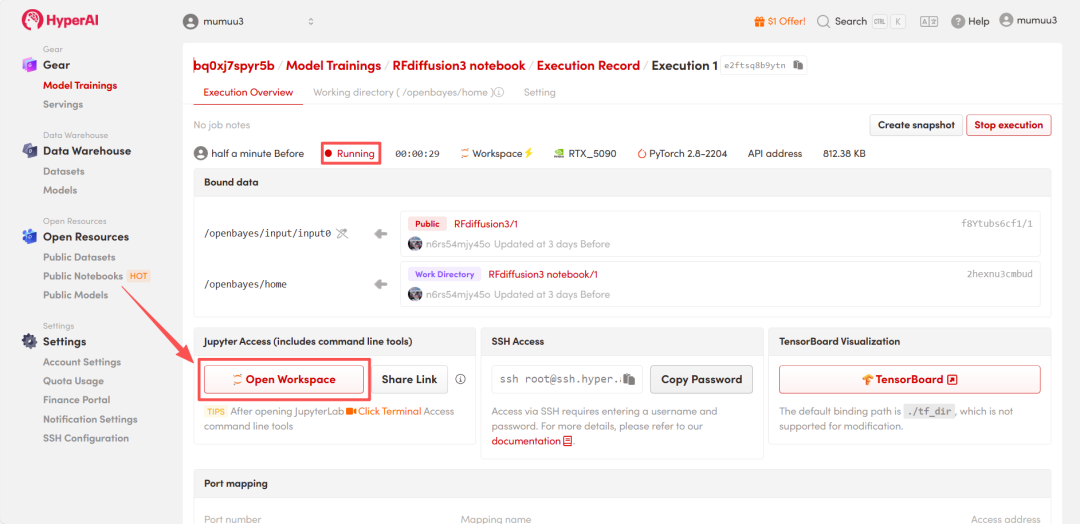
Démonstration d'effet
Une fois la page redirigée, cliquez sur le fichier README à gauche, puis sur « Exécuter » en haut.
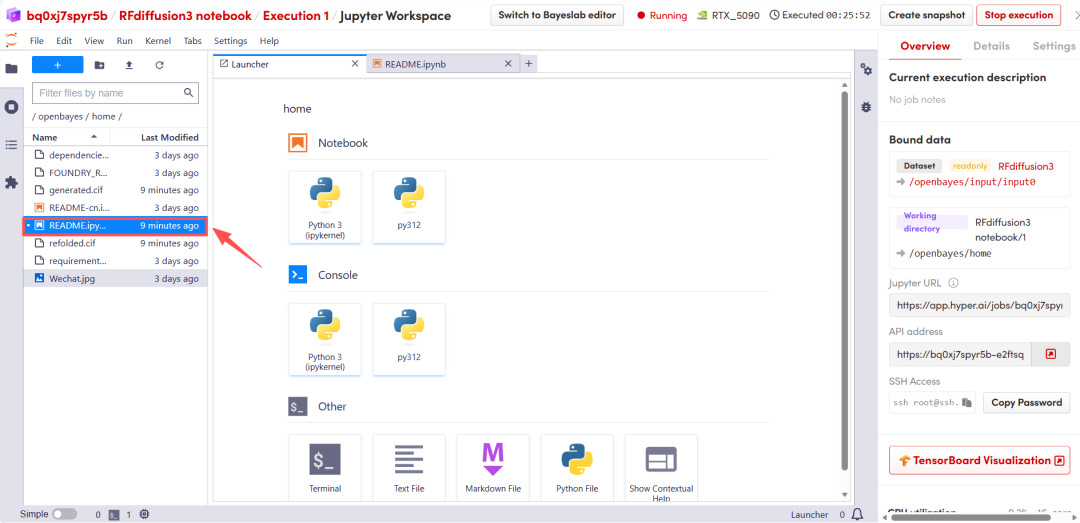
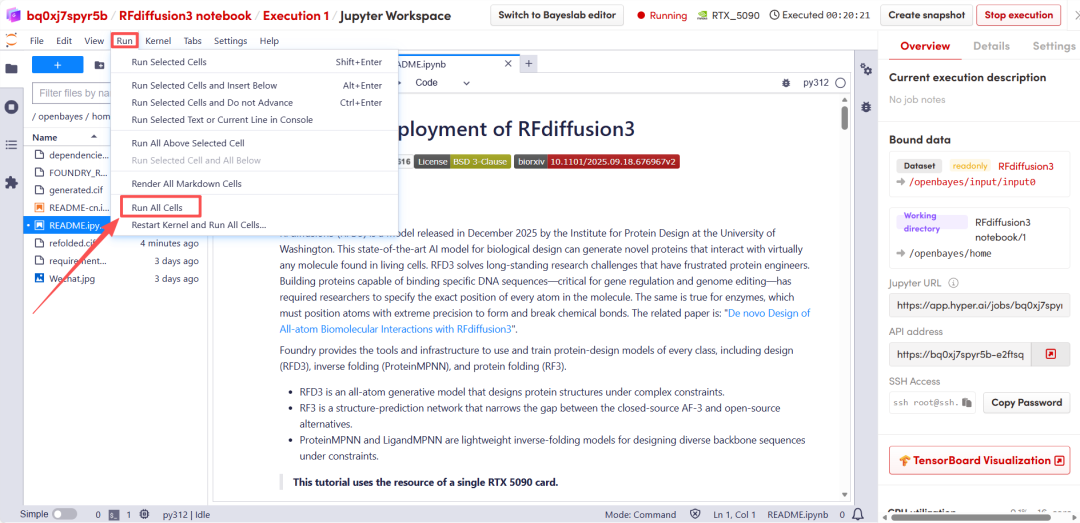
Après une courte attente, faites défiler vers le bas pour afficher les résultats de prédiction structurelle de RFD3.
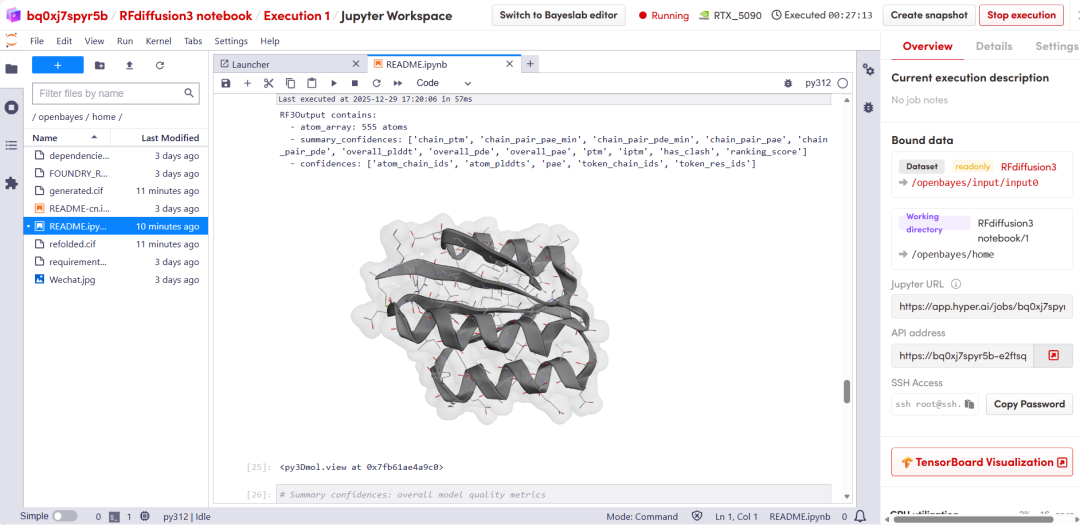
Le tutoriel ci-dessus est celui recommandé par HyperAI cette fois-ci. Bienvenue à tous pour le découvrir !
Lien du tutoriel :