Command Palette
Search for a command to run...
Tutoriel Inclus | Le MIT Et d'autres Lancent BindCraft, Qui Appelle Directement AF2 Pour Réaliser Une Conception Intelligente De Complexes De Liaison Aux Protéines

Dans les systèmes vivants, les protéines remplissent rarement leurs fonctions biologiques seules, mais s'appuient sur des interactions protéine-protéine (IPP) pour exécuter des processus biologiques complexes.La conception de conjugués protéiques capables de cibler et de réguler spécifiquement les IPP présente un énorme potentiel thérapeutique et biotechnologique.Cependant, les méthodes traditionnelles de génération de liants protéiques, telles que l’immunisation, le criblage de bibliothèques d’anticorps ou l’évolution dirigée, sont souvent laborieuses et prennent du temps, et offrent un contrôle limité sur le site cible.
La conception informatique de protéines offre une alternative puissante pour adapter les liants à des cibles et des sites de liaison spécifiques.etBien que les premières méthodes de calcul (telles que Rosetta) aient tenté de combiner la conception d'interface via la modélisation physique et l'optimisation de la chaîne latérale, le taux de réussite était souvent inférieur à 0,1%.À l'ère de l'apprentissage profond, les modèles de prédiction de structure tels qu'AlphaFold2 ont radicalement changé la donne. Ils permettent non seulement de prédire avec précision la structure de repliement tridimensionnelle de protéines individuelles, mais aussi de simuler les interactions spatiales de complexes protéiques.
Mais il ne s'agit pas encore d'une véritable « conception intelligente ». Les méthodes existantes, telles que la diffusion RF ou le ProteinMPNN,Il est toujours nécessaire de définir manuellement le squelette et l'interface d'amarrage, puis d'utiliser AlphaFold pour vérifier si cela est raisonnable.Ainsi, étant donné qu’AlphaFold peut déjà comprendre la structure des protéines, peut-il être autorisé à « penser » à l’envers et à générer directement une nouvelle protéine qui correspond parfaitement à la cible ?
Sur cette base,Une équipe de l'École polytechnique fédérale de Lausanne (EPFL) et du Massachusetts Institute of Technology (MIT) a proposé un processus automatisé open source, BindCraft, permettant de concevoir des liants protéiques de A à Z. L'idée principale est de rétropropager la séquence de liant hallucinée à travers les poids AlphaFold2 et de calculer le gradient d'erreur.L'équipe de recherche a combiné une version multi-corps d'AlphaFold2 avec un réseau neuronal pour générer simultanément la structure, la séquence et l'interface de la protéine par optimisation de gradient. Cela a permis de générer des liants de novo avec une affinité nanomolaire in silico, sans recourir à un criblage à haut débit, à des itérations expérimentales ni même à un site de liaison connu.
Les résultats expérimentaux montrent que BindCraft a obtenu des résultats révolutionnaires sur 12 cibles avec des structures complexes et une grande importance pharmacologique.Le taux de réussite expérimental varie de 10% à 100%, avec un taux de réussite moyen de 46,3%.Cela signifie que les travaux de conception qui nécessitaient auparavant des centaines, voire des milliers de criblages, peuvent désormais obtenir une combinaison utilisable avec un seul calcul.
« BindCraft : Conception de liants protéiques » est désormais disponible sur le site officiel d'HyperAI, dans la section « Tutoriels ». Cliquez sur le lien ci-dessous pour accéder au tutoriel de déploiement en un clic ⬇️
Lien du tutoriel :
Essai de démonstration
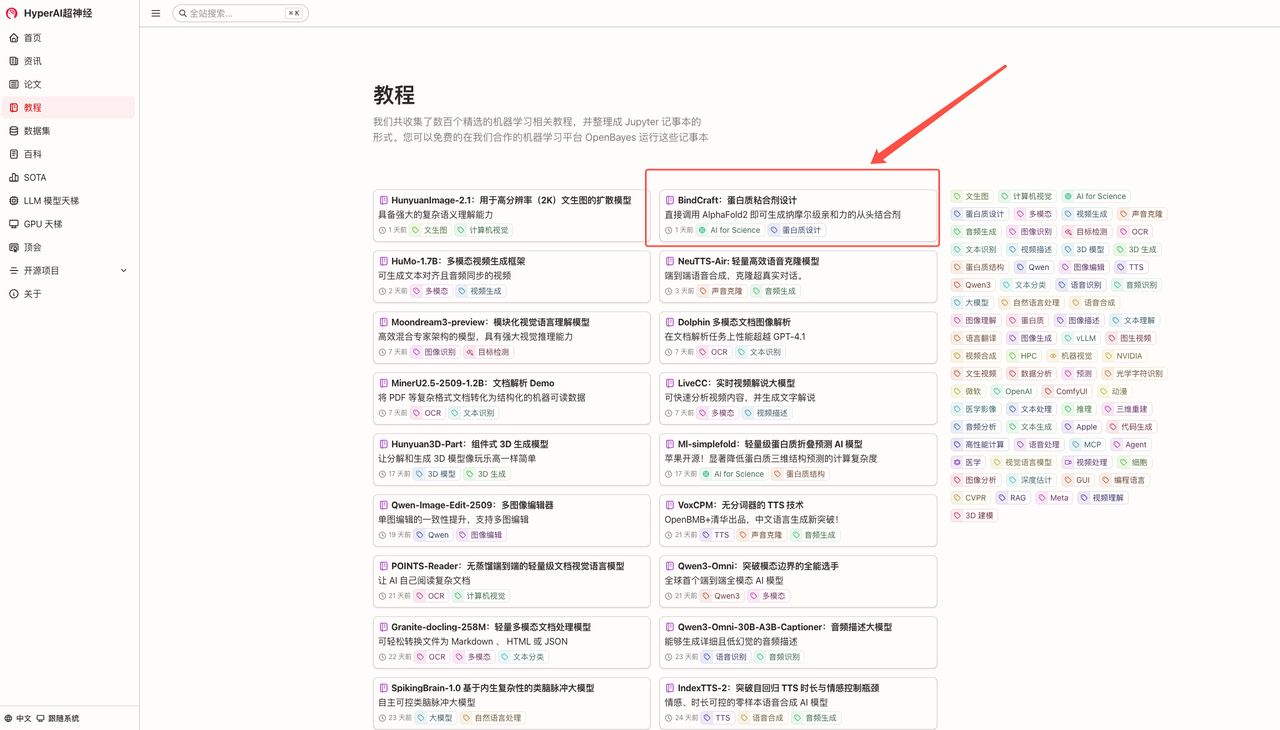
1. Sur la page d'accueil de hyper.ai, sélectionnez la page Tutoriels, choisissez BindCraft : Conception de liants protéiques, puis cliquez sur Exécuter ce didacticiel en ligne.
2. Une fois la page affichée, cliquez sur « Cloner » dans le coin supérieur droit pour cloner le didacticiel dans votre propre conteneur.
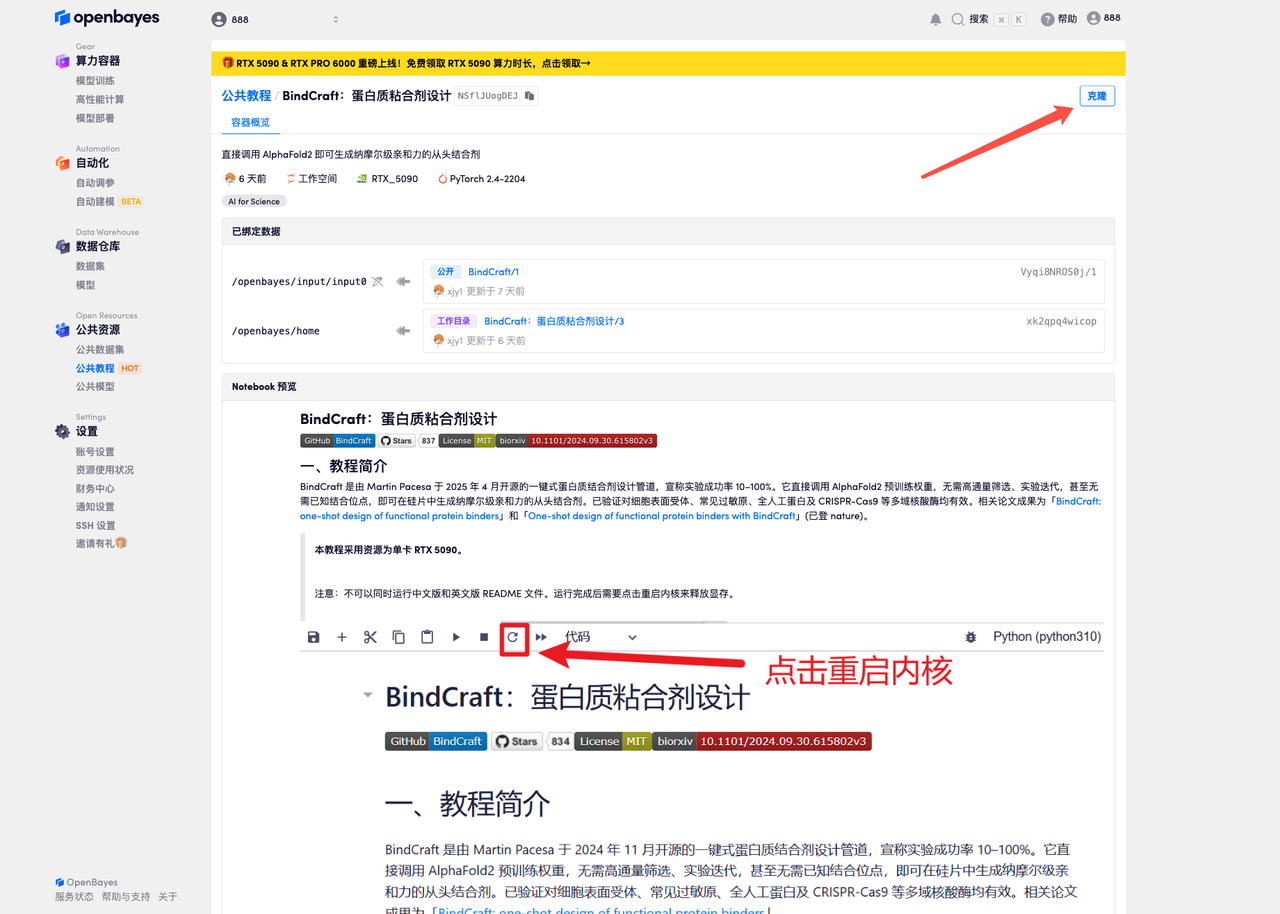
3. Sélectionnez les images NVIDIA GeForce RTX 5090 et PyTorch, puis cliquez sur « Continuer ». La plateforme OpenBayes propose quatre options de facturation : paiement à l'utilisation ou forfait journalier, hebdomadaire ou mensuel. Les nouveaux utilisateurs peuvent s'inscrire via le lien d'invitation ci-dessous pour recevoir 4 heures de RTX 5090 et 5 heures de temps processeur gratuits !
Lien d'invitation exclusif HyperAI (copier et ouvrir dans le navigateur) :
https://openbayes.com/console/signup?r=Ada0322_NR0n
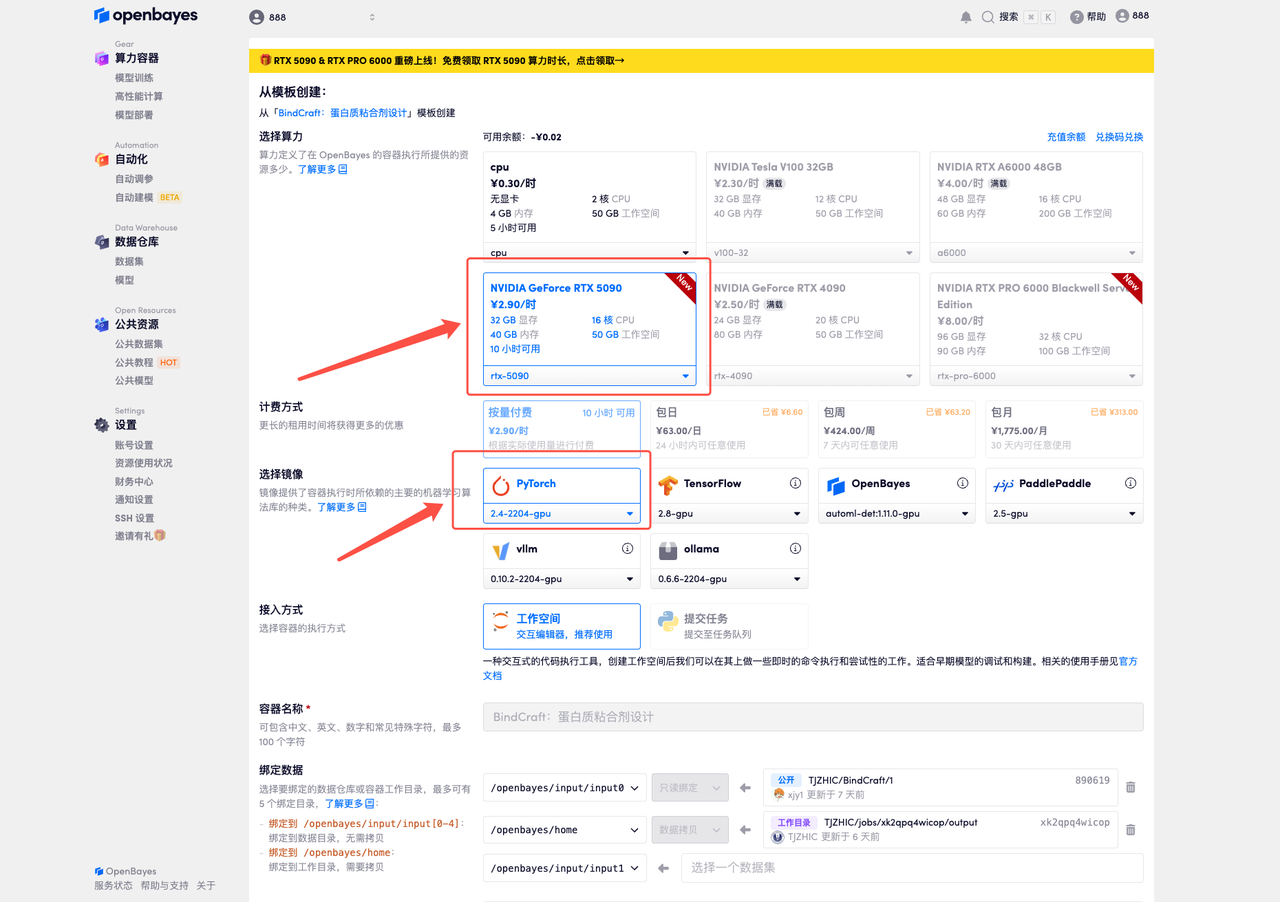
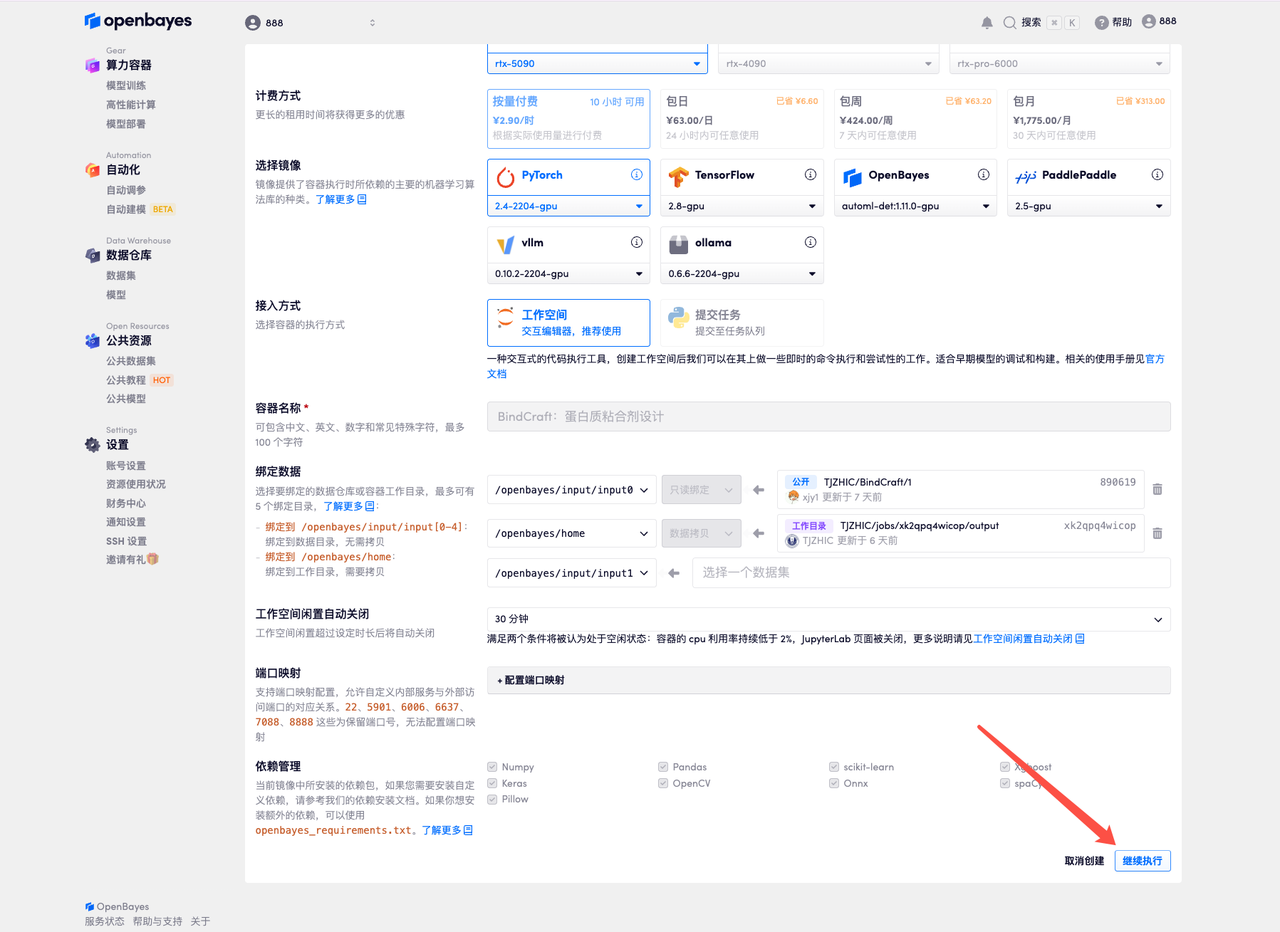
4. Attendez que les ressources soient allouées. Le premier clonage prendra environ 2 minutes. Lorsque le statut passe à « En cours d'exécution », cliquez sur « Ouvrir l'espace de travail » pour accéder à la page de démonstration.
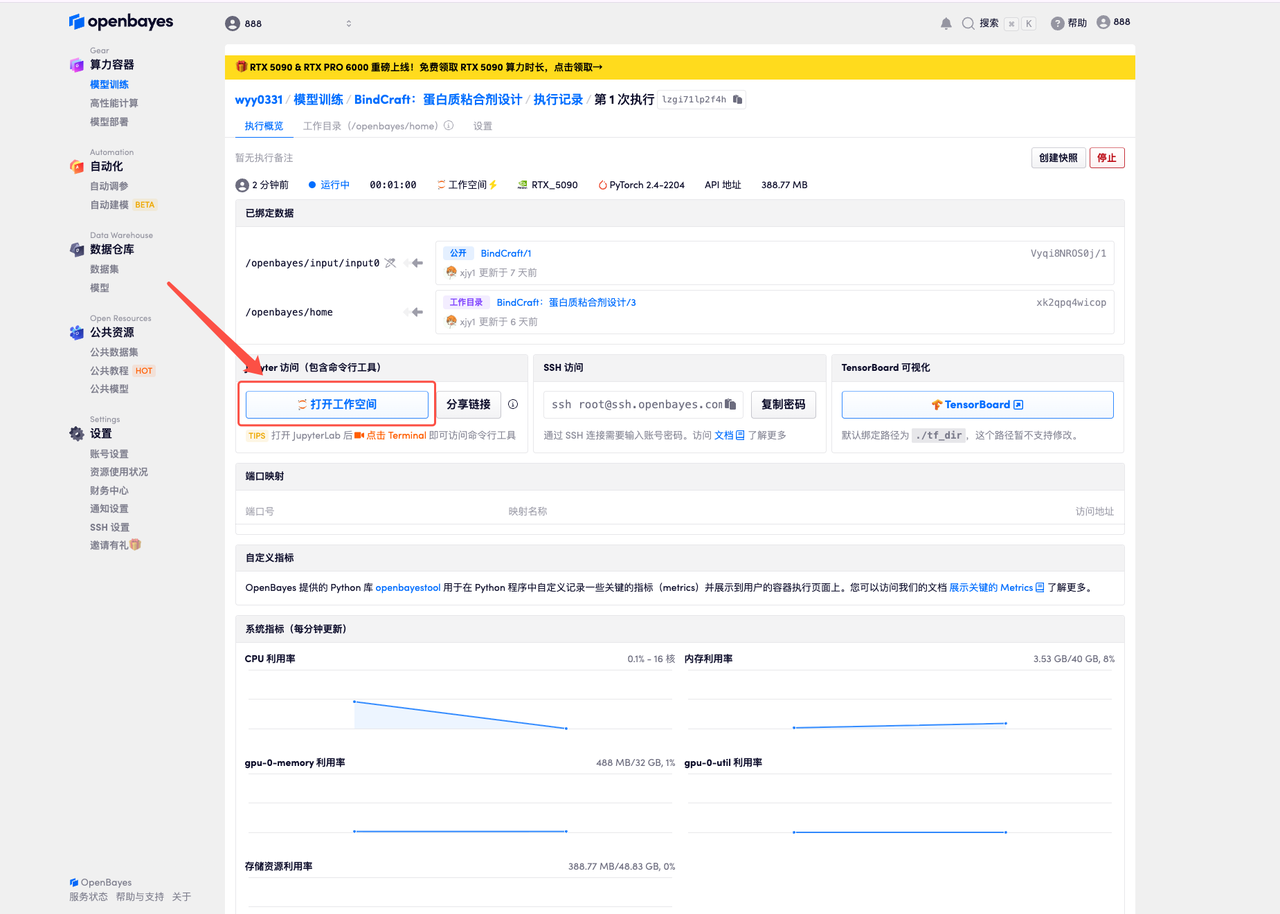
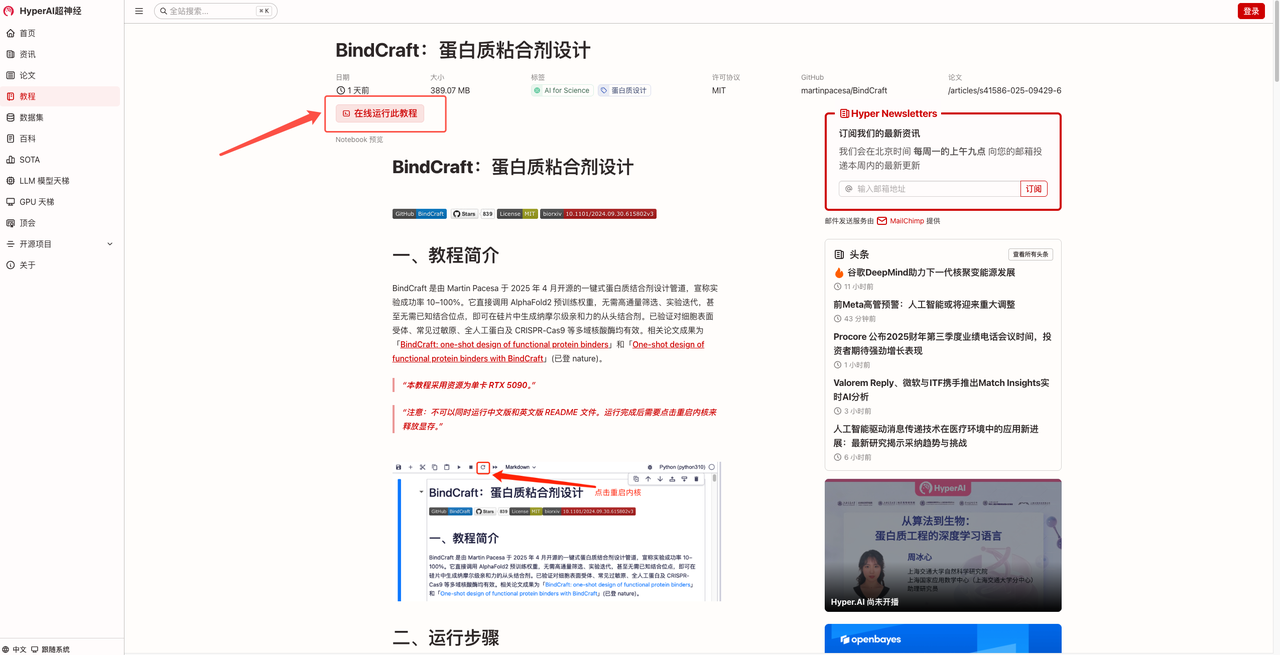
5. Voici la page d'utilisation de BindCraft. Cliquez sur « README » pour accéder à l'interface de génération.
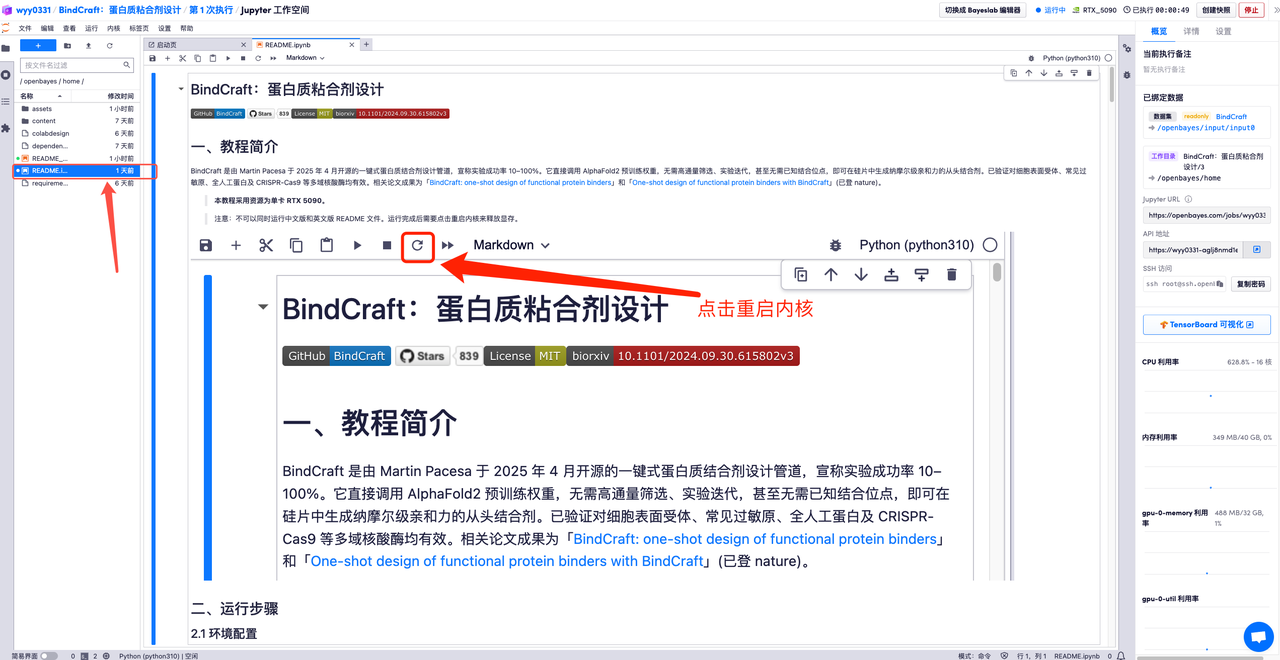
Démonstration d'effet
1. Veuillez compléter les réglages des paramètres pertinents conformément aux instructions des paramètres README
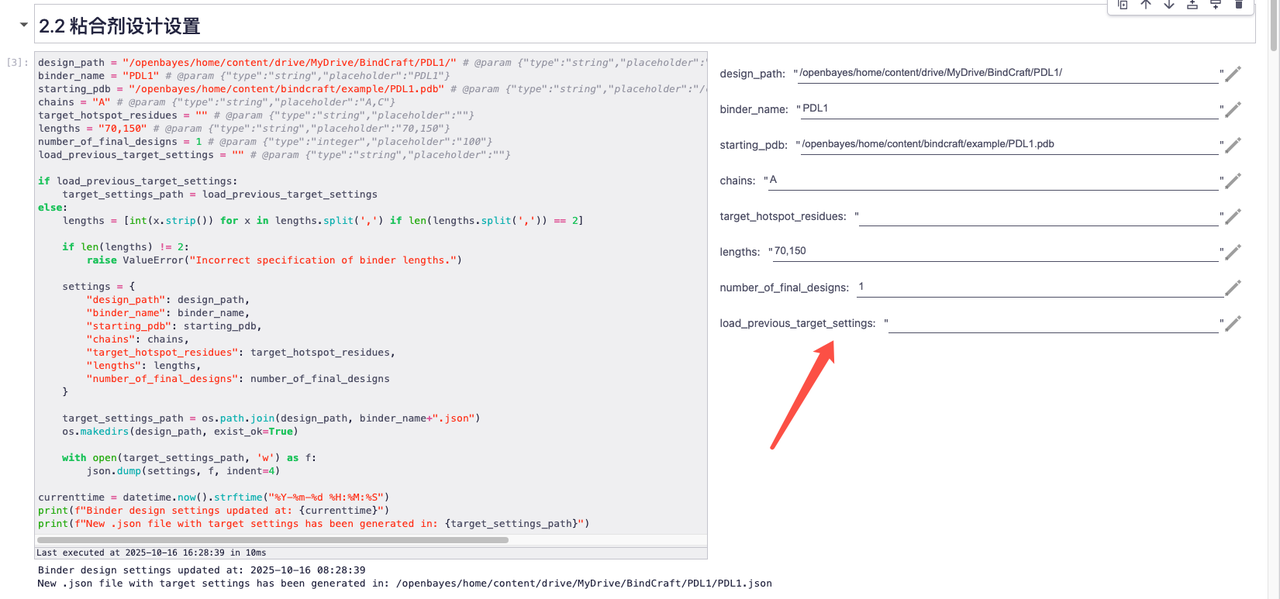
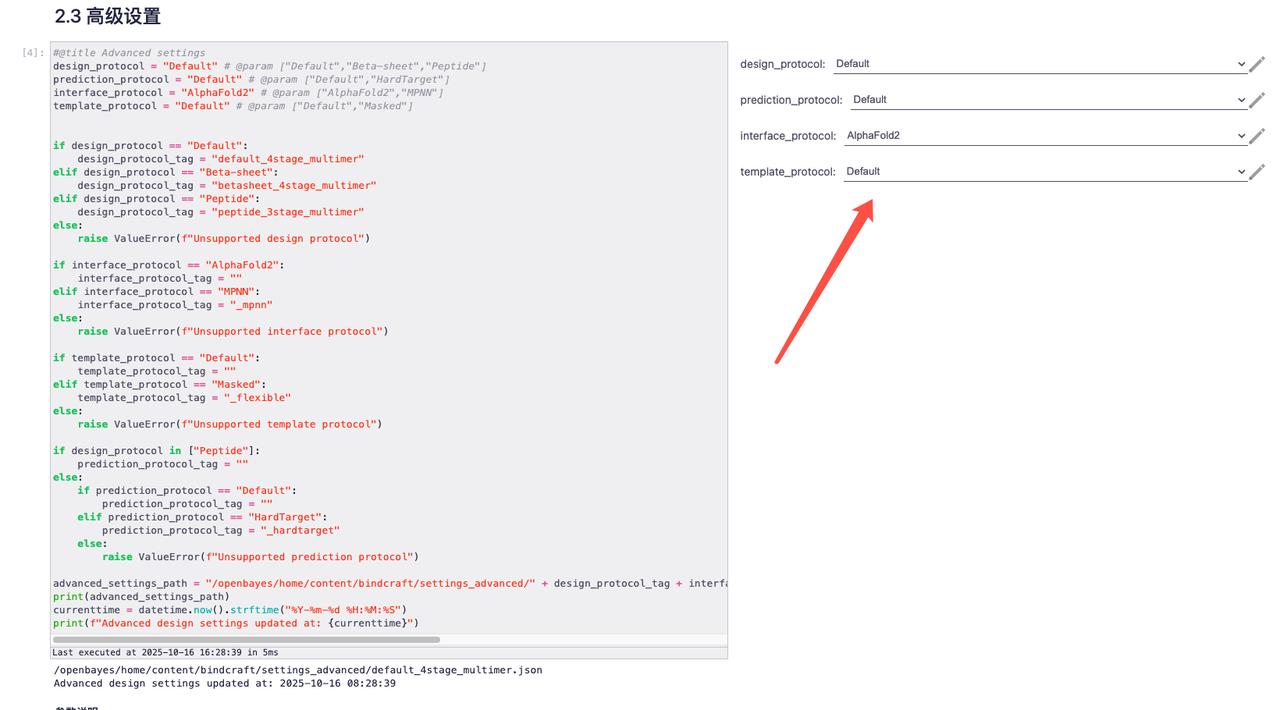
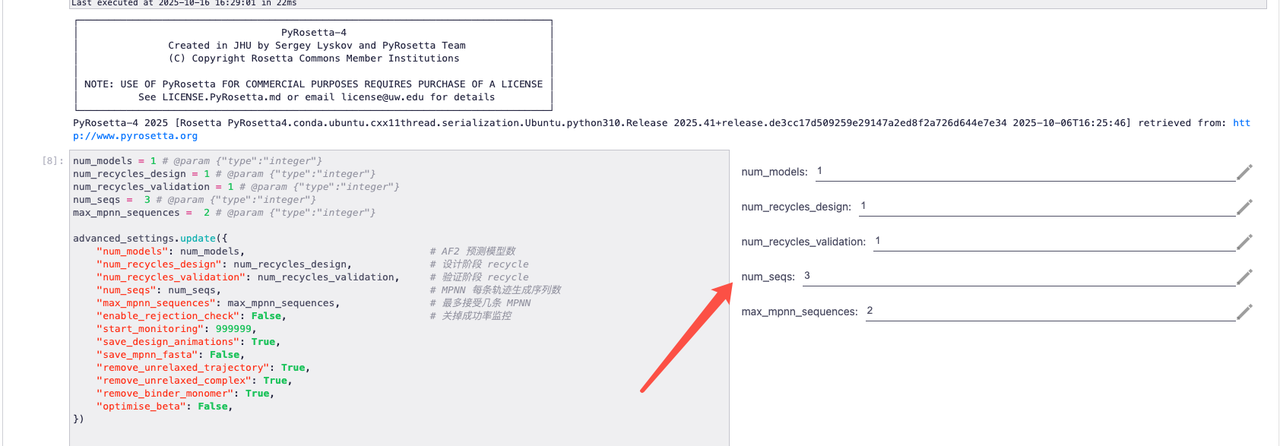
2. En prenant les paramètres par défaut comme exemple, les effets pertinents sont les suivants
L'effet de la « conception de haut niveau » est le suivant :
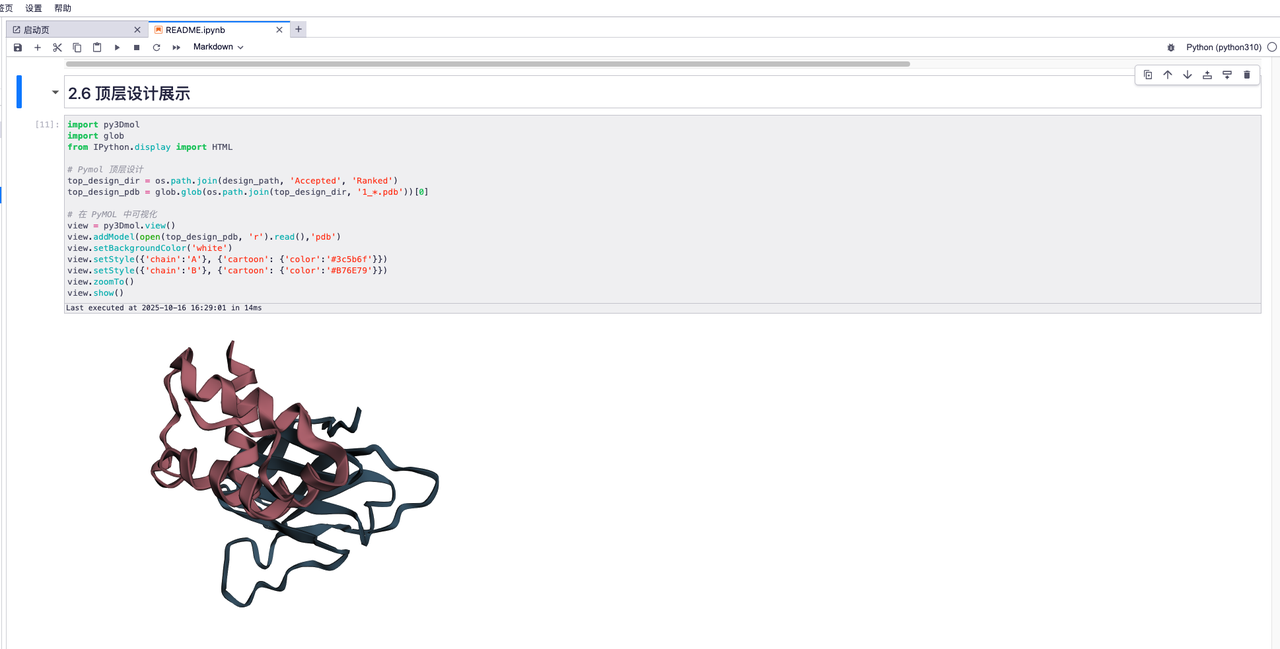
Effet « Afficher l'animation », veuillez regarder la vidéo ci-dessous :
Le tutoriel ci-dessus est celui recommandé par HyperAI cette fois-ci. Bienvenue à tous pour le découvrir !
Lien du tutoriel :