Command Palette
Search for a command to run...
Prédiction De mutation/prédiction De Fonction De Protéines À Guichet Unique, l'atelier d'ingénierie Des Protéines VenusFactory Permet Un Développement Complet
L'amélioration significative de l'efficacité de conception grâce à l'IA transforme le paradigme de recherche traditionnel en matière de conception de protéines. Comparés aux expériences traditionnelles de conception de protéines, les grands modèles d'IA peuvent non seulement prédire et cribler les séquences, les structures et les fonctions des protéines en un temps record, mais aussi concevoir de nouvelles protéines inexistantes dans la nature, en s'appuyant sur des principes physiques et chimiques et des modèles de données. Ils peuvent même prédire simultanément des propriétés protéiques telles que la stabilité, l'affinité de liaison et la cinétique grâce à des modèles d'apprentissage multitâche et d'apprentissage profond.
Cependant,Le cadre informatique complexe du modèle et l’énorme base de données de protéines ont élevé le seuil d’utilisation des outils d’IA.D'une part, la dépendance du domaine de la conception des protéines aux données biologiques oblige les chercheurs à récupérer, télécharger, compiler et convertir des données provenant de multiples bases de données, ce qui représente un travail chronophage. D'autre part, les modèles d'IA protéique ne peuvent actuellement résoudre que des tâches individuelles dans des domaines de niche et ne disposent pas d'un système d'évaluation basé sur des données de référence fiables.
En outre, concernant les défis de la conception des protéines d'IA, le Dr Tan Yang du groupe de recherche du professeur Hong Liang à l'Université Jiao Tong de Shanghai a également indiqué que les modèles d'IA existants dans le domaine de la conception des protéines ont non seulement des difficultés à acquérir des données et à unifier le format, mais ont également des difficultés à ajuster les paramètres et à ralentir la vitesse d'entraînement.Les obstacles causés par les « barrières de données, les barrières de modèles et les barrières d’application » ont entravé la popularisation et l’application des outils d’IA dans une communauté de recherche scientifique plus large.
Parallèlement, les serveurs web, simples et faciles à utiliser, offrent des fonctionnalités limitées, ne peuvent pas être entraînés à partir de données individuelles et leur intelligence est limitée. Les agents réduisent l'intervention humaine, peuvent se concentrer directement sur les objectifs et réaliser de manière autonome une ou plusieurs unités de travail.
Promouvoir l’application généralisée de l’intelligence artificielle dans le domaine de l’ingénierie des protéines,Le groupe de recherche du professeur Hong Liang à l'Université Jiao Tong de Shanghai a développé un banc d'essai d'ingénierie des protéines open source à guichet unique, VenusFactory.La plateforme intègre la récupération de données biologiques, l'analyse comparative de tâches standardisées et des modèles de langage protéique (PLM) pré-entraînés. Elle combine les fonctions d'un serveur web et d'un agent :
* Implémentation de la personnalisation sans code des modèles d'IA à l'aide d'ensembles de données privés, prenant en charge l'exécution en ligne de commande et l'interface sans code basée sur Gradio.
* Fournit des téléchargements open source de plus de 30 grands ensembles de données de référence d'évaluation de modèles, intègre plus de 40 ensembles de données liés aux protéines et plus de 40 PLM populaires, et établit facilement des liens vers les données sur les protéines.
* Il peut réaliser une prédiction de mutation sans échantillon, combiner automatiquement des modèles d'IA pour recommander des mutations en fonction de la demande et intégrer des modules de prédiction supervisés pour prédire les propriétés via des modèles d'IA d'intégration cible.
à l'heure actuelle,La plateforme de conception d'ingénierie des protéines VenusFactory est désormais disponible dans la section tutoriel du site Web officiel d'HyperAI (hyper.ai). Le tutoriel de la plateforme VenusFactory couvre 7 modules fonctionnels, et vous pouvez en faire l'expérience en ligne avec un déploiement en un clic :
* Formation : formation de modèle sans code, prend en charge plus de 40 grands modèles et utilise des ensembles de données privés pour former vos propres modèles.
* Évaluation : Un outil facile à utiliser pour l’évaluation complète des performances des modèles protéiques.
* Prédiction : utilisez le modèle formé pour prédire la fonction de nouvelles séquences protéiques.
* Outils rapides : version facile à utiliser, prenant en charge la prédiction de mutation à échantillon zéro (évolution dirigée) et la prédiction supervisée (prédiction de fonction ou de propriété).
* Outils avancés : version personnalisée avancée, prenant en charge la prédiction de mutation à échantillon zéro (évolution dirigée) et la prédiction supervisée (prédiction de fonction ou de propriété).
* Téléchargement : créez facilement un lien vers les données sur les protéines et prenez en charge le téléchargement multithread des principales bases de données grand public (RCSB, UniProt...).
* VenusAgent : un agent d'ingénierie des protéines qui fonctionne avec DeepSeek pour permettre le calcul des protéines par l'IA.
Lien du tutoriel :
De plus, nous avons préparé des avantages surprises en matière de ressources informatiques pour les nouveaux utilisateurs.Inscrivez-vous avec le code d'invitation « VenusFactory » pour obtenir 2 heures d'utilisation du double SIM A6000 (la période de validité des ressources est de 1 mois).La quantité est limitée, ne la manquez pas !
Essai de démonstration
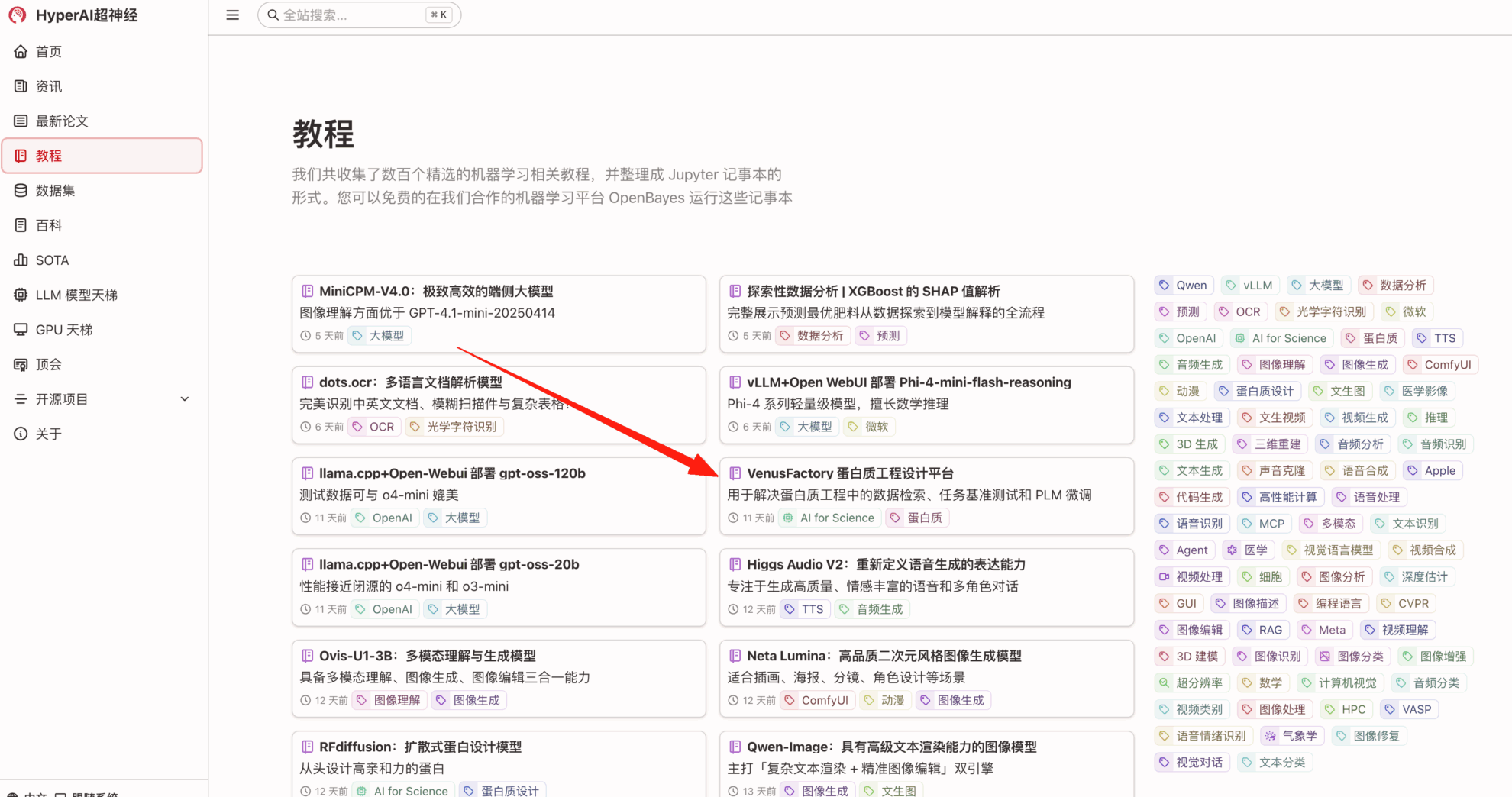
1. Saisissez l'URL hyper.ai dans votre navigateur. Une fois sur la page d'accueil, cliquez sur la page « Tutoriels », sélectionnez « Plateforme d'ingénierie des protéines VenusFactory » et cliquez sur « Exécuter ce tutoriel en ligne ».
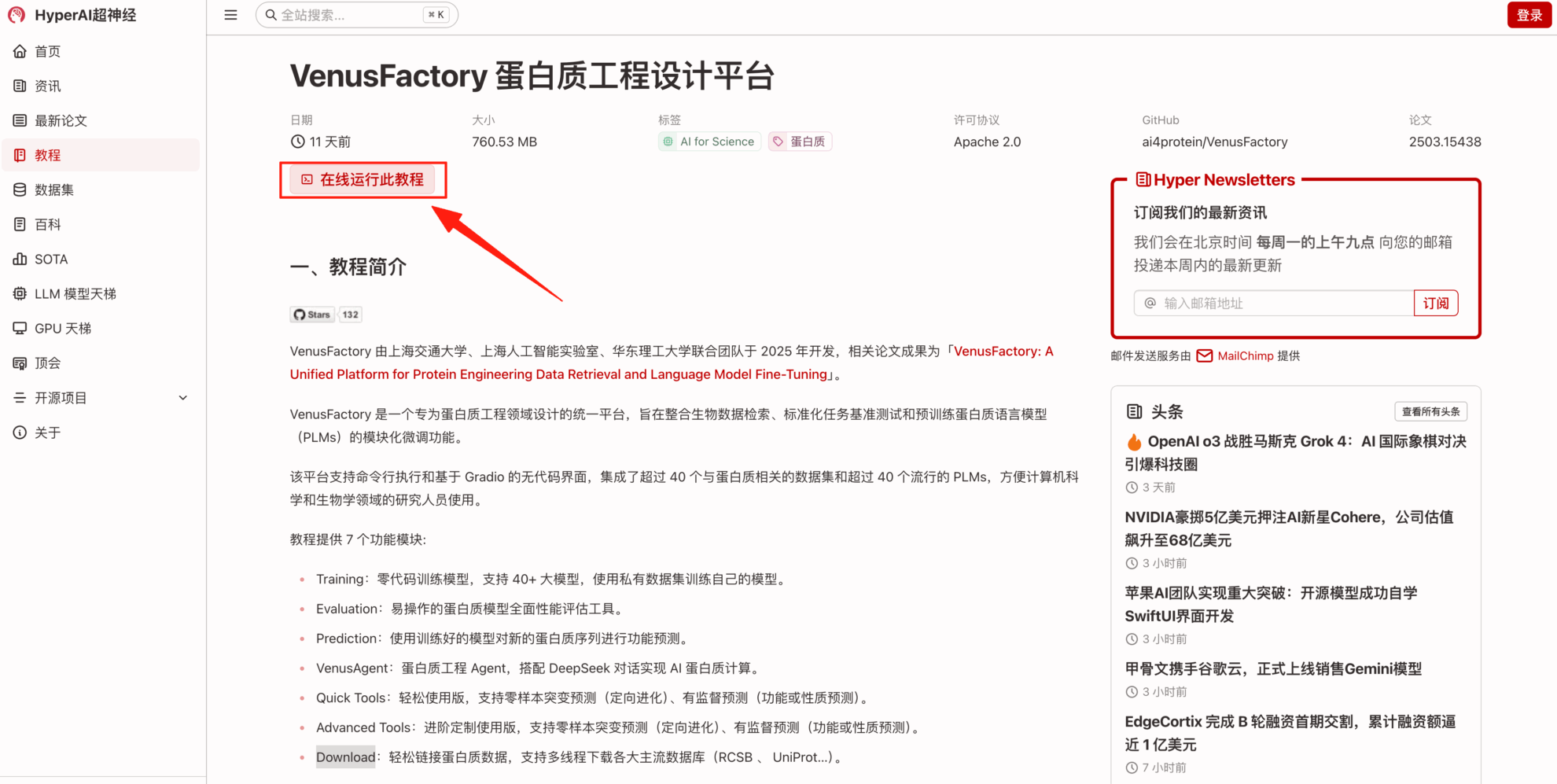
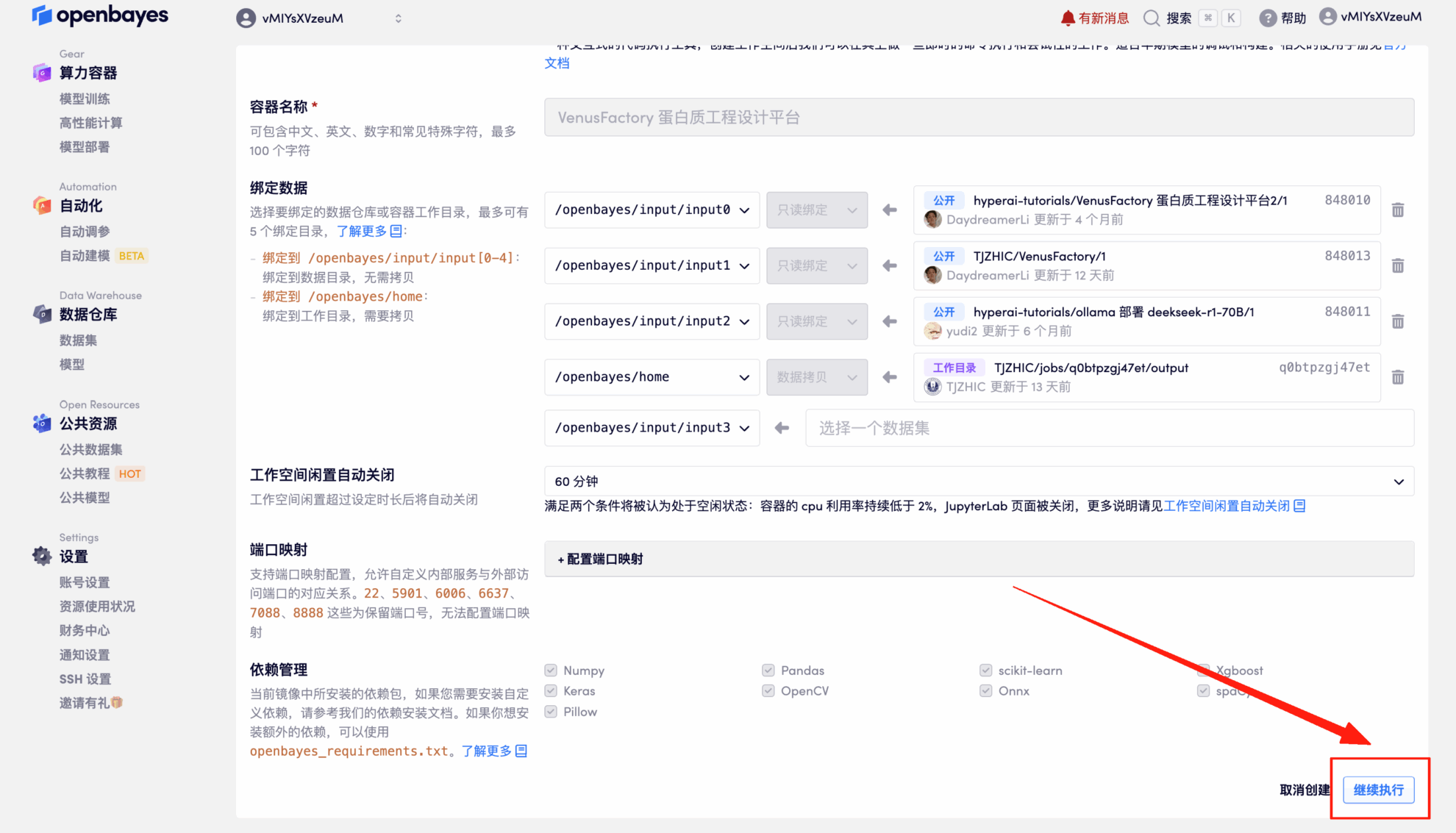
2. Une fois la page affichée, cliquez sur « Cloner » dans le coin supérieur droit pour cloner le didacticiel dans votre propre conteneur.
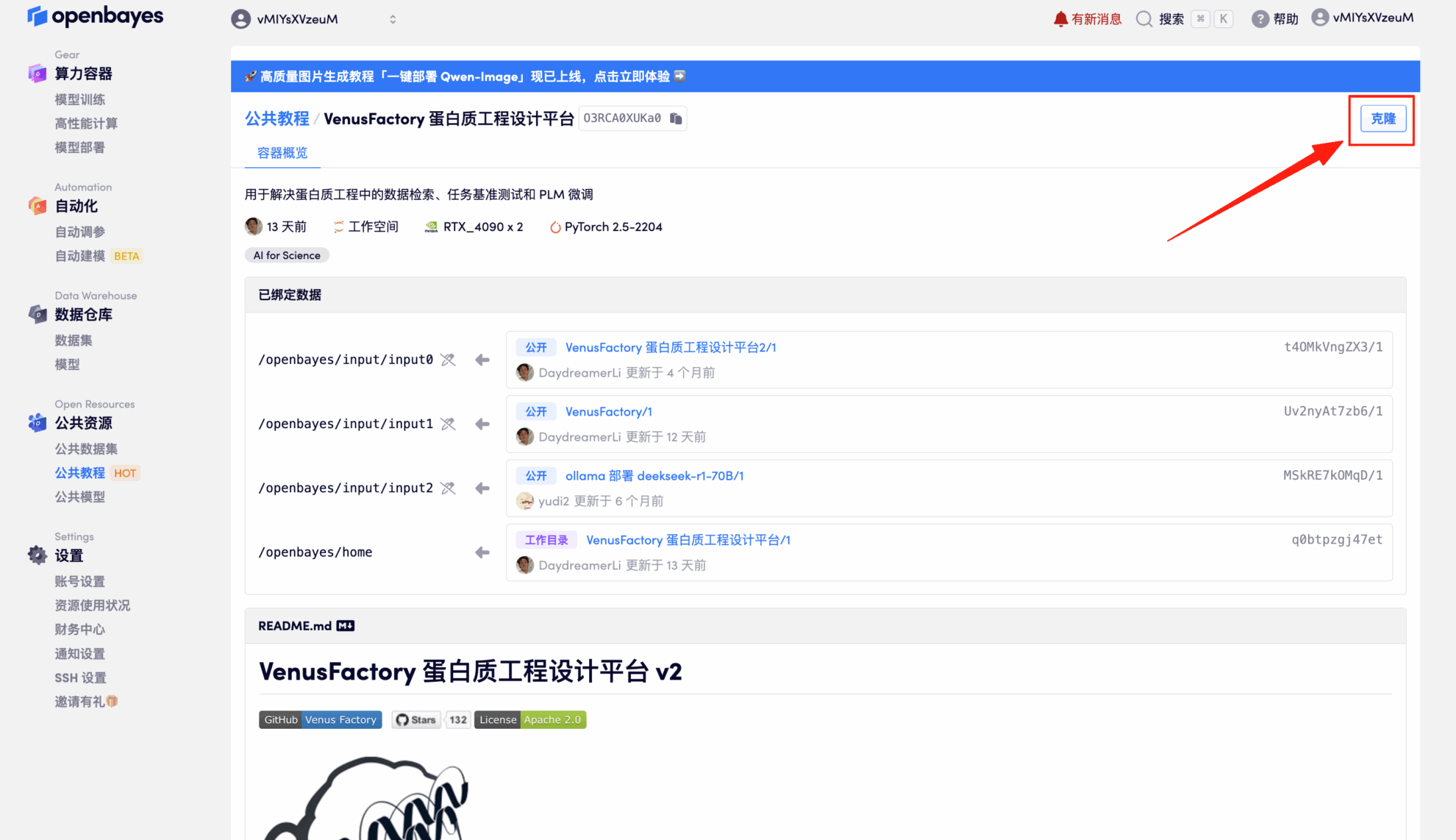
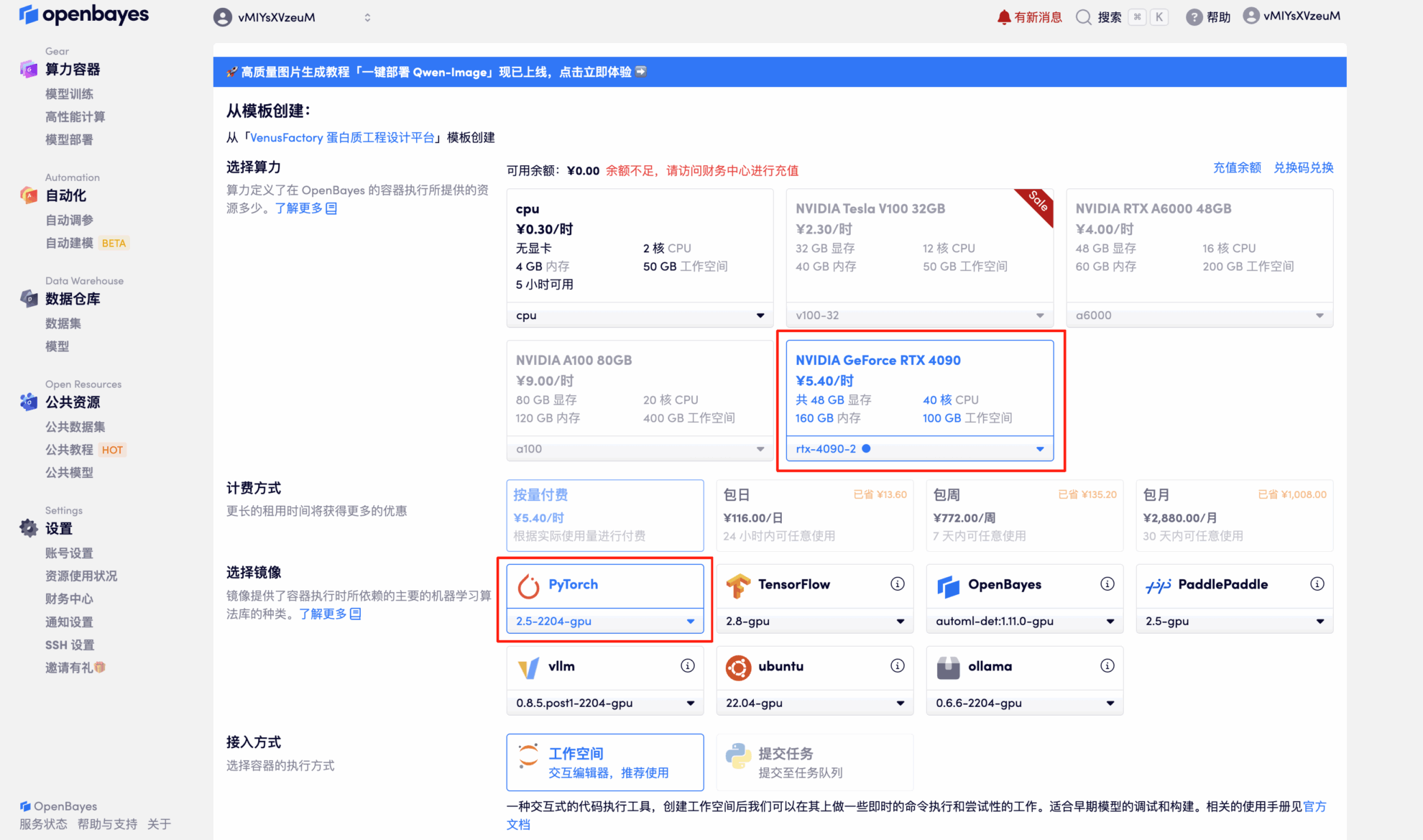
3. Sélectionnez les images NVIDIA GeForce RTX 4090-2 et PyTorch, puis cliquez sur Continuer. La plateforme OpenBayes propose quatre options de facturation : paiement à l'utilisation ou forfait journalier, hebdomadaire ou mensuel. Les nouveaux utilisateurs peuvent s'inscrire via le lien d'invitation ci-dessous pour recevoir 4 heures de RTX 4090 et 5 heures de temps processeur gratuits !
Lien d'invitation exclusif HyperAI (copier et ouvrir dans le navigateur) :
https://openbayes.com/console/signup?r=Ada0322_NR0n
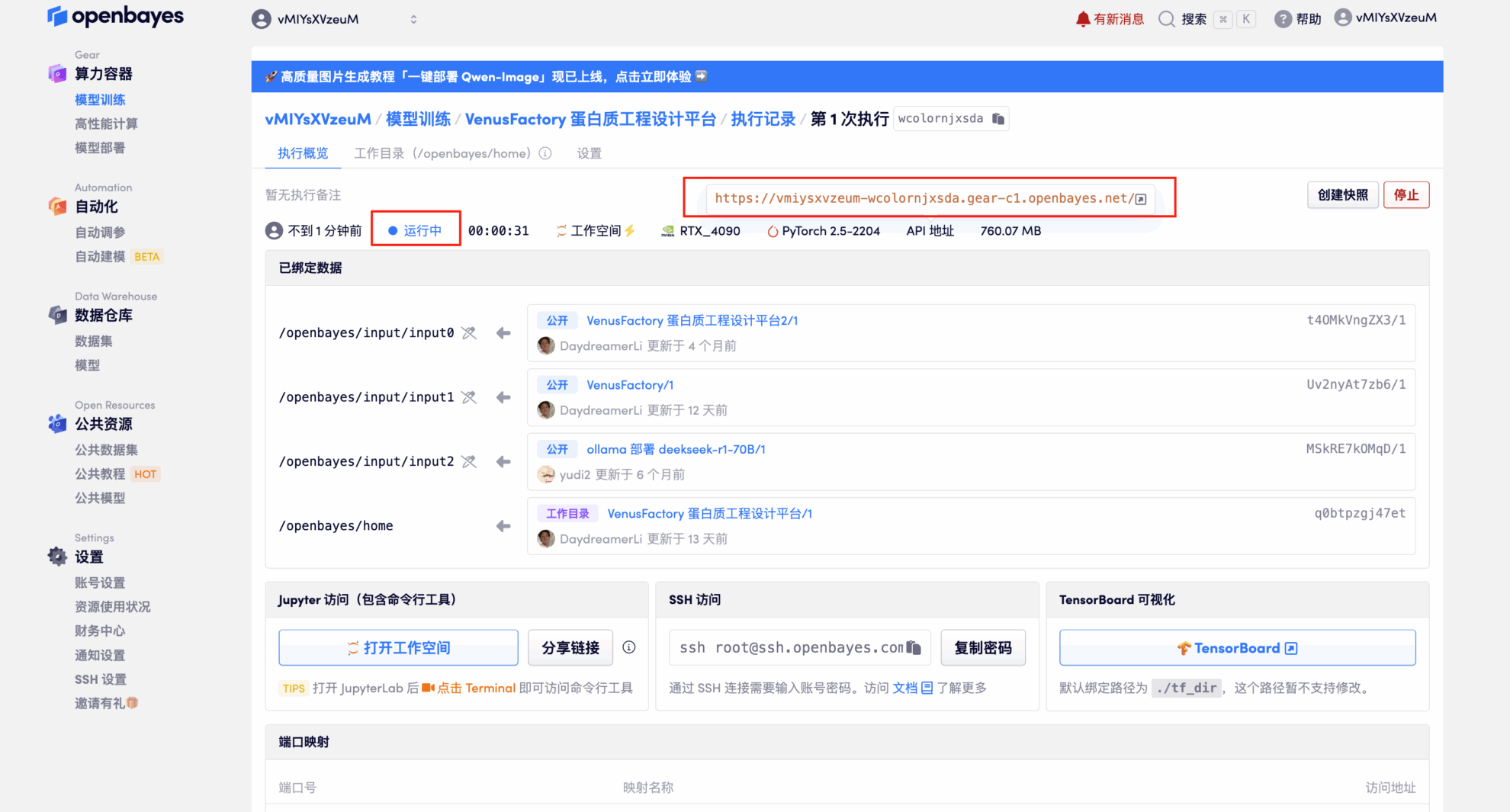
4. Attendez que les ressources soient allouées. Le premier clonage prendra environ 2 minutes. Lorsque le statut passe à « En cours d'exécution », cliquez sur la flèche à côté de « Adresse API » pour accéder à la page de démonstration.Étant donné que le modèle est volumineux, il faut environ 3 minutes pour que l'interface WebUI s'affiche, sinon « Bad Gateway » s'affichera.Veuillez noter que les utilisateurs doivent effectuer l'authentification par nom réel avant d'utiliser la fonction d'accès à l'adresse API.
Démonstration d'effet
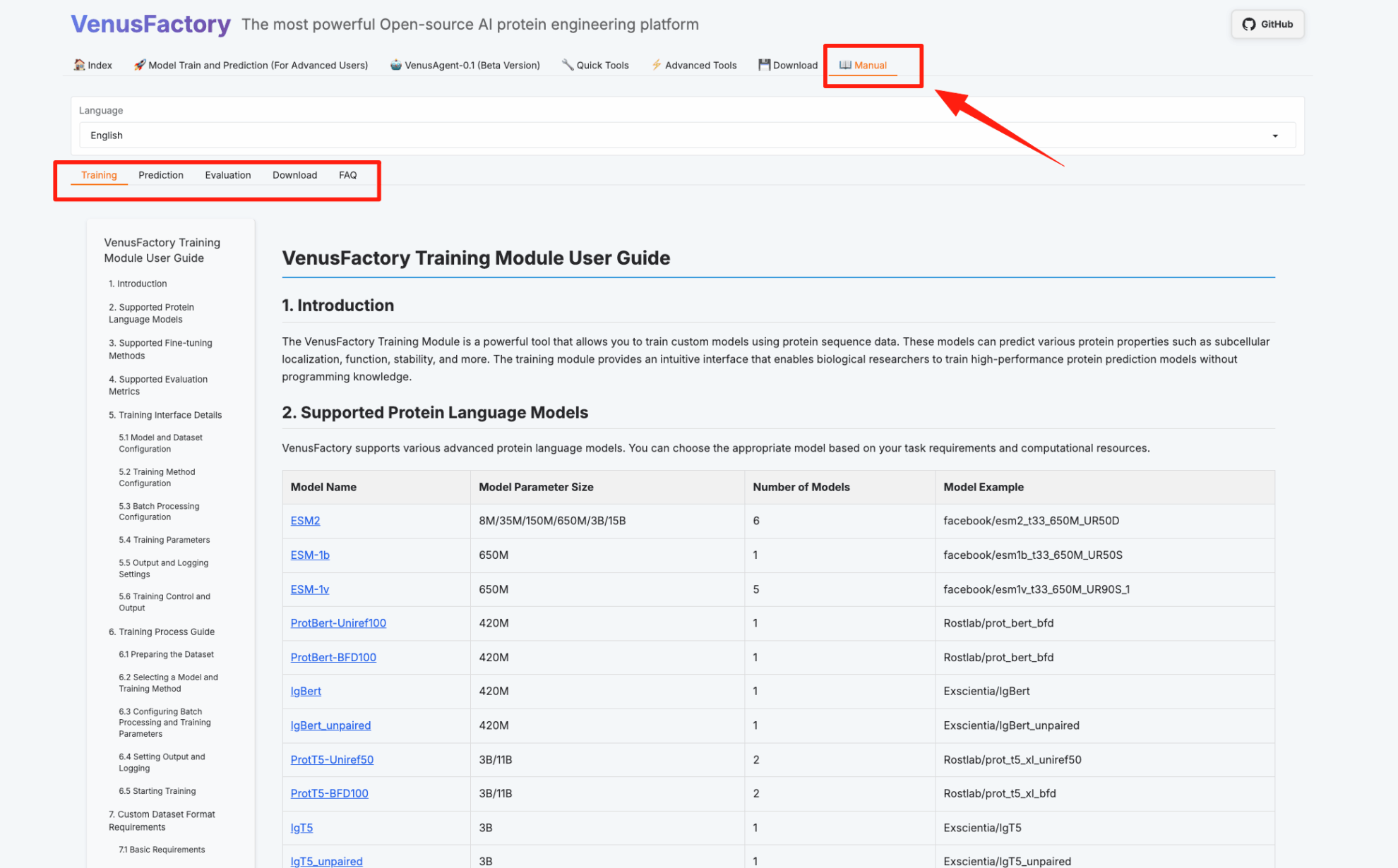
Voici la page d'utilisation de VenusFactory. Cliquez sur « Manuel » pour accéder directement aux guides d'utilisation des modules d'entraînement, de prédiction, d'évaluation et de téléchargement :
Affichage du module de formation
Cliquez sur le module « Formation » dans le module « Formation au modèle et à la prédiction » :
* Sélectionnez le modèle de langage des protéines
* Sélection de l'ensemble de données
* Aperçu du jeu de données
* Configuration de la méthode de formation (se référer au guide d'utilisation pour des informations spécifiques)
* Configuration par lots (voir le Guide de l'utilisateur pour plus de détails)
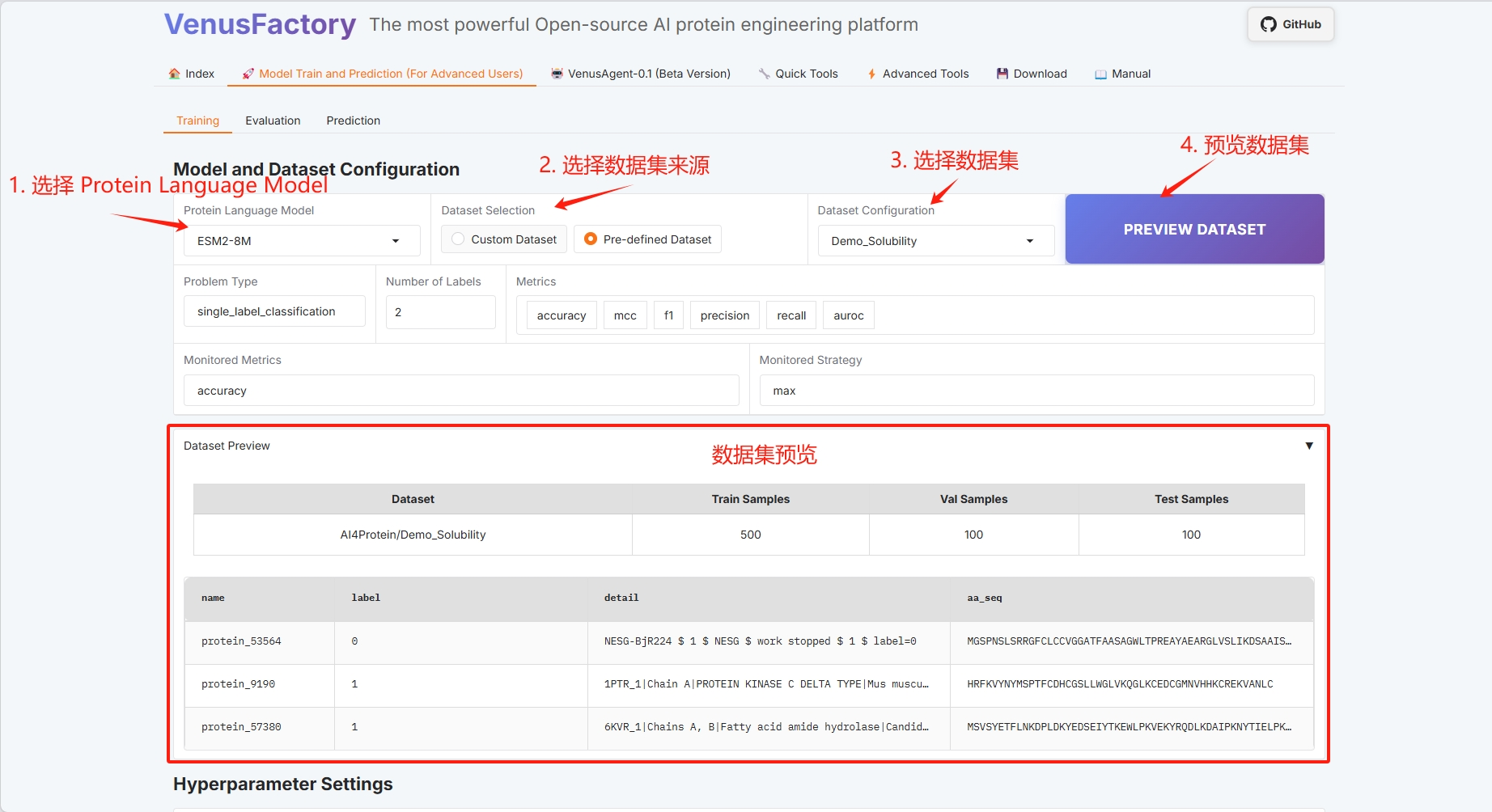
Définissez le chemin d'enregistrement du modèle de formation et cliquez sur « DÉMARRER LA FORMATION » pour démarrer la formation.
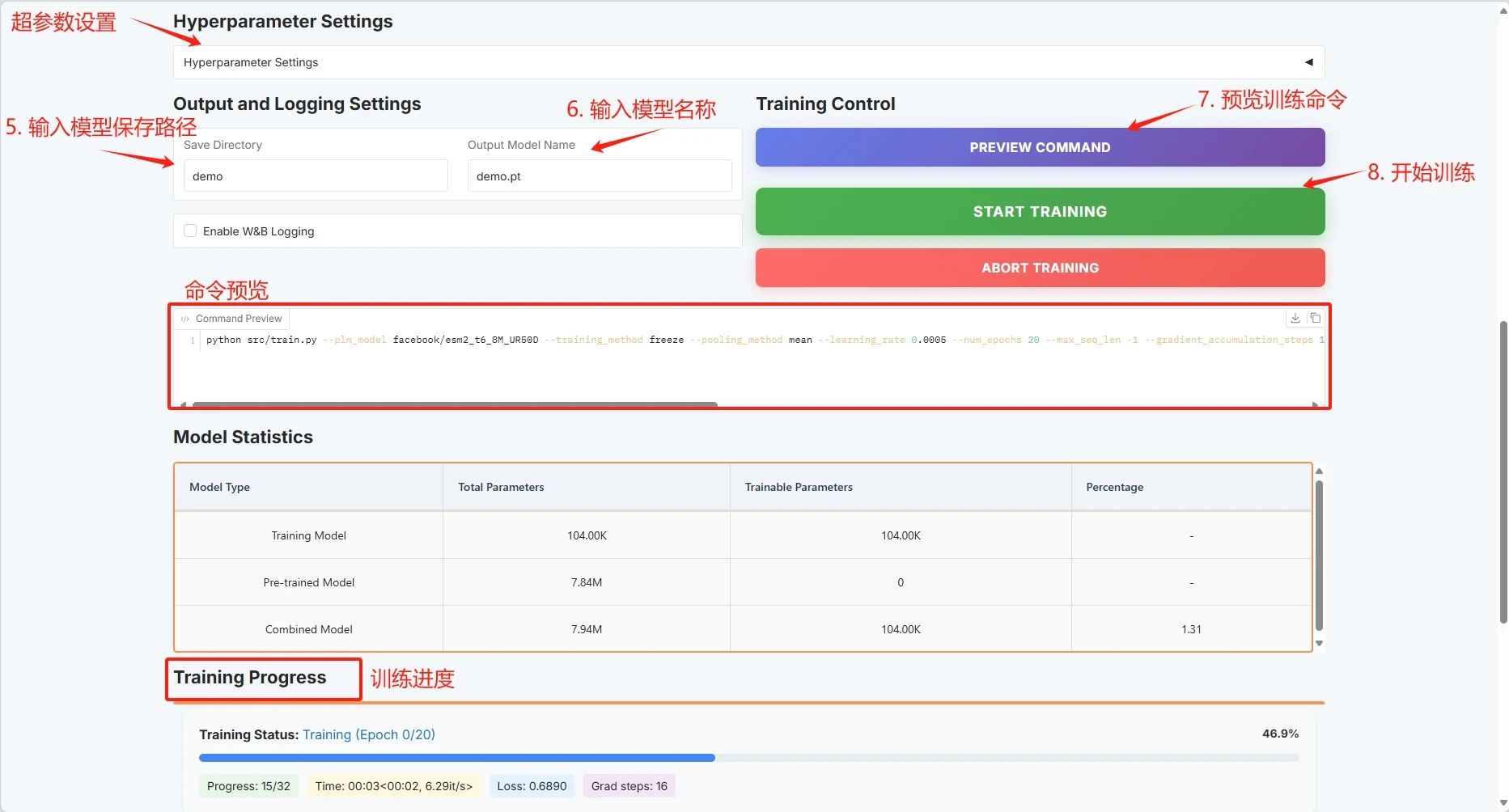
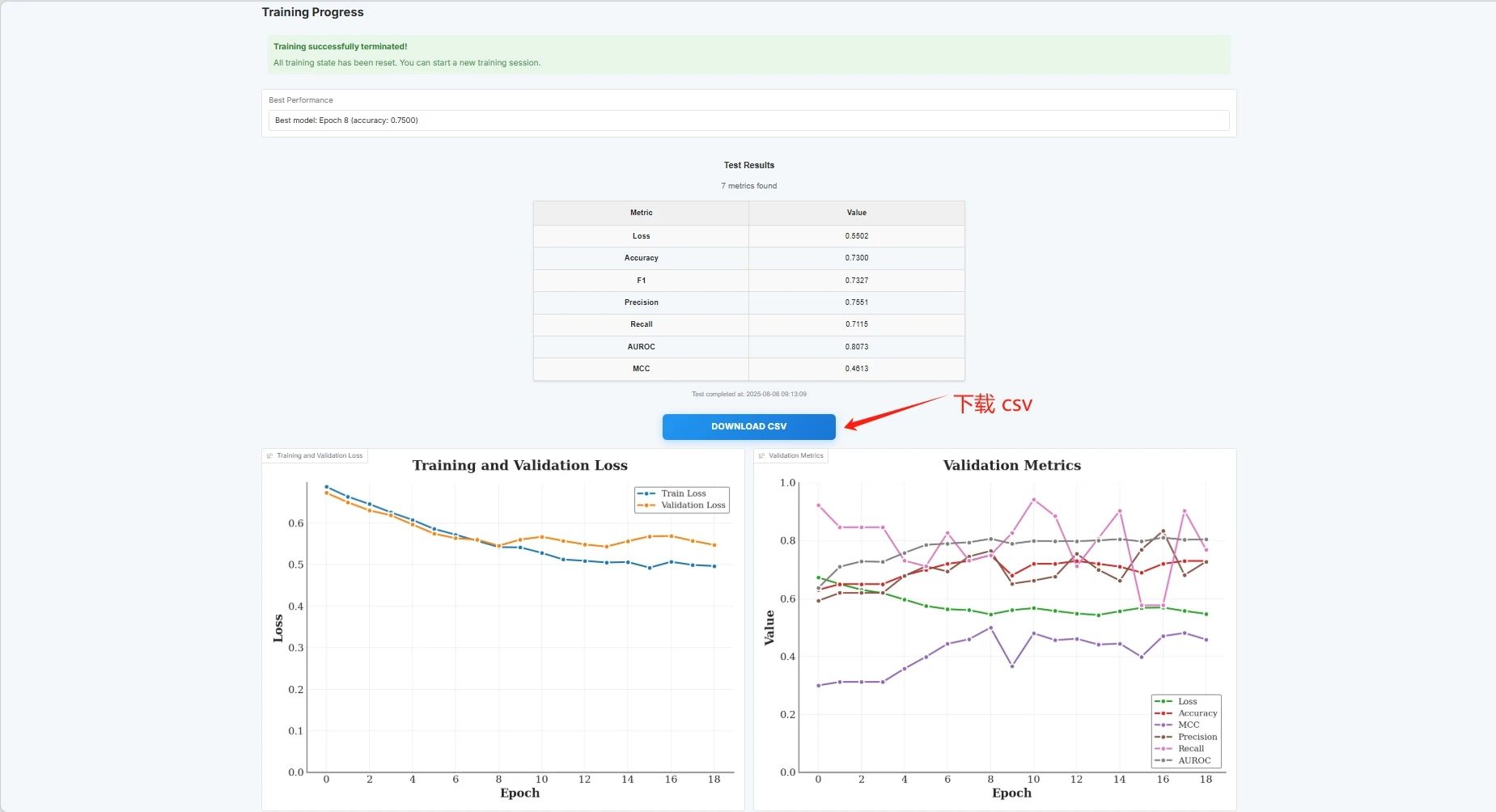
À ce stade, vous pouvez voir les paramètres d’entraînement et la courbe de perte :
Si vous souhaitez utiliser votre propre jeu de données, vous pouvez utiliser la configuration Jeu de données personnalisé. Il vous suffit de renseigner le chemin d'accès à votre jeu de données (consultez la documentation du manuel pour plus de détails).
Affichage du module d'évaluation
Cliquez sur le module « Évaluation » dans le module « Formation de modèles et de prédiction ».
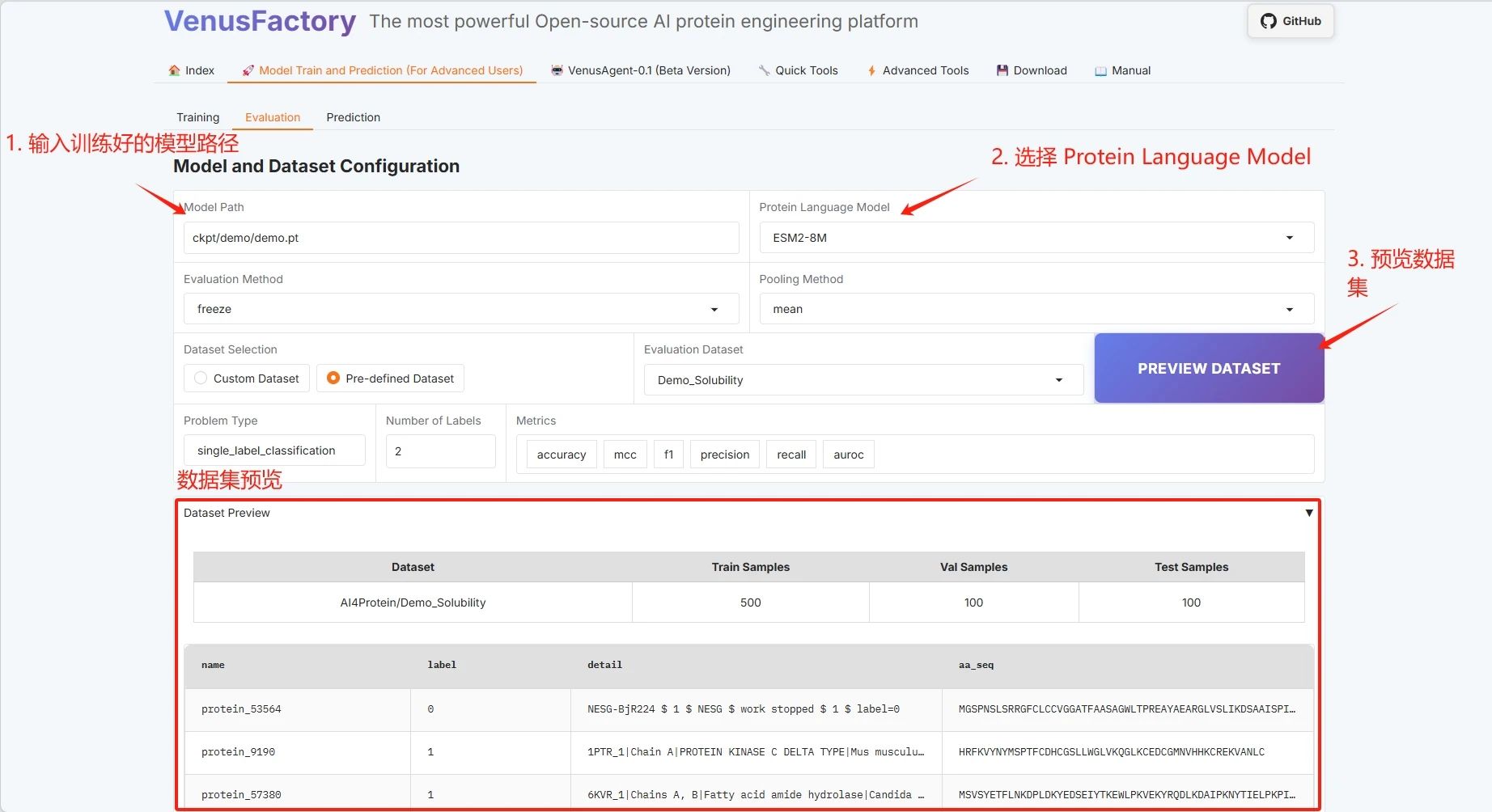
Configuration par lots, cliquez sur « DÉMARRER L'ÉVALUATION » pour démarrer la formation.
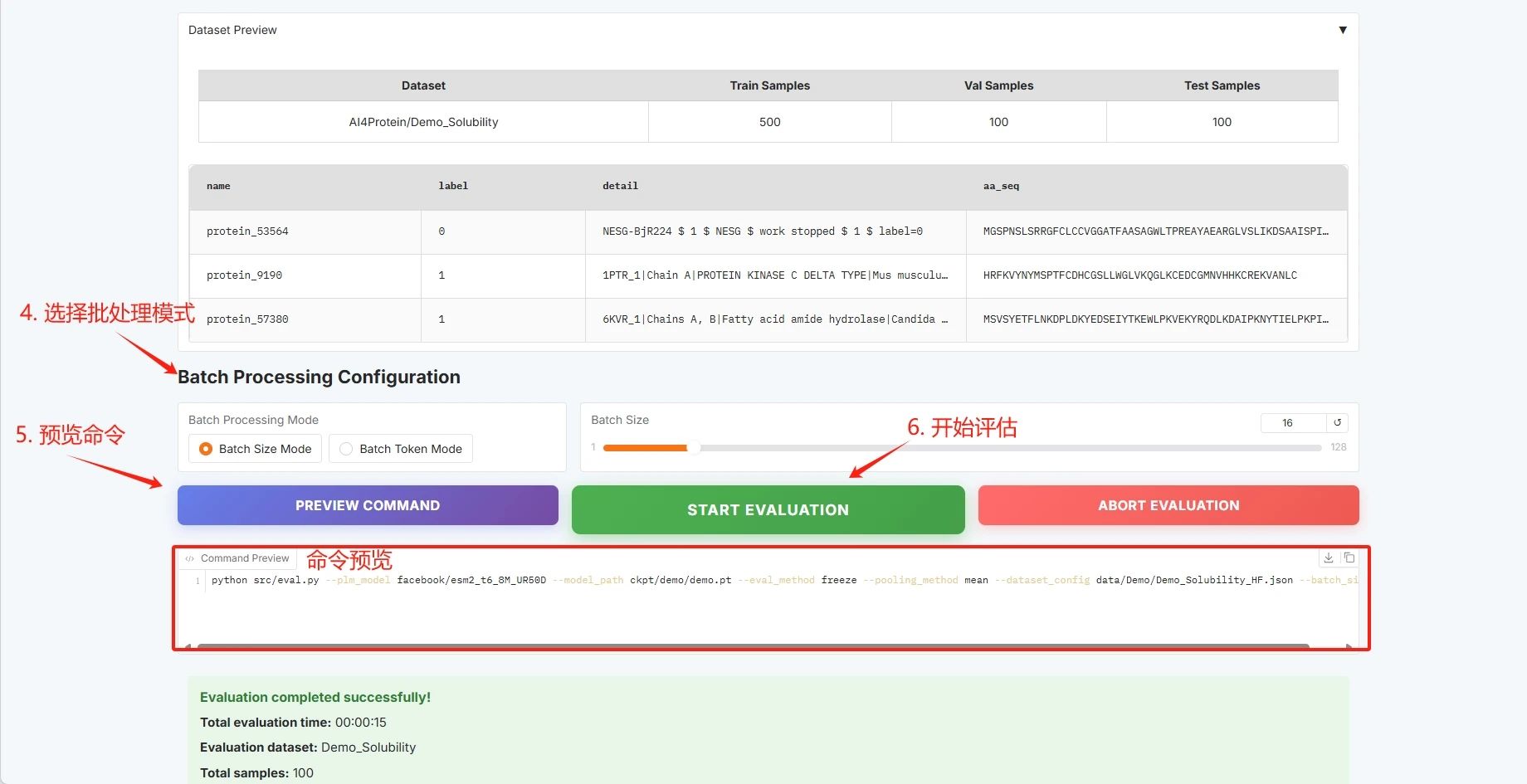
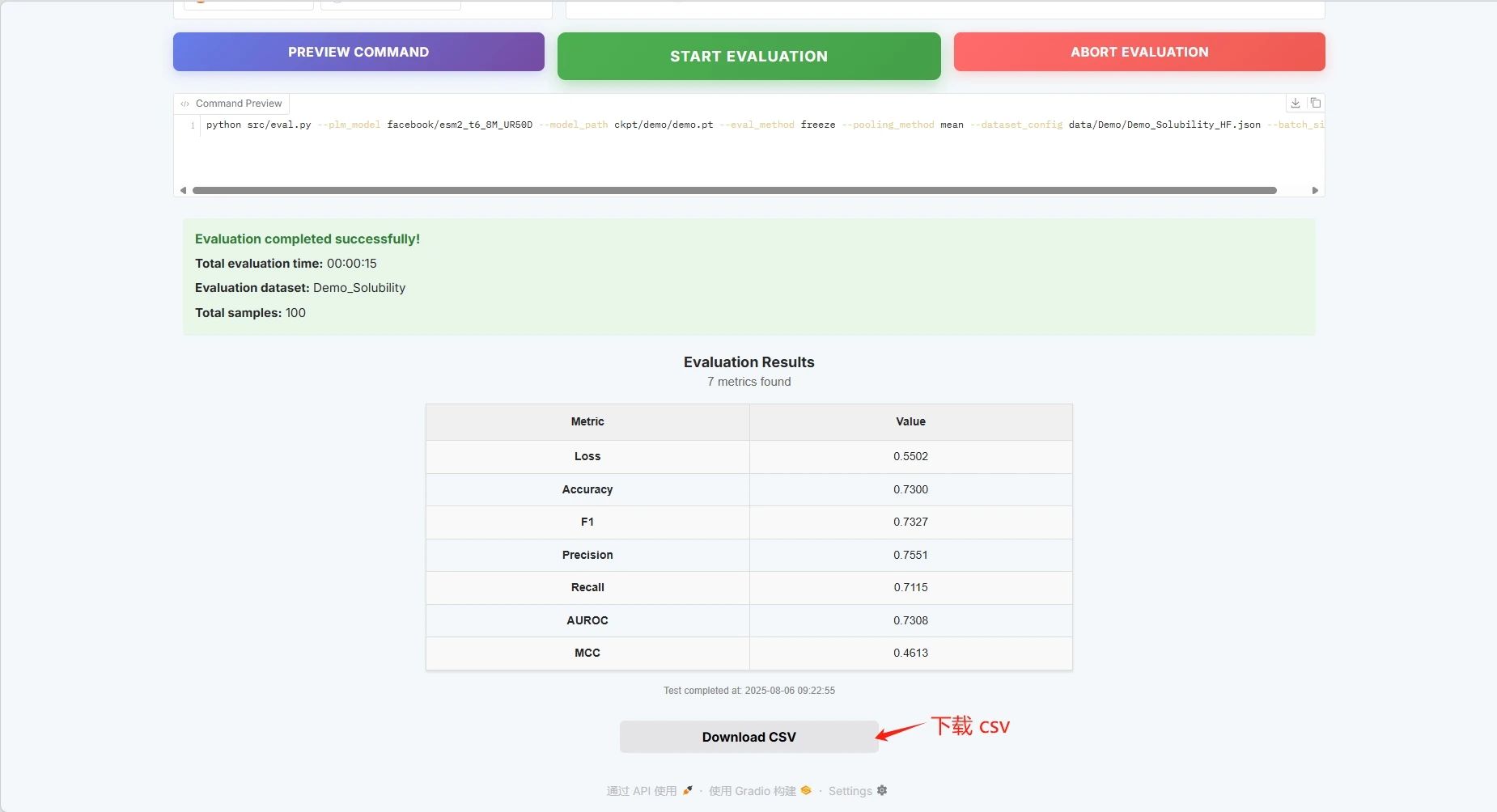
Les résultats de l'évaluation sont les suivants et vous pouvez télécharger le fichier CSV :
Si vous souhaitez utiliser votre propre jeu de données, vous pouvez utiliser la configuration Jeu de données personnalisé. Il vous suffit de renseigner le chemin d'accès à votre jeu de données (consultez la documentation du manuel pour plus de détails).
Affichage du module de prédiction
Cliquez sur le module « Prédiction » dans le module « Formation de modèles et de prédiction », définissez le chemin d'enregistrement du modèle de formation, sélectionnez le modèle de langage protéique et cliquez sur « DÉMARRER LA PRÉDICTION » pour démarrer la formation.
Prenons l’exemple de la prédiction d’une séquence unique :
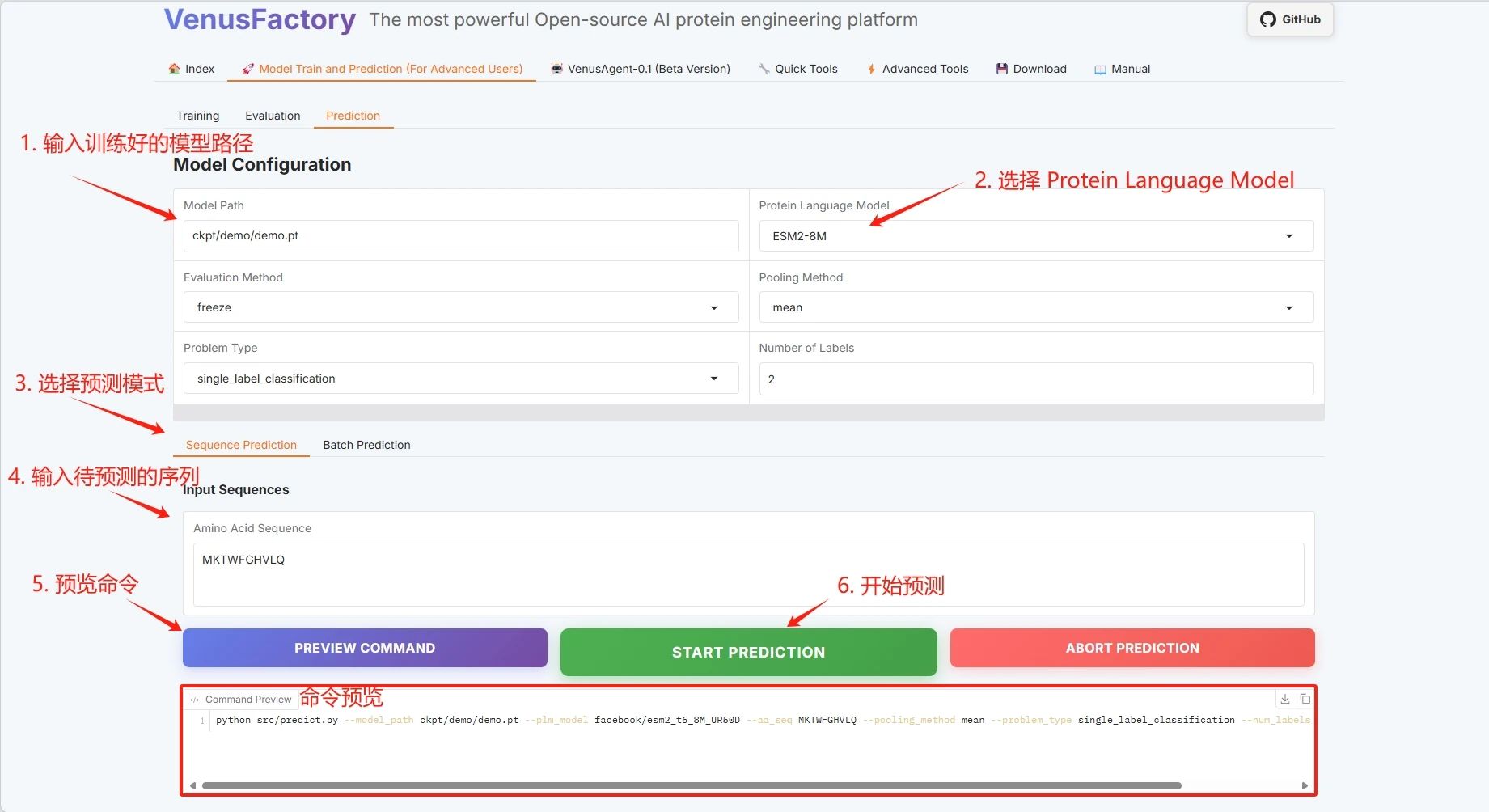
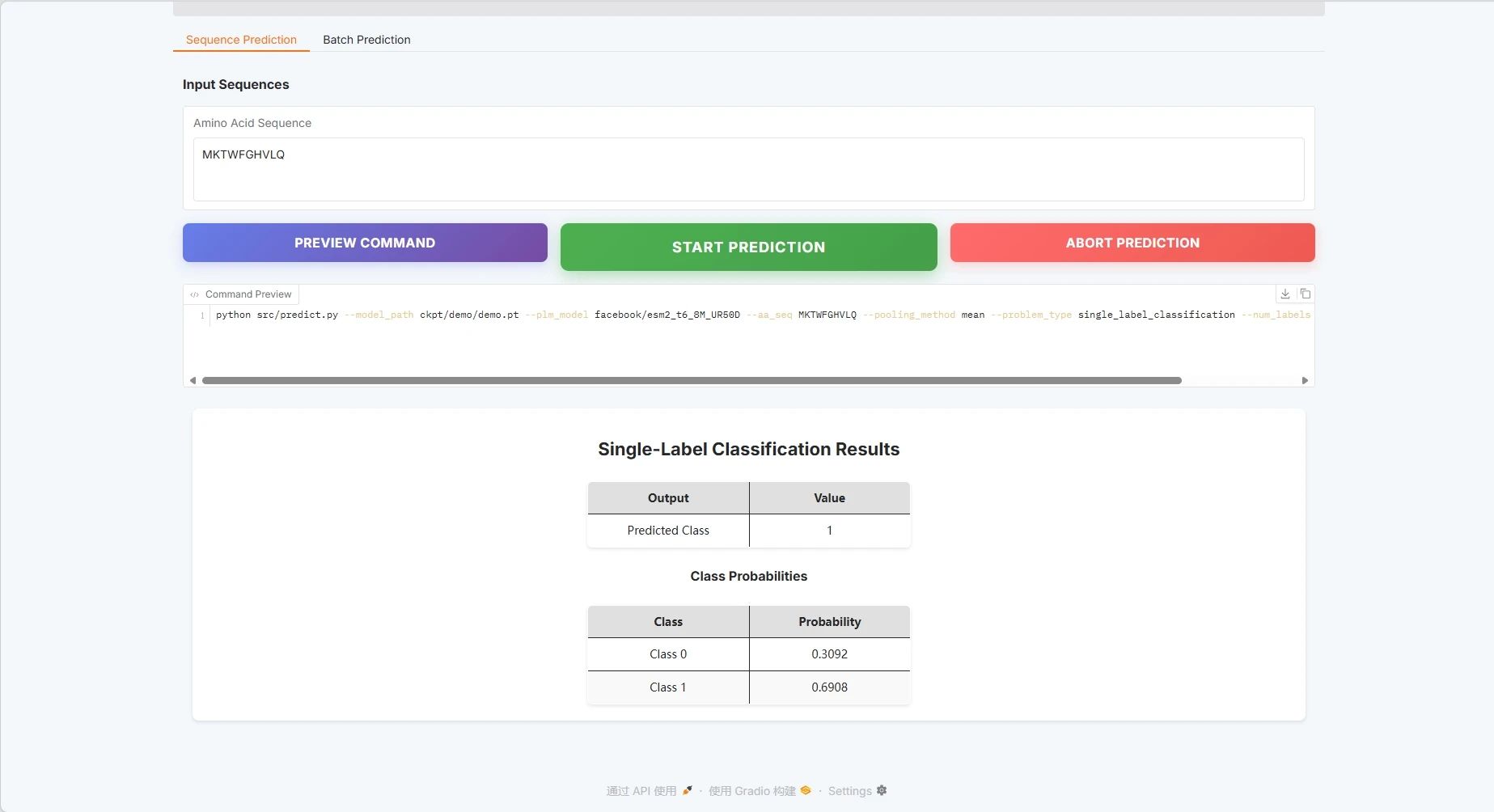
Exemple de séquence protéique : MKTWFGHVLQ
Présentation de VenusAgent
Cliquez sur le module VenusAgent.
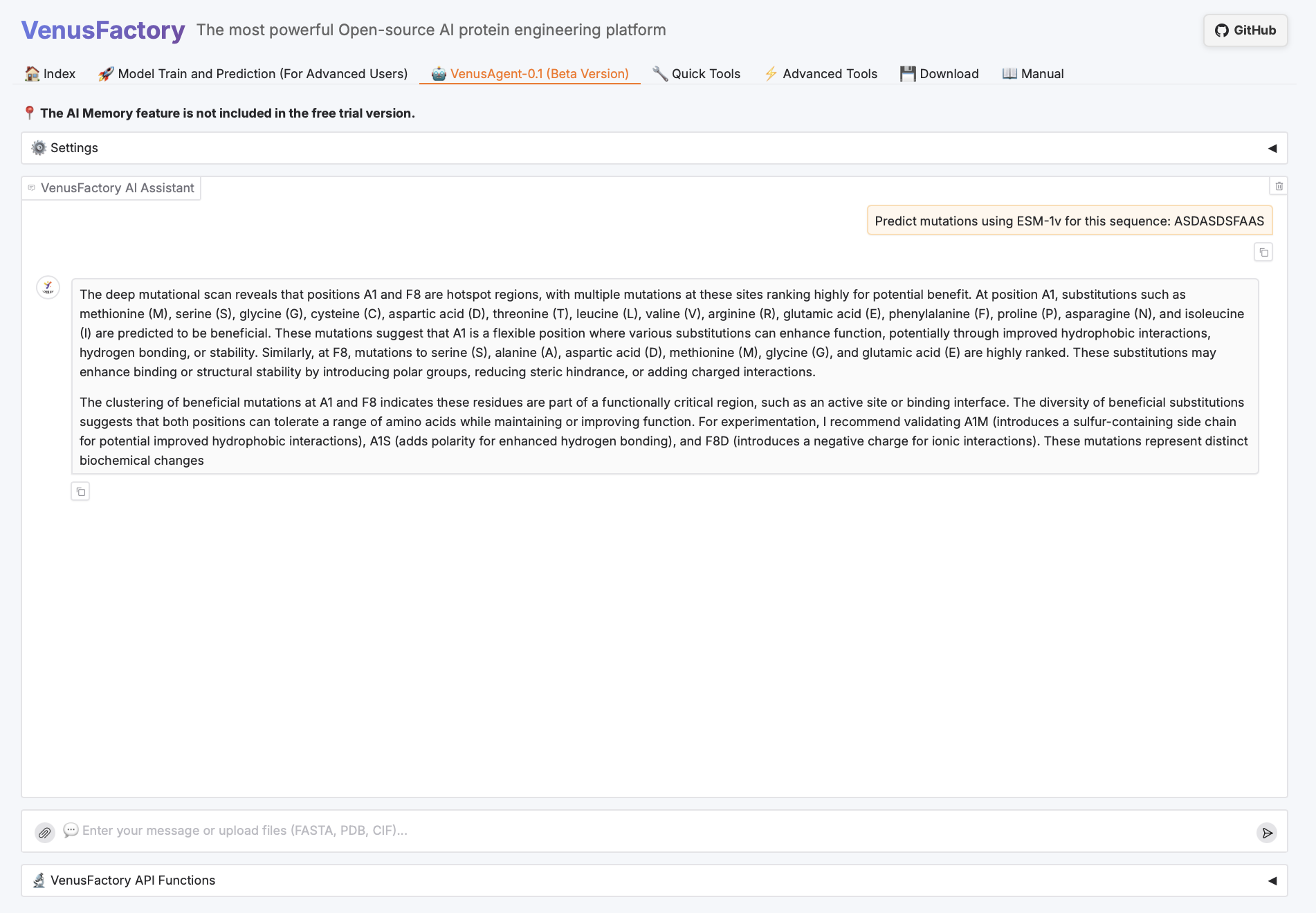
VenusAgent nécessitant des modèles DeepSeek volumineux, ce tutoriel propose deux méthodes d'appel : saisir vous-même la clé API ou utiliser le modèle DeepSeek-R1-70B déployé sur la plateforme. Vous pouvez choisir différentes cartes graphiques selon les fonctionnalités requises. Les instructions de sélection sont les suivantes :
* Si vous utilisez une seule carte graphique RTX 4090, la fonction VenusAgent ne prend pas en charge l'utilisation de services de modèles volumineux déployés localement (l'utilisation de la clé API DeepSeek est illimitée).
* Si vous utilisez deux cartes graphiques RTX 4090, vous ne pouvez pas utiliser d'autres fonctions immédiatement (après 1 à 2 minutes) après avoir utilisé la fonction VenusAgent (il n'y a aucune restriction lors de l'utilisation de la clé API DeepSeek).
* Si vous utilisez deux cartes graphiques RTX A6000, les fonctions de VenusAgent sont illimitées.
* Les utilisateurs peuvent saisir la clé API DeepSeek. Sinon, le service de modèle volumineux déployé localement dans le tutoriel est utilisé par défaut. Avec ce service, le temps de réponse pour la première conversation est d'environ 2 à 3 minutes. Merci de votre patience.
Ce qui précède est un tutoriel détaillé sur la façon d'utiliser la « VenusFactory Protein Engineering Design Platform ». Tout le monde est le bienvenu pour venir en faire l'expérience !
Lien du tutoriel :
Obtenez des articles de haute qualité et des articles d'interprétation approfondis dans le domaine de l'IA4S de 2023 à 2024 en un seul clic⬇️









