Command Palette
Search for a command to run...
Peut Inhiber La Prolifération Des Cellules Cancéreuses ! L'École De Pharmacie De Huihu Et l'Université De Médecine De Tianjin Ont Développé Conjointement Un Nouveau Dégradateur De Protéine Suppresseur De Tumeur dp53m

Peut-être que beaucoup de gens ne le savent pas,En fait, chacun d’entre nous a des cellules cancéreuses dans son corps.Le corps humain subit la création et le remplacement de milliards, voire de dizaines de milliards de cellules chaque jour. Au cours de ce processus métabolique, la réplication de l’ADN va inévitablement mal se passer, comme par exemple des mutations génétiques qui transforment des cellules normales en cellules cancéreuses in situ. mais,Il existe certaines protéines suppressives de tumeurs dans le corps humain.Comme la p53, ces protéines peuvent freiner l’apparition du cancer en régulant le cycle cellulaire et en induisant l’apoptose ou la sénescence des cellules cancéreuses. C’est également une raison importante pour laquelle la plupart d’entre nous peuvent « coexister pacifiquement » avec les cellules cancéreuses.
La protéine suppresseur de tumeur p53, connue sous le nom de « gardien du génome », est codée par le gène TP53 et joue un rôle important dans la prévention de l'apparition du cancer.Cependant, TP53 peut subir des mutations faux-sens dans plusieurs points chauds spécifiques communs, tels que R175, G245, R248, R273 et R282, entraînant des mutants p53 et conduisant à la perte de la fonction normale de la protéine suppressive de tumeur. De plus, en raison de l'effet négatif dominant de certains mutants p53, non seulement le mutant p53 perdra sa fonction suppresseur de tumeur d'origine, mais il interférera également avec l'activité suppresseur de tumeur du p53 de type sauvage normal (p53-WT), augmentant ainsi le risque d'apparition de tumeur.
Comparé à plusieurs autres mutants p53,La protéine mutante p53-R175H présente un potentiel plus élevé de tumorigenèse, de métastase et de résistance aux médicaments.Développer des médicaments ciblant p53-R175H et permettre aux médicaments ciblés d’identifier et de dégrader avec précision p53-R175H est une stratégie efficace pour inhiber l’apparition du cancer. Cependant, comme la plupart des protéines p53 mutantes manquent de sites actifs de médicaments à petites molécules, il devient extrêmement difficile de concevoir des médicaments ciblés capables de reconnaître et d'affecter avec précision les protéines p53 mutantes.
À cet égard,Le professeur Wu Sijin de l'École de pharmacie Huihu de l'Université Xi'an Jiaotong-Liverpool, en compagnie du professeur Xie Songbo et du professeur Zhong Diansheng de l'Hôpital général de l'Université médicale de Tianjin,Un article intitulé « Un PROTAC à base d'aptamère d'ADN conçu pour une thérapie précise du cancer induit par un mutant du point chaud p53-R175H » a été publié dans Elsevier.
Cette étude a développé un dégradateur sélectif de p53-R175H, dp53m.Ce dégradateur peut reconnaître spécifiquement la protéine mutante p53-R175H et utiliser le système naturel de dégradation des protéines dans la cellule - le système de protéase d'ubiquitine pour obtenir une dégradation ciblée de la protéine cible et inhiber l'expression fonctionnelle de la protéine mutante p53.
Le dégradant a une efficacité antitumorale significative et aucune réaction toxique évidente. De plus, le dp53m peut également fonctionner en synergie avec le médicament de chimiothérapie cisplatine pour améliorer la sensibilité des cellules cancéreuses au cisplatine, ce qui est crucial pour le traitement du cancer.
Points saillants de la recherche :
- Les chercheurs ont modifié les bases non essentielles sous la direction de simulations MD et ont guidé l'aptamère d'ARN vers un aptamère d'ADN (p53m-DA) par une méthode post-SELEX guidée par amarrage moléculaire itératif.
- En tant que composant de dp53m, p53m-DA peut reconnaître spécifiquement la protéine p53-R175H. Le CRBN est impliqué dans la dégradation des protéines, donc le dp53m peut reconnaître spécifiquement la protéine p53-R175H et la dégrader.

Adresse du document :
https://pubmed.ncbi.nlm.nih.gov/38811338
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit également des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Ingénierie post-SELEX : Construction d'un aptamère d'ADN haute performance qui reconnaît spécifiquement le mutant p53-R175H
Dans l’étude de p53, les chercheurs ont découvert que la mutation R175H affecte la fonction de liaison à l’ADN en modifiant l’état structurel de p53. La combinaison de plusieurs petites molécules inhibitrices et d'aptamères peut restaurer partiellement l'activité de p53-R175H, mais le mécanisme moléculaire de son influence n'a pas été rapporté.
Cette étude a d’abord utilisé des méthodes de simulation moléculaire pour explorer le mécanisme moléculaire intrinsèque des aptamères dans la restauration de la fonction de p53-R175H.Par rapport au p53-WT normal, dans le mutant p53-R175H, la distance entre L3 et l'hélice C est plus proche et la distance entre L2 et L3 est plus grande. Les changements structurels à ces positions peuvent être la principale raison pour laquelle le mutant p53-R175H perd sa fonction typique et fournit également une base structurelle pour la reconnaissance spécifique de ses aptamères et des molécules PROTAC apparentées.
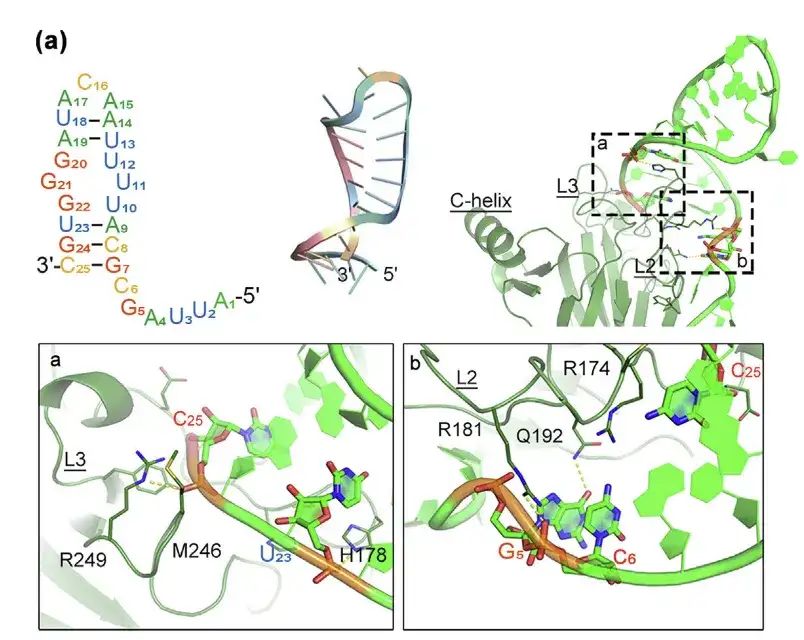
- Prédiction de la structure de p53m-RA et de son interaction avec la protéine p53-R175H à l'aide de la modélisation moléculaire
- L'interface de liaison est représentée dans les régions a et b.
Dans des études précédentes, il a été démontré que l'aptamère d'ARN ciblant p53-R175H (p53m-RA) avait une forte affinité pour p53-R175H, cependant, il est très instable dans le sérum, ce qui limite son application pratique.
Références :
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9884801
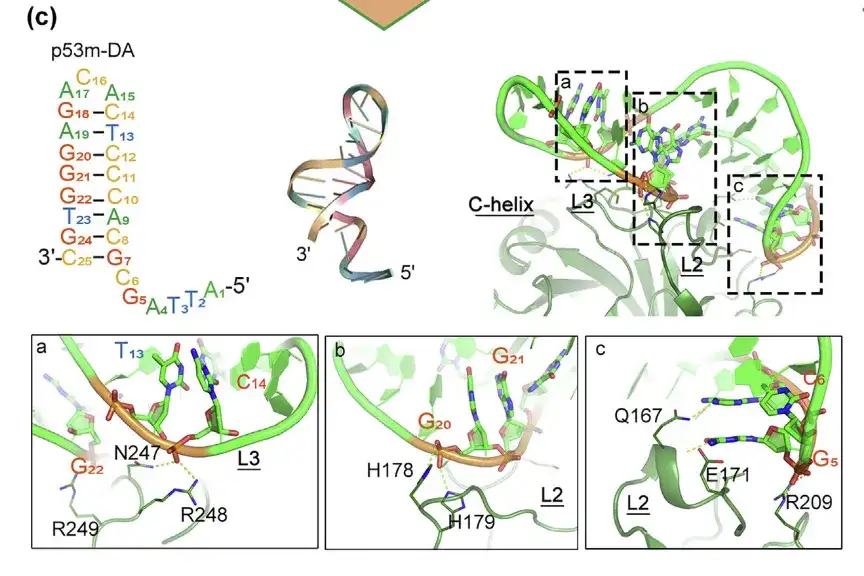
p53m-DA optimisé post-SELEX et son interaction avec p53-R175H
L'interface de liaison se trouve dans les régions a, b et c
Pour améliorer la stabilité structurelle et l'affinité de l'aptamère, les chercheurs ont modifié les bases non essentielles sous la direction de la simulation MD et ont conçu l'aptamère d'ARN en un aptamère d'ADN (p53m-DA) via post-SELEX.
La puissance de calcul des simulations moléculaires dans cette étude a été fournie par OpenBayes fournir
Les résultats ont montré que la structure de la région appariée de p53m-DA était un sillon majeur et que la région de boucle non appariée était un sillon mineur. Le sillon majeur joue un rôle clé dans la liaison avec H178, H179, E171 et Q167 dans L2, et le sillon mineur joue un rôle clé dans l'interaction avec N247, R248 et R249 dans L3.De plus, le p53m-DA a maintenu en permanence une conformation hélicoïdale double brin tout au long de la simulation, ce qui indique la stabilité de sa structure.
L2 et L3 sont des régions présentes dans la protéine mutante
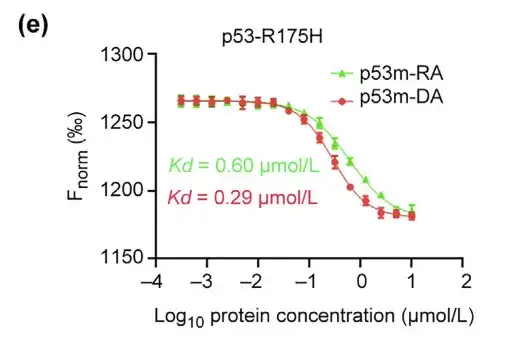
Les chercheurs ont également évalué l’affinité et la sélectivité du p53m-DA pour le p53-R175H. Le test MST a montré que le Kd du p53m-DA était de 0,29 lmol/L, ce qui était environ 2 fois inférieur à celui du p53m-RA.
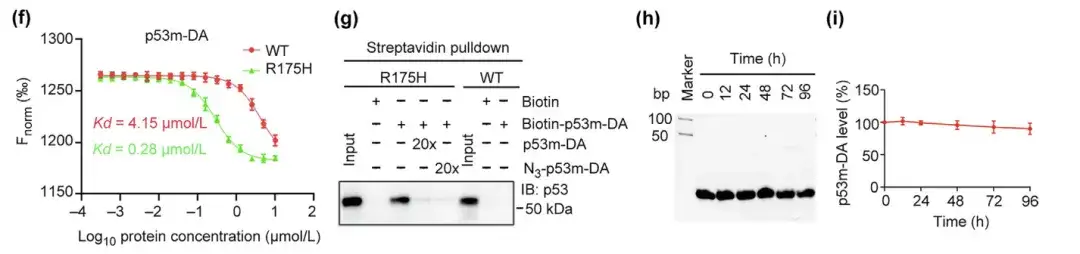
(f) Affinité de p53m-DA pour p53-WT et p53-R175H
(g) Liaison spécifique de p53m-DA à p53-R175H par test de pulldown à la streptavidine
(h,i) Évaluation de la stabilité sérique du p53m-DA et détermination de la quantité résiduelle de p53m-DA
De plus, l'affinité du p53m-DA pour le mutant p53-R175H était 14 fois plus élevée que son affinité pour le p53-WT normal. Dans le test de pulldown à la streptavidine, le p53m-DA s'est spécifiquement lié au p53-R175H mais n'a pas interagi avec le p53-WT, indiquant la spécificité du p53m-DA pour le p53-R175H. Notamment, la stabilité sérique du p53m-DA a été significativement améliorée, avec une dégradation minimale après 96 h.
En résumé,p53m-DA est un aptamère doté d'une spécificité et d'une stabilité élevées.
dp53m : un dégradateur spécifique de p53-R175H
Expérience 1 : Synthèse de DP53M
L'extrémité 5' de l'aptamère p53m-DA est située à l'extérieur de son site de liaison avec p53-R175H. Les chercheurs ont utilisé une réaction de clic pour conjuguer l'alcynyl thalidomide (un ligand de la ligase CRBN E3) à l'extrémité 5' de N3-p53m-DA. La chimère ciblant la dégradation des protéines résultante, PROTAC (nommée dp53m), a pu se lier sélectivement à p53-R175H. La structure du dp53m est illustrée dans la figure ci-dessous :
*PROTAC : se compose d'un ligand ligase E3, d'un ligand ciblant le POI et d'un lieur chimique entre les ligands
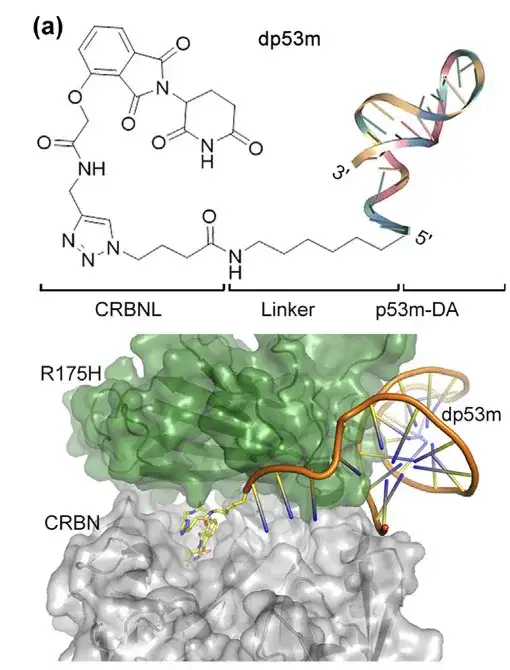
(a) La structure de dp53m a été simulée à l'aide de calculs d'amarrage protéine-protéine et de dynamique moléculaire.
En bas : Son interface de liaison avec p53-R175H et CRBN
Expérience 2 : dp53m peut reconnaître et dégrader spécifiquement le mutant p53-R175H
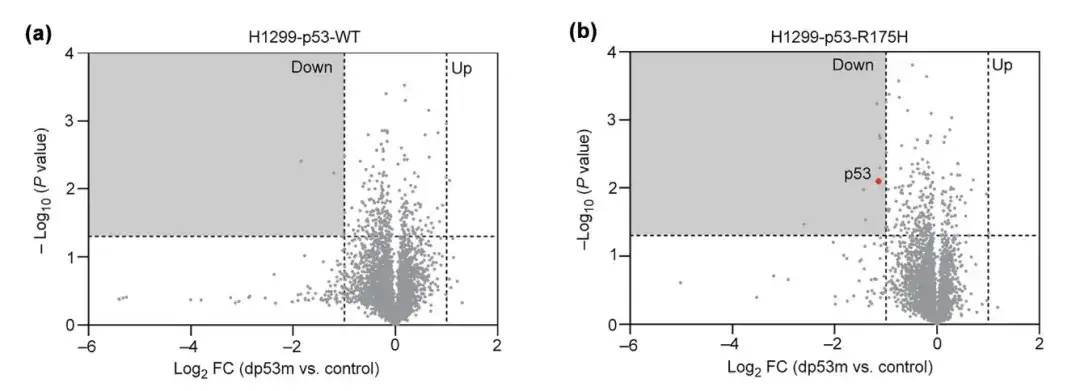
Pour étudier la spécificité cellulaire de dp53m pour p53-R175H, p53-R175H et p53-WT surexprimés dans les cellules H1299 ont été traités avec du PBS ou du dp53m pendant 16 h et leurs niveaux de changement de protéines ont été surveillés. Les résultats ont montré que le dp53m dégradait significativement le p53-R175H mais pas le p53-WT.
*Le PBS est un tampon phosphate salin, utilisé comme groupe témoin
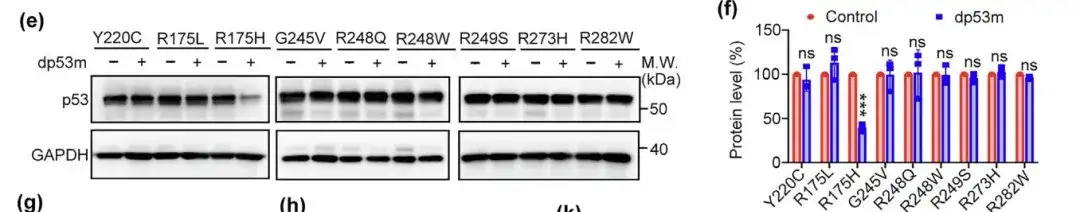
De plus, les chercheurs ont également évalué le potentiel de dégradation du dp53m par rapport à d’autres mutants de points chauds tels que Y220C, R175L, G245V, R248Q, R248W, R249S, R273H et R282W. Les résultats ont montré que le dp53m ne dégradait efficacement que le p53-R175H.Sa spécificité pour la dégradation de p53-R175H a été démontrée.
Comparé au p53WT, le traitement au dp53m a augmenté la polyubiquitination du p53-R175H. Des études ont montré que la dégradation de dp53m peut être bloquée par l’inhibiteur de protéase MG132, le ligand CRBN ou l’ARNsi ciblant CRBN.On suppose que la dégradation de p53-R175H induite par dp53m se produit via le mécanisme ubiquitine-protéasome.
En résumé,dp53m est un dégradateur spécifique de p53-R175H, et son processus de dégradation se produit via le mécanisme ubiquitine-protéasome.
dp53m inhibe la prolifération et la migration des cellules cancéreuses ainsi que la croissance tumorale
Expérience 1 : dp53m inhibe la prolifération et la migration des cellules cancéreuses induites par p53-R175H
Les chercheurs ont traité les cellules H1299-p53-WT, H1299-p53-R175H, A549, Detroit 562 et SKBR3 avec PBS, p53m-DA ou dp53 m, respectivement, ont effectué des tests de viabilité sur elles et ont observé la formation de colonies dans les cellules.
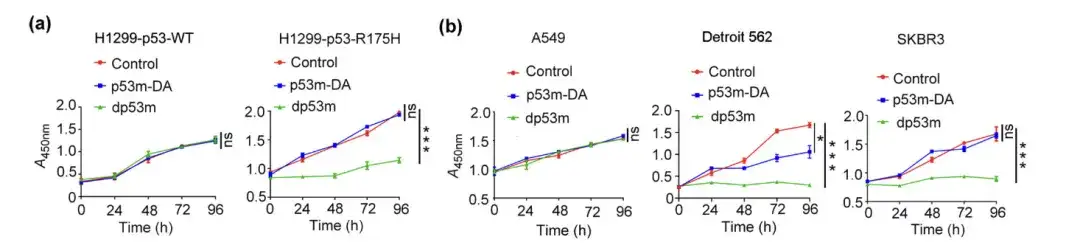
Dosage de la viabilité des cellules H1299-p53-WT, H1299-p53-R175H, A549, Detroit 562 et SKBR3 traitées avec du PBS, du p53m-DA ou du dp53m
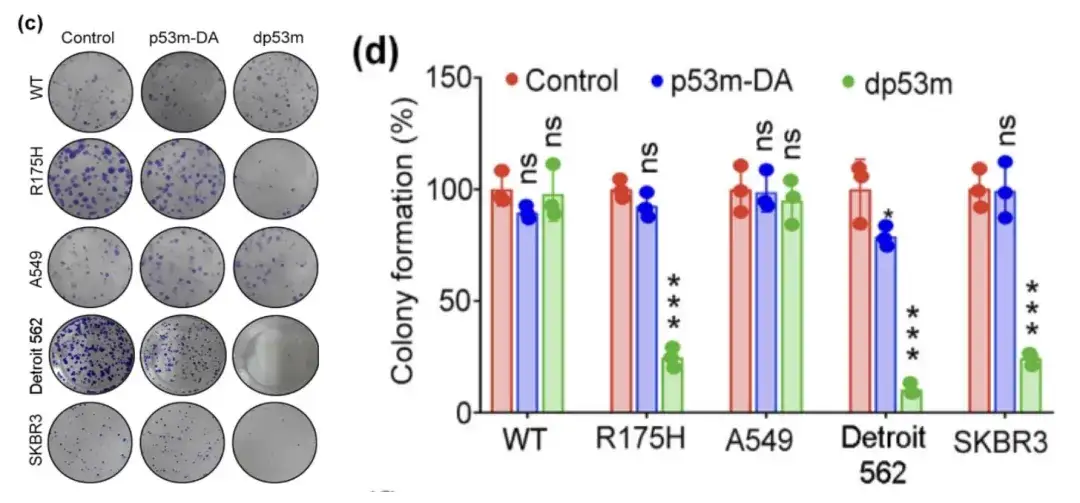
* dp53m inhibe la formation de colonies dans les cellules H1299-p53-R175H, Detroit 562 et SKBR3
* Aucun effet significatif n'a été observé dans les cellules H1299-p53-WT et A549
La figure ci-dessus montre que dp53m présente un effet inhibiteur de prolifération significatif dans les cellules cancéreuses exprimant p53-R175H (H1299-p53-R175H, Detroit 562 et SKBR3), tandis que les cellules exprimant p53-WT ne sont pratiquement pas affectées.Cela suggère que le dp53m a un grand potentiel thérapeutique dans les cellules cancéreuses porteuses de la mutation p53-R175H.
Expérience 2 : dp53m inhibe la croissance des tumeurs induites par p53-R175H in vivo
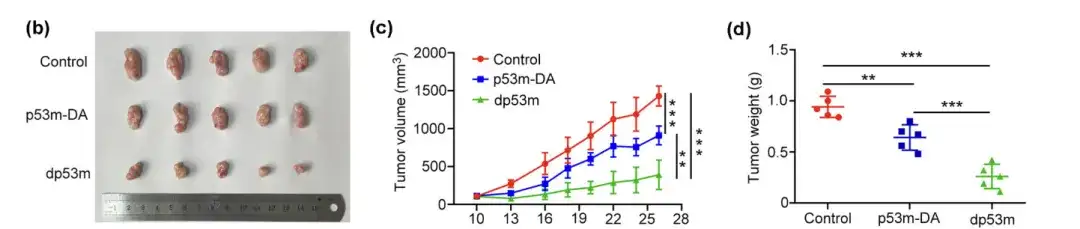
(b) Aspect macroscopique des tumeurs isolées après traitement
(c) Volume tumoral mesuré au moment indiqué
(d) Détermination du poids final de la tumeur
Les chercheurs ont réparti de manière aléatoire des souris BALB/c porteuses de tumeurs en trois groupes et leur ont administré du p53m-DA, du dp53m ou une solution saline comme contrôle par voie intraveineuse. Comparé au groupe témoin ou au p53m-DA,dp53m a inhibé de manière significative la croissance tumorale.
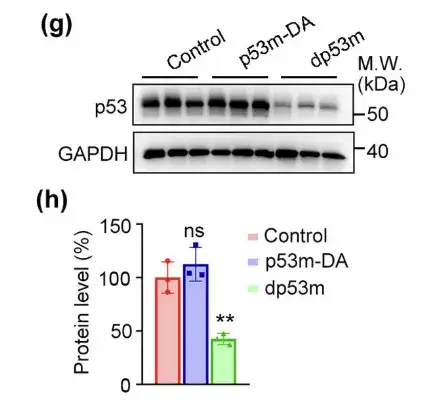
Comme prévu,dp53m a efficacement réduit le niveau d’expression de p53-R175H dans les tumeurs.
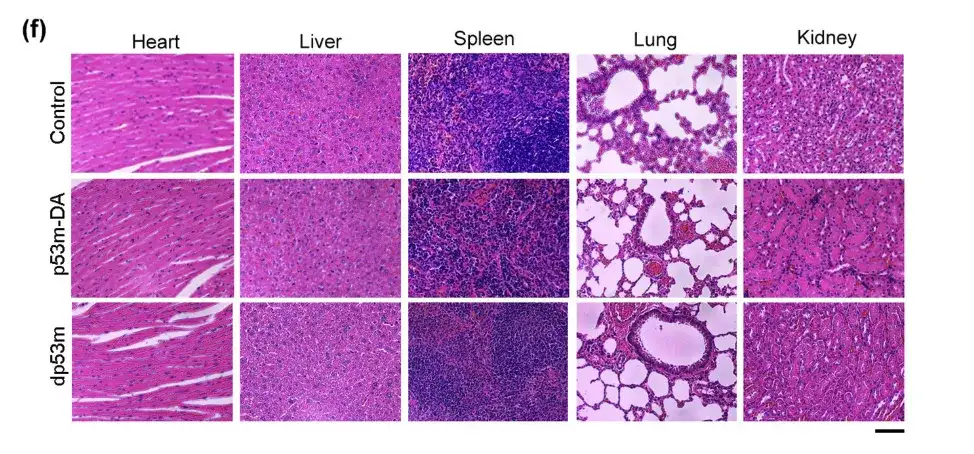
Dans la figure supérieure, il n’y avait aucune différence significative dans l’histologie des tissus du cœur, du foie, des poumons, de la rate et des reins entre les trois groupes de souris.Cela suggère qu’il n’y a aucune toxicité du dp53m in vivo.
En résumé,dp53m peut inhiber la croissance des tumeurs induites par p53-R175H sans toxicité évidente.
De plus, considérant que la mutation p53-R175H est associée à une résistance aux médicaments chimiothérapeutiques tels que le cisplatine, les chercheurs ont également analysé l'effet du dp53m sur l'activité antitumorale du cisplatine. Les résultats ont montré qu’il existait une forte interaction synergique entre le dp53m et le cisplatine.Autrement dit, le dp53m peut améliorer la sensibilité des cellules cancéreuses p53-R175H au cisplatine.Il s’agit d’un « partenaire invincible » pour le traitement du cancer.
En renforçant la ligne de défense contre le cancer, les scientifiques construisent ensemble une route d'espoir
Le cancer est une maladie causée par une croissance cellulaire incontrôlée. Son incidence a augmenté de manière significative avec le vieillissement de la population mondiale, l’augmentation de la pollution environnementale et les changements dans les habitudes de vie. Selon les données du Centre international de recherche sur le cancer (CIRC), le nombre de nouveaux cas de cancer dans le monde devrait passer d’environ 19,29 millions de cas en 2020 à environ 30,23 millions de cas en 2040, soit une augmentation de 56,71 %.
Les chercheurs jouent un rôle essentiel dans la lutte mondiale contre le cancer. Les 3 auteurs correspondants dans cette étude sont :M. Wu Sijin de l'École de pharmacie Huihu de l'Université Xi'an Jiaotong-Liverpool, le professeur Xie Songbo et le professeur Zhong Diansheng de l'Hôpital général de l'Université médicale de Tianjin sont des représentants exceptionnels dans ce domaine.
Parmi eux, le professeur Wu Sijin se concentre sur la recherche et le développement de nouveaux médicaments ciblés. Ses recherches actuelles portent sur l’utilisation de la conception et de la découverte de médicaments assistées par ordinateur pour identifier de nouveaux médicaments thérapeutiques à l’aide de méthodes de simulation telles que la modélisation, l’analyse pharmacophore, l’amarrage et la dynamique moléculaire (MD). Dans son étude de 2022, il a découvert que l'expression régulée à la hausse de SOST était associée à un mauvais pronostic chez les patientes atteintes d'un cancer du sein, et que SOST activait les voies de signalisation en aval pour favoriser la prolifération des cellules cancéreuses du sein et les métastases osseuses. Grâce à un criblage informatique, l’étude a identifié un composé thérapeutique candidat, le S6, qui peut perturber l’interaction entre SOST et STAT3 pour inhiber la phosphorylation de STAT3 et réduire les métastases osseuses du cancer du sein.
Adresse du document :
https://pubmed.ncbi.nlm.nih.gov/36581888
De plus, le professeur Xie Songbo, dont la principale orientation de recherche est la dégradation ciblée des protéines et l'administration de médicaments, a proposé dans son étude de 2023 qu'une nouvelle stratégie puisse être utilisée pour induire la dégradation de protéines « non médicamenteuses » en utilisant des aptamères comme « ogives ciblées ». Pour confirmer ce concept, les chercheurs ont choisi la nucléoline oncogène (NCL) comme cible et ont finalement généré une série de dégradeurs de NCL. dNCL#T1 a induit la dégradation du NCL d'une manière qui dépendait du système de protéase d'ubiquitine, qui pourrait inhiber la prolifération des cellules cancéreuses du sein induite par le NCL. Cette étude a non seulement fourni une nouvelle perspective sur la dégradation des protéines, mais a également posé des bases solides pour le développement de médicaments thérapeutiques pour des maladies telles que le cancer.
Adresse du document :
https://pubmed.ncbi.nlm.nih.gov/36608275
Enfin, le professeur Zhong Diansheng a également mené des recherches approfondies dans les domaines du diagnostic précoce, de la chimiothérapie, de la thérapie ciblée et de la thérapie anti-angiogénique du cancer du poumon, et a publié plus de 90 articles universitaires. Dans son étude de 2024, il a découvert que CBX4 favorisait la prolifération de l'adénocarcinome pulmonaire (LUAD) en régulant positivement l'expression de PHGDH et la biosynthèse de la sérine, tout en inhibant les métastases du LUAD en supprimant la transcription de ZEB2. Cette découverte nous aide à comprendre l’interaction entre CBX4 et les facteurs épigénétiques et fournit des informations sur les approches thérapeutiques potentielles pour le LUAD.
Adresse du document :
https://www.nature.com/articles/s41419-024-06745-z
En plus du professeur Wu Sijin, du professeur Xie Songbo et du professeur Zhong Diansheng, il y a de nombreux scientifiques et médecins qui se consacrent silencieusement et travaillent dur. Nous attendons avec impatience le jour où le cancer ne sera plus un cauchemar pour l’humanité, grâce aux progrès de la science et de la technologie.








