Command Palette
Search for a command to run...
AlphaFold 3 Est Là ! Source Fermée Mais Disponible Pour La Recherche Scientifique, La Filiale De DeepMind Est À Portée De Main Et Avance d'abord Vers Une Utilisation Commerciale ?

Le 8 mai, heure locale, Google DeepMind et sa filiale Isomorphic Labs ont publié conjointement AlphaFold 3.
DeepMind a déclaré qu'AlphaFold 3 a prédit avec succès les structures et les interactions de toutes les molécules vivantes (protéines, ADN, ARN, ligands, etc.) avec une précision sans précédent. Par rapport aux méthodes de prédiction existantes,AlphaFold 3 a trouvé au moins 50% interactions supplémentaires entre les protéines et d'autres types moléculaires, et pour certaines catégories d'interactions importantes, sa précision de prédiction a doublé.
Les lecteurs qui suivent AI for Science connaissent certainement AlphaFold, qui a été salué comme une « avancée majeure » lors de sa première sortie. AlphaFold 2 a été nommé « Percée de l'année » par Science et « Méthode de l'année » par Nature en 2021, ouvrant la voie et repoussant constamment les limites d'application de l'IA + biologie.
Aujourd’hui, AlphaFold 3 a une fois de plus rafraîchi la cognition des gens. Comment parvient-on à ce que « toutes les molécules de la vie soient prévisibles » ? Quelles avancées ont été réalisées en matière de technologie et de performance ? Quel rôle joue Isomorphic Labs, né de DeepMind ?
Le module Pairformer remplace Evoformer, le module de diffusion remplace le module de structure
Les chercheurs ont apporté des améliorations significatives à la structure et aux procédures de formation d’AlphaFold 2 pour s’adapter à des structures chimiques plus générales et améliorer l’efficacité des données d’apprentissage du modèle. Maintenant, étant donné une liste d'entrées moléculaires, AlphaFold 3 peut générer leur structure 3D conjointe et révéler comment elles s'assemblent.

Comme le montre la figure ci-dessus, la structure globale d'AlphaFold 3 est similaire à celle d'AlphaFold 2, tous deux dotés d'une grande épine dorsale pour l'évolution des représentations d'appariement de complexes chimiques. AlphaFold 3 remplace l'Evoformer d'AlphaFold 2 par un module Pairformer plus simple.Cela réduit la quantité de traitement d'alignement de séquences multiples (MSA) et le nombre de modules MSA est réduit à 4. La représentation MSA de AF3 est traitée à l'aide de la méthode de moyenne pondérée par paire la moins chère, et seule la représentation par paire est utilisée dans les étapes de traitement suivantes.

Les chercheurs ont observé dans AlphaFold 2 que la suppression de la majeure partie de la complexité du module de structure avait peu d'effet sur la précision de la prédiction, tandis que la conservation de la structure principale et de la représentation de la torsion de la chaîne latérale augmentait la complexité des graphiques moléculaires généraux.
Donc dans AlphaFold 3,Les ingénieurs ont utilisé le module de diffusion pour prédire directement les coordonnées atomiques brutes, remplaçant le module de structure AlphaFold 2 qui fonctionne sur la base de structures spécifiques aux acides aminés et d'angles de torsion de la chaîne latérale.Ce dernier utilise une représentation par paires pour générer des positions atomiques explicites. La nature multi-échelle du processus de diffusion (réseau induit à faible niveau de bruit avec structure locale améliorée) permet également l'élimination des pertes stéréochimiques et le traitement spécial des motifs de liaison dans le réseau, s'attaquant ainsi facilement à des compositions chimiques arbitraires.
Spécifiquement,Le module de diffusion est capable d'opérer directement sur des coordonnées atomiques brutes et des représentations d'étiquetage abstrait grossières, sans avoir besoin de cadres de rotation ou de traitement isovariant.Les chercheurs ont d’abord entraîné le modèle de diffusion à recevoir des coordonnées atomiques « bruyantes », puis à prédire les vraies coordonnées. Ce processus nécessite que le modèle apprenne les structures des protéines à différentes échelles de longueur, où la tâche de débruitage sous un faible bruit met l'accent sur la compréhension de la stéréochimie très locale, tandis que la tâche de débruitage sous un bruit élevé met l'accent sur la structure à grande échelle du système.
Au moment de l'inférence, le modèle échantillonne d'abord le bruit aléatoire, puis effectue un débruitage récurrent pour générer la structure finale. Il convient de mentionner qu’AlphaFold 3 est une procédure de formation générative qui produit une distribution de réponses. Cela signifie que pour chaque réponse, même si le modèle est incertain quant à sa position, il est également capable de déterminer sa structure locale (comme la géométrie des liaisons latérales). donc,AlphaFold 3 évite à la fois la paramétrisation des résidus basée sur la torsion et les pénalités de violation structurelle tout en gérant la complexité des ligands généraux.

Comme le montre la figure ci-dessus, dans la comparaison des performances de prédiction de l'interaction protéine-dsADN (protéine-ADN double brin), le taux de réussite d'AlphaFold 3 a atteint 64,8%, tandis que RosettaAlphaFold2NA n'avait que 28,3%. Dans la prédiction protéine-anticorps (interaction entre protéine et anticorps), AlphaFold 3 a un taux de réussite précis de 62,9%, tandis que les autres systèmes n'ont que 29,6%.
* RosettaAlphaFold2NA combine la technologie de modélisation classique de Rosetta avec AlphaFold 2.
Historique de croissance d'AlphaFold : un excellent élève avec quatre bonds consécutifs en 6 ans
Après qu'AlphaGo ait vaincu le maître international de Go Lee Sedol, la « série Alpha » a officiellement attiré l'attention du public. Selon DeepMind, en 2016, presque immédiatement après qu'AlphaGo soit devenu célèbre, l'équipe a commencé à étudier le problème du repliement des protéines.
Lors du 13e CASP (Critical Assessment of protein Structure Prediction) fin 2018,AlphaFold est arrivé premier parmi 98 concurrents, prédisant avec précision les structures de 25 des 43 protéines.Le candidat arrivé deuxième dans le même groupe n'a prédit correctement que 3 options.
À cette époque, l'AlphaFold de « première génération » avait déjà démontré une résistance étonnante. AlphaFold 1 a été formé sur la base de milliers de protéines connues, en utilisant des réseaux neuronaux pour prédire les distances entre les paires d'acides aminés et les angles entre les liaisons chimiques qui les relient, puis en ajustant la structure préliminaire pour trouver l'agencement le plus économe en énergie.
Mais l'équipe a découvert que l'approche d'AlphaFold 1, qui combine la physique locale et les potentiels de guidage dérivés de la reconnaissance de formes, tend à surestimer les interactions entre les résidus qui sont proches dans une séquence, par rapport aux interactions entre les résidus qui sont éloignés le long de la chaîne. Par conséquent, AlphaFold 1 a tendance à sélectionner des modèles avec une structure secondaire légèrement plus importante (hélices α et feuillets β) que ce n'est réellement le cas (une sorte de surajustement).
D'un point de vue technique, la conception logicielle utilisée par AlphaFold 1 se compose de plusieurs modules, chacun étant formé individuellement pour générer un potentiel guidé, puis combiné avec un potentiel énergétique basé sur la physique.
Ainsi, AlphaFold 2, sorti en 2020, combine des sous-réseaux avec un seul modèle de bout en bout différentiable.Le système est entièrement basé sur la reconnaissance de formes et est formé de manière globale en tant que structure d'ensemble unique.
AlphaFold 2 a obtenu des prédictions très précises des structures des monomères protéiques, et l'équipe DeepMind a ensuite porté son attention sur la prédiction des complexes. En octobre 2021, DeepMind a publié une mise à jour appelée AlphaFold-Multimer.Il s'agit d'une extension d'AlphaFold 2 et peut modéliser des complexes de diverses protéines.
Tutoriel de déploiement en un clic d'AlphaFold 2 :
https://openbayes.com/console/public/tutorials/m6k2bdSu30C
Les chercheurs ont testé 4 433 complexes protéiques et examiné la précision de prédiction d'AlphaFold-Multimer aux interfaces de contact des hétéropolymères et des homopolymères, atteignant respectivement 67% et 69%, avec des prédictions très précises représentant respectivement 23% et 34%.
Par la suite, AlphaFold, qui était resté silencieux pendant deux ans, a de nouveau étonné tout le monde. En plus d'améliorer encore la précision de la prédiction de la structure des protéines, il a également ajouté des capacités de prédiction RAN. Le dernier jour d'octobre 2023,DeepMind a publié les dernières avancées d'AlphaFold (le document l'appelait AlphaFold-latest, qui semble maintenant être AlphaFold 3).

Adresse du document :https://storage.googleapis.com/deepmind-media/DeepMind.com/Blog/a-glimpse-of-the-next-generation-of-alphafold/alphafold_latest_oct2023.pdf
DeepMind a déclaré que le modèle AlphaFold de nouvelle génération peut prédire presque toutes les molécules de la Protein Data Bank (PDB) avec une précision de prédiction au niveau atomique, ce qui ouvre non seulement une nouvelle compréhension de plusieurs catégories clés de macromolécules biologiques, mais améliore également considérablement la précision de prédiction. Ces classes de biomacromolécules comprennent des ligands (petites molécules), des protéines, des acides nucléiques (ADN et ARN) et des biomacromolécules avec des modifications post-traductionnelles (MPT).
Après beaucoup d'attente, la nouvelle génération d'AlphaFold 3, qui a aiguisé l'appétit de tous depuis fin 2023, est enfin apparue. Ses capacités n'ont évidemment pas déçu l'industrie et le monde universitaire, mais il existe encore des questions plus importantes qui nécessitent des efforts conjoints de toutes les parties pour les promouvoir -Comment faire sortir AlphaFold 3 du laboratoire et l'intégrer à la chaîne de production pharmaceutique, comment permettre à davantage d'équipes de recherche scientifique d'utiliser cet outil avancé pour optimiser le processus de recherche, etc.Une attention et des investissements continus sont toujours nécessaires.
Isomorphic Labs, filiale de DeepMind, voit le jour
Il convient de noter qu'une équipe incontournable est également apparue dans la version d'AlphaFold 3 - Isomorphic Labs.
Cette société, issue de DeepMind, a été fondée en novembre 2021.Son nom s’inspire de la cartographie isomorphe potentielle entre la biologie et les sciences de l’information. S'appuyant sur AlphaFold, Isomorphic Labs se concentre sur le domaine de la médecine de l'IA, avec pour mission d'utiliser l'intelligence artificielle et les méthodes d'apprentissage automatique pour accélérer et améliorer le processus de découverte de médicaments afin de trouver des traitements pour certaines des maladies les plus dévastatrices de l'humanité.
En mai 2022, Isomorphic Labs a annoncé la première phase de son équipe de direction, qui est composée de stars.
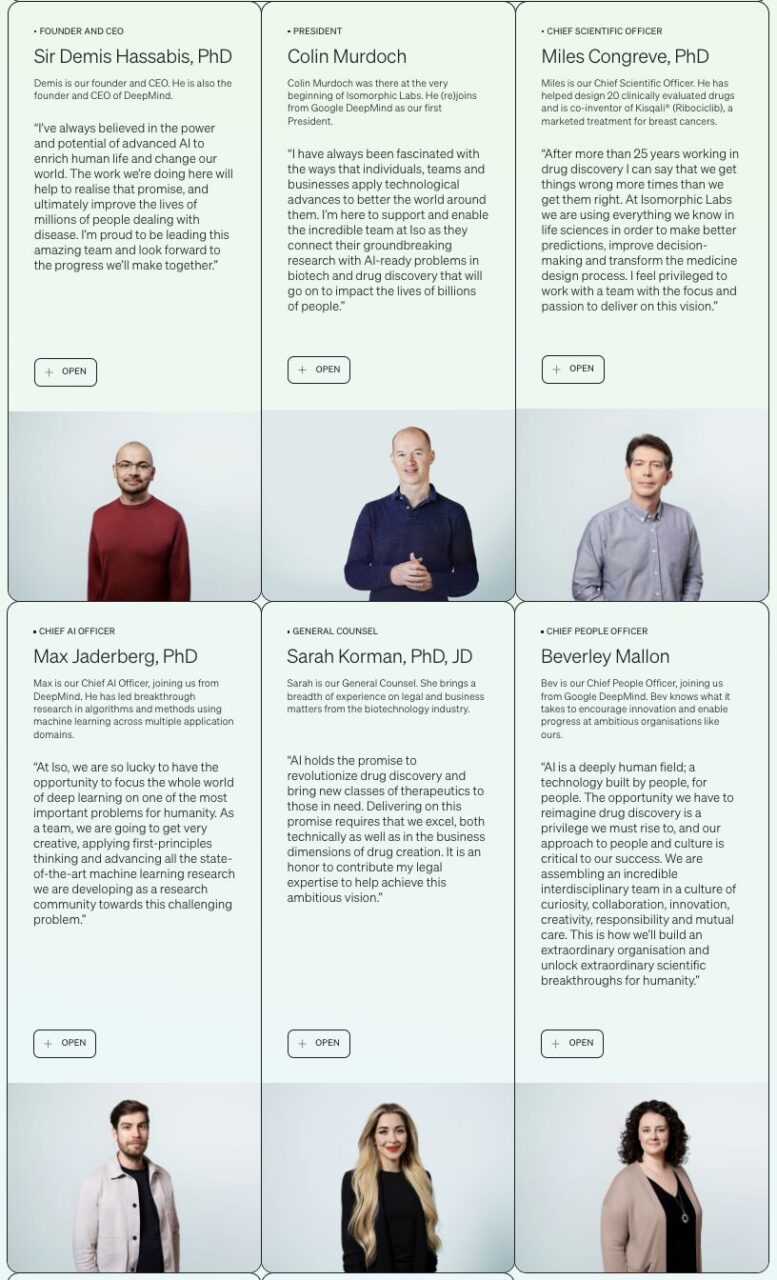
Demis Hassabis, connu comme le « père d'AlphaGo », est le PDG de la société ; Le directeur scientifique est Miles Congreve, qui a participé à la conception de 20 médicaments cliniquement évalués et est le co-inventeur de Kisqali® (Ribociclib), un médicament commercialisé pour le traitement du cancer du sein ; Sergei Yakneen, directeur de la technologie, a plus de 20 ans d'expérience dans l'ingénierie, l'apprentissage automatique, les produits, les sciences de la vie et la recherche médicale ; Max Jaderberg, titulaire d'un doctorat de l'Université d'Oxford, est directeur de l'apprentissage automatique...
Au cours des deux années environ qui se sont écoulées depuis sa création, Isomorphic, basé à Londres, n'a publié aucune nouvelle sur son travail, à l'exception de l'équipe de direction initialement annoncée. Ce n'est qu'à l'occasion d'une interview exclusive avec Endpoints News en 2023 que Demis Hassabis a révélé pour la première fois sa vision d'Isomorphic.
Dans l'interview, en parlant du travail principal de l'entreprise depuis novembre 2021, Demis Hassabis a déclaré :Nous nous concentrons d'abord sur le domaine des petites molécules, où nous pouvons avoir un impact rapide et maximal. Nous nous intéressons également de près au domaine des interactions : interactions protéine-ligand, interactions protéine-protéine, et à la nature dynamique de la biologie. Nous nous intéressons également au domaine chimique, pour comprendre les structures des composés, leur liaison aux cibles, leur affinité de liaison, etc.
Concernant la collaboration actuelle avec l'industrie pour développer un pipeline, Demis Hassabis a déclaré : « Nous sélectionnerons les cibles et les programmes que nous jugeons particulièrement adaptés à notre feuille de route technologique et collaborerons avec de grandes sociétés pharmaceutiques sur des cibles intéressantes. De plus amples informations pourront être communiquées ultérieurement. »
En janvier 2024, Isomorphic Labs a annoncé deux accords de développement de médicaments de 3 milliards de dollars avec Eli Lilly et Novartis. Demis Hassabis a révélé que « bien qu'Isomorphic ait déjà eu des discussions préliminaires avec d'autres grandes sociétés pharmaceutiques, Eli Lilly et Novartis ont une grande sincérité dans leur coopération. » Dans le même temps, l’objectif premier de cette coopération est clair : fabriquer des médicaments, plutôt que de se limiter à des projets pilotes ou à une coopération universitaire.
Plus précisément, la collaboration avec Eli Lilly porte sur le développement de thérapies ciblant de multiples protéines et voies liées à la maladie. À cet égard, Demis Hassabis a souligné : « L'application de notre plateforme technologique exclusive, AlphaFold de nouvelle génération, et de nos capacités informatiques à grande échelle aux projets de développement d'Eli Lilly permettra de faire progresser conjointement la conception de médicaments révolutionnaires. »
La collaboration avec Novartis se concentre sur la découverte de petites molécules thérapeutiques contre trois cibles non divulguées. Fiona Marshall, présidente de Novartis BioMedical Research, a déclaré : « Cette collaboration combine les atouts uniques des deux entreprises, de l'intelligence artificielle et de la science des données à la chimie médicinale et à l'expertise approfondie dans certains domaines thérapeutiques, pour faire progresser de nouvelles possibilités dans la découverte de médicaments basée sur l'IA. »
Aujourd'hui, la sortie d'AlphaFold 3 devrait encore renforcer la force technique d'Isomorphic Labs, mais du point de vue des applications, le boom des investissements dans les produits pharmaceutiques basés sur l'IA a diminué ces dernières années. Face aux coûts élevés de R&D, la manière de transformer la technologie de pointe en production réelle est une question importante liée à l'orientation future de l'entreprise. Avec le soutien solide de DeepMind, nous espérons également voir AlphaFold 3 véritablement débarquer dans l’industrie dès que possible.
Encore une chose : AlphaFold Server

AlphaFold 3 est le premier système d'IA à surpasser les outils de prédiction de structure biomoléculaire basés sur la physique, et il n'est actuellement pas prévu d'ouvrir le code source complet.Cependant, l'équipe de recherche a publié une interface publique pour le modèle, le serveur AlphaFold, qui prend en charge la recherche non commerciale, ouvrant la porte aux chercheurs du monde entier.
Visitez le site officiel d'AlphaFold Server :
alphafoldserver.com
En quelques clics de souris, les biologistes peuvent utiliser AlphaFold 3 pour modéliser des structures composées de protéines, d'ADN, d'ARN et de ligands, d'ions et de modifications chimiques sélectionnés, et prédire comment les protéines interagissent avec d'autres molécules de la cellule. La plateforme peut aider les scientifiques à élaborer de nouvelles hypothèses à tester en laboratoire, à accélérer les flux de travail et peut être utilisée indépendamment du fait que les scientifiques disposent de ressources informatiques suffisantes ou d'une expertise en apprentissage automatique.
À cet égard, Céline Bouchoux, chercheuse au laboratoire Uhlmann de l'Institut Francis Crick, a salué : « Avec AlphaFold Server, il ne s'agit plus seulement de prédire des structures, mais de fournir généreusement un accès : permettre aux chercheurs de poser des questions audacieuses et d'accélérer la découverte. »
Il ne fait aucun doute que l’avènement d’AlphaFold 3 constitue non seulement un grand pas en avant dans l’exploration scientifique, mais également un outil clé pour ouvrir une nouvelle ère de recherche et de développement biomédical. Il a réalisé des avancées significatives dans la simulation de nombreux types différents d’interactions moléculaires, ce qui est crucial pour les projets de recherche et développement tels que l’identification précise des cibles médicamenteuses.
L'équipe de DeepMind est pleine d'attentes à son égard : « Nous venons tout juste de commencer à explorer le potentiel d'AlphaFold 3 et nous sommes impatients de voir ce qui se passera dans le futur. »
Références :
1.https://cloud.tencent.com/developer/article/2017961
2.https://hub.baai.ac.cn/view/31181
3.https://zh.wikipedia.org/wiki/AlphaFold
4.https://mp.weixin.qq.com/s/18cNw-E-5vU3vKb1J4WWKg








