Command Palette
Search for a command to run...
Nature誌に掲載された、遺伝子配列解析と機械学習に基づく廃水疫学的評価により、最大4週間早くウイルスを検出できる。

ここ数年、世界の公衆衛生安全保障は深刻な課題に直面しています。特にCOVID-19パンデミックの発生以降、その傾向は顕著です。病原体である重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)は進化を続け、複数の優勢な変異株が出現しています。これらの変異株は感染力や免疫反応の回避能力が異なり、感染症の予防と制御の難易度と医療システムへの負担を著しく増大させています。
公衆衛生検査と SARS-CoV-2 ゲノム配列解析は、流行している変異株を包括的に検出するための重要な手段です。ただし、このタイプの臨床モニタリングは、多くの場合、大量の研究室リソースに大きく依存しており、個人がテストに積極的に参加する必要があります。SARS-CoV-2変異株の出現と拡散を完全に追跡することは困難です。特に医療資源が比較的限られている地域や検査への意欲が低い地域では、臨床モニタリングは検出バイアスの影響を受けやすく、予防と制御における盲点を生み出します。
補完的なアプローチとして、廃水ベースの疫学(WBE)は、1940 年代にコミュニティ感染を評価するために初めて提案されて以来、病気の発生の警告において重要な役割を果たしてきました。 WBE は主に、人体から廃水中に排出されたウイルスの痕跡を分析することで、ウイルスの組成と動的な変化を検出し追跡します。臨床モニタリングと比較すると、WBE は個別の積極的検査に頼ることなく対象エリア内の集団感染状況を客観的かつ公平に反映し、早期警告を実現し、費用対効果も大幅に向上します。
しかし、現在主流となっている廃水モニタリング方法 (線形回帰に基づく Freyja や COJAC など) には依然として限界があります。検出は、既知の変異体の変異パターン(GISAIDまたはUshERデータベースの参照配列など)に基づいて行う必要がある。臨床文献に記載されていない、または特徴付けられていない新しい変異体が出現した場合、それを正確に識別することが困難な場合が多く、WBE の検出効率がある程度制限されます。
これに対処するため、ネバダ大学ラスベガス校の研究チームは、ICA-Var(Independent Component Analysis of Variants)と呼ばれる多変量解析手法を提案しました。この方法は、教師なし機械学習プロセス設計に基づいており、独立成分分析 (ICA) を使用して廃水データから共変動と時間とともに変化する変異パターンを抽出します。より早期かつ正確な変異検出が実現します。
研究チームはこの方法を用いて、2021年末から2023年の間にデルタ変異株、オミクロン変異株、組み換えXBB変異株を正確に検出しました。この方法は、流行予防と制御の早期警報のための廃水モニタリングの有効性を再確認するだけでなく、臨床モニタリングがない場合でもウイルスの変異と拡散を包括的に追跡するための新しいツールを提供します。
関連研究は「ゲノム配列解析と機械学習による廃水からのSARS-CoV-2変異体の早期検出」というタイトルでネイチャー・コミュニケーションズ誌に掲載された。
研究のハイライト:
* この方法は、都市部と農村部におけるウイルス変異の時空間的ダイナミクスを明らかにし、都市部から農村部へのウイルス伝播の法則を確認し、医療アクセスが悪い地域や臨床配列データが不足している地域に効果的で低コストの変異検出パラダイムを提供します。
* 現在のゴールドスタンダードツールであるFreyjaと比較して、ICA-Varの多変量解析法には大きな利点があり、Delta、Omicron、最新のEG.5、HV.1、BA.2.86変異体の検出時間は平均1~4週間早くなります。

用紙のアドレス:
https://www.nature.com/articles/s41467-025-61280-5
長期にわたる多地点データ収集
この研究では、実験に使用された廃水サンプルは2021年8月から2023年11月まで収集されました。南ネバダ州の都市部と農村部から 3,659 個の廃水サンプルが採取されました。収集後、廃水サンプルは現場で氷の上に置かれ、処理されるまで冷蔵保存され、保管時間は 36 時間以内となります。
核酸抽出プロセスでは、研究チームはまず、規制要件に従い、Promega Wizard Enviro Total Nucleic Kit(カタログ番号A2991)を用いて廃水サンプルから核酸を単離した。次に、Promegaのプロトコルを改変し、廃水をプロテアーゼ溶液で溶解し、Macherey-Nagel NucleoMag Beads(カタログ番号744970)を用いて遊離核酸を結合させた。10 ngを超えるRNAについては、New England BioLabs LunaScript RT SuperMix Kitを用いてファーストストランドcDNAを合成した。
シーケンシングライブラリの構築とシーケンシング、研究チームは、Paragon Genomics の CleanPlex SARS-CoV-2 FLEX Panel を使用してアンプリコン シーケンス ライブラリを構築し、その後、300 サイクル フロー セルを使用して Illumina NextSeq 500 または NextSeq 1000 プラットフォームでシーケンスしました。
シーケンスデータ処理に関しては、研究チームはまず、cutadaptソフトウェア(バージョン4.2)を用いて、シーケンスリードペアからIlluminaアダプター配列を除去しました。次に、bwa memソフトウェア(バージョン0.7.17-r1188)を用いて、シーケンスリードペアをSARS-CoV-2リファレンスゲノム(NC_045512.2)にマッピングしました。次に、fgbio TrimPrimersツール(バージョン2.1.0、ハードトリミングモード)を用いて、アラインメントされたリードからParagon Genomics CleanPlex SARS-CoV-2 FLEXアンプリコンプライマー配列を除去しました。最後に、iVar variantsソフトウェア(バージョン1.4.1)を用いてバリアントを検出し(元の2020年リファレンスゲノムと比較したアレル頻度の差に基づきます)、samtoolsソフトウェア(バージョン1.16.1)を用いてゲノムカバレッジとリード深度を計算しました。
重複サンプルと陽性/陰性コントロールを除去した後、残りの 2,684 個のサンプルは品質管理 (QC) 分析に使用されました。厳格な品質管理の後、シーケンス深度が50倍で、SARS-CoV-2ゲノムの少なくとも80%をカバーする廃水サンプルのみがその後の分析のために保持されました。
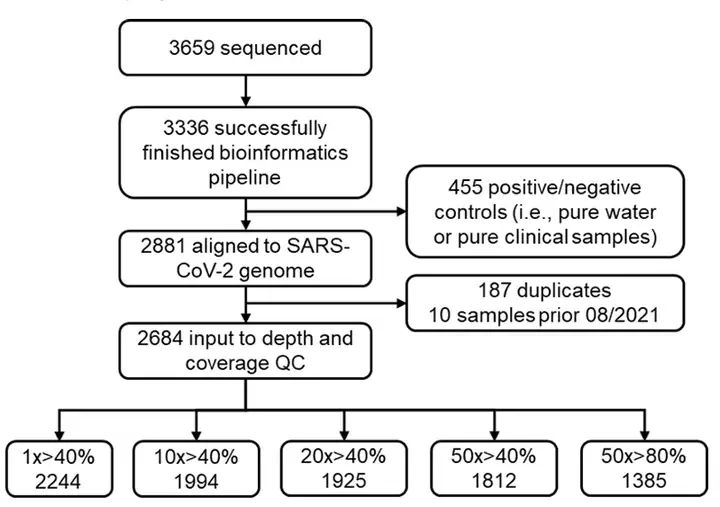
ファイナル、この研究では 1,385 件の高品質サンプルが使用されました。その後の分析のために、SARS-CoV-2 変異体の 59,422 個の変異部位をカバーします。
ICA-Var法の有効性の検証を支援するために、研究チームは臨床データを対照および参照基準として使用し、2021年9月から2023年11月までの期間をカバーする、GISAIDデータベースからダウンロードしたネバダ州の8,810件の高カバレッジ臨床SARS-CoV-2配列データを分析しました。
ICAを核として、二重回帰法を導入し、COVID-19検出のための新しいツールを作成する
ICA-Varのコアプロセスは独立成分分析を通じて廃水サンプル内の変異頻度を処理し、独立した共変動変異パターンを抽出します。これらのパターンは、下の図に示すように、二重回帰を通じて元のサンプルと関連付けられ、ウイルスの変異体を追跡します。
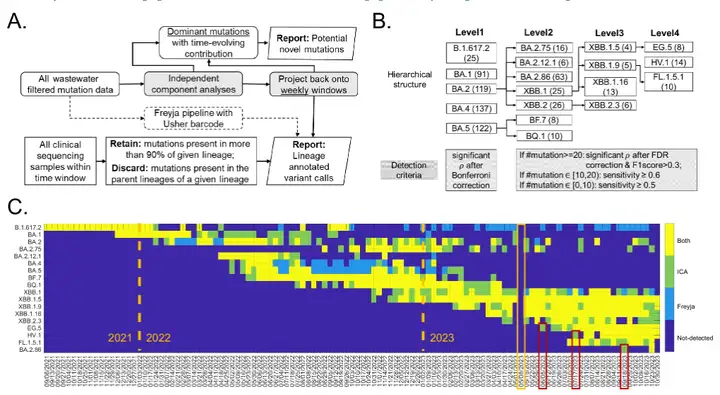
* 図中のAは独立成分分析のプロセスです。2つのマトリックスは、毎週のSARS-CoV-2系統検出(下段)と潜在的な新しい変異(上段)です。
* 図 B は、懸念される 18 の変異体の階層構造を示しています。各変異の主な変異部位(すなわち、系統を定義する部位)は、 http://covspectrum.org 要約された臨床データ。括弧内に主要な変異の数が表示され、網掛けのボックスは提案されたワークフローでテストされる基準を示しています。
*図 C は、ICA-Var 法と最先端ツール Freyja の比較を示しています。新たに出現した変異株 EG.5、HV.1、および BA.2.86 については、赤いボックスは ICA-Var の検出時間が早かったことを示し、黄色いボックスは技術的な問題により廃水サンプルの採取が行われなかった週を示しています。
具体的には、廃水サンプル中のSARS-CoV-2ゲノムシグナルは複数の変異体が混ざった結果であり、サンプルの劣化や配列エラーなどの影響を受けているため、従来の方法では単一の変異体の特性を直接分析することが困難です。ICA-Varの核となる考え方は、独立成分分析を使用することです。——このブラインドソース分離技術は、混合変異信号が複数の「独立したソース」の線形結合であると仮定し、数学モデルを使用してこれらの独立したパターンを混合データから分離します。
研究チームはまずデータを前処理しました。下水サンプルから得られたSARS-CoV-2ゲノムシーケンシングデータの品質管理を行い、低品質のリードやノイズの多い変異を除外することで、「変異頻度マトリックス」を構築しました。行はサンプル、列は変異部位、値はサンプル内の各部位の変異頻度を表します。次に、この変異頻度マトリックスに対して独立成分分析を行い、混合信号を独立成分に分解しました。各成分は「共変変異パターン」のセット、つまり、特定の変異に特徴的な変異の組み合わせを表し、時間の経過とともにサンプル間で同期して出現または消失します。
ここ、この研究では、最小記述長 (MDL) 基準を使用して独立成分の数を決定し、fastICA アルゴリズムを使用して独立成分分解を実行しました。結果の信頼性を確保するために、異なる初期値で ICA 分析を 50 回繰り返し、ICASSO ソフトウェアを使用して各実行で得られたコンポーネントをクラスター化して視覚化し、最終的に密接なクラスターに対応する信頼性の高い推定値のみをソース マトリックスとして保持しました。
その後、週ごとの変異株の状況をさらに把握するために、研究チームは二重回帰法を使用して、独立成分分析から得られたソース マトリックスを元のサンプルに再投影しました。各サンプル内の各独立成分の「寄与」、つまりサンプル内の変異体の相対的存在量を計算して、出現時期、流行傾向、都市と農村の分布の違いなど、時間と空間におけるさまざまな変異体の動的な変化を定量化します。
研究チームは、全サンプルのソース行列を一般線形モデル(GLM)のソース回帰変数として用い、全サンプルのソース行列に関連する各週次サンプルの信号分解パターンを求めた。次に、各週次サンプルの信号分解パターンを2つ目のGLMの回帰変数として用い、全サンプルのソース行列に関連する週固有のソース行列を求めた。このプロセスにより、双対空間を構成する推定値のペアが生成され、これらを組み合わせることで、各週次サンプルにおける元の全サンプル独立成分分析ソース行列への最良の近似値が得られました。
やっと、研究チームは、分離された独立したコンポーネントを臨床配列データ内の既知の変異と比較し、注釈を付けました。これにより、対応する変異株を正確に特定したり、一致しない共変動変異パターンを除外して、新しい変異株の可能性を警告したりすることができます。
ICA-Var メソッドは、「事前定義された参照バリアント バーコード」に依存する従来の方法の欠点を克服します。突然変異の共変動パターンを捉えることで、従来の方法よりも早く、より正確に新しい変異体を特定することが可能になります。この手法は、双回帰分析と組み合わせることで、都市部と農村部における感染状況の違いや変異部位の経時的変化も明らかにします。まとめると、ICA-Varは、COVID-19検出において、より感度が高く、包括的で、費用対効果の高いツールを提供します。
検出効率は現在のゴールドスタンダードツールであるFreyjaを超えており、新しい変異体を予測する可能性を秘めている。
ICA-Varの性能を検証・評価するため、研究チームは、廃水中のSARS-CoV-2系統の相対的存在量を推定するツールである、現在のゴールドスタンダードツールであるFreyjaと比較しました。Freyjaは、系統を定義する変異からなる「バーコード」ライブラリを用いて、既知のSARS-CoV-2系統を全て一意に識別し、重み付け最小絶対偏差回帰法を用いて系統の存在量を算出します。実験により、ICA-Var 多変量解析法にはより大きな利点があることが確認されました。
下の図に示すように、モデル方法とアーキテクチャのセクションでは、ICA-Var が新しいバリアント EG.5、HV.1、および BA.2.86 を検出する方法について簡単に説明しましたが、このセクションでは主要な内容を詳しく説明します。
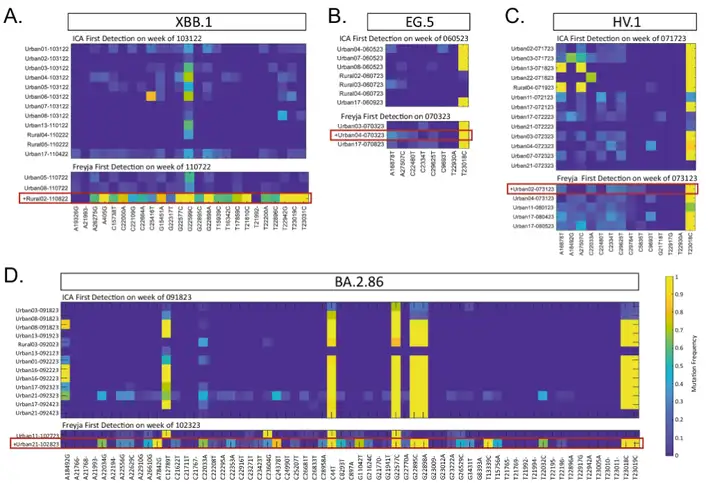
具体的には、2022年には、ICA-Var は、BA.2、BA.4、BA.5、BF.7、BQ.1、XBB.1、XBB.1.5 などの変異体を Freyja よりも 1 週間以上早く検出することが示されています。EG.5の検出において、ICA-Varは6月5日の週にこの変異体を検出しましたが、Freyjaは7月3日までEG.5のシグナルを特定しませんでした。その時点では、廃水サンプルの含有量は23.08%に達し、EG.5の優性変異部位8箇所のうち5箇所が既に検出されていました。同様に、XBB.1、HV.1、BA.2.86などの変異体についても、ICA-Var も Freyja より数週間早く検出されました。
これは、ICA-Var が、信頼性は高いが発生率の低い変異部位の複数のサンプルからの情報を統合するためです。これにより、統計的検出力が向上し、より早期の検出が可能になります。つまり、単一サンプル中の優性変異の割合が高いことに依存しないということです。複数のサンプルからの弱いシグナルを集約するだけで検出力を高めることができます。一方、Freyjaは、優性変異部位を明確に示すために、少なくとも1つの個別のサンプルを必要とします。これはまた、単一サンプル中の十分に強い変異シグナルへの依存度が高く、弱いシグナルや散在したシグナルに対する感度が低いことを意味します。
この実験では、都市部と農村部のサンプルにおける変異体の動態的傾向をさらに調査しました。研究チームは2022年初頭から、ネバダ州南部の農村部から採取した下水サンプルの配列解析と分析を行い、都市部と農村部の包括的な疫学比較を実施しました。この比較では、都市部と農村部のサンプルを毎週別々に分析しました。
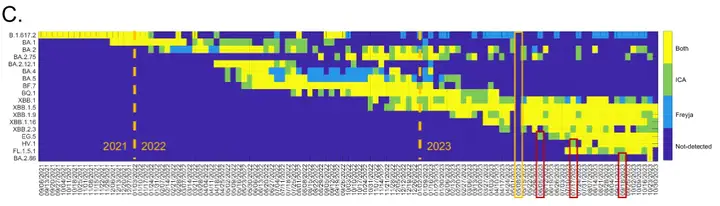
調査結果によると、懸念される18の変異株のうち、ICA-VarとFreyjaの両社は、都市部の廃水サンプルで16のSARS-CoV-2変異株を最初に検出し、その後農村部のサンプルでも検出された。これは、ウイルスの変異株は通常、最初に都市部で出現し、その後農村部へ広がることを示唆している。下図の通り:
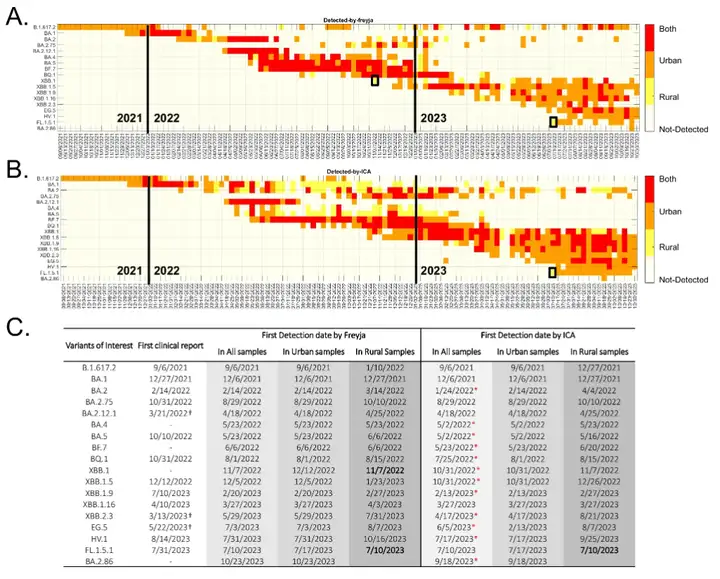
例外は、Freyja が最初に農村部の廃水サンプルで XBB.1 を検出したのに対し、ICA-Var は 1 週間前に都市部の廃水サンプルでこの変異体を発見したことです。両方のツールとも農村部の廃水サンプルで FL.1.5.1 を検出しましたが、同時期の都市部の廃水サンプルではこの変異体の優性突然変異の代替対立遺伝子の頻度と有病率ははるかに低かったです。
この研究では、変異部位の時系列的な進化傾向も明らかにされました。研究チームは、2021年8月から2023年11月の間に顕著な時系列的進化を示した177の変異部位を、B.1.617.2、BA.1、XBB.1の各変異株の主要な変異部位と比較しました(下図参照)。
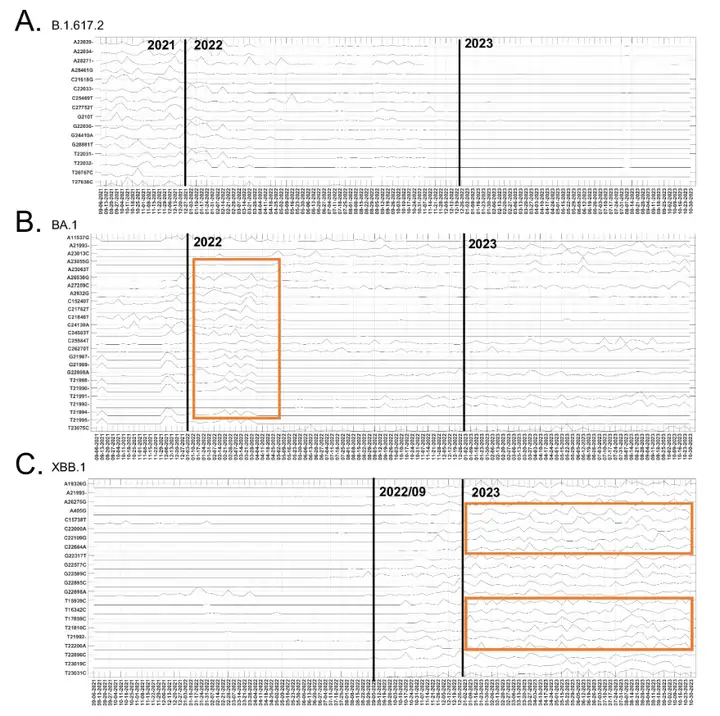
Delta変異体(B.1.617.2)の主要な変異部位25箇所のうち、16箇所は2021年末に寄与度が大きく変動し、その後2022年には徐々に減少しました。OmicronサブタイプBA.1の関連変異の寄与度は2021年末に大幅に増加し、2022年初頭にピークに達しました。BA.1の一部の変異部位の寄与度は2023年も変動を続け、XBB.1などの他のOmicronサブ系統でも確認されました。XBB.1変異体の25箇所の主要な変異のうち、22箇所は時間的に大きな変動を示し、2022年9月以降に大きな影響を与えました。複数の変異部位が同様の変動パターンを示し、共変動を示唆しており、XBB.1の組み換え特性を反映しています。
これらの分析により、ICA-Var によって特定された変異部位の時間的進化的寄与は Delta、Omicron、XBB.1 変異体の臨床所見と一致していることが実証され、ICA-Var 結果の信頼性がさらに示され、新しい変異体の出現につながる可能性のある新しい変異パターンを特定する可能性が実証されました。
この実験では、この点を詳細に検証しました。研究チームは、15の主要な変異体における優勢な変異部位と交差比較することで、113の潜在的な新規変異部位をスクリーニングしました。そして、階層的クラスタリングアルゴリズムを用いて、これらの変異部位を6つの特徴的なクラスターに分類しました。下図をご覧ください。

これらの特徴的なクラスターのうち、4つのクラスター(クラスター2~5)の変異部位は、2023年末に出現した変異株と重複しています。クラスター1とクラスター6には、既知の変異部位と重複する変異はありません。その中で、クラスター1の変異部位は、2023年8月以降、明らかな共変動パターンを示しました。GISAIDの臨床シークエンシングデータでは、8つの変異部位が検証され、臨床検体における報告頻度が低いことが確認されました。そのため、これらの変異は新たなコロナウイルス変異体の出現につながる可能性があり、臨床試験によるさらなる検証が必要となる。綿密な監視が必要です。
機械学習を活用した廃水監視は、高品質なウイルス予防と制御を推進するために進化し続けています。
冒頭で述べたように、WBEは新しい方法ではありません。環境ウイルス学者は、1940年代初頭から、廃水中の細胞培養実験を通じてポリオウイルスを採取することの価値を認識していました。それ以来、WBEは継続的に改良され、疾病発生の早期警戒に効果的なツールとなっています。COVID-19パンデミックの発生以来、WBEは再び伝染病の予防と制御において積極的な役割を果たしてきました。
例えば、2023年末には、スウェーデンの研究チームが下水のゲノム検査とCOVID-19症例のゲノム検査を統合することで、新たなSARS-CoV-2変異体BA.2.86の出現を検出することに成功したとの報告がありました。さらに、新型コロナウイルス変異体の検出にWBEをより効果的かつ積極的に活用するため、多くの研究室が関連モデルの開発や改良を行い、より費用対効果の高いWBEツールを提供しています。
例えば、清華大学、河北科技大学、天津生態環境モニタリングセンターの研究者らは、「廃水中のSRAS-CoV-2 RNAの濃縮および検出方法の検証」と題した研究論文を共同で発表しました。この研究では、限外濾過と共有結合親和性樹脂分離という2つの濃縮技術と、逆転写定量PCR(RT-qPCR)と逆転写デジタルPCR(RT-dPCR)という2つの検出方法を比較し、廃水中ウイルスモニタリングにおける性能を評価しました。
やっと、この研究では、逆転写デジタルPCR(RT-dPCR)法が、廃水中の低濃度のSARS-CoV-2 RNAを検出するのにより適した選択肢であることが示されました。検出率が高く、PCR 阻害剤に対する耐性も優れています。
* 紙のアドレス:
https://link.springer.com/article/10.1007/s10311-025-01843-6
さらに、カナダのアルバータ大学病理学・臨床検査医学部のXingfang Li教授率いる研究チームは、「監視のための廃水中のSARS-CoV-2変異体の定量化と識別」と題した研究を発表しました。研究チームは、臨床検体用に開発されたガンマ(ABG)法とデルタ法によるマルチプレックスRT-qPCR検出法に基づき、オミクロン亜変異体を標的とし、その特有の変異を利用しました。Omicron 変異体の 5 つの主要なサブ系統を区別できる Omicron トリプレックス RT-qPCR アッセイが開発されました。これは、単一チューブ RT-qPCR トリプレックスアッセイを使用して、1 年間にわたって廃水サンプル内のすべてのオミクロン サブバリアントを検出および識別した最初の研究です。
* 紙のアドレス:
https://pubs.acs.org/doi/10.1021/envhealth.3c00089
つまり、世界は今日、深刻な公衆衛生と安全の課題に直面しており、人口モニタリングの非常に効果的な手段として、廃水モニタリングはかけがえのない役割を果たしています。技術の継続的な進歩に伴い、廃水モニタリングは、既知の変異パターンに基づく早期標的検出から、全ゲノム配列解析のブレークスルー、そして未知の病原体の特定に至るまで、進化を続けていきます。感度とカバー範囲の継続的な向上により、廃水モニタリングは、疫病の警告、追跡、そして政策立案のためのより正確で重要なデータを提供し、公衆衛生と安全の防衛線を補完する重要な手段となるでしょう。
参考文献:
1.https://www.nature.com/articles/s41467-025-61280-5
2.https://mp.weixin.qq.com/s/ZzzZt-uNNc5DsD-ib3Ww8g








