Command Palette
Search for a command to run...
En Reliant Les Données D’expression Génétique Aux Images De Morphologie Cellulaire, Des Chercheurs De L’université Chinoise De Hong Kong Et D’autres Ont Développé Un Modèle De Diffusion Guidé Par Le Transcriptome Pour Accélérer Le Développement De Médicaments phénotypiques.
La morphologie cellulaire est un domaine de recherche essentiel en biologie unicellulaire. Son intérêt réside dans l'analyse systématique des changements dynamiques de la morphologie cellulaire sous l'effet de perturbations génétiques ou médicamenteuses, grâce à l'analyse d'images à haut débit. Cette recherche améliore non seulement significativement la précision des prédictions du mécanisme d'action (MOA) des composés, mais aussi celle des évaluations de leur bioactivité. Enfin, elle fournit des données pour les étapes clés de la découverte phénotypique de médicaments, telles que le criblage des composés candidats et la vérification du mécanisme d'action, accélérant ainsi efficacement le processus de R&D.
Cependant, observer et analyser les changements de morphologie cellulaire suite à des perturbations génétiques ou médicamenteuses n'est pas chose aisée. Le nombre de composés pouvant être criblés dépasse les millions, et le nombre de gènes pouvant être modifiés se chiffre également en dizaines de milliers. Utiliser des méthodes expérimentales traditionnelles pour vérifier chaque individu individuellement est non seulement extrêmement inefficace, mais engendre également des coûts importants en termes de temps et d'argent. Bien que diverses méthodes informatiques aient été proposées et appliquées à la prédiction de la morphologie cellulaire, leur précision et leur fidélité ne répondent toujours pas aux besoins de la recherche pratique.
Spécifiquement,Les limites des méthodes existantes se reflètent principalement sur deux points :Premièrement, les performances des modèles avancés, tels que l'IMA (IMage Perturbation Autoencoder), dépendent fortement des connaissances biologiques connues ou d'ensembles de données spécifiques, ce qui entraîne une faible capacité de généralisation et un manque d'applicabilité. Deuxièmement, les données de morphologie cellulaire sont facilement affectées par des facteurs d'interférence expérimentaux tels que les effets de lot et de position des puits, et présentent un niveau de bruit élevé, ce qui complique la capture efficace des véritables caractéristiques morphologiques cellulaires, ce qui affecte directement la stabilité et la fiabilité des données et limite la précision des résultats d'analyse ultérieurs.
Pour relever les défis ci-dessus, des chercheurs de l'Université chinoise de Hong Kong, de l'Université Mohamed bin Zayed d'intelligence artificielle et d'autres institutions ont proposé un modèle de diffusion évolutif guidé par le transcriptome, MorphDiff.Ce modèle est spécialement conçu pour la simulation haute fidélité des réponses morphologiques cellulaires aux perturbations. Il repose sur l'architecture du modèle de diffusion latente (LDM) et utilise le profil d'expression génétique L1000 comme entrée conditionnelle pour l'apprentissage du débruitage.
Les résultats de l’étude ont confirmé queLe principal avantage de MorphDiff est sa capacité à générer avec précision des morphologies cellulaires dans des « conditions de perturbation invisibles ».Cette capacité offre deux avantages clés : d'une part, elle permet aux chercheurs d'explorer efficacement le vaste champ du criblage des perturbations phénotypiques, réduisant ainsi considérablement le recours aux expériences de terrain à grande échelle, diminuant ainsi les coûts expérimentaux et améliorant l'efficacité du criblage ; d'autre part, elle contribue à élucider les mécanismes d'action de molécules médicamenteuses structurellement diverses, fournissant ainsi un support essentiel à la validation des mécanismes des composés. MorphDiff peut donc servir d'outil performant pour accélérer le développement de médicaments phénotypiques.
Les résultats de la recherche ont été publiés dans Nature Communications sous le titre « Prédiction des changements de morphologie cellulaire sous perturbations avec un modèle de diffusion guidé par le transcriptome ».
Points saillants de la recherche :
* Cette étude applique de manière innovante des modèles de diffusion pour prédire la morphologie cellulaire pour la première fois, ouvrant de nouvelles voies et fournissant de nouveaux outils pour le développement de médicaments phénotypiques.
* Des tests de référence approfondis démontrent l'efficacité de MorphDiff, en particulier dans la récupération de MOA, où il peut atteindre une précision comparable à la morphologie de vérité terrain, surpassant les méthodes de base de 16,9% et 8% respectivement.

Adresse du document :
https://www.nature.com/articles/s41467-025-63478-z
Suivez le compte officiel et répondez « diffusion guidée par le transcriptome » pour obtenir le PDF complet
Autres articles sur les frontières de l'IA :
https://hyper.ai/papers
Ensemble de données : Un ensemble de données multi-omiques à grande échelle vérifie son efficacité
Pour vérifier systématiquement l'efficacité et la généralisation du modèle MorphDiff dans la prédiction de la morphologie cellulaire dans des conditions de perturbation,Cette étude a construit un système de données multi-lignée cellulaire et multi-source basé sur la double dimension de « perturbation génétique-perturbation médicamenteuse ».Pour chaque échantillon de l'expérience, deux types de données ont été collectées : un profil d'expression génétique L1000 et une image de morphologie cellulaire, formant ainsi des données appariées. La première servait de « donnée d'entrée pour les caractéristiques moléculaires » et la seconde de « donnée d'entrée pour les caractéristiques phénotypiques ». Cette méthode assure la corrélation entre le signal de perturbation au niveau génétique et la réponse phénotypique au niveau morphologique, déterminée uniquement par la perturbation cible. Cela élimine les interférences dues à des variables non pertinentes, telles que les différences entre les lignées cellulaires et les lots expérimentaux.
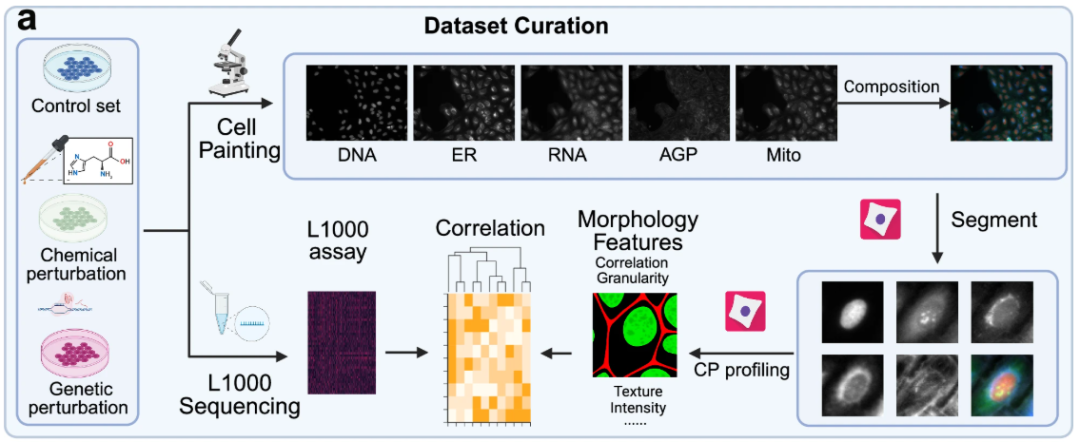
En termes d'ensembles de données d'images de morphologie cellulaire,L'étude a utilisé trois jeux de données d'images de morphologie cellulaire à grande échelle pour l'entraînement, l'évaluation et l'analyse du modèle : un jeu de données de perturbation génétique et deux jeux de données de perturbation médicamenteuse. Le jeu de données de perturbation génétique, dérivé du jeu de données JUMP basé sur la lignée cellulaire U2OS, contient 130 perturbations génétiques. Les jeux de données de perturbation médicamenteuse, dérivés du jeu de données CDRP basé sur la lignée cellulaire U2OS et du jeu de données LINCS basé sur la lignée cellulaire A549, contiennent respectivement 1 028 et 61 perturbations médicamenteuses.
Les trois ensembles de données d'images de morphologie cellulaire ont été prétraités et segmentés. CellProfiler 4.2.5 a été utilisé pour segmenter les images de plaques cellulaires en masse en images de cellules individuelles pour une analyse plus fine.Les images de morphologie cellulaire obtenues par la technologie Cell Painting contiennent 5 canaux principaux.C'est-à-dire l'ADN (noyau), l'ARN (nucléole et cytoplasme), le RE (réticulum endoplasmique), l'AGP (appareil de Golgi/membrane cellulaire/squelette d'actine) et les Mitochondries.
en plus,L'expérience a également planifié l'ensemble de données L1000 de « aucune image morphologique correspondante ».Cet ensemble de données est principalement utilisé pour explorer davantage l'application du modèle dans le scénario de « l'obtention uniquement de données d'expression génétique » et peut jeter les bases d'une vérification ultérieure des mécanismes, d'un criblage de médicaments, etc. pour fournir des hypothèses « basées sur les données ».
Architecture et méthodes du modèle : lier les données d'expression génétique aux images de morphologie cellulaire
L'objectif principal de MorphDiff est de réaliser une cartographie précise de bout en bout à partir des profils d'expression génétique L1000 vers des images de morphologie cellulaire via un cadre de modèle de diffusion latente guidé par le transcriptome.En termes simples, il s'agit de concevoir et de former un modèle, qui est comme un « pont » - entrer les données d'expression du gène L1000 correspondant à une certaine « perturbation », puis sortir l'image morphologique ou la morphologie de perturbation de la cellule sous cette perturbation.
Le cœur du modèle MorphDiff se compose de deux modules principaux :Autoencodeur variationnel de morphologie (MVAE) et modèle de diffusion latente (LDM), comme illustré dans la figure b ci-dessous.
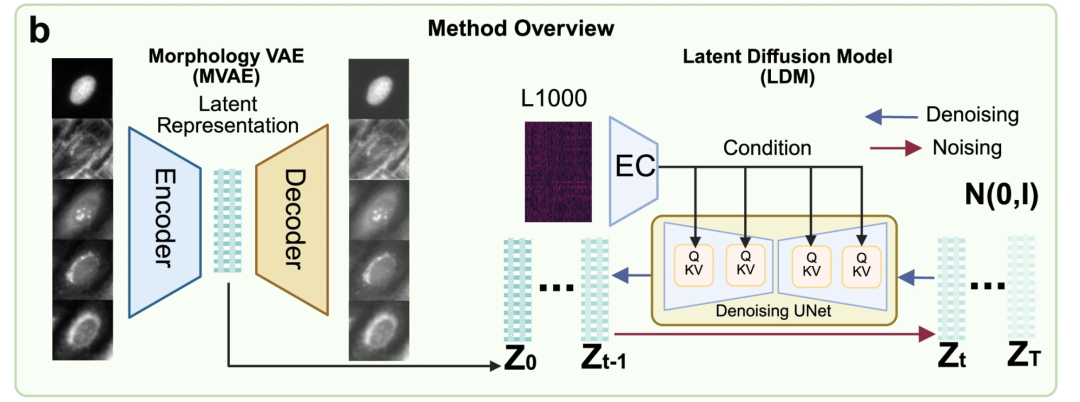
Parmi eux, MVAE est le « moteur de compression d'image » du modèle, qui est responsable de la conversion d'images de morphologie cellulaire multicanaux à haute résolution en représentations potentielles interprétables à faible dimension.Cette approche permet de pallier le coût de calcul élevé et l'instabilité de l'apprentissage associés à l'apprentissage direct de modèles de diffusion sur des images de grande dimension. Structurellement, le MVAE se compose de deux parties : un encodeur et un décodeur. L'encodeur compresse des images morphologiques complexes à 5 canaux en une représentation plus simple de faible dimension ; le décodeur restaure l'image morphologique d'origine à partir de cette représentation de faible dimension.
Le LDM est principalement responsable de la liaison entre « l'expression des gènes » et les « caractéristiques morphologiques compressées », aidant le modèle à compléter la relation « des gènes aux caractéristiques morphologiques ». Le LDM implique un processus de débruitage. Ce processus ajoute progressivement un bruit gaussien aux caractéristiques morphologiques compressées jusqu'à ce qu'elles soient complètement aléatoires. Ce processus permet au modèle de restaurer progressivement le bruit aléatoire aux caractéristiques morphologiques d'origine, compte tenu de l'expression connue des gènes L1000. Le modèle utilise une architecture réseau U-Net et intègre un mécanisme d'attention pour relier plus précisément les informations génétiques et morphologiques clés.
La figure c ci-dessous illustre deux applications du modèle MorphDiff pré-entraîné : G2I et I2I. Le premier débruite l'image morphologique cellulaire correspondante à partir d'une distribution de bruit aléatoire, conditionnée par l'expression du gène L1000, afin de générer l'image morphologique cellulaire correspondante. Le second, conditionné par l'expression du gène L1000 sous une perturbation spécifique, transforme l'image morphologique de la cellule témoin en image morphologique perturbée prédite, permettant ainsi une prédiction de « morphologie normale à perturbée ».
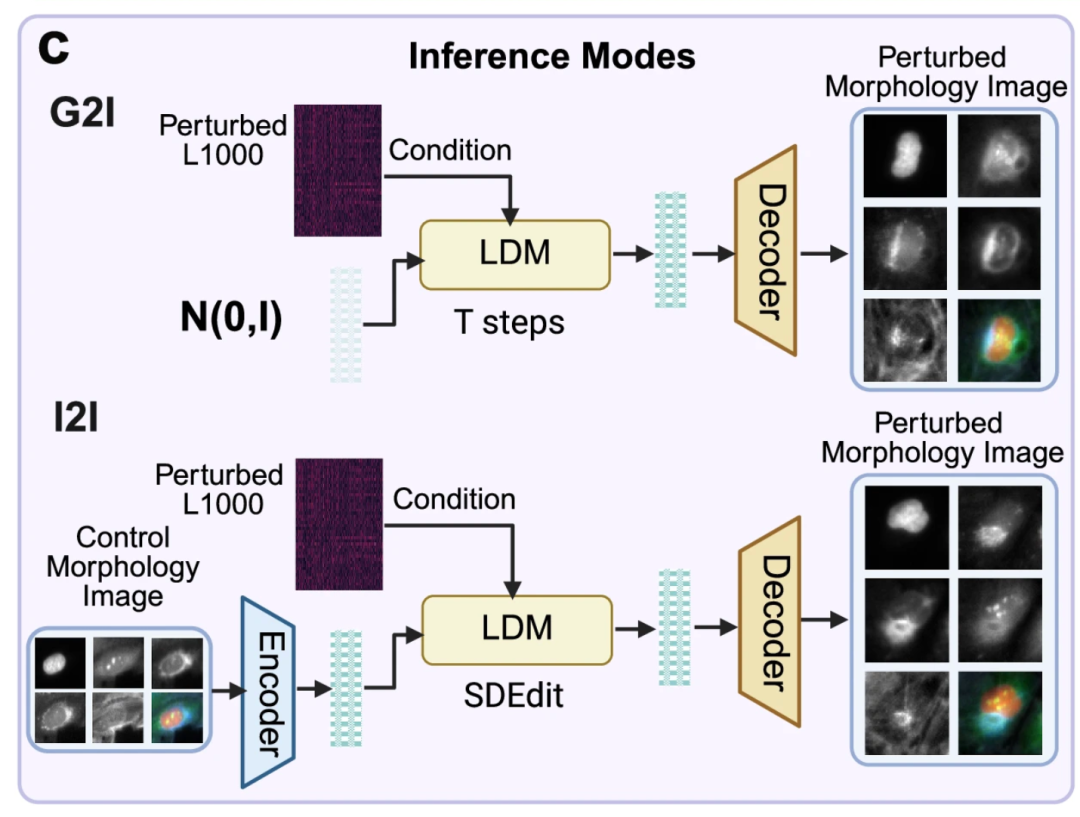
La figure d ci-dessous montre la valeur du modèle MorphDiff dans les applications pratiques.Premièrement, le modèle est capable de prédire les changements morphologiques cellulaires causés par des « perturbations invisibles » qui n’ont pas été rencontrées pendant la formation.Cela permet aux chercheurs de simuler l'état des changements cellulaires sous l'effet de la perturbation de nouveaux médicaments par ordinateur sans avoir à mener d'expériences physiques, les aidant ainsi à explorer davantage de possibilités rapidement et à faible coût ; deuxièmement,Le cadre du modèle combine des outils tels que CellProfiler et DeepProfiler.Il peut aider à identifier le mode d’action des médicaments, favorisant ainsi le développement de médicaments phénotypiques.
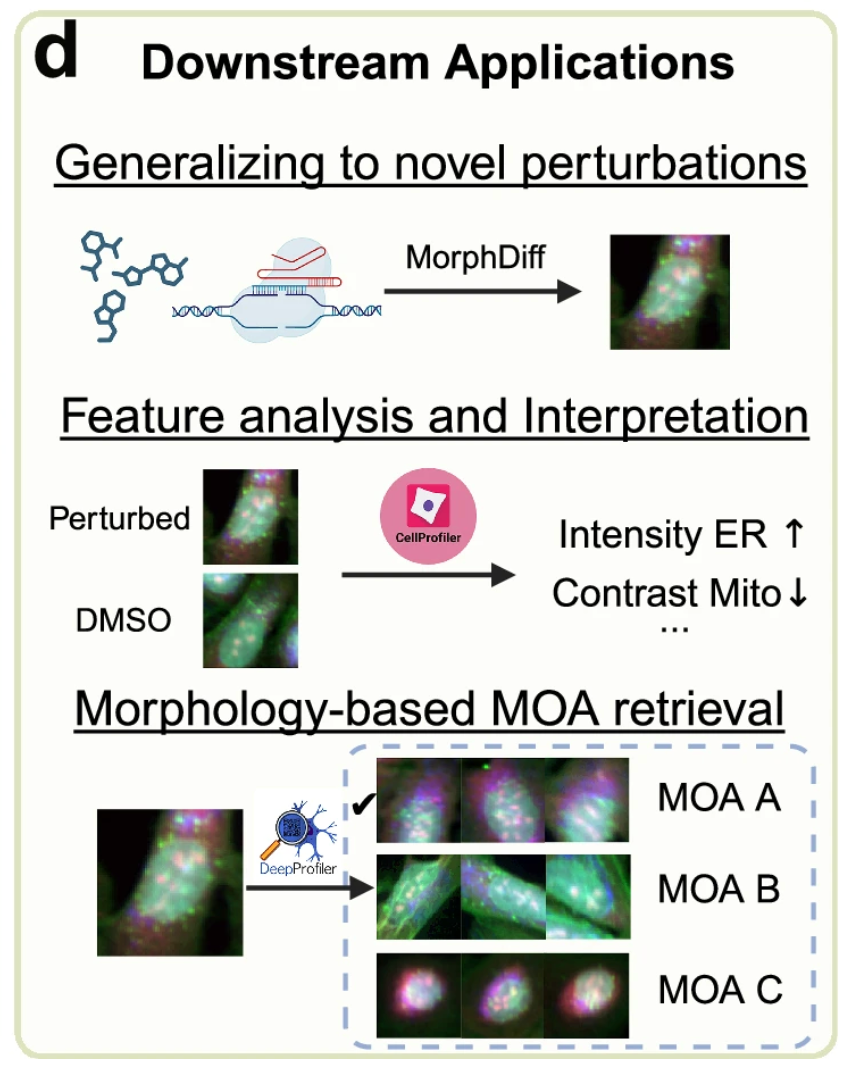
Les chercheurs affirment que MorphDiff est actuellement le seul outil qui prend en charge la génération d’images morphologiques à partir de l’expression génétique et la conversion de la morphologie non perturbée en morphologie perturbée.
Résultats expérimentaux : les performances dépassent celles de l'IMPA, accélérant le développement de médicaments phénotypiques
Afin de vérifier l’efficacité du modèle MorphDiff, les chercheurs ont conçu une série d’expériences à des fins différentes.Grâce à une comparaison expérimentale avec des outils avancés, l’efficacité et la praticité de MorphDiff sont systématiquement vérifiées.
Premièrement, les expériences ont vérifié les prédictions de perturbation génétique.Les chercheurs ont effectué des tests de référence sur l'ensemble de données JUMP OOD et les ont comparés à plusieurs méthodes de base, notamment MorphNet, DMIT (Disentanglement for Multi-mapping Image-to-Image Translation), DRIT++ (Disentangled Representation for Image-to-Image Translation), StarGANv1, IMPA, VQGAN (Vector Quantized Generative Adversarial Network) et MDTv2 (Masked Diffusion Transformers).
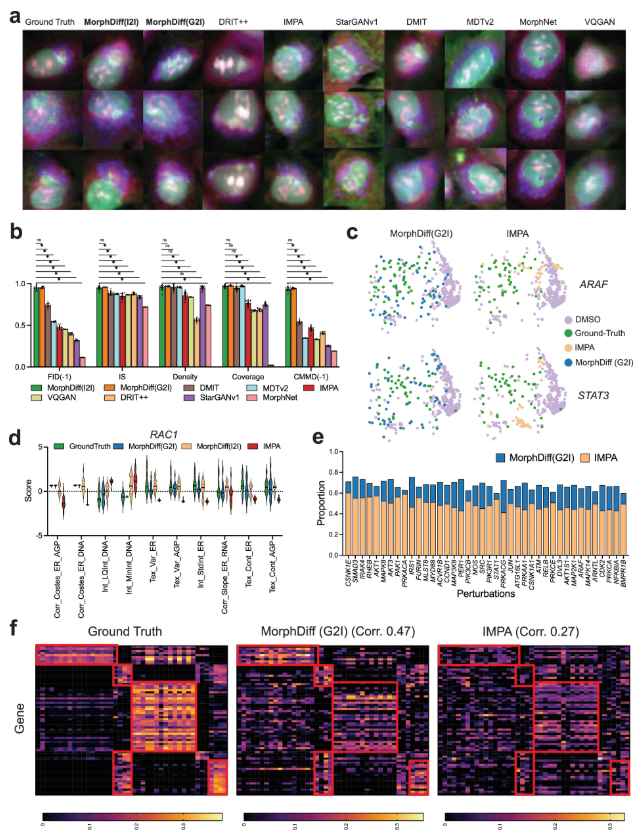
Les résultats montrent queLes résultats générés par les deux modes de MorphDiff sont plus proches de la ligne de base réelle en termes de qualité visuelle et de morphologie structurelle.En termes de mesures quantitatives, notamment le FID, l'IS (Inception Score), le CMMD, la densité et la couverture, les deux modes MorphDiff ont surpassé les méthodes de référence en termes de généralisation, de fidélité et de diversité, obtenant ainsi une qualité de sortie supérieure. Pour prédire les changements de morphologie cellulaire, les résultats de MorphDiff (G2I) sont plus diversifiés et plus proches de la véritable référence ; tandis que les caractéristiques générées par MorphDiff (I2I) présentent un chevauchement plus important avec les caractéristiques de référence réelles, ce qui indique que la précision de sa prédiction est significativement cohérente avec la véritable morphologie de perturbation.
Des expériences ont ensuite été menées pour vérifier la prédiction des perturbations médicamenteuses. Les chercheurs ont d'abord comparé toutes les méthodes à l'aide de l'ensemble de données CDRP OOD.MorphDiff atteint des performances impressionnantes par rapport aux autres méthodes de base dans la plupart des métriques, démontrant ses capacités de génération complètes plus puissantes et plus stables.Les chercheurs ont ensuite procédé à une évaluation plus rigoureuse du modèle sur l’ensemble de données LINCS, en étudiant plus en détail la réponse de la morphologie cellulaire aux composés à petites molécules en comparant les résultats générés avec les fonctionnalités de CellProfiler.
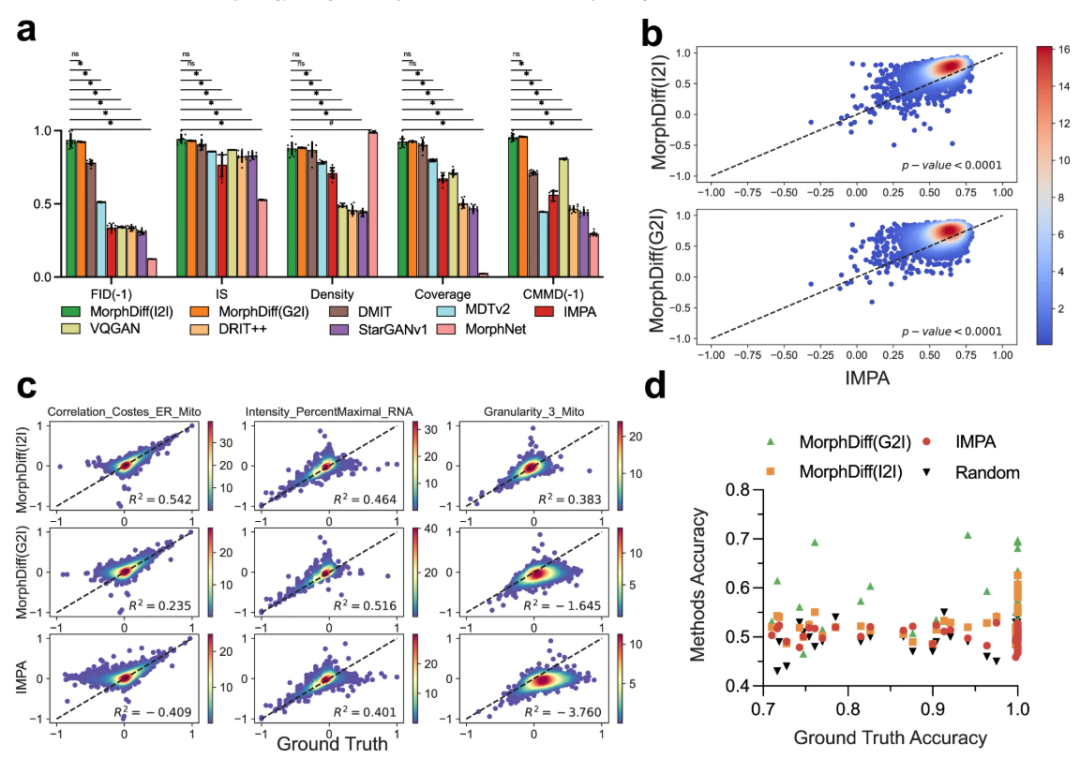
Les résultats montrent que les deux modes de MorphDiff surpassent considérablement la ligne de base.Français Sous G2I, 87,61 échantillons TP3T ont obtenu un score supérieur à 0,5 et 16,21 ont dépassé 0,8. Sous I2I, 891 échantillons TP3T ont obtenu un score supérieur à 0,5 et 27,21 ont dépassé 0,8. En comparaison, 78,31 échantillons TP3T de l'IMPA ont obtenu un score supérieur à 0,5, mais aucun n'a dépassé 0,8. La même analyse sur l'ensemble de données CDRP OOD et l'ensemble de données leave-one-out LINCS a révélé que les deux modèles MorphDiff ont surpassé la méthode de base, avec des valeurs p inférieures à 0,0001, démontrant la généralisabilité de la méthode.
Dans l'analyse d'intégration DeepProfiler,MorphDiff (G2I) peut capturer de manière très fiable et précise les modèles morphologiques cellulaires spécifiques aux perturbations.Au niveau pharmaceutique,Le résultat généré par le modèle G2I présente une spécificité de perturbation supérieure à celle du modèle I2I. Ces démonstrations illustrent le potentiel de MorphDiff dans le criblage de médicaments.
Enfin, l'expérience a également permis de vérifier les capacités de MorphDiff dans le développement de médicaments. Les chercheurs ont sélectionné le jeu de données CDRP Target_MOA pour comparer les deux modes d'application de MorphDiff et IMPA.
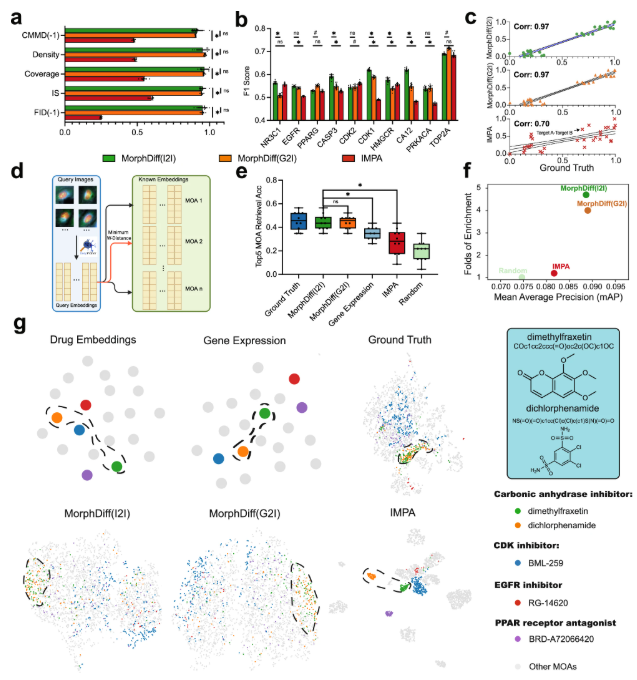
Les résultats montrent que la morphologie de perturbation générée par MorphDiff est très cohérente avec la véritable ligne de base.Cela a démontré sa capacité à capturer la diversité liée à la cible et à comprendre efficacement la relation complexe entre les perturbations des médicaments et leur morphologie. Dans la tâche de récupération du mode d'action (MOA), la précision moyenne des résultats générés par MorphDiff était respectivement de 16,91 TP3T supérieure à celle de la récupération basée sur l'IMPA et de 81 TP3T supérieure à celle de la récupération basée sur l'expression génique. De plus, des expériences ont démontré que MorphDiff peut découvrir que la morphologie cellulaire contient des informations complémentaires et peut identifier des médicaments ayant le même MOA mais des structures différentes, ce qui peut contribuer à accélérer le développement de médicaments phénotypiques.
Les simulations à base de silicium deviennent courantes, accélérant le développement de médicaments en aval
La découverte phénotypique de médicaments diffère de la découverte ciblée de médicaments en ce qu'elle explore et développe des médicaments en observant leurs effets sur des systèmes biologiques globaux ou des phénotypes cellulaires. Cette approche offre des avantages significatifs pour la découverte de nouveaux mécanismes d'action et de nouvelles cibles, ainsi que pour le traitement de maladies complexes. De nombreux laboratoires et instituts de recherche étudient activement ce sujet, intégrant les technologies informatiques à la biomédecine pour ouvrir un nouveau chapitre de la découverte phénotypique de médicaments.
Comme mentionné dans l’article, le modèle IMPA est un modèle génératif profond proposé par une équipe de l’Université technique de Munich en Allemagne et de l’Université d’Oxford au Royaume-Uni.En utilisant la méthode de transfert de style, les images de cellules sont décomposées en « style » (représentation de perturbation/lot) et « contenu » (représentation de cellule), ce qui peut prédire la réponse de la cellule aux perturbations et supprimer les effets de lot.L'article, intitulé « Prédire les réponses morphologiques des cellules aux perturbations à l'aide de la modélisation générative », a également été publié dans Nature Communications.
De plus, dans un article publié par l'équipe de l'Université du Michigan intitulé « MorphNet prédit la morphologie cellulaire à partir de l'expression génétique d'une seule cellule »,a proposé une méthode informatique appelée MorphNet qui peut dessiner des images morphologiques de cellules en fonction de leurs profils d'expression génétique.La méthode utilise des données morphologiques et moléculaires appariées pour former un réseau neuronal à prédire la morphologie du noyau ou de la cellule entière en fonction de l’expression des gènes.
En résumé, promouvoir le développement de médicaments phénotypiques et la recherche biologique par l'observation et l'analyse des changements d'état cellulaire sous l'effet de perturbations génétiques ou médicamenteuses est devenu un sujet important. Concernant MorphDiff, bien qu'il présente encore de nombreuses lacunes, comme la difficulté de prédire des perturbations invisibles face à un grand nombre de nouvelles perturbations extérieures aux données d'apprentissage, il est clair que MorphDiff s'améliore constamment par rapport à ses prédécesseurs, les surpassant en termes de praticité, de généralisation, de simplicité d'utilisation et d'évolutivité.
Obtenez des articles de haute qualité et des articles d'interprétation approfondis dans le domaine de l'IA4S de 2023 à 2024 en un seul clic⬇️









