Command Palette
Search for a command to run...
Le Groupe De Recherche De Yu Xiang À l'Université Jiao Tong De Shanghai a Publié Un Modèle d'apprentissage Profond Transférable Pour Identifier Plusieurs Types De Modifications De l'ARN Et Réduire Considérablement Les Coûts De calcul.

En 2021, grâce à l'appel retentissant de Gao Fu, académicien de l'Académie chinoise des sciences, les vaccins à ARNm sont devenus célèbres du jour au lendemain et sont devenus l'espoir des gens lors de l'épidémie du nouveau coronavirus. Aujourd’hui, ce passé particulier est devenu de l’histoire ancienne, mais la modification de l’ARN derrière les vaccins à ARNm continue de se développer à un rythme rapide.
La modification dite de l'ARN est un type important de régulation post-transcriptionnelle, qui peut être largement impliquée dans divers traitements post-transcriptionnels et voies métaboliques de l'ARN.
La modification de l’ARN mérite l’attention car elle joue une fonction biologique vitale dans la croissance et le développement des organismes eucaryotes.Par exemple, des études récentes ont montré que l’effet déstabilisateur de la N⁶-méthyladénosine (m⁶A) dans les cellules souches embryonnaires de mammifères est lié à diverses maladies, et que la 5-méthylcytosine (m⁵C) est liée à la tolérance du riz aux températures élevées.
Cependant, l’ARN présente de nombreux types de modifications et, à ce jour, plus de 160 types de modifications ont été découverts dans l’ARN naturel. Auparavant, la technologie de séquençage direct d'ARN nanopore (DRS) développée par Oxford Nanopore Technologies (ONT) combinée à des méthodes d'apprentissage en profondeur peut réaliser l'identification de modifications de bases uniques.Cependant, cette méthode a du mal à détecter plusieurs types de modifications simultanément dans un seul échantillon.
En réponse aux questions ci-dessus, le groupe de recherche de Yu Xiang, professeur associé titulaire à l'École des sciences de la vie et de la technologie de l'Université Jiao Tong de Shanghai, et l'équipe de Yang Jun/Wang Hongxia du Jardin botanique de Chenshan de Shanghai ont publié un article de recherche intitulé « L'apprentissage par transfert permet l'identification de plusieurs types de modifications d'ARN à l'aide du séquençage direct de l'ARN par nanopores » dans Nature Communications.Un modèle d’apprentissage profond transférable, TandemMod, a été développé pour permettre l’identification de plusieurs types de modifications d’ARN dans DRS.
Points saillants de la recherche :
* À condition de garantir les mêmes performances, réduire considérablement les coûts de calcul tels que la quantité de données de l'ensemble d'entraînement et le temps d'entraînement du modèle
* TandemMod fournit un support technique important pour l'identification de divers types de sites de modification d'ARN et d'études d'épitranscriptome chez les animaux, les plantes et les micro-organismes
* TandemMod peut également être utilisé pour détecter l'ARN modifié artificiellement, comme les vaccins à ARN

Adresse du document :
https://www.nature.com/articles/s41467-024-48437-4
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Ensemble de données : formation ciblée avec plusieurs ensembles de données
Afin de former et d’évaluer les performances du modèle TandemMod, l’équipe de recherche a utilisé plusieurs ensembles de données pour les expériences.
d'abord,L'équipe de recherche a utilisé l'ensemble de données de transcription in vitro ELIGOS généré par le laboratoire Nookaew.Cinq caractéristiques de niveau de base (moyenne, médiane, écart type, longueur du signal et qualité de base) ont été calculées pour six bases modifiées (m¹A, m⁶A, m⁵C, hm⁵C, m⁷G et Ψ) et comparées aux bases non modifiées.
Deuxièmement, l’équipe de recherche a choisi d’étudier les performances de TandemMod en fonction des deux modifications les plus courantes de l’ARNm eucaryote, m⁵C et m⁶A.Les chercheurs ont formé le modèle TandemMod m⁵C sur l’ensemble de données Curlcake.L'ensemble de données est dérivé de séquences transcrites in vitro contenant tous les 5-mères possibles et est divisé en ensembles d'entraînement et de test dans un rapport de 4:1.
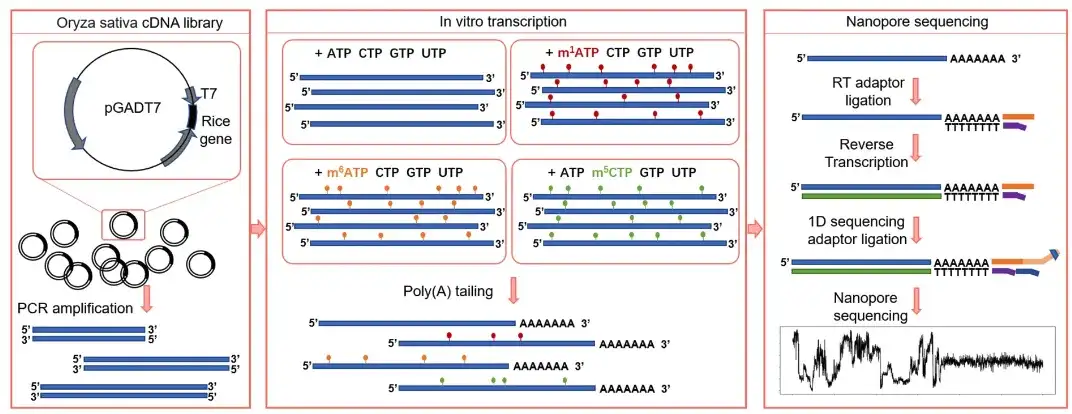
Parallèlement, afin de résoudre le problème selon lequel l'ARN transcrit à partir de séquences synthétiques in vitro ne peut pas couvrir toute la gamme des séquences naturelles, l'équipe de recherche a effectué une transcription in vitro sur une bibliothèque d'ADNc de riz contenant un promoteur T7 et a obtenu des milliers de transcrits avec différentes étiquettes de modification. Après avoir ajouté des queues polyA, quatre ensembles d'entraînement (m¹A, m⁶A, m⁵C et bases non modifiées) ont été construits via DRS.Il s'agit de l'ensemble de données du transcriptome apparent in vitro (IVET).
Architecture du modèle : un cadre d'apprentissage profond
Sur cette base, l’équipe de recherche a utilisé les signaux électriques attribués à chaque 5 bases et leurs caractéristiques statistiques comme entrée pour former le modèle d’apprentissage par transfert TandemMod, qui peut détecter simultanément plusieurs types de modifications d’ARN.
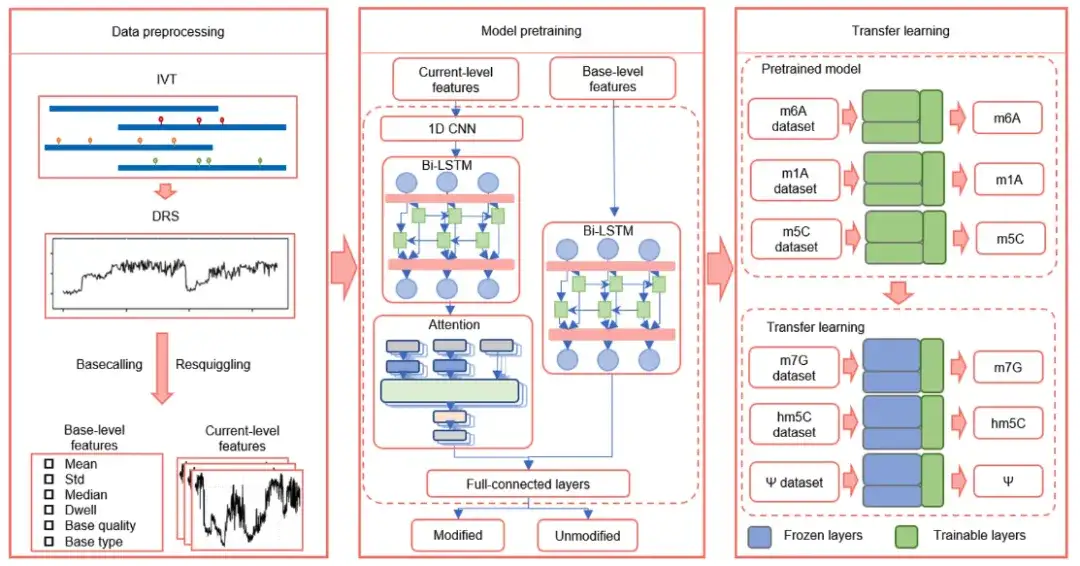
Comme le montre la figure ci-dessus,TandemMod comprend le prétraitement des données, le pré-entraînement du modèle et l'apprentissage par transfert.
Parmi eux, la pré-formation du modèle se compose de 4 composants principaux :
* Réseau neuronal convolutif unidimensionnel (1D-CNN) pour extraire les caractéristiques locales du signal d'intensité de courant d'origine ;
* Bi-LSTM (Bi-LSTM), qui est utilisé pour capturer les corrélations à long terme entre les signaux adjacents et améliorer la capacité à comprendre le contexte dans un processus plus long ;
* Mécanisme d’attention, qui est utilisé pour pondérer l’importance de chaque caractéristique à différents moments et améliorer la capacité du modèle à capturer des signaux importants ;
* Les classificateurs dans les couches entièrement connectées sont chargés de faire des prédictions basées sur la combinaison de toutes les fonctionnalités.
De plus, pour vérifier si l’apprentissage par transfert peut être appliqué aux données DRS pour détecter plusieurs types de modifications d’ARN,Les chercheurs ont formé TandemMod sur l’ensemble de données IVET m5C et ont obtenu un modèle pré-entraîné.Dans le modèle TandemMod, la couche supérieure agit comme un extracteur de fonctionnalités et la couche inférieure agit comme un classificateur. Les chercheurs ont gelé les couches supérieures du modèle pré-entraîné et ont réentraîné les couches inférieures sur l'ensemble d'entraînement ELIGOS (hm5C, m7G, Ψ et I) pour minimiser l'erreur de classification.

Après 2 époques, tous les modèles ont atteint une grande précision.Les ROC-AUC de hm⁵C, m⁷G, Ψ et I ont atteint respectivement 0,98, 0,95, 0,96 et 0,97. Comme le montrent les figures a, b, c et d ci-dessus.
Résultats expérimentaux : TandemMod réduit considérablement la quantité de données d'ensemble d'entraînement et le temps d'entraînement du modèle
Au cours de la phase expérimentale, l'équipe de recherche a comparé le modèle TandemMod avec des algorithmes d'apprentissage automatique classiques pour évaluer ses performances, à savoir XGBoost, la machine à vecteurs de support (SVM) et le k-plus proche voisin (KNN). Dans le cas de l'ensemble de données de test Curlcake m⁶A, reconnaissance,TandemMod surpasse les autres algorithmes avec une précision de 0,90.De même, pour l’identification de m⁵C, TandemMod a atteint une précision de 0,95, et cette comparaison met en évidence l’efficacité de TandemMod dans l’identification des modifications à l’aide des données DRS.
TandemMod a également montré une meilleure supériorité que tombo et xPore dans l'identification d'échantillons avec différents niveaux de taux de modification in vivo.Cela indique que TandemMod peut prédire avec précision des échantillons avec différents taux de modification sans avoir besoin d'échantillons de contrôle négatifs.
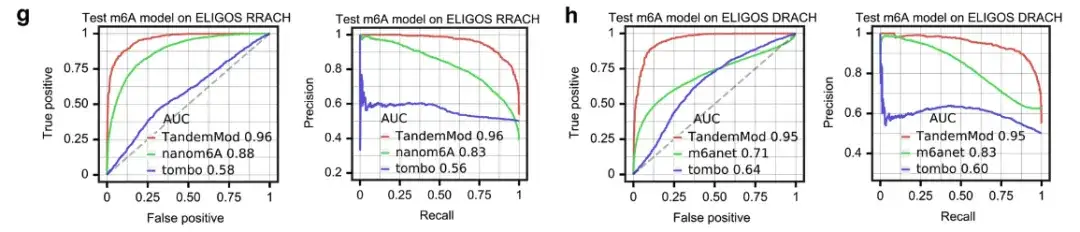
De plus, l'équipe de recherche a également comparé le modèle TandemMod m⁶A avec tombo, nanom6A et m6Anet, comme le montre la figure ci-dessus.
Sur le motif d'éruption cutanée ELIGOS (RA ou G, HA ou C ou U), les ROC-AUC de TandemMod, nanom6A et tombo étaient respectivement de 0,96, 0,88 et 0,52. Sur le motif ELIGOS DRACH (DA, G ou U), les ROC-AUC de TandemMod, m6Anet et tombo étaient respectivement de 0,95, 0,71 et 0,64.
Ces résultats indiquent queFormé à l'aide de l'ensemble de données DRS in vitro, TandemMod fournit les prédictions de niveau de lecture les plus précises parmi les outils existants.
L'équipe de recherche a vérifié les performances de classification, les données de formation requises et l'utilisation des ressources informatiques de l'apprentissage par transfert du modèle TandemMod m⁵C dans la détection m⁶A, et les a comparées au modèle TandemMod m⁶A de l'instance standard. Les résultats montrent que l’apprentissage par transfert peut réduire considérablement les coûts tels que la quantité de données d’ensemble d’entraînement et le temps d’entraînement du modèle tout en garantissant les mêmes performances.
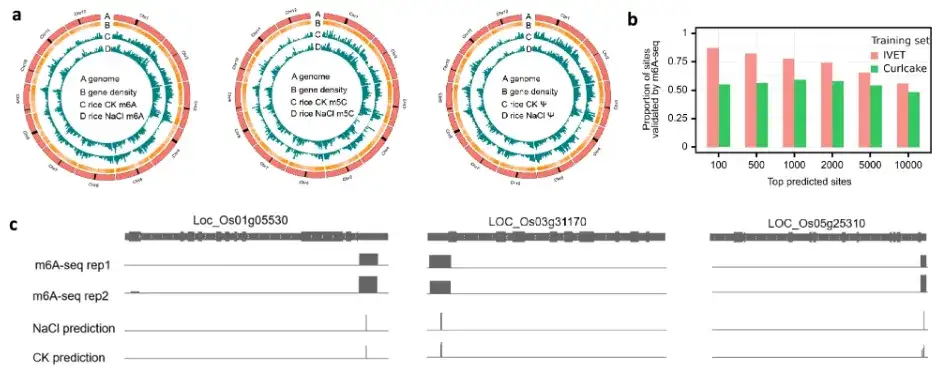
Enfin, l'équipe de recherche a testé la capacité du modèle TandemMod à être étendu à de nouvelles espèces pour le séquençage des données DRS et a vérifié davantage la fiabilité de TandemMod en utilisant des lignées cellulaires humaines (2 échantillons de suppression d'enzymes de modification et 5 échantillons de type sauvage). Dans le même temps, l'équipe de recherche a également utilisé TandemMod pour cartographier les cartes de modification épigénétique de m⁶A, m⁵C et Ψ dans les semis de riz soumis à un stress salin élevé, et a révélé la co-modification de m⁶A et m⁵C dans l'ARNm et les changements dans leurs taux de modification dans des environnements à forte teneur en sel. Comme le montre l'image ci-dessus.
La modification de l'ARN ouvre de nouvelles portes pour explorer la vie
À travers les âges, les hommes n’ont jamais cessé d’explorer la vie. Après que l’hypothèse du monde de l’ARN a été proposée, l’argument selon lequel l’ARN est à l’origine de la vie est sans aucun doute devenu l’une des réponses les plus convaincantes à l’heure actuelle. Depuis la découverte de la première modification de l’ARN en 1960, elle est depuis longtemps une priorité absolue pour la recherche scientifique et continue de recevoir une grande attention dans les études récentes.
Outre le groupe de recherche de Yu Xiang et l'équipe de Yang Jun/Wang Hongxia dans cet article, ainsi que la société ONT mentionnée dans l'article, de nombreuses autres équipes et sociétés mènent également des recherches sur la modification de l'ARN.

Par exemple, en 2021, l'équipe du professeur Meng Jia de l'Université Xi'an Jiaotong-Liverpool a publié un article intitulé « Réseaux neuronaux multi-étiquettes basés sur l'attention pour la prédiction et l'interprétation intégrées de douze modifications d'ARN largement répandues » dans la revue Nature Communications.
Adresse du document :https://www.nature.com/articles/s41467-021-24313-3
L'article mentionne un modèle MultiRM basé sur un framework d'apprentissage profond multi-étiquettes avec mécanisme d'attention.Non seulement 12 sites de transcriptome largement existants peuvent être prédits simultanément, mais les séquences clés du processus de prédiction sont également extraites et analysées, révélant une forte corrélation entre différents types de modifications d'ARN, ce qui permet de mieux analyser et comprendre de manière exhaustive les mécanismes de modification d'ARN basés sur les séquences.

Par coïncidence, dans un article de 2021 intitulé « Identification des modifications différentielles de l'ARN à partir du séquençage direct de l'ARN par nanopore avec xPore » publié dans Nature Biotechnology,L'équipe de recherche a utilisé xPore pour identifier les modifications de l'ARN avec une grande précision à partir des données Direct RNA-seq et analyser les modifications et l'expression différentielles à partir d'une seule expérience à haut débit.
Adresse du document :https://www.nature.com/articles/s41587-021-00949-w
Ces études nous aident à ouvrir davantage la porte au monde de l’ARN, nous permettant d’explorer davantage le « vrai sens de la vie ». Bien qu'il reste encore de nombreux obstacles à surmonter dans l'avancement de diverses recherches, les défis continus des « pionniers » ont déjà ouvert la porte à la recherche sur l'ARN.
Références :
1. https://news.sjtu.edu.cn/jdzh/2








