Command Palette
Search for a command to run...
نُشرت في مجلة Cell! طورت مجموعة البحث التي يقودها Zhang Qiangfeng من جامعة Tsinghua خوارزمية SPACE، التي تتمتع بقدرة اكتشاف الوحدة التنظيمية الرائدة بين الأدوات المماثلة
على الرغم من أن الخلايا في الكائنات متعددة الخلايا تشترك في نفس الجينوم، إلا أنها تظهر تنوعًا كبيرًا في الشكل والتعبير الجيني والوظيفة بسبب الاختلافات في شبكات تنظيم الجينات الداخلية وتبادل الإشارات الخارجية مع الخلايا المجاورة في البيئة المحيطة. من أجل ربط معلومات نوع الخلية بموقعها المكاني داخل الأنسجة، ظهرت تقنية النسخ المكاني (ST).لا تستطيع هذه التقنية الحصول على بيانات النسخ عالية الدقة فحسب، بل تتوافق أيضًا مع المعلومات الموضعية لتحديد التوزيع المكاني والعلاقة الموضعية لأنواع الخلايا المختلفة أو الحالات النسخية. ويلعب دورًا رئيسيًا في إعادة فهم بنية الحياة، والتطور الفردي، وتطور الحياة، وتحديد الأمراض.
في السنوات الأخيرة، ومع التطوير المستمر لتكنولوجيا النسخ المكاني، أصبح الباحثون قادرين على الحصول على ملف التعبير الجيني للخلايا بدقة الخلية الواحدة مع الاحتفاظ بمعلومات الموقع المكاني للخلايا داخل الأنسجة. أصبحت كيفية استخدام هذه المعلومات المكانية بشكل فعال لتحديد الأنواع الفرعية للخلايا المكانية واكتشاف وحدات الأنسجة مهمة أساسية في تحليل بيانات النسخ المكاني.
في الوقت الحالي، يواجه تحليل بيانات النسخ المكاني التحديين التاليين: أولاً، لتحديد أنواع الخلايا المكانية، تستخدم العديد من الدراسات ملفات تعريف التعبير الجيني للخلايا فقط وتتجاهل معلومات الموقع المكاني للخلايا. أظهرت الأبحاث في السنوات الأخيرة أن أنواع الخلايا، التي كان يُعتقد في الأصل أنها متجانسة، يمكن تقسيمها إلى أنواع فرعية متعددة اعتمادًا على موقعها في الأنسجة. ثانياً، بالنسبة لنوع الشعر في وحدات الأنسجة، بما أن خصائص التعبير الجيني للخلايا المختلفة التي تشكل الأنسجة قد تكون غير متجانسة إلى حد كبير، فقد فشلت طرق التحليل السابقة في الاستفادة الكاملة من تباين أنواع الخلايا في بيانات النسخ المكاني بدقة الخلية الواحدة.
وبناء على هذا،مجموعة الأبحاث التابعة للأستاذ المشارك تشانغ تشيانغفينج في كلية علوم الحياة، جامعة تسينغهوا/مركز الابتكار المتقدم في علم الأحياء البنيوي/مركز تسينغهوا-بكين المشترك لعلوم الحياة،نُشرت مؤخرًا ورقة بحثية بعنوان "اكتشاف وحدة الأنسجة في بيانات النسخ المكاني بدقة خلية واحدة عبر تضمين الخلايا مع مراعاة التفاعل بين الخلايا" على الإنترنت في مجلة Cell Systems.
طورت الدراسة خوارزمية الذكاء الاصطناعي SPACE (تحليل بيانات النسخ المكاني عبر تضمين الخلايا "الواعي بالتفاعل") استنادًا إلى إطار التعلم العميق للمشفر التلقائي للرسم البياني.يمكن استخدام القدرة على تحديد أنواع الخلايا المكانية واكتشاف وحدات الأنسجة من بيانات النسخ المكاني بدقة الخلية الواحدة لدراسات النسخ المكاني واسعة النطاق.
أبرز الأبحاث:
* تم تطوير SPACE، وهي أداة تحليل النسخ المكاني القائمة على الذكاء الاصطناعي والتي يمكنها تحديد أنواع الخلايا المكانية واكتشاف وحدات الأنسجة من بيانات النسخ المكاني بدقة خلية واحدة
* يتفوق SPACE بشكل كبير على الأدوات الأخرى في تحديد نوع الخلية واكتشاف وحدات الأنسجة، وخاصة في الأنسجة المعقدة التي تحتوي على أنواع متعددة من الخلايا
* يمكن استخدام SPACE في دراسات النسخ المكاني واسعة النطاق لفهم كيفية تأثير التفاعلات بين الخلايا المجاورة مكانيًا على الوظائف البيولوجية لأنواع الخلايا ووحدات الأنسجة
عنوان الورقة:
https://www.cell.com/cell-systems/fulltext/S2405-4712(24)00124-8
يجمع المشروع المفتوح المصدر "awesome-ai4s" أكثر من مائة تفسير ورقي لـ AI4S ويوفر مجموعات وأدوات ضخمة من البيانات:
https://github.com/hyperai/awesome-ai4s
مجموعات البيانات: مجموعات بيانات متعددة تتحقق من قدرات SPACE
من أجل التحقق من قدرات SPACE، تم استخدام مجموعات بيانات متعددة في الدراسة، والتي تم تلخيصها على النحو التالي:
عنوان تنزيل مجموعة البيانات:
https://go.hyper.ai/CBJfX
مجموعة بيانات PMC للفأر MERFISH
بالنسبة لمجموعة بيانات PMC الخاصة بفئران MERFISH، تم الحصول على مصفوفة الجينات الخلوية الطبيعية المحولة باللوغاريتم من مكتبة صور الدماغ، وتم إزالة الخلايا التي تم تصنيفها على أنها "أخرى" أو تقع خارج منطقة العينة الرئيسية.
رابط مجموعة البيانات:
مجموعة بيانات PLA للفأر STARmap
بالنسبة لمجموعة بيانات PLA الخاصة بفأر STARmap، تم توفير مصفوفة الخلية الجينية الطبيعية بواسطة الورقة الأصلية وتم تحويلها لوغاريتميًا.
رابط مجموعة البيانات:
https://drive.google.com/file/d/1DDCowUuZ7PPFUSZsjvSqntWkYJMjf1Na/view?usp=sharing
مجموعة بيانات فئران MERFISH AB
بالنسبة لمجموعة بيانات فئران MERFISH AB، تم الحصول على مصفوفة تعداد الجينات من مكتبة CELL x GENE. تم تطبيع العدد الإجمالي لكل خلية إلى 10000، وتم تحويل مصفوفة الخلية الجينية الطبيعية لوغاريتميًا.
رابط مجموعة البيانات:
https://cellxgene.cziscience.com/collections/31937775-06024e52-a799-b6acdd2ba2e
مجموعة بيانات MERFISH فئران WB
بالنسبة لمجموعة بيانات WB الخاصة بفأر MERFISH، تم الحصول على مصفوفة الجينات الخلوية الطبيعية المحولة بالسجل من مستودع GitHub.
رابط مجموعة البيانات:
https://github.com/AllenInstitute/abc_atlas_access
مجموعة بيانات Xenium Human BC
بالنسبة لمجموعة بيانات سرطان الثدي البشري Xenium، تم الحصول على مصفوفة تعداد الجينات من موقع 10x genomics. تم تطبيع العدد الإجمالي لكل خلية إلى 10000، وتم تحويل مصفوفة الخلية الجينية الطبيعية لوغاريتميًا.
رابط مجموعة البيانات:
https://www.10xgenomics.com/products/xenium-in-situ/preview-dataset-human-breast
مجموعة بيانات سرطان الرئة ذو الخلايا غير الصغيرة البشري CosMx
بالنسبة لمجموعة بيانات سرطان الرئة ذو الخلايا غير الصغيرة البشري CosMx، تم الحصول على مصفوفة الجينات الخلوية الطبيعية المحولة لوغاريتميًا من موقع nanoString.
رابط مجموعة البيانات:
https://nanostring.com/products/cosmx-spatial-molecular-imager/ffpe-dataset/nsclc-ffpe-dataset
مجموعة بيانات الدماغ البشري Visium
بالنسبة لمجموعة بيانات الدماغ البشري Visium، تم الحصول على مصفوفة تعداد الجينات باستخدام حزمة Bioconductor spatialLIBD. تم التعرف على أفضل 3000 جين شديد التغير في كل عينة من مجموعة بيانات الدماغ البشري Visium باستخدام دالة scanpy.pp.highly_variable_genes() (flavor = "seurat_v3") من حزمة Python SCANPY (v1.9.1). تم بعد ذلك تطبيع العدد الإجمالي لكل خلية إلى 10000، وتم تحويل مصفوفة الخلية الجينية الطبيعية لوغاريتميًا.
رابط مجموعة البيانات:
https://bioconductor.org/packages/release/data/experiment/html/spatialLIBD.html
هندسة النموذج: نموذج مدمج في الخلايا يراعي التفاعل بين الخلايا
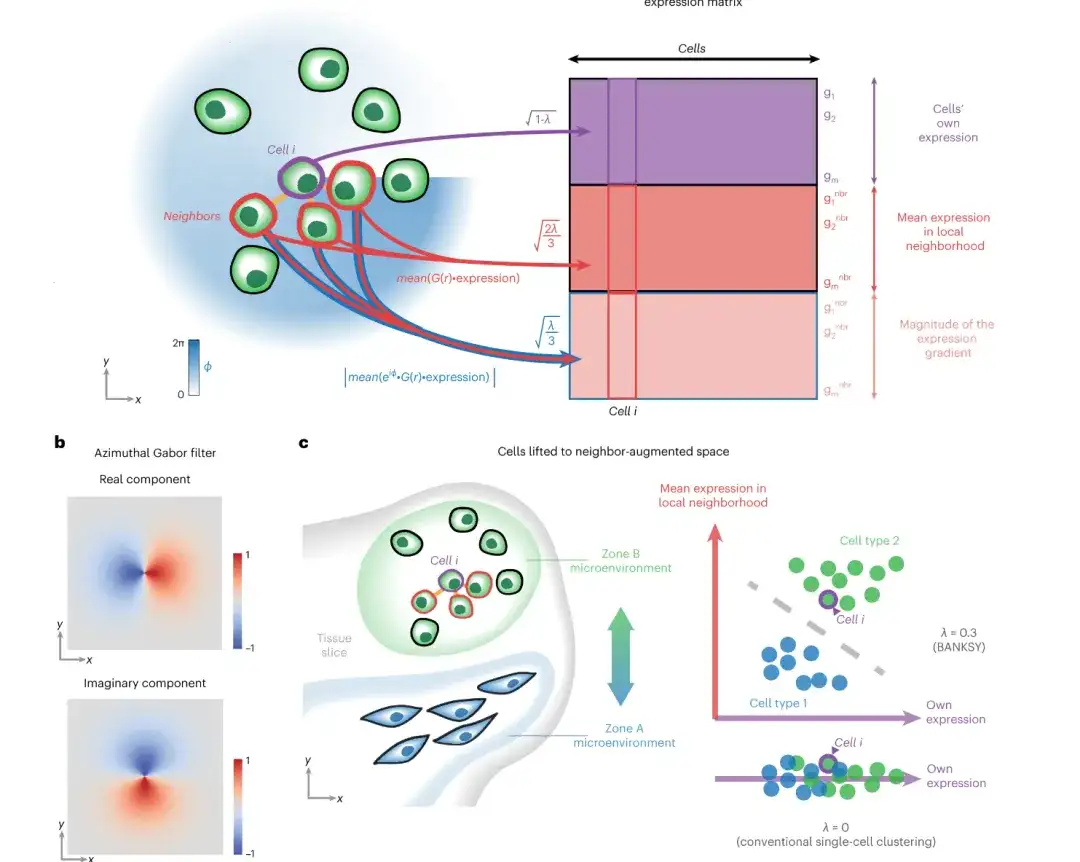
يستخدم SPACE إطار عمل ترميز تلقائي للرسم البياني لتعلم تضمينات الخلايا منخفضة الأبعاد التي تصف معلومات التعبير الجيني لكل خلية في بيانات النسخ المكاني بالإضافة إلى معلومات تفاعلها مع الخلايا المجاورة المكانية (ومن ثم يُطلق على تضمين الخلايا اسم تضمين الخلايا الواعي للتفاعل بين الخلايا). وبناءً على تضمين الخلايا هذا، يستخدم SPACE بعد ذلك خوارزميات التجميع لتحديد الأنواع الفرعية للخلايا المكانية واكتشاف وحدات الأنسجة.
من منظور الهندسة المعمارية،يتكون نموذج SPACE من ثلاثة أجزاء: المشفر (شبكة انتباه الرسم البياني ثلاثية الطبقات)، وفك تشفير الرسم البياني المجاور، وفك تشفير التعبير الجيني.الشكل التالي يوضح الإطار العام للنموذج:
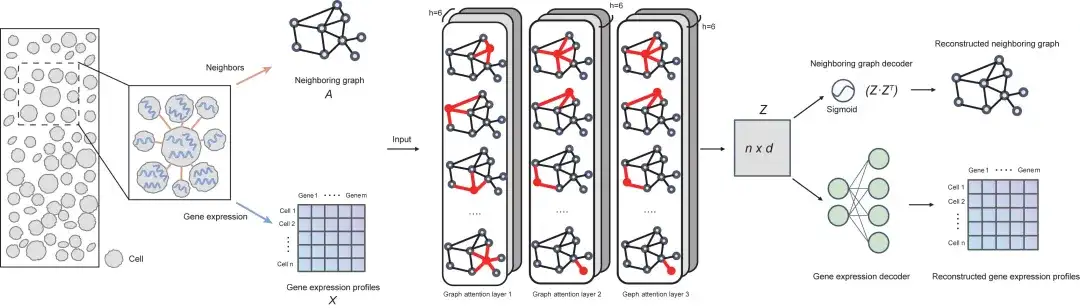
أولاً، يقوم SPACE ببناء رسم بياني للتجاور من خلال ربط كل خلية بأقرب k خلية مجاورة لها بناءً على القرب المكاني؛ بعد ذلك، يستخدم SPACE شبكة انتباه بيانية ثلاثية الطبقات (GAT) كمشفر لتحويل مدخلات ملف تعريف التعبير الجيني ورسم الجوار إلى تمثيلات خلوية منخفضة الأبعاد، والتي يتم استخدامها لاحقًا لإعادة بناء ملف تعريف التعبير الجيني ورسم الجوار لكل خلية من خلال شبكتين فك تشفير مستقلتين.
لتدريب نموذج GAE، يستخدم SPACE التعلم الذاتي الإشرافي، بهدف تقليل الخسارة الكلية لإعادة بناء ملفات تعريف التعبير الجيني والرسوم البيانية المجاورة. يمكن بعد ذلك استخدام تمثيلات الخلايا المكتسبة لتحديد نوع الخلية واكتشاف الوحدة التنظيمية باستخدام خوارزميات التجميع المختلفة.
استخدمت أدوات التعلم العميق التي تم تطويرها سابقًا شبكات التفافية بيانية (GCNs) (على سبيل المثال، SpaGCN، وSpaceFlow، وGraphST، وSEDR) أو مشفرات انتباه الرسم البياني التلقائية (على سبيل المثال، STAGATE) لتوليد تضمينات "واعية بالجوار" تكتشف وحدات التنظيم من خلال تجميع ملفات تعريف التعبير الجيني للخلايا وجيرانها. يختلف SPACE عن هذه الأدوات في ثلاثة أمور رئيسية:
أولاً، يحتاج مشروع SPACE إلى إعادة بناء كل من ملفات تعريف التعبير الجيني والرسوم البيانية المجاورة من نفس تمثيل الخلايا منخفض الأبعاد (عبر فكين مستقلين).يتيح هذا التصميم لبرنامج SPACE تذكر ملفات تعريف التعبير الجيني والتفاعلات المكانية للخلية التي تم تحليلها والخلايا المجاورة لها بشكل فردي. على النقيض من ذلك، تأخذ الطرق الأخرى الرسم البياني المجاور كمدخل ولكنها لا تعيد بناء الرسم البياني. ولتأكيد هذا التمييز، أطلقت هذه الدراسة على تضمينات الخلايا التي تم إنشاؤها بواسطة SPACE اسم "تضمينات الخلايا الواعية للتفاعل بين الخلايا".
ثانيًا، يحدد SPACE نسبة المجال الاستقبالي لتحديد الأوزان النسبية لخسائر إعادة بناء ملف التعبير الجيني والرسم البياني المجاور.تتيح هذه النسبة القابلة للتعديل لـSPACE تخصيص التركيز على التعلم لتلبية احتياجات بحثية محددة، مع التركيز على ملف التعبير الجيني لكل خلية تم تحليلها أو تفاعلات الخلايا المجاورة مكانيًا.
ثالثًا، يستخدم SPACE أيضًا آلية الانتباه في مشفر GAT لتعلم وزن كل حي بشكل تكيفي أثناء عملية تجميع معلومات الحي.يأخذ هذا النهج في الاعتبار تلقائيًا المساهمات الخاصة بالأحياء المختلفة في إعادة بناء ملفات تعريف التعبير الجيني.
نتائج البحث: يتفوق SPACE على الأدوات المماثلة الأخرى في تحديد نوع الخلية واكتشاف وحدات الأنسجة
تم اختبار SPACE باستخدام مجموعات بيانات النسخ المكانية المتعددة، مما يدل على أن مجتمعات الخلايا التي اكتشفها SPACE كانت مماثلة في خصائص التوزيع المكاني للهياكل النسيجية الموضحة يدويًا.
تقييم قدرة SPACE على تحديد أنواع الخلايا ذات المعلومات المكانية
لقد استخدمنا في البداية مجموعة بيانات ST المميزة بـ MERFISH للقشرة الحركية الأولية للفأر (PMC) (بدءًا من الشريحة 153) للتحقيق في قدرة SPACE على تحديد أنواع الخلايا. وتظهر النتائج أنوقد تطابقت أنواع الخلايا التي حددها SPACE بشكل جيد مع تلك المذكورة في الدراسة الأصلية.كما هو موضح في الشكل أدناه؛ بالإضافة إلى ذلك، يوفر SPACE أيضًا تعليقات على أنواع الخلايا بدقة أعلى لأنواع معينة من الخلايا (مثل الخلايا النجمية والخلايا القليلة التغصن).
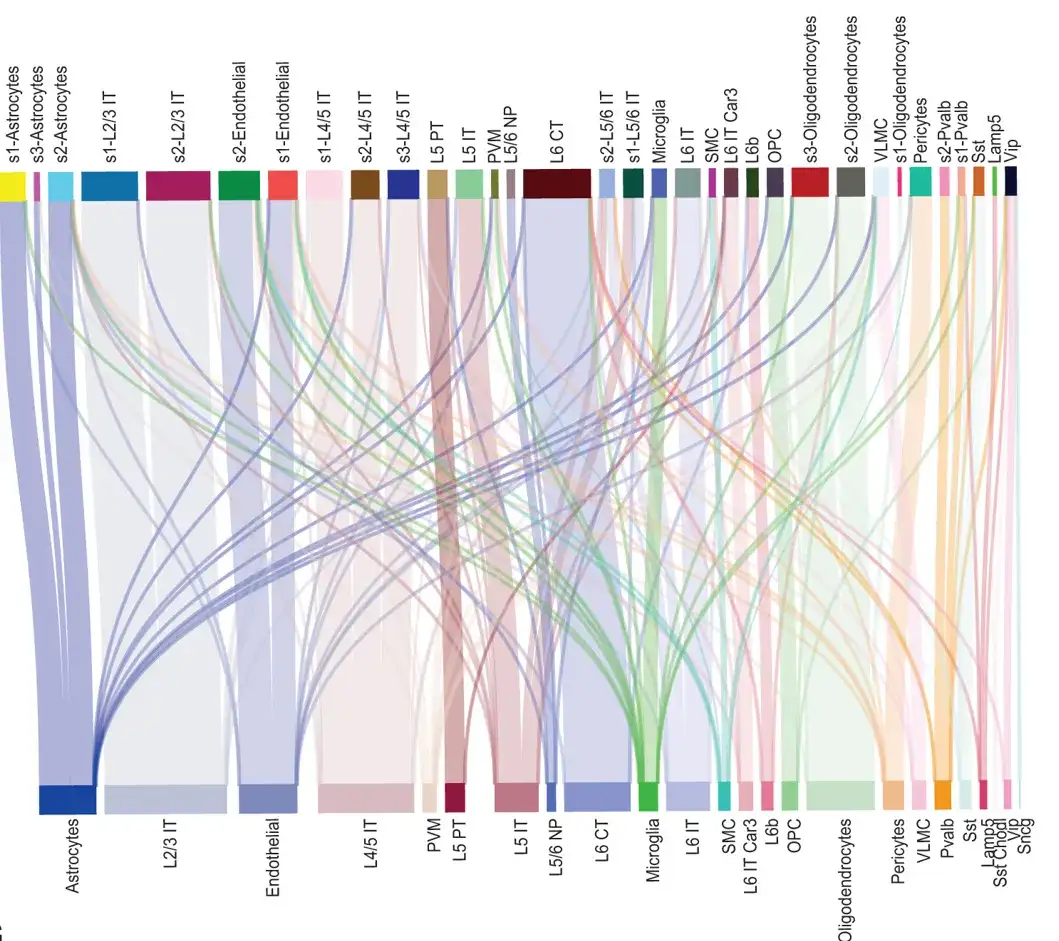
مخطط سانكي يوضح المراسلات بين أنواع الخلايا ذات الصلة وأنواع الخلايا الأصلية في المعلومات المكانية لجميع الخلايا في الشريحة 153 من مجموعة بيانات الخلايا البلعمية العصبية الفأرية MERFISH
وركز الباحثون بعد ذلك على الأنواع الفرعية التي تم تحديدها من الخلايا النجمية (الخلايا الدبقية في القشرة المخية) والخلايا القليلة التغصن (خلايا غمد الميالين في الجهاز العصبي المركزي). كانت الخلايا النجمية تعتبر في السابق نوعًا متجانسًا من الخلايا، لكن دراسات ST الحديثة أفادت أن لها وظائف مميزة في مناطق مختلفة من الدماغ.
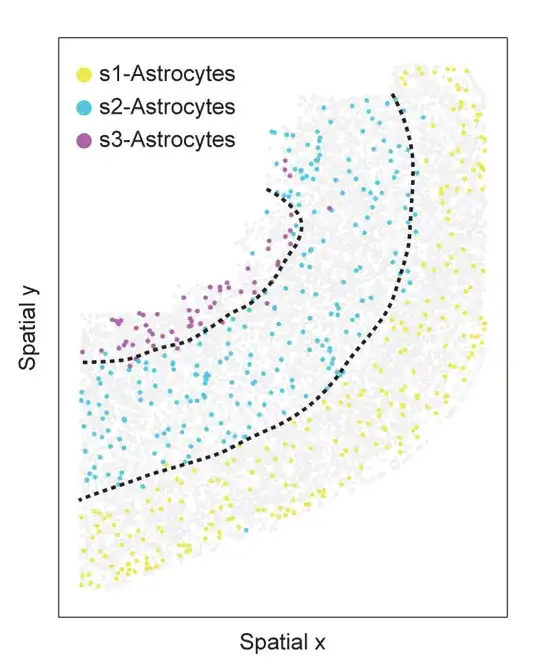
الأنواع الفرعية للخلايا النجمية المرتبطة بالمعلومات المكانية في الشريحة 153 من مجموعة بيانات الخلايا النجمية الأولية للفأر MERFISH. يتم تلوين الخلايا حسب النوع الفرعي للخلايا النجمية، وتمثل النقاط الرمادية الفاتحة الخلايا الأخرى. تشير الخطوط المتقطعة إلى المادة العلوية والعميقة والبيضاء
في التجربة، وجد SPACE ثلاثة أنواع فرعية مختلفة في شريحة PMC 153، كما هو موضح في الشكل أعلاه، حيث تم توزيع كل نوع فرعي مكانيًا في طبقة قشرية مختلفة. على غرار الخلايا النجمية، يصنف SPACE أيضًا الخلايا القليلة التغصن إلى ثلاثة أنواع فرعية ذات معلومات مكانية مع أنماط توزيع مكانية مميزة.

كما طبق الباحثون أيضًا SPACE على مجموعة بيانات المشيمة الفأرية (PLA) التي تم إنشاؤها بواسطة STARmap، وهي تقنية ST أخرى. وتظهر النتائج أن SPACE قام بتصنيف الخلايا إلى 16 نوعًا من الخلايا، وهو ما يتطابق جيدًا مع أنواع الخلايا في الدراسة الأصلية، كما هو موضح أعلاه. وقد حدد برنامج SPACE نوعين فرعيين من الخلايا الغاذية السكرية، وقد تم تصنيف كل منهما على أنهما خلايا "الخلايا الغاذية الضخمة 2" في الدراسة الأصلية. يتمركز النوعان الفرعيان في مناطق مميزة من المشيمة ولديهما أنواع مميزة من الخلايا المجاورة المتفاعلة.
باختصار، فإن تحليل مجموعتين مستقلتين من البيانات استنادًا إلى أساليب ST والمنظمات المختلفة يدعم الاستنتاجات التالية:يتمكن SPACE من تحديد أنواع الخلايا المتميزة بيولوجيًا استنادًا إلى المعلومات المكانية في مجموعة بيانات ST.
تقييم أداء SPACE في تحديد نوع الخلية
قام الباحثون بمقارنة SPACE بأداتين حاليتين لتحديد أنواع الخلايا من بيانات النسخ المكانية، BANKSY وFICT، والتي تأخذ في الاعتبار المعلومات المكانية بالإضافة إلى التعبير الجيني. وفي تحليلهم، أدرج الباحثون أيضًا أداة SCANPY، وهي أداة تستخدم على نطاق واسع لتحديد نوع الخلية، على الرغم من أنها تأخذ في الاعتبار التعبير الجيني فقط.
للمقارنة، استخدم الباحثون مجموعة بيانات PMC الخاصة بالفأر MERFISH المذكورة سابقًا ومجموعة بيانات PLA الخاصة بالفأر STARmap. كما هو موضح في الشكل أدناه، فإن SPACE قادر على تحديد أنواع فرعية مختلفة من الخلايا النجمية والخلايا القليلة التغصنات ذات المعلومات المكانية، ولكن لا يمكن لـ SCANPY ولا FICT تعريف الأنواع الفرعية من الخلايا النجمية والخلايا القليلة التغصنات باستخدام أنماط التوزيع المكاني المحددة حسب الطبقة القشرية.
بالنسبة لمجموعة بيانات PLA الخاصة بفئران STARmap، على الرغم من أن SPACE وBANKSY نجحا في تحديد النوعين الفرعيين من الخلايا الغاذية السكرية، إلا أن SCANPY وFICT فشلا في تحديد النوعين الفرعيين من الخلايا الغاذية السكرية، وقد يكون ذلك بسبب الاختلافات الواضحة في أنواع الخلايا المحيطة بين النوعين الفرعيين من الخلايا الغاذية السكرية.
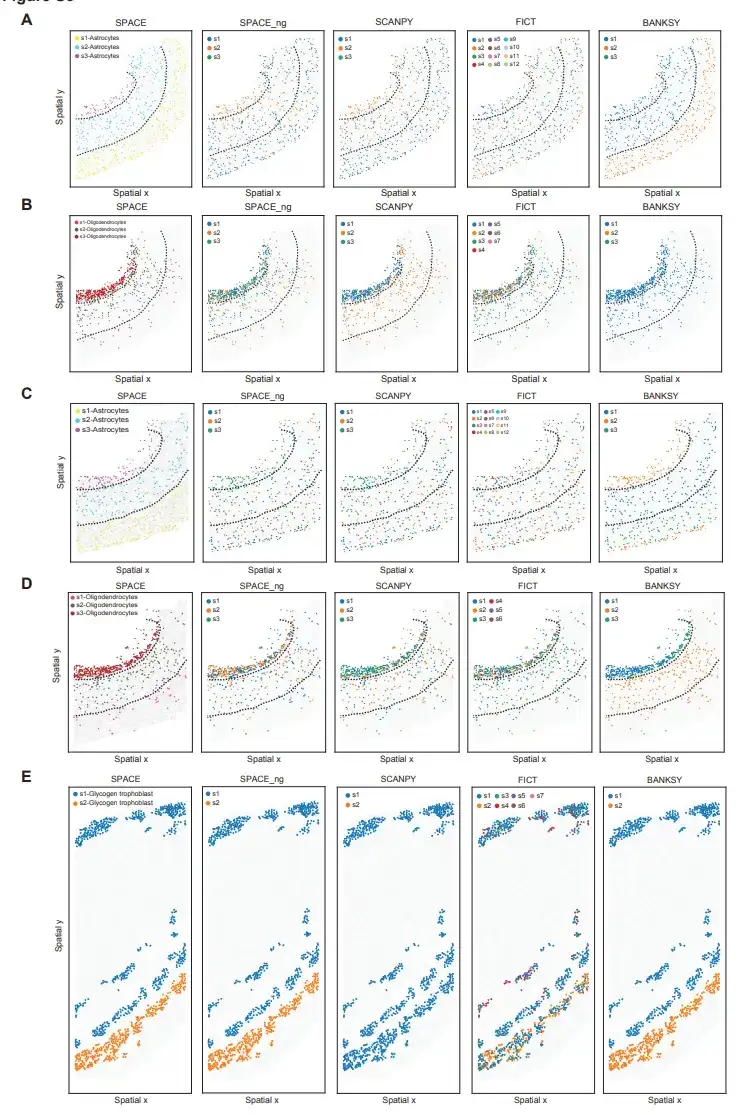
تشير هذه النتائج مجتمعة إلى أنيتفوق SPACE على الأدوات المتاحة حاليًا للتمييز بين أنواع الخلايا ذات المعلومات المكانية من بيانات ST.
يتفوق SPACE على الأدوات الحديثة في اكتشاف وحدات الأنسجة
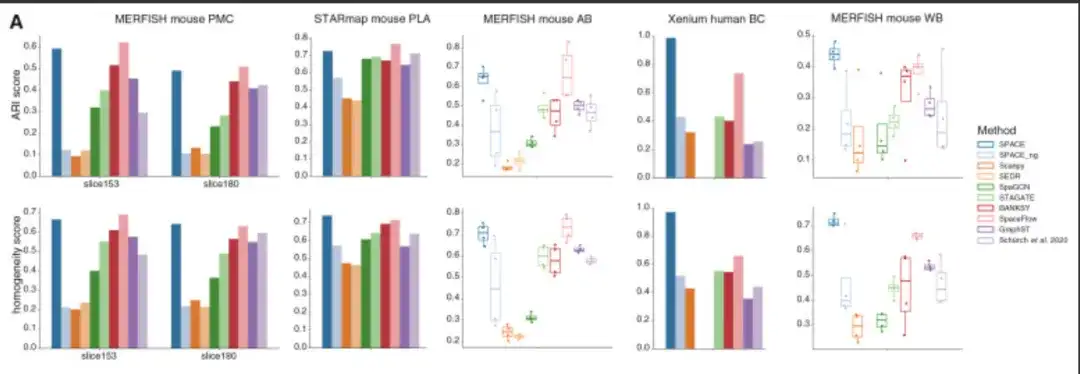
إن إحدى المهام المهمة في دراسات النسخ المكانية هي اكتشاف الوحدات التنظيمية داخل نسيج معين. لتقييم قدرات SPACE في هذا الصدد، قارن الباحثون SPACE مع SEDR وSpaGCN وSTAGATE وBANKSY وSpaceFlow وGraphST وطرق Schürch et al. بالإضافة إلى SCANPY وSPACE_ng، واستخدموا مجموعتين من مجموعات بيانات ST المذكورة أعلاه (مجموعة بيانات MERFISH mouse PMC ومجموعة بيانات STARmap mouse PLA)، بالإضافة إلى ثلاث مجموعات بيانات إضافية مع وحدات الأنسجة الموضحة، بما في ذلك مجموعة بيانات دماغ الفأر المسن (AB) MERFISH ومجموعة بيانات دماغ الفأر الكامل (WB) MERFISH ومجموعة بيانات سرطان الثدي البشري (BC) Xenium، والتي تمثل بيانات ST التي تم الحصول عليها من أنسجة مختلفة وفي ظل ظروف مختلفة.
إجمالي،يتفوق SPACE على الأدوات المنافسة بهامش كبير في 2 من مجموعات البيانات الخمس ويؤدي أداءً جيدًا تقريبًا مثل أفضل الأدوات أداءً (مقارنةً بأفضل الأدوات المقابلة) في مجموعات البيانات الثلاث الأخرى.كما هو موضح في الشكل التالي:
التغلب على تحديات تحليل بيانات النسخ المكاني
تُعد تقنية النسخ المكاني أحد أهم الإنجازات في مجال المعلوماتية الحيوية في السنوات الأخيرة، وقد تم تسميتها بتقنية العام من قبل Nature Method في عام 2020.تعوض هذه التقنية عن العيب المتمثل في صعوبة تقنية تسلسل الخلية الفردية في قياس العلاقة الموضعية بين الخلايا الفردية من خلال قياس الموضع المكاني لعدد كبير من الخلايا وعدد النسخ داخل الخلايا في وقت واحد، وبالتالي توفير قاعدة بيانات جديدة لفهم التفاعلات بين الخلايا المتعددة. يعد تطوير أساليب التحليل الأساسية لبيانات النسخ المكانية إحدى القضايا الرائدة الحالية في مجال المعلوماتية الحيوية.
أدى اقتران معلومات تحديد الموقع المكاني الخلوي مع طيف خصائصه الجزيئية إلى إنتاج نوع جديد من موارد البيانات عالية الإنتاجية ومتعددة الوسائط، مما يفرض العديد من التحديات أمام تطوير أساليب تحليل البيانات الفعالة واستخراج المعلومات. يقدم الذكاء الاصطناعي أفكارًا جديدة لحل هذه التحديات.

في يوليو 2022، نشرت مجموعة البحث المكونة من البروفيسور شين هونغبين والأستاذ المساعد يوان يي من قسم الأتمتة، كلية المعلومات الإلكترونية والهندسة الكهربائية، جامعة شنغهاي جياو تونغ، ورقة بحثية بعنوان "تجميع الخلايا لبيانات النسخ المكانية باستخدام الشبكات العصبية الرسومية" في مجلة Nature Computational Science، وهي شركة تابعة لـ Nature.
رابط الورقة:https://www.nature.com/articles/s43588-022-00266-5
تقترح الورقة طريقة تجميع الخلايا النسخية المكانية (Cell Clustering for Spatial Transcriptomics, CCST) تعتمد على الشبكة العصبية التلافيفية البيانية.ويوفر حلاً جديدًا لمعالجة بيانات النسخ المكاني ولديه القدرة على تطبيقه في أبحاث المشاكل الأساسية متعددة المستويات في العلوم الحياتية والطبية، بما في ذلك نمذجة التوزيع المكاني للتعبير الجيني، وتحليل ديناميكيات الخلايا، واكتشاف التفاعلات الفرعية الرئيسية للخلايا وآلياتها الجزيئية.
أبريل 2023قام فريق بحثي بجامعة جونز هوبكنز بتطوير SpaceMarkers.هذه خوارزمية معلوماتية حيوية يمكنها استخدام تحليل الفضاء الكامن لبيانات ST لاستنتاج التغيرات الجزيئية في التفاعلات بين الخلايا. استخدم الباحثون هذا النهج لاستنتاج التغيرات الجزيئية في التفاعلات بين الورم والمناعة في الآفات النقيلية والغزوية والسابقة، وكذلك في الاستجابة للعلاج المناعي باستخدام بيانات النسخ المكاني Visium.
نُشرت الدراسة في مجلة Cell Systems تحت عنوان "كشف المشهد المكاني للتفاعلات الجزيئية داخل البيئة الدقيقة للورم من خلال المساحات الكامنة".
في أبريل/نيسان من هذا العام، نُشر تقرير بحثي بعنوان "BANKSY يوحد تصنيف الخلايا وتقسيم نطاق الأنسجة لتحليل بيانات الوميكس المكانية القابلة للتطوير" في المجلة الدولية Nature Genetics.أفاد علماء من معهد أبحاث A*STAR في سنغافورة ومؤسسات أخرى عن خوارزمية تسمى BBANKSY (بناء الكتل باستخدام نواة الحي والمقياس المكاني).باعتبارها أداة مبتكرة لتحليل بيانات الجينوم المكاني، فإن الوظيفة الرئيسية لهذه الخوارزمية هي تصنيف الخلايا بشكل فعال في بيانات الجينوم المكاني وفقًا للنوع ومجال الأنسجة.
رابط الورقة:https://www.nature.com/articles/s41588-024-01664-3
من الواضح أنه بدعم من تكنولوجيا الذكاء الاصطناعي في المستقبل، ستكشف تكنولوجيا النسخ المكاني بشكل أفضل عن التوزيع المكاني لأنواع الخلايا المختلفة في الأنسجة، والتفاعلات بين مجموعات الخلايا المختلفة، ورسم خرائط التعبير الجيني لمناطق الأنسجة المختلفة، والتي لها قيمة تطبيقية بعيدة المدى لفهم آلية حدوث الأمراض والسرطانات.
مراجع:
1.https://www.cell.com/cell-systems/fulltext/S2405-4712(24)00124-8#secsectitle0030
2.https://www.tsinghua.edu.cn/info/1175/112190.htm
3.https://news.bioon.com/article/367a820e60b9.html
4.https://www.sohu.com/a/677912398_12