Command Palette
Search for a command to run...
من خلال ربط بيانات التعبير الجيني بصور مورفولوجيا الخلايا، تمكن الباحثون في الجامعة الصينية في هونج كونج وآخرون من تطوير نموذج انتشار موجه بالنسخ الجيني لتسريع تطوير الأدوية الظاهرية.
يُعدّ مورفولوجيا الخلايا مجالًا بحثيًا أساسيًا في بيولوجيا الخلية الواحدة. وتكمن قيمته في التحليل المنهجي للتغيرات الديناميكية في مورفولوجيا الخلايا في ظل الاضطرابات الجينية أو الدوائية من خلال تحليل الصور عالي الإنتاجية. لا يقتصر هذا البحث على تحسين دقة تنبؤات آلية عمل المركبات (MOA) بشكل ملحوظ، بل يعزز أيضًا دقة تقييمات النشاط الحيوي للمركبات. وفي نهاية المطاف، يوفر دعمًا بالبيانات للخطوات الرئيسية في اكتشاف الأدوية النمطية، مثل فحص المركبات المرشحة والتحقق من آلية عملها، مما يُسرّع عملية البحث والتطوير بشكل فعال.
مع ذلك، فإن مراقبة وتحليل التغيرات في مورفولوجيا الخلايا الناتجة عن الاضطرابات الجينية أو الدوائية ليست بالمهمة السهلة. فعدد المركبات التي يمكن فحصها يتجاوز الملايين، وعدد الجينات التي يمكن تعديلها يصل أيضًا إلى عشرات الآلاف. إن استخدام الأساليب التجريبية التقليدية للتحقق من كل مركب على حدة ليس فقط غير فعال للغاية، بل يتطلب أيضًا تكاليف زمنية ومالية باهظة. على الرغم من اقتراح وتطبيق العديد من الأساليب الحسابية للتنبؤ بمورفولوجيا الخلايا، إلا أن دقتها ودقتها لا تزالان غير كافيتين لتلبية احتياجات البحث العملي.
خاصة،تتجلى قيود الأساليب الحالية بشكل رئيسي في نقطتين:أولاً، يعتمد أداء النماذج المتقدمة، مثل IMPA (مُرمِّز اضطراب الصورة التلقائي)، بشكل كبير على المعرفة البيولوجية المعروفة أو مجموعات بيانات محددة، مما يُضعف قدرات التعميم ويقلِّل من نطاق تطبيقها. ثانياً، تتأثر بيانات مورفولوجيا الخلايا بسهولة بعوامل التداخل التجريبية، مثل تأثيرات الدفعات وتأثيرات مواقع الآبار، وتُظهِر مستوىً عالياً من التشويش، مما يُصعِّب التقاط الخصائص المورفولوجية الحقيقية للخلايا بفعالية، مما يؤثر بدوره بشكل مباشر على استقرار البيانات وموثوقيتها، ويُقيِّد دقة نتائج التحليلات اللاحقة.
ولمعالجة التحديات المذكورة أعلاه، اقترح باحثون من الجامعة الصينية في هونج كونج وجامعة محمد بن زايد للذكاء الاصطناعي ومؤسسات أخرى نموذج انتشار موجه بالنسخ الجيني قابل للتطوير، يُعرف باسم MorphDiff.صُمم هذا النموذج خصيصًا لمحاكاة عالية الدقة لاستجابات مورفولوجيا الخلايا للاضطرابات. ويعتمد على بنية نموذج الانتشار الكامن (LDM)، ويستخدم ملف تعريف التعبير الجيني L1000 كمدخل مشروط لتدريب إزالة الضوضاء.
وأكدت نتائج الدراسة أنالميزة الأساسية لـ MorphDiff هي قدرتها على توليد أشكال الخلايا بدقة في ظل "ظروف اضطراب غير مرئية".توفر هذه الإمكانية فائدتين رئيسيتين: أولاً، تُساعد الباحثين على استكشاف المجال الواسع لفحص الاضطرابات الظاهرية بكفاءة، مما يُقلل بشكل كبير من الاعتماد على التجارب الميدانية واسعة النطاق، وبالتالي يُقلل من تكاليف التجارب ويُحسّن من كفاءة الفحص؛ ثانياً، تُساعد في توضيح آليات عمل جزيئات الأدوية المتنوعة هيكلياً، مما يُوفر دعماً أساسياً للتحقق من صحة آلية المُركب. لذلك، يُمكن أن يُمثل MorphDiff أداةً عالية الأداء لتسريع تطوير الأدوية الظاهرية.
نُشرت نتائج البحث في مجلة Nature Communications تحت عنوان "التنبؤ بتغيرات مورفولوجيا الخلايا تحت الاضطرابات باستخدام نموذج الانتشار الموجه بالنسخ الجيني".
أبرز الأبحاث:
* تطبق هذه الدراسة نماذج الانتشار بشكل مبتكر للتنبؤ بمورفولوجيا الخلايا لأول مرة، مما يفتح آفاقًا جديدة ويوفر أدوات جديدة لتطوير الأدوية الظاهرية.
* أظهرت اختبارات المقارنة الشاملة فعالية MorphDiff، وخاصة في استرجاع MOA، حيث يمكنها تحقيق دقة مماثلة لدقة مورفولوجيا الحقيقة الأساسية، متفوقة على طرق الأساس بمقدار 16.9% و8% على التوالي.

عنوان الورقة:
https://www.nature.com/articles/s41467-025-63478-z
اتبع الحساب الرسمي ورد "الانتشار الموجه بالنسخ" للحصول على ملف PDF كامل
مزيد من أوراق البحث الرائدة في مجال الذكاء الاصطناعي:
https://hyper.ai/papers
مجموعة البيانات: مجموعة بيانات متعددة الأوميكس واسعة النطاق للتحقق من فعاليتها
للتحقق بشكل منهجي من فعالية وتعميم نموذج MorphDiff في التنبؤ بمورفولوجيا الخلية في ظل ظروف الاضطراب،قامت هذه الدراسة ببناء نظام بيانات متعدد الخلايا ومتعدد المصادر يعتمد على الأبعاد المزدوجة "الاضطراب الجيني - اضطراب الدواء".لكل عينة في التجربة، جُمِع نوعان من البيانات: ملف تعريف التعبير الجيني L1000 وصورة مورفولوجيا الخلية، مما شكل بيانات مقترنة. كان النوع الأول بمثابة "مدخل للسمات الجزيئية"، وكان النوع الثاني بمثابة "مدخل للسمات الظاهرية". تضمن هذه الطريقة الارتباط بين إشارة الاضطراب على المستوى الجيني والاستجابة الظاهرية على المستوى المورفولوجي، مدفوعةً فقط بالاضطراب المستهدف. هذا يُزيل التداخل الناتج عن متغيرات غير ذات صلة، مثل اختلافات سلالات الخلايا والدفعات التجريبية.
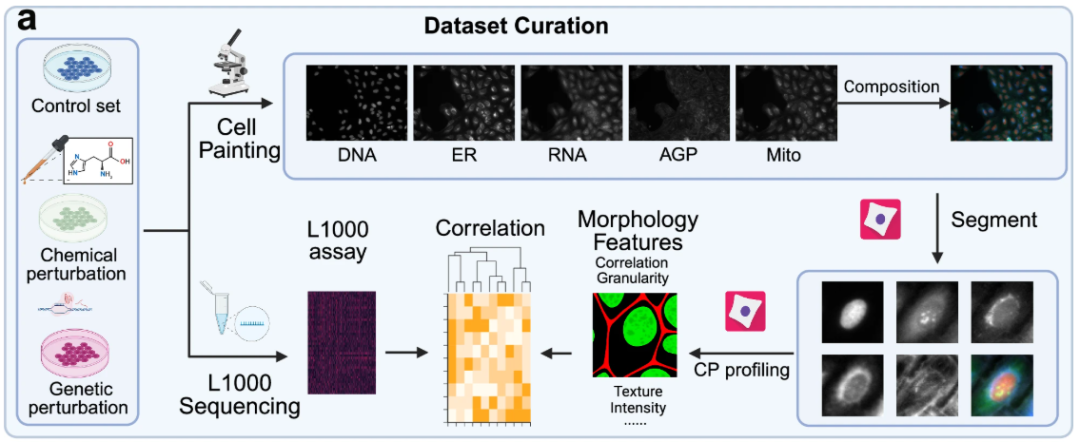
من حيث مجموعات بيانات صور مورفولوجيا الخلايا،استخدمت الدراسة ثلاث مجموعات بيانات واسعة النطاق لصور مورفولوجيا الخلايا لتدريب النموذج وتقييمه وتحليله: مجموعة بيانات واحدة للاضطرابات الجينية ومجموعتان لاضطرابات الأدوية. تحتوي مجموعة بيانات الاضطرابات الجينية، المستمدة من مجموعة بيانات JUMP المستندة إلى سلالة خلايا U2OS، على 130 اضطرابًا جينيًا. أما مجموعتا بيانات اضطرابات الأدوية، المستمدة من مجموعة بيانات CDRP المستندة إلى سلالة خلايا U2OS ومجموعة بيانات LINCS المستندة إلى سلالة خلايا A549، فتحتويان على 1028 اضطرابًا دوائيًا و61 اضطرابًا دوائيًا على التوالي.
خضعت مجموعات بيانات صور مورفولوجيا الخلايا الثلاث لمعالجة مسبقة وتجزئة. واستُخدم برنامج CellProfiler 4.2.5 لتجزئة صور صفائح الخلايا الكلية إلى صور خلية واحدة لتحليل أكثر دقة.تحتوي صور مورفولوجيا الخلايا التي تم الحصول عليها بواسطة تقنية رسم الخلايا على 5 قنوات أساسية.وهذا يشمل، DNA (النواة)، وRNA (النوية والسيتوبلازم)، وER (الشبكة الإندوبلازمية)، وAGP (جهاز جولجي/غشاء الخلية/هيكل الأكتين)، وميتو (الميتوكوندريا).
بجانب،خططت التجربة أيضًا لمجموعة البيانات L1000 التي تحتوي على "صور مورفولوجية لا تتوافق مع المعايير".تُستخدم مجموعة البيانات هذه بشكل أساسي لاستكشاف تطبيق النموذج بشكل أكبر في سيناريو "الحصول على بيانات التعبير الجيني فقط"، ويمكن أن تشكل الأساس للتحقق اللاحق من الآلية، وفحص الأدوية، وما إلى ذلك لتوفير فرضيات "مبنية على البيانات".
هندسة النموذج والطرق: ربط بيانات التعبير الجيني بصور مورفولوجيا الخلايا
الهدف الأساسي من MorphDiff هو تحقيق رسم خرائط دقيقة من البداية إلى النهاية من ملفات تعريف التعبير الجيني L1000 إلى صور مورفولوجيا الخلايا من خلال إطار نموذج الانتشار الكامن الموجه بالنسخ الجيني.ببساطة، يتعلق الأمر بتصميم وتدريب نموذج، وهو بمثابة "جسر" - إدخال بيانات التعبير الجيني L1000 المقابلة لاضطراب معين، ثم إخراج الصورة المورفولوجية أو مورفولوجيا الاضطراب للخلية تحت هذا الاضطراب.
يتكون جوهر نموذج MorphDiff من وحدتين رئيسيتين:نموذج التشفير التلقائي المتغير المورفولوجي (MVAE) ونموذج الانتشار الكامن (LDM)، كما هو موضح في الشكل ب أدناه.
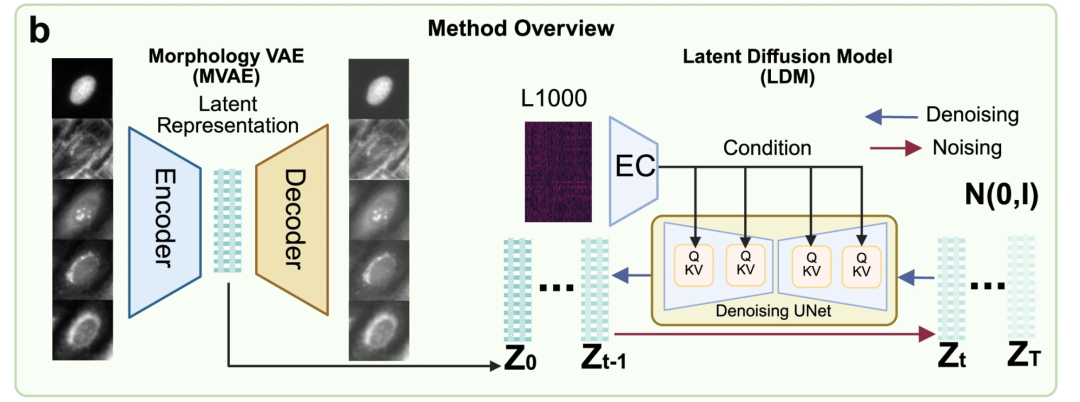
ومن بينها، MVAE هو "محرك ضغط الصور" للنموذج، والذي يتحمل مسؤولية تحويل صور مورفولوجيا الخلايا متعددة القنوات وعالية الدقة إلى تمثيلات محتملة منخفضة الأبعاد وقابلة للتفسير.يعالج هذا النهج التكلفة الحسابية العالية والتدريب غير المستقر المرتبط بتدريب نماذج الانتشار مباشرةً على صور عالية الأبعاد. من الناحية الهيكلية، يتكون MVAE من جزأين: مُشفِّر وفكّ تشفير. يضغط المُشفِّر الصور المورفولوجية المعقدة ذات الخمس قنوات في تمثيل أبسط منخفض الأبعاد؛ بينما يستعيد فكّ التشفير الصورة المورفولوجية الأصلية من هذا التمثيل منخفض الأبعاد.
يعد LDM مسؤولًا بشكل أساسي عن ربط "التعبير الجيني" و"السمات المورفولوجية المضغوطة"، مما يساعد النموذج على استكمال العلاقة "من الجينات إلى السمات المورفولوجية". يتضمن نموذج LDM عملية إزالة ضوضاء. تُضيف عملية إزالة الضوضاء تدريجيًا ضوضاء غاوسية إلى السمات المورفولوجية المضغوطة حتى تصبح عشوائية تمامًا. تُمكّن عملية إزالة الضوضاء النموذج من استعادة الضوضاء العشوائية تدريجيًا إلى السمات المورفولوجية الأصلية، مع الأخذ في الاعتبار التعبير المعروف لجينات L1000. يستخدم النموذج بنية شبكة U-Net، ويتضمن آلية انتباه لربط المعلومات الجينية والمورفولوجية الرئيسية بدقة أكبر.
يوضح الشكل ج أدناه تطبيقين لنموذج MorphDiff المُدرَّب مسبقًا: G2I وI2I. يُزيل الأول الضوضاء من صورة مورفولوجيا الخلية المقابلة من توزيع ضوضاء عشوائي، مُشروطًا بتعبير جين L1000، لتوليد صورة مورفولوجيا الخلية المقابلة. أما الثاني، المُشروط بتعبير جين L1000 تحت اضطراب مُحدد، فيُحوِّل صورة مورفولوجيا الخلية من مورفولوجيا الخلية الضابطة إلى صورة مورفولوجيا الخلية المُضطربة المُتوقعة، مُحققًا بذلك إمكانيات تنبؤ "بالتحول من مورفولوجيا طبيعية إلى مورفولوجيا مُضطربة".
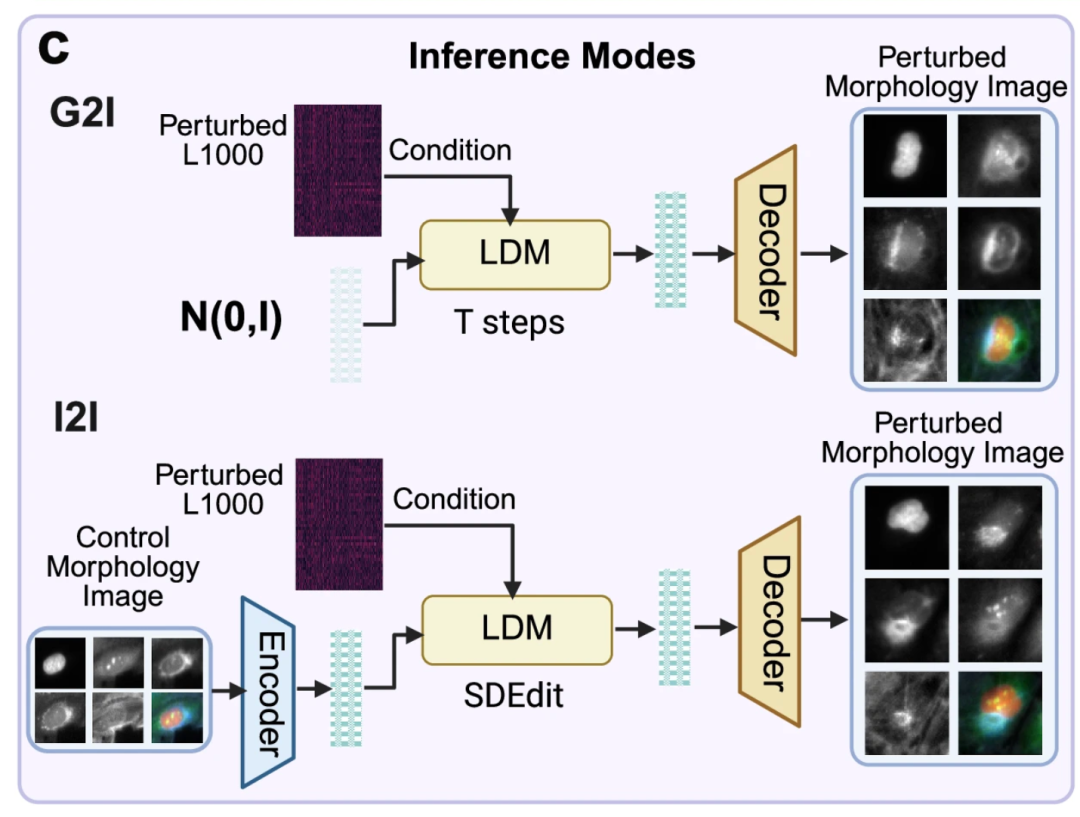
يوضح الشكل د أدناه قيمة نموذج MorphDiff في التطبيقات العملية.أولاً، يتمكن النموذج من التنبؤ بالتغيرات المورفولوجية للخلايا الناجمة عن "اضطرابات غير مرئية" لم يتم مواجهتها أثناء التدريب.وهذا يسمح للباحثين بمحاكاة حالة التغيرات الخلوية تحت تأثير الأدوية الجديدة بواسطة أجهزة الكمبيوتر دون الحاجة إلى إجراء تجارب فيزيائية، مما يساعدهم على استكشاف المزيد من الاحتمالات بسرعة وبتكلفة منخفضة؛ ثانيًا،يجمع إطار النموذج بين أدوات مثل CellProfiler و DeepProfiler.يمكن أن يساعد في تحديد آلية عمل الأدوية، وبالتالي تعزيز تطوير الأدوية الظاهرية.
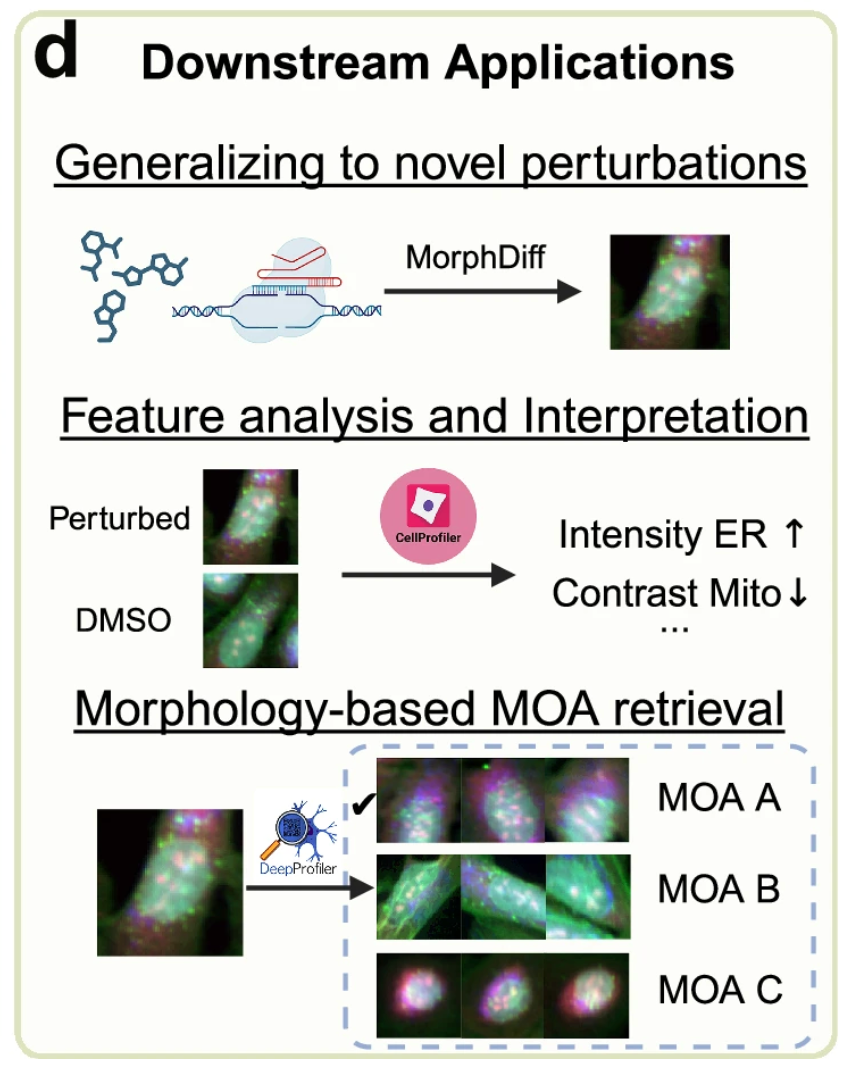
ويقول الباحثون إن MorphDiff هو حاليًا الأداة الوحيدة التي تدعم إنشاء صور مورفولوجية من التعبير الجيني وتحويلها من مورفولوجيا غير مضطربة إلى مورفولوجيا مضطربة.
النتائج التجريبية: الأداء يتفوق على IMPA، مما يؤدي إلى تسريع تطوير الأدوية النمطية
ومن أجل التحقق من فعالية نموذج MorphDiff، قام الباحثون بتصميم سلسلة من التجارب لأغراض مختلفة.من خلال المقارنة التجريبية مع الأدوات المتقدمة، يتم التحقق بشكل منهجي من فعالية وفائدة MorphDiff.
أولاً، أثبتت التجارب صحة تنبؤات الاضطرابات الجينية.أجرى الباحثون اختبارات معيارية على مجموعة بيانات JUMP OOD وقارنوها بطرق أساسية متعددة، بما في ذلك MorphNet، وDMIT (فك التشابك للترجمة متعددة الخرائط من صورة إلى صورة)، وDRIT++ (التمثيل المفكك للترجمة من صورة إلى صورة)، وStarGANv1، وIMPA، وVQGAN (شبكة متجهية توليدية مضادة)، وMDTv2 (محولات الانتشار المقنعة).
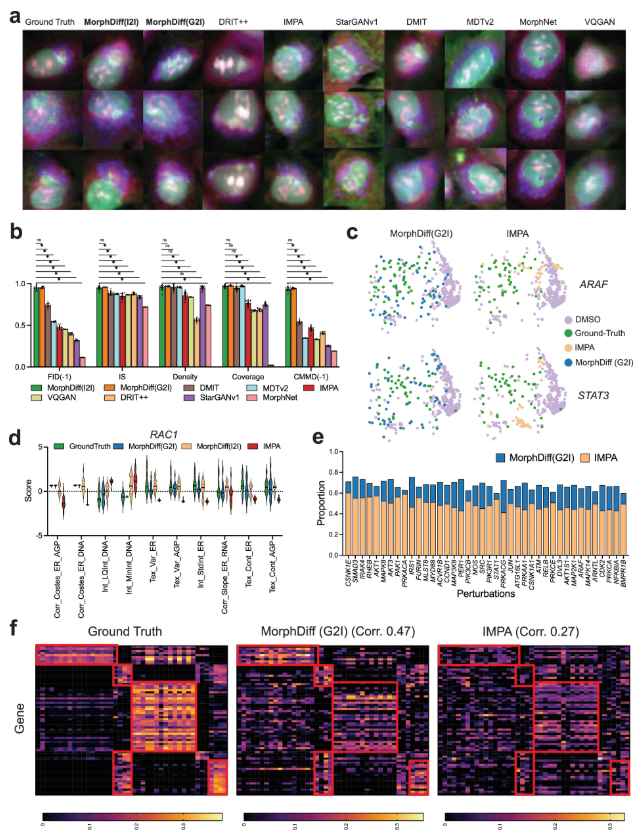
وتظهر النتائج أنالنتائج التي تم إنشاؤها بواسطة وضعي MorphDiff أقرب إلى خط الأساس الحقيقي من حيث الجودة البصرية والشكل البنيوي.من حيث المقاييس الكمية، بما في ذلك FID وIS (درجة البداية) وCMMD والكثافة والتغطية، تفوقت طريقتا MorphDiff على طرق خط الأساس في التعميم والدقة والتنوع، محققتين جودة نتائج أعلى. في التنبؤ بتغيرات مورفولوجيا الخلية، تكون مخرجات MorphDiff (G2I) أكثر تنوعًا وأقرب إلى خط الأساس الحقيقي؛ بينما تُظهر السمات الناتجة عن MorphDiff (I2I) تداخلًا أعلى مع سمات خط الأساس الحقيقية، مما يشير إلى أن دقة توقعها تتوافق بشكل كبير مع مورفولوجيا الاضطراب الحقيقية.
بعد ذلك، أُجريت تجارب للتحقق من تنبؤ اضطراب الدواء. وقارن الباحثون أولًا جميع الطرق باستخدام مجموعة بيانات CDRP OOD.يحقق MorphDiff أداءً رائعًا مقارنةً بطرق الأساس الأخرى في معظم المقاييس، مما يوضح قدراته الشاملة الأكثر قوة واستقرارًا.ثم انتقل الباحثون إلى إجراء تقييم أكثر صرامة للنموذج على مجموعة بيانات LINCS، وإجراء مزيد من التحقيق في استجابة مورفولوجيا الخلية للمركبات الجزيئية الصغيرة من خلال مقارنة النتائج الناتجة مع ميزات CellProfiler.
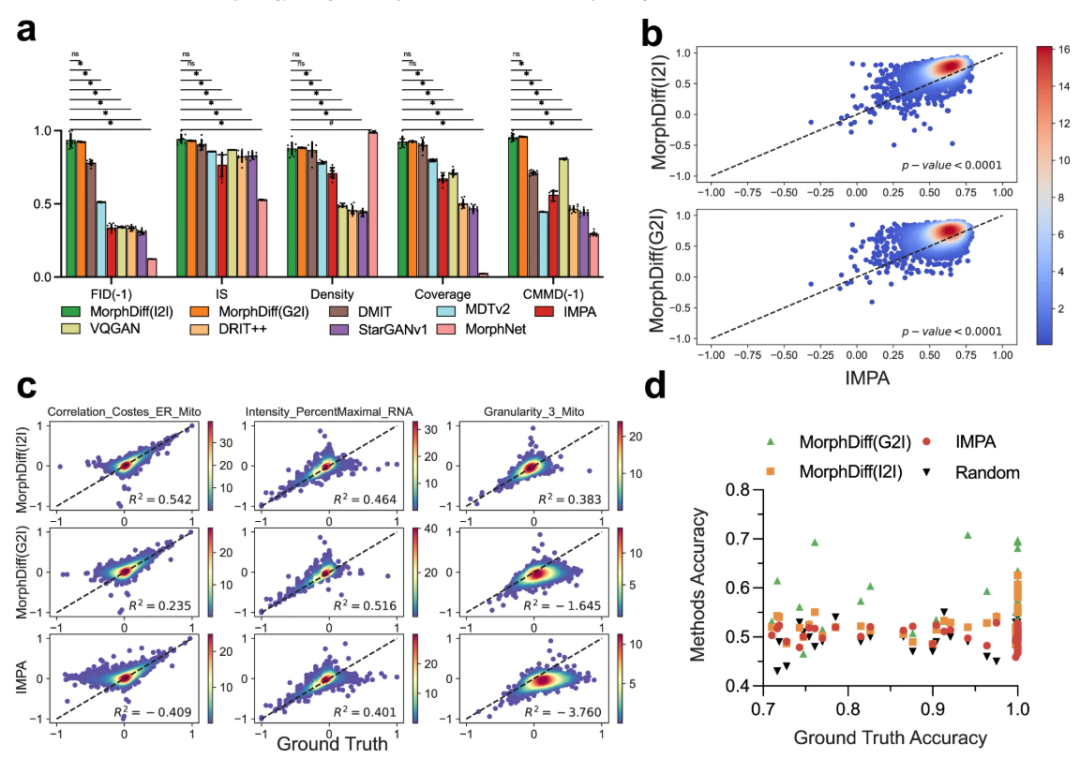
تظهر النتائج أن كلا الوضعين من MorphDiff يتفوقان بشكل كبير على خط الأساس.في اختبار G2I، سجلت 87.61 عينة TP3T أعلى من 0.5، وتجاوزت 16.21 عينة 0.8. وفي اختبار I2I، سجلت 891 عينة TP3T أعلى من 0.5، وتجاوزت 27.21 عينة 0.8. وبالمقارنة، سجلت 78.31 عينة TP3T من IMPA أعلى من 0.5، ولكن لم تتجاوز أي منها 0.8. وكشف التحليل نفسه على مجموعة بيانات CDRP OOD ومجموعة بيانات LINCS التي تُركت فيها البيانات على شكل استبعاد أن كلا نموذجي MorphDiff تفوقا على الطريقة الأساسية، حيث كانت قيم الاحتمالية أقل من 0.0001، مما يُظهر قابلية تعميم هذه الطريقة.
في تحليل تضمين DeepProfiler،يمكن لـ MorphDiff (G2I) التقاط الأنماط المورفولوجية للخلايا الخاصة بالاضطراب بشكل أكثر موثوقية ودقة.وعلى المستوى الصيدلاني،يتميز الناتج الناتج عن نمط G2I بخصوصية اضطراب أعلى من I2I. توضح هذه العروض التوضيحية بشكل أكبر إمكانات MorphDiff في فحص الأدوية.
أخيرًا، أثبتت التجربة أيضًا قدرات MorphDiff في تطوير الأدوية. اختار الباحثون مجموعة بيانات CDRP Target_MOA لمقارنة وضعي تطبيق MorphDiff وIMPA.
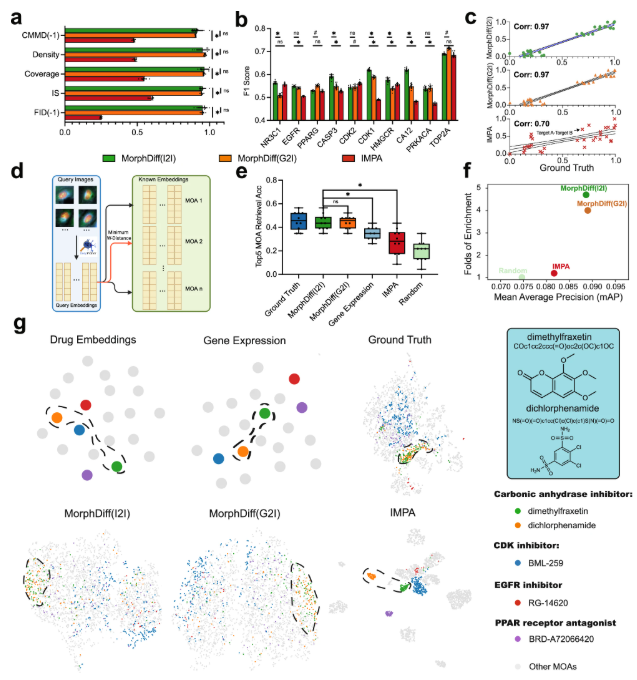
تظهر النتائج أن مورفولوجيا الاضطراب التي يولدها MorphDiff تتوافق إلى حد كبير مع خط الأساس الحقيقي.أثبت هذا قدرته على رصد التنوع المرتبط بالهدف، وفهم العلاقة المعقدة بين اضطرابات الدواء وشكله بفعالية. في مهمة استرجاع MOA، كان متوسط دقة النتائج الناتجة عن MorphDiff أعلى بمقدار 16.9% من متوسط دقة الاسترجاع القائم على IMPA، وأعلى بمقدار 8% من متوسط دقة الاسترجاع القائم على التعبير الجيني، على التوالي. علاوة على ذلك، أظهرت التجارب أن MorphDiff قادر على اكتشاف أن شكل الخلية يحتوي على معلومات متكاملة، ويمكنه تحديد الأدوية التي لها نفس MOA ولكن بهياكل مختلفة، مما يُسرّع من تطوير الأدوية النمطية.
أصبحت عمليات المحاكاة القائمة على السيليكون شائعة، مما أدى إلى تسريع تطوير الأدوية اللاحقة
يختلف اكتشاف الأدوية الظاهرية عن اكتشاف الأدوية القائمة على الأهداف في أنه يستكشف الأدوية ويطورها من خلال مراقبة آثارها على الأنظمة البيولوجية العامة أو الأنماط الظاهرية الخلوية. يوفر هذا النهج مزايا كبيرة في اكتشاف آليات عمل وأهداف جديدة، وفي علاج الأمراض المعقدة. وتسعى العديد من المختبرات ومؤسسات البحث بنشاط إلى هذا الموضوع، حيث تدمج التكنولوجيا الحاسوبية مع الطب الحيوي لفتح آفاق جديدة في اكتشاف الأدوية الظاهرية.
كما ذكر في الورقة البحثية، فإن نموذج IMPA هو نموذج توليدي عميق اقترحه فريق من الجامعة التقنية في ميونيخ في ألمانيا وجامعة أكسفورد في المملكة المتحدة.باستخدام طريقة نقل الأسلوب، يتم تحليل صور الخلايا إلى "أسلوب" (تمثيل الاضطرابات/الدفعات) و"محتوى" (تمثيل الخلايا)، وهو ما يمكنه التنبؤ باستجابة الخلية للاضطرابات وإزالة تأثيرات الدفعات.نُشرت المقالة أيضًا، بعنوان "التنبؤ باستجابات الخلايا المورفولوجية للاضطرابات باستخدام النمذجة التوليدية"، في مجلة Nature Communications.
بالإضافة إلى ذلك، في مقال نشره فريق جامعة ميشيغان بعنوان "MorphNet يتنبأ بمورفولوجيا الخلية من خلال التعبير الجيني للخلية الفردية"،واقترح طريقة حسابية تسمى MorphNet يمكنها رسم صور مورفولوجية للخلايا استنادًا إلى ملفات تعريف التعبير الجيني الخاصة بها.تستخدم الطريقة بيانات مورفولوجية وجزيئية مقترنة لتدريب شبكة عصبية للتنبؤ بشكل مورفولوجيا النواة أو الخلية بأكملها بناءً على التعبير الجيني.
باختصار، أصبح تعزيز تطوير الأدوية الظاهرية والبحوث البيولوجية من خلال مراقبة وتحليل التغيرات في حالات الخلايا في ظل الاضطرابات الجينية أو الدوائية موضوعًا مهمًا. فيما يتعلق بـ MorphDiff، على الرغم من وجود العديد من العيوب فيه، مثل صعوبة التنبؤ بالاضطرابات غير المرئية في ظل وجود عدد كبير من الاضطرابات الجديدة خارج بيانات التدريب، فمن الواضح أن MorphDiff يتطور باستمرار على أساس سابقاته، متفوقًا عليها في التطبيق العملي والتعميم وسهولة الاستخدام وقابلية التوسع.
احصل على أوراق بحثية عالية الجودة ومقالات تفسيرية متعمقة في مجال AI4S من عام 2023 إلى عام 2024 بنقرة واحدة⬇️









