Command Palette
Search for a command to run...
VenusFactory:一个用于蛋白质工程数据检索与语言模型微调的统一平台
VenusFactory:一个用于蛋白质工程数据检索与语言模型微调的统一平台
摘要
自然语言处理(NLP)已对人类语言以外的科学领域产生深远影响,特别是在蛋白质工程领域,预训练蛋白质语言模型(Protein Language Models, PLMs)已展现出卓越的性能。然而,由于数据收集困难、任务基准测试不统一以及实际应用门槛高等问题,跨学科的推广应用仍面临局限。为此,本文提出VenusFactory——一个多功能集成引擎,能够实现生物数据检索、标准化任务基准测试以及PLMs的模块化微调。VenusFactory同时面向计算机科学与生物学研究社区,提供命令行执行模式与基于Gradio的无代码交互界面两种使用方式,集成超过40个蛋白质相关数据集及40余个主流PLMs。所有代码与实现均已开源,访问地址为:https://github.com/tyang816/VenusFactory。
一句话摘要
上海交通大学、上海人工智能实验室和华东理工大学的研究团队提出了 VENUSFACTORY,一个统一平台,集成了生物数据检索、标准化基准测试以及40多个蛋白质语言模型的模块化微调功能,支持基于代码和无代码两种方式,适用于跨学科的蛋白质工程应用。
主要贡献
-
针对跨学科协作中的空白,VENUSFACTORY 集成了来自 RCSB PDB、UniProt 和 AlphaFold DB 等主要数据库的生物数据检索功能,支持高效批量下载与标准化格式化,满足计算与生物研究人员的共同需求。
-
该平台提出了一种统一的框架,用于对40多个预训练蛋白质语言模型(PLMs)进行标准化任务基准测试和模块化微调,支持多种方法,包括 LoRA、冻结微调与全量微调、以及 SES-Adapter,覆盖40多个蛋白质相关下游任务。
-
VENUSFACTORY 提供了易用的接口——包括无代码 Gradio 网页界面和命令行工具——实现模型训练、评估与应用的无缝衔接,所有组件均在 GitHub 和 Hugging Face 上开源,采用 Apache 2.0 许可证。
引言
作者利用蛋白质语言模型(PLMs)在酶工程中日益增长的成功,解决计算机科学与生物学之间长期存在的跨学科协作障碍。尽管 ESM2 等 PLMs 在功能预测和序列设计等任务中取得进展,但以往工作存在数据访问碎片化、基准测试不一致以及技术门槛高等问题,限制了非专家用户的使用。现有工具或仅聚焦于数据整合,或缺乏对多样化蛋白质任务的微调与评估的全面支持。为克服这些挑战,作者提出 VENUSFACTORY,一个统一平台,集成了来自主要生物数据库的高通量数据检索、40多个数据集上的标准化基准测试,以及通过无代码 Gradio 界面和命令行工具实现的40多个 PLMs 的模块化微调。通过基于 PyTorch 构建、托管于 Hugging Face 的可复现、开源框架,VENUSFACTORY 降低了各领域研究人员的入门门槛,推动了最先进的 AI 方法在蛋白质工程中的无缝应用。
数据集
-
数据集由四个主要蛋白质数据库构成:RCSB PDB(超过20万条实验确定的3D结构)、UniProt(超过2.5亿条蛋白质序列及功能注释)、InterPro(约4.1万条具有家族、结构域和功能位点注释的蛋白质)以及 AlphaFold DB(与 UniProt ID 关联的 AlphaFold2 预测结构)。
-
每个基准子集均针对特定生物工程任务进行筛选:
- 定位:包含 DeepLocBinary(二分类膜关联)、DeepLocMulti(多类定位)和 DeepLoc2Multi(多标签、多类),均包含序列及 AlphaFold2/ESMFold 预测结构。
- 溶解度:包含三个二分类数据集(DeepSol、DeepSoluE、ProtSolM)和一个回归基准(eSol),使用 ESMFold 预测结构;eSol 还包含 AlphaFold2 预测结构。
- 注释:四个多类、多标签基准(EC、GO-CC、GO-BP、GO-MF),基于酶学委员会和基因本体注释,结构来自 AlphaFold2 和 ESMFold。
- 突变:19个具有数值标签的回归数据集,包括酶溶解度(PETA_TEM_Sol、PETA_CHS_Sol、PETA_LGK_Sol)、荧光/稳定性(TAPE_Fluorescence、TAPE_Stability)以及病毒适应度(FLIP_AAV)和核苷酸结合(FLIP_GB1)基准,每个数据集均有明确的划分规则。
- 其他性质:五个额外数据集用于热稳定性、最适酶温(DeepET_Topt)、金属离子结合和分选信号检测,均使用 AlphaFold2 预测结构,其中热稳定性、DeepET_Topt 和分选信号任务使用 ESMFold。
-
数据通过多线程下载处理,采用模拟 HTTP 请求与用户代理伪装,并行执行,实现从 UniProt、AlphaFold DB 和 RCSB PDB 高效获取数据,支持 .cif、.pdb、.xml 和 .json 等格式。元数据按 RCSB ID 或 UniProt ID 索引,以结构化 JSON 格式存储。
-
蛋白质结构通过三种方法转换为离散标记:DSSP(3类或8类二级结构)、FOLDSEEK(通过 VQ-VAE 生成的20D 3Di 标记)和 ESM3(基于局部子图的4096D 整数表示)。
-
该数据集用于训练和评估40多个预训练蛋白质语言模型(PLMs),包括 ESM2、ESM-1b、ProtBERT、ANKH 和 ProtT5。训练采用标准序列截断方法,以及一种非截断方法,动态确定每批最优标记限制,以保持序列完整性。
-
所有基准数据格式统一,包含准确率、F1 分数和斯皮尔曼相关系数等评估指标。AlphaFold2 和 ESMFold 的置信度分数(pLDDT)以均值和标准差形式报告。
-
元数据、序列和结构采用分层存储,便于访问:InterPro 数据按结构域细节、访问号元数据和 UniProt ID 列表拆分;UniProt 序列以 FASTA 格式保存,AlphaFold 结构按 ID 前缀组织。
-
系统具备健壮的错误处理机制,失败下载记录于 "failed.txt" 以供重试,并使用缓存和自适应速率限制防止 API 过载,提升获取效率。
方法
作者在 VenusFactory 中采用模块化架构,支持多样化的生物系统分析任务,围绕三个核心组件构建:数据收集(Collection)、基准测试(Benchmarking)和应用(Application)。框架从数据收集模块开始,对接 PDB、UniProt、InterPro 和 AlphaFold 等外部生物数据库。该模块采用多线程下载实现高效数据获取,并包含数据处理模块,用于数据序列化与格式转换。如图所示,收集的数据随后被导向基准测试模块,按下游任务组织为结构化格式。这包括用于定位、溶解度、突变效应、功能注释及其他性质预测任务的数据获取,输入按序列、结构、属性和标签分类。
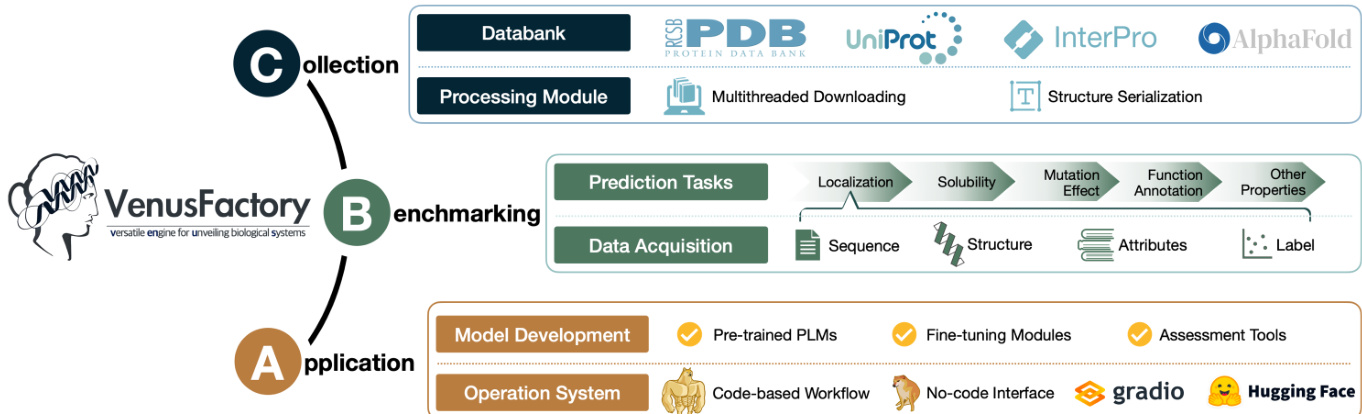
应用层整合了模型开发与运行系统组件。模型开发涵盖预训练蛋白质语言模型(PLMs)、微调模块和评估工具。在微调方面,VenusFactory 支持经典方法——冻结微调(仅更新读出层,固定 PLM 参数)和全量微调(更新整个模型)。此外,还集成参数高效方法,如 LoRA 及其变体,以及专为蛋白质设计的 SES-ADAPTER,该方法利用 PLM 表示与序列-结构嵌入(如 FOLDSEEK 提供)之间的交叉注意力,提升任务特定性能。框架还支持多种分类头,包括:两层全连接网络(含平均池化、Dropout 和 GeLU 激活);轻量级头(结合1D卷积特征提取与注意力加权池化);以及 ATTENTION1D,采用掩码1D卷积注意力池化和非线性投影层,用于多分类任务。运行系统提供基于代码的工作流和无代码界面,集成 Gradio 和 Hugging Face 等平台,实现部署与访问的便捷化。
实验
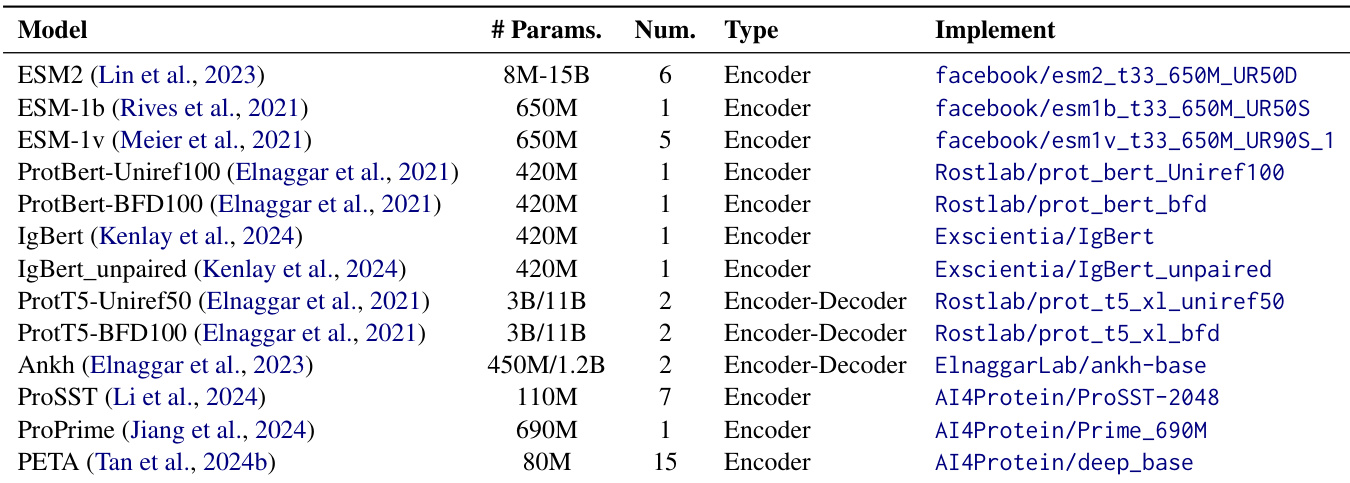
- 在多种下游任务(包括溶解度预测、突变效应分析、定位和功能注释)中,评估了多个预训练语言模型(PLMs)与三种微调策略——冻结、LoRA 和 SES-ADAPTER。
- SES-ADAPTER 在所有任务中持续优于其他方法,尤其在溶解度预测(DSE, PSM)和突变效应预测(AAV, GB1)中表现最佳,展现出更强的鲁棒性。
- PROT5-XL-U50 整体表现最佳,在注释和突变预测任务中尤为突出,而 PROTBERT 在突变和部分注释任务中表现受限。
- LoRA 在定位任务中表现强劲(如 DLB 上得分最高),但在溶解度和注释基准中缺乏一致性。
- 冻结方法表现最差,尤其在注释任务(BP, MF)中,凸显了全量或轻量级微调对模型性能优化的必要性。
- 在多个数据集上,SES-ADAPTER 达到最先进水平,关键指标如多标签分类的 F1-max 和排序任务的斯皮尔曼 ρ 显著提升。
- 训练使用 ADAMW 优化器,学习率为 0.0005,批次大小限制为 12,000 标记,梯度累积为 8,早停策略在连续 10 个 epoch 无提升时终止,确保可复现性,随机种子设为 3407。
- 实验在 20 块 RTX 3090 GPU 上运行两个月,结构输入来自 FOLDSEEK 和 DSSP 8类表示,用于 SES-ADAPTER。
作者采用标准化实验设置,评估多种蛋白质语言模型在多个任务上的表现,微调方法包括冻结、LoRA 和 SES-ADAPTER。结果表明,SES-ADAPTER 在所有模型和任务中持续优于其他方法,尤其在溶解度和突变预测方面表现突出,而模型选择与任务特定的微调策略显著影响性能。
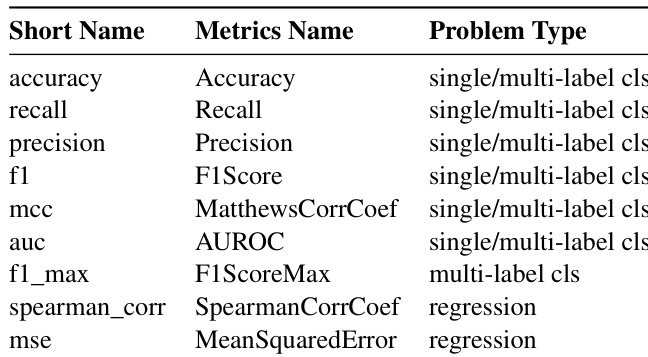
作者采用多种评估指标衡量模型在不同类型问题上的表现,包括分类和回归任务。结果显示,分类任务使用准确率、精确率、召回率、F1 分数和 AUROC,回归任务则采用斯皮尔曼相关系数和均方误差。
结果表明,SES-Adapter 在所有模型和任务中持续优于冻结和 LoRA,尤其在突变预测及其他下游应用中表现优异。在各模型中,ProtT5-XL-U50 达到最高整体性能,而 SES-Adapter 在不同微调策略下均展现出鲁棒性与高效性。
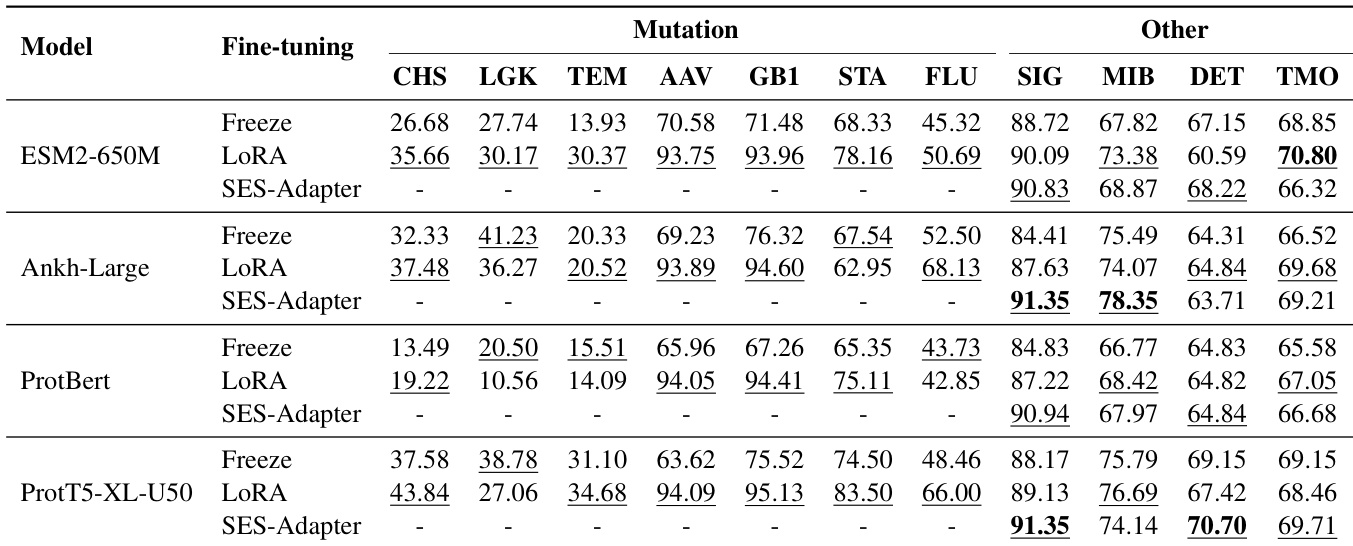
作者在多种下游任务中评估多个蛋白质语言模型,采用不同微调策略,结果表明 SES-ADAPTER 持续优于其他方法,尤其在溶解度和突变预测方面表现突出。表格显示,PROT5-XL-U50 整体性能最佳,而冻结方法结果最低,尤其在注释任务中,表明自适应微调对实现最优模型性能至关重要。
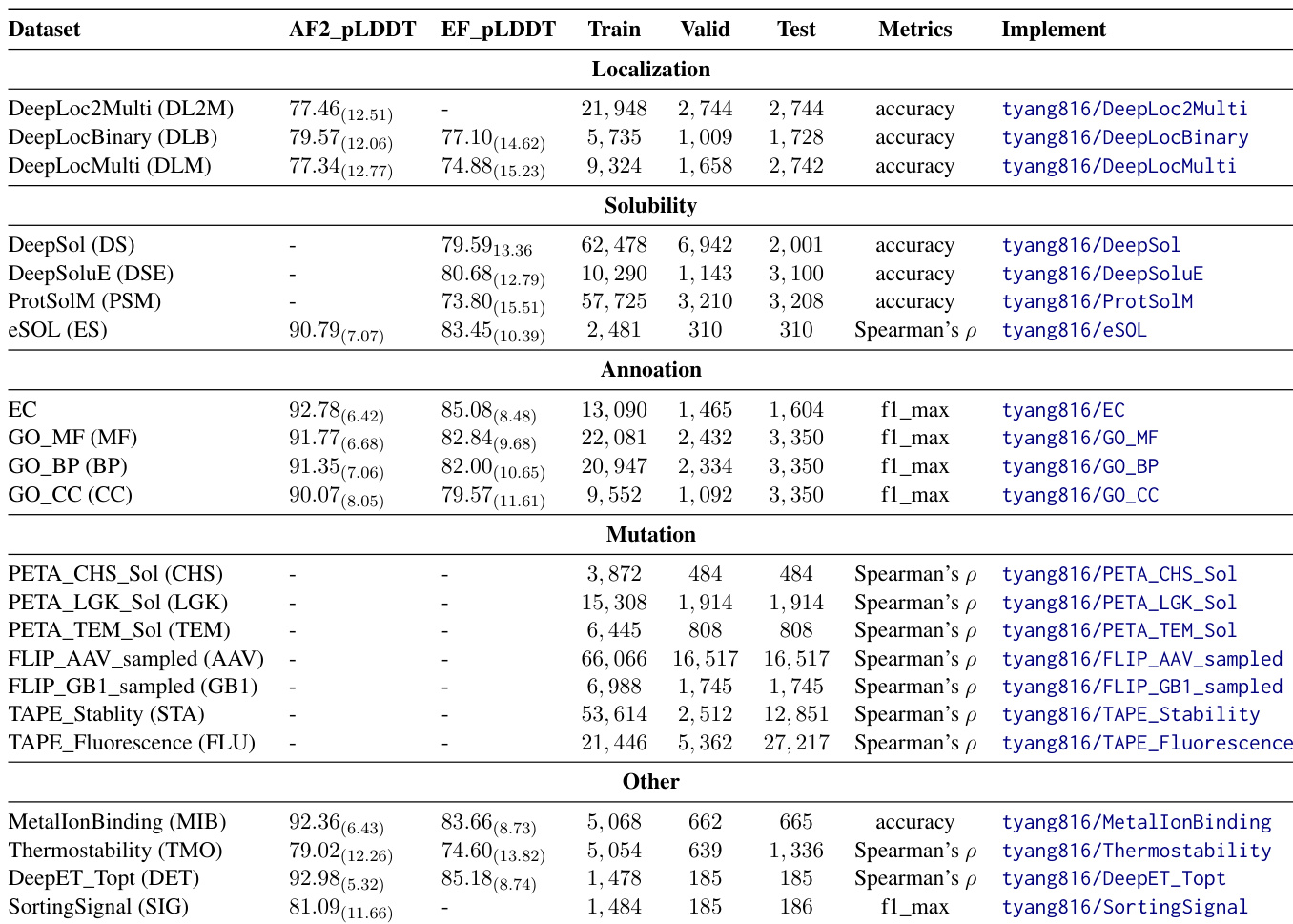
作者采用三种微调方法——冻结、LoRA 和 SES-Adapter——在定位、溶解度和注释任务中评估四个蛋白质语言模型。结果表明,SES-Adapter 在所有任务中持续优于其他方法,尤其在溶解度和突变预测方面表现突出;LoRA 在定位任务中得分最高;而冻结方法在多数基准中表现最差。