Command Palette
Search for a command to run...
أصدرت مجموعة أبحاث يو شيانغ في جامعة شنغهاي جياو تونغ نموذجًا للتعلم العميق قابلاً للتحويل لتحديد أنواع متعددة من تعديلات الحمض النووي الريبي (RNA) وتقليل التكاليف الحسابية بشكل كبير.

في عام 2021، وبفضل الدعوة القوية التي أطلقها قاو فو، الأكاديمي في الأكاديمية الصينية للعلوم، اكتسبت لقاحات mRNA شهرة بين عشية وضحاها وأصبحت أمل الناس أثناء تفشي فيروس كورونا الجديد. اليوم، أصبح هذا الماضي الخاص تاريخًا، لكن تعديل الحمض النووي الريبي وراء لقاحات mRNA لا يزال يتطور بوتيرة سريعة.
يعد ما يسمى بتعديل الحمض النووي الريبي (RNA) نوعًا مهمًا من التنظيم ما بعد النسخ، والذي يمكن أن يشارك على نطاق واسع في مختلف عمليات معالجة الحمض النووي الريبي ما بعد النسخ والمسارات الأيضية.
تستحق تعديلات الحمض النووي الريبوزي (RNA) الاهتمام لأنها تلعب وظيفة بيولوجية حيوية في نمو وتطور الكائنات حقيقية النواة.على سبيل المثال، وجدت دراسات حديثة أن تأثير زعزعة استقرار N⁶-ميثيل أدينوسين (m⁶A) في الخلايا الجذعية الجنينية الثديية مرتبط بمجموعة متنوعة من الأمراض، وأن 5-ميثيل سيتوزين (m⁵C) مرتبط بتحمل الأرز لدرجات الحرارة العالية.
ومع ذلك، فإن الحمض النووي الريبوزي (RNA) يحتوي على العديد من أنواع التعديلات، وحتى الآن تم اكتشاف أكثر من 160 نوعًا من التعديلات في الحمض النووي الريبوزي الطبيعي. في السابق، كان من الممكن تحقيق التعرف على تعديل القواعد الفردية من خلال تقنية التسلسل المباشر للحمض النووي الريبي النانوي (DRS) التي طورتها شركة Oxford Nanopore Technologies (ONT) جنبًا إلى جنب مع أساليب التعلم العميق.ومع ذلك، تواجه هذه الطريقة صعوبة في اكتشاف أنواع متعددة من التعديلات في وقت واحد في عينة واحدة.
ردًا على الأسئلة المذكورة أعلاه، نشرت مجموعة البحث التابعة ليو شيانغ، الأستاذ المشارك الدائم في كلية العلوم الحياتية والتكنولوجيا بجامعة شنغهاي جياو تونغ، وفريق يانغ جون/وانغ هونغشيا من حديقة شنغهاي تشينشان النباتية ورقة بحثية بعنوان "التعلم بالنقل يمكّن من تحديد أنواع متعددة من تعديلات الحمض النووي الريبي باستخدام تسلسل الحمض النووي الريبي المباشر النانوي" في مجلة Nature Communications.تم تطوير نموذج التعلم العميق القابل للتحويل، TandemMod، لتمكين التعرف على أنواع متعددة من تعديلات RNA في DRS.
أبرز الأبحاث:
* في ظل شرط ضمان نفس الأداء، يتم تقليل تكاليف الحوسبة بشكل كبير مثل كمية بيانات مجموعة التدريب ووقت تدريب النموذج
* يوفر TandemMod دعمًا فنيًا مهمًا لتحديد أنواع مختلفة من مواقع تعديل الحمض النووي الريبي ودراسات النسخ الجيني في الحيوانات والنباتات والكائنات الحية الدقيقة
* يمكن أيضًا استخدام TandemMod للكشف عن الحمض النووي الريبي المعدل صناعيًا مثل لقاحات الحمض النووي الريبي

عنوان الورقة:
https://www.nature.com/articles/s41467-024-48437-4
يجمع المشروع المفتوح المصدر "awesome-ai4s" أكثر من مائة تفسير ورقي لـ AI4S ويوفر مجموعات وأدوات ضخمة من البيانات:
https://github.com/hyperai/awesome-ai4s
مجموعة البيانات: تدريب مستهدف مع مجموعات بيانات متعددة
ومن أجل تدريب وتقييم أداء نموذج TandemMod، استخدم فريق البحث مجموعات بيانات متعددة للتجارب.
أولاً،استخدم فريق البحث مجموعة بيانات النسخ المختبري ELIGOS التي أنشأها مختبر Nookaew.تم حساب خمس ميزات على مستوى القاعدة (المتوسط، والوسيط، والانحراف المعياري، وطول الإشارة، وجودة القاعدة) لستة قواعد معدلة (m¹A، وm⁶A، وm⁵C، وhm⁵C، وm⁷G، وΨ) ومقارنتها بالقواعد غير المعدلة.
ثانيًا، اختار فريق البحث دراسة أداء TandemMod بناءً على التعديلين الأكثر شيوعًا في mRNA حقيقية النواة، m⁵C وm⁶A.قام الباحثون بتدريب نموذج TandemMod m⁵C على مجموعة بيانات Curlcake.تم استخلاص مجموعة البيانات من تسلسلات منقولة في المختبر تحتوي على جميع 5-mers الممكنة، وتم تقسيمها إلى مجموعات تدريب واختبار بنسبة 4:1.
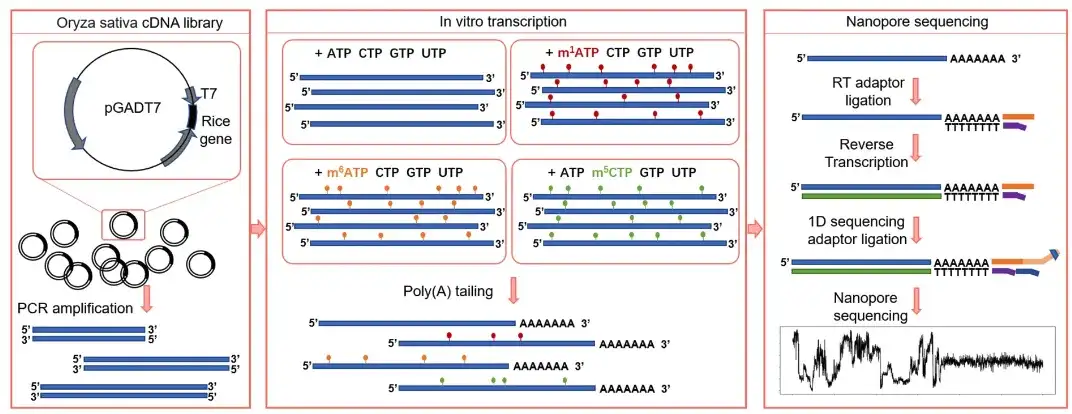
وفي الوقت نفسه، من أجل حل مشكلة عدم قدرة الحمض النووي الريبي المنقول من تسلسلات اصطناعية في المختبر على تغطية النطاق الكامل للتسلسلات الطبيعية، أجرى فريق البحث نسخًا في المختبر على مكتبة cDNA للأرز تحتوي على مُحفز T7، وحصلوا على آلاف النسخ مع علامات تعديل مختلفة. بعد إضافة ذيول polyA، تم إنشاء أربع مجموعات تدريب (m¹A، m⁶A، m⁵C، وقواعد غير معدلة) من خلال DRS.يُطلق عليها اسم مجموعة بيانات النسخ الظاهري في المختبر (IVET).
هندسة النموذج: إطار عمل التعلم العميق
وبناءً على ذلك، استخدم فريق البحث الإشارات الكهربائية المخصصة لكل 5 قواعد وخصائصها الإحصائية كمدخلات لتدريب نموذج التعلم النقلي TandemMod، والذي يمكنه اكتشاف أنواع متعددة من تعديلات الحمض النووي الريبي في وقت واحد.
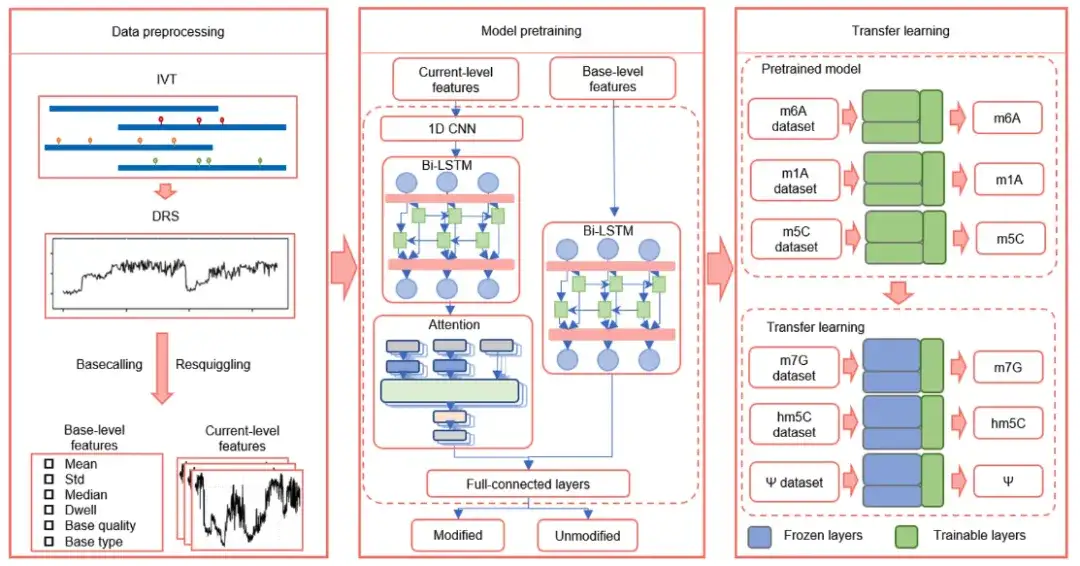
كما هو موضح في الشكل أعلاه،يتكون TandemMod من معالجة البيانات مسبقًا، والتدريب المسبق للنموذج، ونقل التعلم.
من بينها، يتكون نموذج التدريب المسبق من 4 مكونات رئيسية:
* شبكة عصبية ملتوية أحادية البعد (1D-CNN) لاستخراج السمات المحلية لإشارة شدة التيار الأصلية؛
* Bi-LSTM (Bi-LSTM)، والذي يستخدم لالتقاط الارتباطات طويلة الأمد بين الإشارات المتجاورة وتحسين القدرة على فهم السياق في عملية أطول؛
* آلية الاهتمام، التي تستخدم لترجيح أهمية كل ميزة في أوقات مختلفة وتحسين قدرة النموذج على التقاط الإشارات المهمة؛
* المصنفات في الطبقات المتصلة بالكامل مسؤولة عن إجراء التنبؤات بناءً على مجموعة جميع الميزات.
بالإضافة إلى ذلك، للتحقق مما إذا كان من الممكن تطبيق التعلم بالنقل على بيانات DRS للكشف عن أنواع متعددة من تعديلات الحمض النووي الريبي،قام الباحثون بتدريب TandemMod على مجموعة بيانات IVET m5C وحصلوا على نموذج مدرب مسبقًا.في نموذج TandemMod، تعمل الطبقة العلوية كمستخرج للميزات وتعمل الطبقة السفلية كمصنف. قام الباحثون بتجميد الطبقات العليا من النموذج المدرب مسبقًا وإعادة تدريب الطبقات السفلية على مجموعة تدريب ELIGOS (hm5C، m7G، Ψ، وI) لتقليل خطأ التصنيف.

بعد حقبتين، حققت جميع النماذج دقة عالية.وصلت ROC-AUCs لـ hm⁵C وm⁷G وΨ وI إلى 0.98 و0.95 و0.96 و0.97 على التوالي. كما هو موضح في الأشكال (أ)، (ب)، (ج)، و(د) أعلاه.
النتائج التجريبية: يقلل TandemMod بشكل كبير من كمية بيانات مجموعة التدريب ووقت تدريب النموذج
خلال المرحلة التجريبية، قارن فريق البحث نموذج TandemMod مع خوارزميات التعلم الآلي الكلاسيكية لتقييم أدائها، وهي XGBoost وآلة الدعم المتجه (SVM) وk-nearest neighbor (KNN). في حالة التعرف على مجموعة بيانات اختبار Curlcake m⁶A،يتفوق TandemMod على الخوارزميات الأخرى بدقة 0.90.وعلى نحو مماثل، بالنسبة لتحديد m⁵C، حقق TandemMod دقة بلغت 0.95، وتسلط هذه المقارنة الضوء على فعالية TandemMod في تحديد التعديلات باستخدام بيانات DRS.
وأظهر TandemMod أيضًا تفوقًا أفضل من tombo وxPore في تحديد العينات ذات مستويات معدل التعديل المختلفة في الجسم الحي.يشير هذا إلى أن TandemMod يمكنه التنبؤ بدقة بالعينات ذات معدلات التعديل المختلفة دون الحاجة إلى عينات تحكم سلبية.
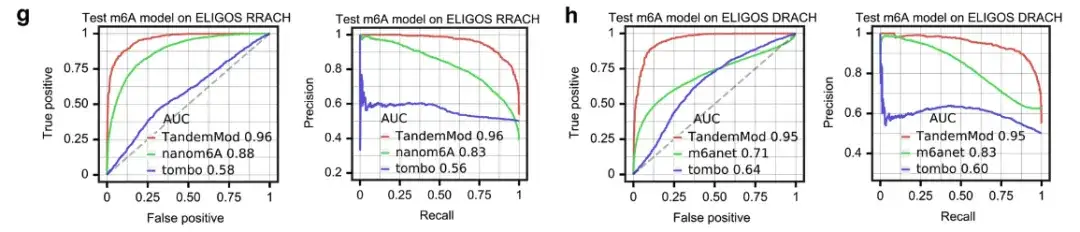
بالإضافة إلى ذلك، قام فريق البحث أيضًا بمقارنة نموذج TandemMod m⁶A مع tombo وnanom6A وm6Anet، كما هو موضح في الشكل أعلاه.
في شكل طفح ELIGOS (RA أو G أو HA أو C أو U)، كانت ROC-AUCs لـ TandemMod وnanom6A وtombo 0.96 و0.88 و0.52 على التوالي. في نموذج ELIGOS DRACH (DA أو G أو U)، كانت ROC-AUCs الخاصة بـ TandemMod وm6Anet وtombo 0.95 و0.71 و0.64 على التوالي.
وتشير هذه النتائج إلى أنتم تدريب TandemMod باستخدام مجموعة بيانات DRS المختبرية، ويوفر تنبؤات مستوى القراءة الأكثر دقة بين الأدوات الموجودة.
قام فريق البحث بالتحقق من أداء التصنيف وبيانات التدريب المطلوبة واستخدام موارد الحوسبة لتعلم النقل لنموذج TandemMod m⁵C في الكشف عن m⁶A، وقارنوه بنموذج TandemMod m⁶A للمثال القياسي. وتظهر النتائج أن التعلم بالنقل يمكن أن يقلل بشكل كبير من التكاليف مثل كمية بيانات مجموعة التدريب ووقت تدريب النموذج مع ضمان نفس الأداء.
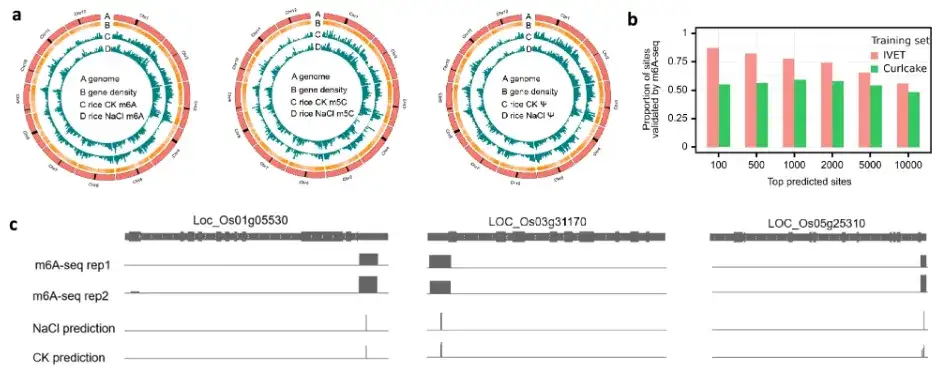
وأخيرًا، اختبر فريق البحث قدرة نموذج TandemMod على التوسع ليشمل أنواعًا جديدة من أجل تسلسل بيانات DRS، كما تحقق بشكل أكبر من موثوقية TandemMod باستخدام سلالات الخلايا البشرية (عينتان من تعديل إنزيم الإخراج و5 عينات من النوع البري). وفي الوقت نفسه، استخدم فريق البحث أيضًا TandemMod لرسم خرائط التعديل الجيني لـ m⁶A وm⁵C وΨ في شتلات الأرز تحت ضغط الملح العالي، وكشف عن التعديل المشترك لـ m⁶A وm⁵C في mRNA والتغيرات في معدلات تعديلها تحت البيئات ذات الملح العالي. كما هو موضح في الصورة أعلاه.
تعديل الحمض النووي الريبوزي يفتح أبوابًا جديدة لاستكشاف الحياة
على مر العصور، لم يتوقف الناس أبدًا عن استكشاف الحياة. بعد طرح فرضية عالم الحمض النووي الريبوزي (RNA)، أصبحت الحجة القائلة بأن الحمض النووي الريبوزي هو أصل الحياة بلا شك واحدة من الإجابات الأكثر إقناعاً في الوقت الحاضر. منذ اكتشاف التعديل الأول للحمض النووي الريبوزي (RNA) في عام 1960، أصبح لفترة طويلة أولوية قصوى للبحث العلمي واستمر في تلقي مستوى عالٍ من الاهتمام في الدراسات الحديثة.
بالإضافة إلى مجموعة البحث الخاصة بـ Yu Xiang وفريق Yang Jun/Wang Hongxia في هذه الورقة، بالإضافة إلى شركة ONT المذكورة في المقالة، هناك العديد من الفرق والشركات الأخرى التي تجري أيضًا أبحاث تعديل الحمض النووي الريبي.

على سبيل المثال، في عام 2021، نشر فريق البروفيسور منج جيا من جامعة شيآن جياوتونغ-ليفربول مقالاً بعنوان "الشبكات العصبية متعددة العلامات القائمة على الاهتمام للتنبؤ والتفسير المتكامل لاثني عشر تعديلاً واسع الانتشار في الحمض النووي الريبي" في مجلة Nature Communications.
عنوان الورقة:https://www.nature.com/articles/s41467-021-24313-3
تذكر المقالة نموذج MultiRM يعتمد على إطار عمل التعلم العميق متعدد العلامات مع آلية الاهتمام.لا يمكن التنبؤ بـ 12 موقعًا من مواقع النسخ المنتشرة على نطاق واسع في وقت واحد فحسب، بل يتم أيضًا استخراج التسلسلات الرئيسية في عملية التنبؤ وتحليلها، مما يكشف عن وجود ارتباط قوي بين أنواع مختلفة من تعديلات الحمض النووي الريبي، مما يساعد على تحليل وفهم آليات تعديل الحمض النووي الريبي القائمة على التسلسل بشكل أفضل وشامل.

بالمصادفة، في ورقة بحثية عام 2021 بعنوان "تحديد تعديلات الحمض النووي الريبي التفاضلية من تسلسل الحمض النووي الريبي المباشر للنانوبور باستخدام xPore" نُشرت في مجلة Nature Biotechnology،استخدم فريق البحث برنامج xPore لتحديد تعديلات الحمض النووي الريبي بدقة عالية من بيانات Direct RNA-seq وتحليل التعديل التفاضلي والتعبير من تجربة واحدة عالية الإنتاجية.
عنوان الورقة:https://www.nature.com/articles/s41587-021-00949-w
وتساعدنا هذه الدراسات على فتح الباب بشكل أكبر أمام عالم الحمض النووي الريبي، مما يسمح لنا باستكشاف "المعنى الحقيقي للحياة" بشكل أكبر. على الرغم من أنه لا يزال هناك العديد من الاختناقات التي يتعين التغلب عليها في تقدم الأبحاث المختلفة، إلا أن التحديات المستمرة التي يواجهها "الرواد" قد فتحت بالفعل الباب أمام أبحاث الحمض النووي الريبي.
مراجع:
1. https://news.sjtu.edu.cn/jdzh/2








