Command Palette
Search for a command to run...
Die Für NeurIPS 2025 Ausgewählte Universität Von Toronto Und Andere Schlugen Ein Ctrl-DNA-Framework Vor, Um Eine „gezielte Kontrolle“ Der Genexpression in Bestimmten Zellen Zu erreichen.

Die präzise Regulierung der Genexpression in spezifischen Zellen ist entscheidend für Fortschritte in Bereichen wie der Gentherapie und der synthetischen Biologie. Dieser Prozess basiert auf einer Klasse von DNA-Sequenzen, den sogenannten „cis-regulatorischen Elementen (CRE)“, wie Promotoren und Enhancer. Sie fungieren als „Schalter“ für Gene und bestimmen, ob Gene in Zielzellen „an-“ oder „abgeschaltet“ werden, während sie eine abnormale Aktivierung in anderen normalen Zellen verhindern. AllerdingsDie Anzahl der natürlich vorkommenden wirksamen CREs ist begrenzt und lässt sich nur schwer genau mit verschiedenen biomedizinischen Anwendungsszenarien abgleichen.Noch wichtiger ist, dass die Möglichkeiten von DNA-Sequenzen exponentiell wachsen. Beispielsweise weist eine 100-Basen-Sequenz 4¹⁰⁰-Kombinationen auf. Es ist äußerst schwierig, sie einzeln experimentell zu verifizieren. Dies ist nicht nur zeit- und arbeitsintensiv, sondern kann auch den praktischen Anforderungen nicht gerecht werden.
Aktuelle Deep-Learning-basierte Methoden haben die experimentelle Effizienz erheblich verbessert, bestehende Methoden stehen jedoch noch immer vor zahlreichen Herausforderungen.Einige Methoden basieren beispielsweise auf Mutationen in vorhandener DNA oder zufälliger Sequenzoptimierung, die leicht in die Falle der „lokalen Optimalität“ tappen können, was zu unzureichender Diversität in den generierten effektiven Sequenzen führt. Ansätze auf Basis autoregressiver Sprachmodelle können zwar DNA-Sequenzmuster erfassen, aber sie können nur „bekannte Sequenzen nachahmen“ und sind nicht in der Lage, neue zellspezifische CREs zu erforschen. Während auf Reinforcement Learning (RL) basierende Methoden die regulatorischen Effekte in Zielzellen verbessern, übersehen sie die Kontrolle von „Nebenwirkungen“ auf andere Zellen. Darüber hinaus übersehen diese standardmäßigen Design-Frameworks oft Überlegungen zur biologischen Plausibilität. Die generierten Sequenzen passen möglicherweise nicht zu wichtigen Transkriptionsfaktor-Bindungsstellen (TFBSs), was zum Versagen tatsächlicher regulatorischer Funktionen führt.
Um die Lücke im präzisen Design zellspezifischer CRE zu schließen, hat ein Team der Universität Toronto in Zusammenarbeit mit dem Changping Laboratory und anderen Institutionen ein Framework für eingeschränktes bestärkendes Lernen namens Ctrl-DNA entwickelt.Dieses Framework basiert auf einem vortrainierten DNA-Sprachmodell und nutzt einen Reinforcement-Learning-Algorithmus, um während des Optimierungsprozesses zwei Ziele gleichzeitig zu erreichen: die Maximierung der regulatorischen Aktivität von CREs in Zielzellen bei gleichzeitiger strikter Begrenzung ihrer Aktivität in Nicht-Zielzellen. Darüber hinaus wird das mathematische Werkzeug der Lagrange-Multiplikatoren eingesetzt, um diese beiden Anforderungen in Einklang zu bringen. Die Verteilung der TFBSs in realer DNA wird herangezogen, um die biologische Validität der generierten Sequenzen sicherzustellen.
Die Ergebnisse der Studie zeigten, dassBei den Designaufgaben mit 6 menschlichen Zellen übertraf das von Ctrl-DNA generierte CRE die bestehenden Methoden bei zwei Schlüsselindikatoren deutlich: „hohe Aktivität in Zielzelltypen“ und „Einschränkung in Nicht-Zielzelltypen“.Darüber hinaus wird eine erhebliche Vielfalt bewahrt, indem neue Lösungen für die synthetische Biologie bereitgestellt werden, um „kontrollierbare Systeme zu schaffen“, für die Gentherapie, um „Off-Target-Risiken zu vermeiden“, und für die Präzisionsmedizin, um „individuelle Anpassungen auf Zellebene durchzuführen“.
Die entsprechenden Forschungsergebnisse wurden auf der Preprint-Plattform arXiv unter dem Titel „Ctrl-DNA: Constrained Reinforcement Learning for Cell-Specific Cis-Regulatory Element Design“ veröffentlicht und für NeurIPS 2025 ausgewählt.
Forschungshighlights:
* Es wird ein neuartiges Framework für einschränkungsbewusstes bestärkendes Lernen vorgeschlagen, um Werkzeuge für die Entwicklung von CREs für eine präzise zelltypspezifische Genexpression bereitzustellen.
* Vereinfachter Optimierungsprozess, verbesserte experimentelle Effizienz und reduzierte Rechenkosten
* Experimente haben bestätigt, dass Ctrl-DNA sowohl funktionelle Wirksamkeit als auch biologische Plausibilität besitzt

Papieradresse:
https://arxiv.org/abs/2505.20578
Folgen Sie dem offiziellen Konto und antworten Sie mit „Strg-DNA“, um das vollständige PDF zu erhalten
Weitere Artikel zu den Grenzen der KI:
Datensatz: Basierend auf realen menschlichen Promotor- und Enhancer-Datensätzen
In dieser Studie verwendeten die Forscher echte menschliche Promotor- und Enhancer-Datensätze, um Ctrl-DNA zu bewerten und zu validieren.
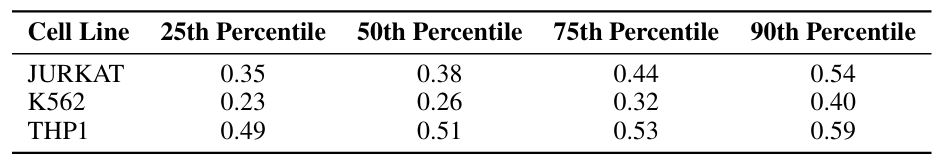
In,Der Human Promoter Dataset enthält Daten zur Promotoraktivität von drei aus Leukämie stammenden Zelllinien.Die drei Zelllinien sind: Jurkat, K562 und THP1. Alle drei sind mesoderm-abgeleitete hämatopoetische Zelllinien mit hoher biologischer Ähnlichkeit. Jede Sequenz in diesem Datensatz ist 250 Basenpaare lang. Siehe die folgende Tabelle:
Der Human Enhancer Dataset enthält CRE-Aktivitätsdaten von drei Zelllinien, die mit einem massiv parallelen Reporter-Assay (MPRA) gemessen wurden.Die drei Zelllinien sind: HepG2 (Leberzelllinie), K562 (Erythroidzelllinie) und SK-N-SH (Neuroblastomzelllinie). Jede Sequenz in diesem Datensatz ist 200 Basenpaare lang. Wie in der folgenden Tabelle dargestellt:
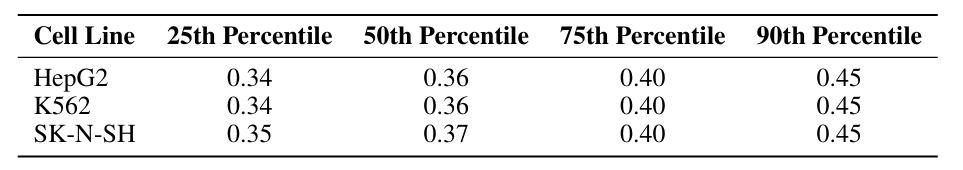
Bemerkenswert ist, dass in der THP1-Zelllinie die 25. Perzentile der Aktivität 0,49 erreichten, was eine rechtsschiefe Verteilung zeigt. Diese Verteilungsverzerrung könnte teilweise dafür verantwortlich sein, dass es in der THP1-Zelllinie schwieriger ist, die Aktivität einzuschränken.
Modellarchitektur: Basierend auf einem vortrainierten DNA-Sprachmodell, kombiniert mit Lagrange-Relaxation
Ctrl-DNA ist ein Framework für die regulatorische DNA-Sequenzgestaltung, das auf eingeschränktem Verstärkungslernen basiert und dessen Hauptziel die Generierung von CREs mit kontrollierbarer Zelltypspezifität ist.Im Hinblick auf die funktionale Umsetzung muss die Fitness des CRE in Zielzellen maximiert werden, d. h. die Genexpression gesteigert werden, während die Fitness in Off-Target-Zellen innerhalb eines voreingestellten Schwellenwerts streng kontrolliert wird. Gleichzeitig muss sichergestellt werden, dass die generierten Sequenzen den tatsächlichen biologischen Gesetzen entsprechen, um Situationen zu vermeiden, in denen die experimentellen Ergebnisse den Anforderungen entsprechen, die Anwendung jedoch ineffektiv ist.
Zu diesem Zweck berücksichtigten die Forscher die Benutzerfreundlichkeit, Rationalität und andere Aspekte des Frameworks und führten einen detaillierten Entwurf des Frameworks durch, wie in der folgenden Abbildung dargestellt:
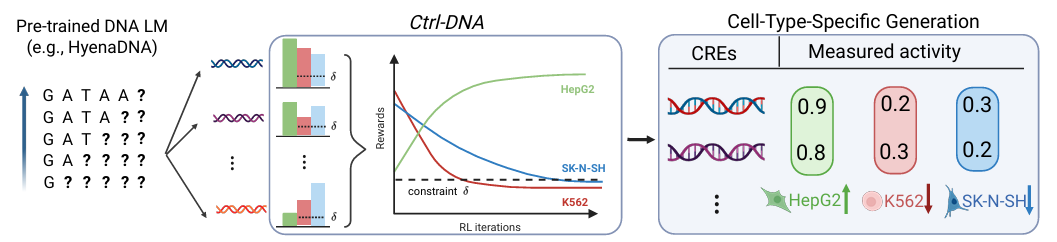
In Bezug auf Modelle und EingabenCtrl-DNA optimiert das autoregressive genomische Sprachmodell HyenaDNA, das auf dem menschlichen Genom als anfängliches Richtlinienmodell vortrainiert wurde, und verwendet die Enformer-Architektur, um das zelltypspezifische Belohnungsmodell zu trainieren.In Kombination mit den durch massiv parallele Berichtsexperimente gemessenen „Sequenzfitness“-Daten werden die Belohnungen für Zielzellen und die Belohnungen für Nicht-Zielzellen separat berechnet.
Auf der Ebene der ProblemmodellierungDie Forscher wandelten das DNA-Sequenzdesign in einen eingeschränkten Markov-Entscheidungsprozess (CMDP) um. Der zentrale Optimierungsmechanismus von Ctrl-DNA nutzt die Constrained Batch-wise Relative Policy Optimization (CBROP). Dieser Mechanismus transformiert ein eingeschränktes Optimierungsproblem durch Lagrange-Relaxation in ein uneingeschränktes primalduales Optimierungsproblem. Der Optimierungsprozess ist iterativ, wobei die Richtlinienaktualisierungen dem Gradienten der Lagrange-Zielfunktion mit der Lernrate folgen. Die Belohnungen für Off-Target-Zellen werden durch Anpassung des Lagrange-Multiplikators eingeschränkt. Durch Erhöhen des Lagrange-Multiplikators wird die Einschränkung für Off-Target-Zellen, die einen Schwellenwert überschreiten, verstärkt, und durch Verringern des Lagrange-Multiplikators wird die Einschränkung für Off-Target-Zellen, die den Schwellenwert erreichen, abgeschwächt.
Um die Komplexität des Trainings zu reduzieren, verzichtet Ctrl-DNA auf die Abhängigkeit von Wertmodellen im traditionellen bestärkenden Lernen.Der normalisierte Vorteil wird direkt auf Grundlage der Batchdatenstatistik berechnet, um die Strategieoptimierung zur Auswahl von Sequenzen mit „hoher Zielbelohnung + geringer Off-Target-Belohnung“ zu steuern.
Beim Entwurf der Zielfunktion zur Strategieaktualisierung verwendeten die Forscher eine Kombination aus „Pruning-Ersatzzielen“ und „KL-Regularisierung“. Durch das Pruning schränkten sie Strategiemutationen ein und führten die KL-Divergenz zwischen der aktuellen Strategie und der ursprünglichen Referenzstrategie ein, um die Übereinstimmung der generierten Sequenz mit dem natürlichen DNA-Muster sicherzustellen und so letztendlich eine Zielfunktion zur Strategieaktualisierung zu bilden.
Um die biologische Plausibilität weiter zu gewährleisten, führt Ctrl-DNA die TFBS-Frequenzkorrelation als zusätzliche Einschränkung ein. Zunächst werden TFBSs mit dem FIMO-Tool aus realen, hochspezifischen CRE-Sequenzen gescannt, um einen echten TFBS-Frequenzvektor zu erstellen. Anschließend wird für jede generierte Sequenz der entsprechende TFBS-Frequenzvektor berechnet. Der Pearson-Korrelationskoeffizient wird dann als zusätzliche Einschränkungsbelohnung verwendet, während der entsprechende Lagrange-Multiplikator auf [0, λmax] (λmax ≤ 1) begrenzt wird. Dies gleicht biologische Plausibilität mit objektiver Optimierung aus und vermeidet übermäßige Einschränkungen, die die Möglichkeiten der Modellerkundung einschränken könnten.
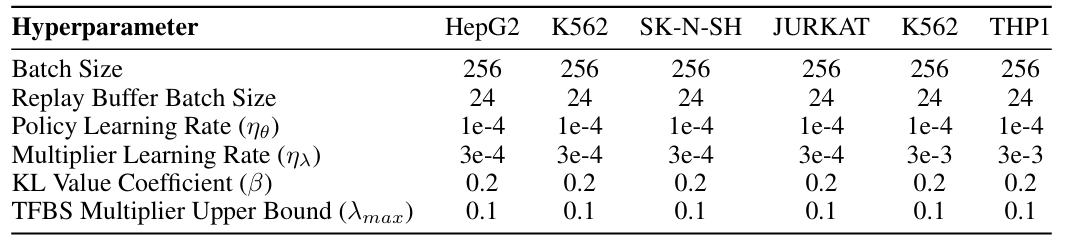
Um die Stabilität des Modelltrainings zu gewährleisten, demonstrierten die Forscher die in den Experimenten verwendeten Hyperparametereinstellungen. Alle Modelle wurden mit dem Adam-Optimierer mit einer Policy-Learning-Rate von 1e-4, einer Batchgröße von 256 und 100 Trainingsepochen trainiert. Die Experimente wurden auf einer einzelnen NVIDIA A100-GPU mit 40 GB Speicher trainiert, wie in der folgenden Abbildung dargestellt.
Experimentelle Ergebnisse: Im Vergleich zu 8 Arten von Basismethoden hat Ctrl-DNA offensichtliche Vorteile
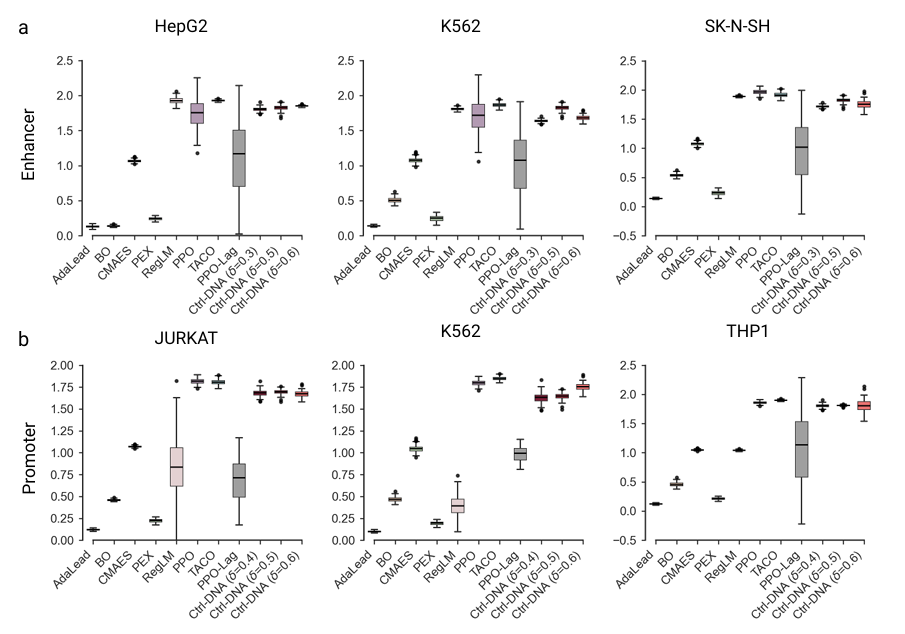
Das Leistungsbewertungsexperiment von Ctrl-DNA dreht sich um zwei wichtige Designaufgaben: menschliche Enhancer und Promotoren, die die sechs oben genannten Zelllinien abdecken. Es wird mit acht Arten von Basismethoden verglichen, darunter Evolutionsalgorithmen (einschließlich AdaLead, Bayesianische Optimierung (BO), CMA-ES, PEX), generative Modelle (RegLM) und Methoden des bestärkenden Lernens (einschließlich TACO, PPO und PPO-Lagrange), um seine Wirksamkeit und Praktikabilität in mehreren Dimensionen wie Zelltypspezifität, biologischer Plausibilität und Sequenzdiversität zu überprüfen.
Ctrl-DNA weist erhebliche Vorteile hinsichtlich der zelltypspezifischen Eingrenzung auf.Wie in der folgenden Abbildung dargestellt, stellt die horizontale Achse die Fitness der Off-Target-Zelltypen dar, während die vertikale Achse die Fitness der Zielzelltypen darstellt. Die in der oberen rechten Ecke dargestellte Methode stellt das beste Gleichgewicht zwischen der Maximierung der Zielzellfitness und der Minimierung der Off-Target-Expression dar.
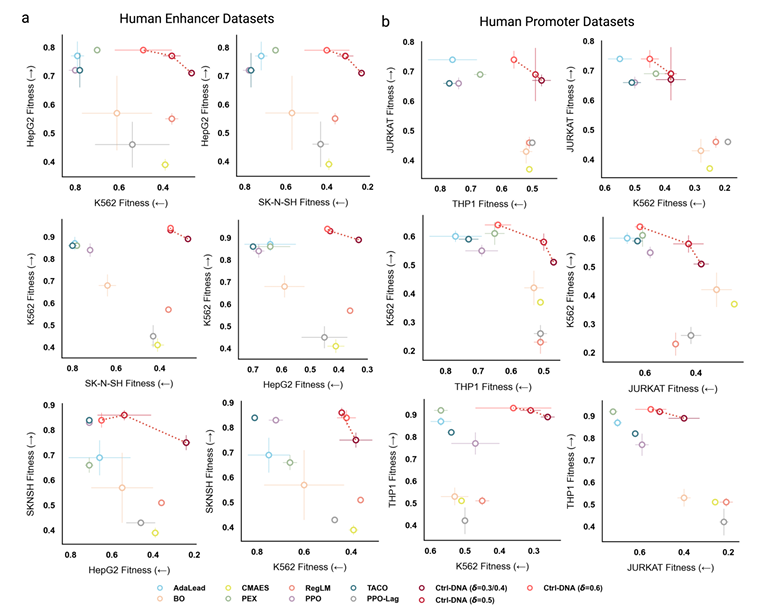
Beim Enhancer-Design erreichte Ctrl-DNA durchweg die höchste Zielzellfitness und erfüllte gleichzeitig die Off-Target-Bedingungen bei allen verschiedenen Grenzwerten (δ = 0,3, 0,5 und 0,6). Dies bedeutet, dass die Zielzellfitness maximiert und gleichzeitig die Off-Target-Bedingungen strikt eingehalten wurden. Darüber hinaus erreichten Methoden wie TACO und CMAES zwar eine hohe Expression in Zielzellen, konnten jedoch die Off-Target-Zellfitness nicht unterdrücken, was zu einer schlechten Zelltypspezifität führte.
Da alle drei Zielzelltypen hämatopoetische Zellen mesodermalen Ursprungs sind, weisen sie bei der Promotorgestaltung große transkriptionelle Ähnlichkeiten auf, was eine erhebliche Herausforderung für diese Aufgabe darstellt, aber Ctrl-DNA schneidet trotzdem gut ab. Im Experiment wurden drei verschiedene Grenzwerte (δ=0,4, 0,5 und 0,6) zum Testen festgelegt. Ctrl-DNA übertraf alle Basiswerte, wenn es die Fitness des Zielzelltyps maximierte und die Grenzwerte δ=0,5 und 0,6 einhielt. Es ist auch erwähnenswert, dass in Fällen wie THP1-Zellen, bei denen die Aktivitätsverteilung rechtsschief ist (wie im Abschnitt zum Datensatz oben erwähnt, hat die Aktivität des 25. Perzentils 0,49 erreicht), keine Methode die Off-Target-Aktivität auf den strengen Grenzwert von δ=0,4 unterdrücken kann, aber Ctrl-DNA ist die Methode, die dieser Anforderung von allen am nächsten kommt.
Bei der biologischen Plausibilitätsvalidierung erzielte Ctrl-DNA, wie in der folgenden Abbildung dargestellt, die höchste Belohnungsdifferenz (ΔR) über alle Zelltypen hinweg für menschliche Promotoren und Enhancer. Dies deutet darauf hin, dass es die zellspezifische Fitness von DNA-Sequenzen besser optimiert. Auch hinsichtlich der Motivrelevanz erzielte Ctrl-DNA in den meisten Zelltypen eine stärkere Leistung, mit Ausnahme des THP1-Promotordesigns.
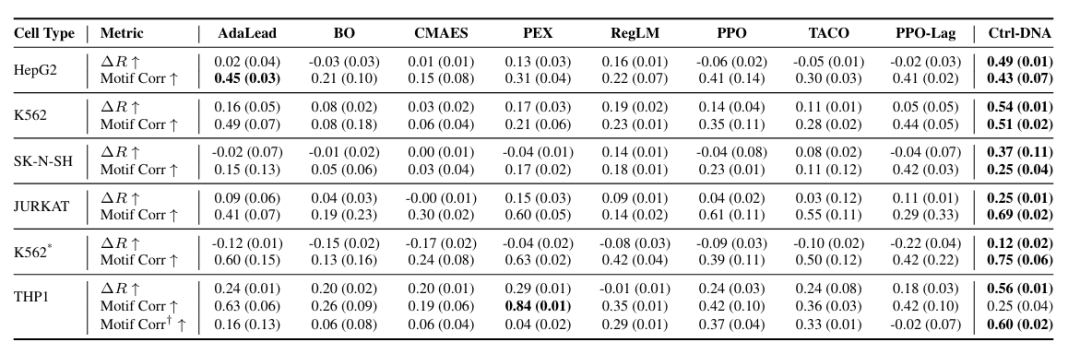
Um diese Diskrepanz weiter zu untersuchen, extrahierten die Forscher Motive aus Promotorsequenzen im 90. Perzentil der THP1-Fitness. Um falsch positive Ergebnisse zu vermeiden, verwendeten sie einen Schwellenwert von q < 0,05 und überprüften die Motivkorrelation zwischen den generierten Sequenzen und dem Referenzsatz, dargestellt als Motiv Corr† in der obigen Abbildung. Die Ergebnisse zeigten, dass Ctrl-DNA selbst unter diesen strengen Bedingungen alle Basislinien übertraf. Der Korrelationskoeffizient stieg auf 0,60, während die Korrelationen für die meisten Basislinien sanken. Dies belegt die Fähigkeit des Ctrl-DNA-Systems, bevorzugt funktionell relevante regulatorische Motive zu erfassen.
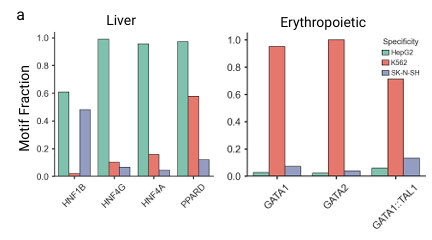
Um die Häufigkeit der gefundenen spezifischen TFBSs weiter zu analysieren, untersuchten die Forscher insbesondere die Sequenzen, die für Motive generiert wurden, die spezifisch für die Hepatozytenzelllinie HepG2 und die Erythroidzelllinie K562 sind.Wie in der folgenden Abbildung dargestellt, weist die von Ctrl-DNA generierte HepG2-Sequenz die höchste Frequenz leberspezifischer Motive wie HNF4A und HNF4G auf. Ebenso enthält die für K562 generierte Sequenz die höchste Frequenz erythroidspezifischer Motive wie GATA1 und GATA2. Dies zeigt, dass Ctrl-DNA nicht nur die Fitness der Zielzelle optimiert, sondern auch regulatorische Muster lernt, die die zugrunde liegende Zelltypspezifität widerspiegeln.
In Bezug auf die Sequenzvielfalt erreichte Ctrl-DNA eine vergleichbare oder höhere Vielfalt als die meisten Basislinien, was seine Fähigkeit bestätigt, vielfältige Sequenzen zu erzeugen, ohne die regulatorische Kontrolle zu opfern.Wie in der folgenden Abbildung dargestellt:
Abschließend validierten die Forscher die Wirksamkeit des Ctrl-DNA-Kernmoduls durch Ablationsexperimente. Auch die Rolle des TFBS-Regularisierungsmoduls wurde bestätigt, da es Sequenzen effektiv in Richtung biologisch realistischer Muster lenkt.
KI-gesteuertes DNA-„Schalter“-Design schlägt ein neues Kapitel auf
In der Vergangenheit beruhte die Entwicklung regulatorischer DNA-Sequenz-„Schalter“ meist auf dem Prinzip „Versuch und Irrtum“ durch eine große Zahl wiederholter manueller Screenings.Durch die Kombination mit KI-Technologie können wir nun mithilfe von Algorithmen vorhersagen, „welche DNA-Sequenzen die höchste Übereinstimmung mit dem Zielregulationsprotein aufweisen“, was die Designeffizienz und -genauigkeit erheblich verbessert.Dies ist auch der Hauptgrund, warum das KI-gesteuerte Design von DNA-Schaltern eine neue Richtung eingeschlagen hat, die wiederum Bereiche wie Gentherapie und synthetische Biologie direkt von „umfangreich“ zu „präzise“ vorantreibt.
Dieses Papier ist nur eine der großen Früchte am großen Baum des „KI-gesteuerten DNA-Schalterdesigns“. Rückblickend haben viele Labore bereits entsprechende Forschungen durchgeführt.
So veröffentlichte beispielsweise ein Team des Jackson Laboratory, des Broad Institute und der Yale University in Nature eine Studie mit dem Titel „Maschinengeführtes Design von cis-regulatorischen Elementen, die auf Zelltypen abzielen“.Im Rahmen der Studie wurde künstliche Intelligenz eingesetzt, um Tausende neuer DNA-Schalter zu entwerfen.Diese Schalter können die Genexpression in verschiedenen Zelltypen präzise steuern. Konkret konstruierten die Forscher ein tiefes Convolutional Neural Network (Malinois), das die CRE-Aktivität präzise vorhersagen kann, und entwickelten eine modulare Plattform (CODA) für die Entwicklung von CREs mit spezifischen Funktionen. Diese Plattform bietet leistungsstarke Werkzeuge für die Entwicklung von Reportergenen, CRISPR-Therapien, Genersatzmethoden und mehr.
Papieradresse:
https://www.nature.com/articles/s41586-024-08070-z
Darüber hinaus gibt es das im obigen Artikel erwähnte RegLM von Genentec. In der Studie mit dem Titel „Designing realistic regulatory DNA with autoregressive language models“Wir stellen ein Framework namens RegLM vor, das auf einem autoregressiven Sprachmodell in Kombination mit einem überwachten Sequenz-Funktionsmodell basiert, um synthetische CREs mit spezifischen Eigenschaften zu entwerfen.RegLM basiert ebenfalls auf dem HyenaDNA-Framework. Es kodiert funktionale Markierungen als Hinweis-Token und fügt sie dem DNA-Sequenzpräfix hinzu, trainiert oder optimiert das Modell für die nächste Token-Vorhersage und generiert so DNA-Sequenzen mit gewünschten Funktionen. Gleichzeitig kombiniert es ein überwachtes Sequenz-Aktivitäts-Regressionsmodell, um die generierten Sequenzen zu screenen.
Papieradresse:
https://genome.cshlp.org/content/34/9/1411.full#aff-1
Zusammenfassend lässt sich sagen, dass die Entwicklung von Ctrl-DNA zweifellos einen weiteren Fortschritt im DNA-Schalter-Design darstellt. Obwohl es noch einige Probleme oder Bereiche gibt, die dringend verbessert werden müssen, wie z. B. die Einbeziehung zusätzlicher biologischer Einschränkungen zur weiteren Verbesserung der Rationalität und Funktionalität der generierten Sequenz, und die Anpassung des Lagrange-Multiplikators noch eine Frage der Erfahrung ist, haben die Entwicklung und Verbesserung dieser Werkzeuge zweifellos ein neues Kapitel im DNA-Schalter-Design aufgeschlagen und gleichzeitig die kontinuierliche Weiterentwicklung der interdisziplinären Wissenschaft der künstlichen Intelligenz und Biologie gefördert.