Command Palette
Search for a command to run...
Meta AI Et al. Haben Ein Neues Framework Zur Charakterisierung Dynamischer Proteinfusionen Namens FusionProt Vorgeschlagen, Das Einen Iterativen Informationsaustausch Ermöglicht Und Bei Mehreren Aufgaben Eine SOTA-Leistung erreicht.
Proteine sind die Ausführenden der Lebensfunktionen und ihre Geheimnisse liegen in zwei Dimensionen:Eine davon ist die eindimensionale (1D) Sequenz, die aus den aneinandergereihten Aminosäuren besteht, und die andere ist die dreidimensionale (3D) Struktur, die durch das Falten und Winden der Sequenz entsteht.Bisherige Modelle spezialisieren sich typischerweise auf eines der beiden: entweder auf die Beherrschung der Sequenzsprache, wie Proteinsprachenmodelle (PLMs) wie ProteinBERT und ESM, oder auf die Erkennung der Strukturmorphologie, wie dreidimensionale Proteindarstellungstechnologien wie GearNet. Selbst wenn Modelle versuchen, beides zu kombinieren, verwenden sie oft einen vereinfachten, gespleißten Ansatz, der es scheinbar jedem Spezialisten ermöglicht, unabhängig statt kollaborativ zu arbeiten.
In diesem Zusammenhang haben die Forschungsteams des Technion-Israel Institute of Technology und von Meta AI gemeinsam das Proteinrepräsentations-Lernframework FusionProt vorgeschlagen.Ziel ist es, gleichzeitig eine einheitliche Darstellung der eindimensionalen Sequenz und der dreidimensionalen Struktur von Proteinen zu erlernen.Diese Forschung führt auf innovative Weise ein lernbares Fusionstoken als adaptive Brücke zwischen dem Proteinsprachenmodell (PLM) und dem 3D-Strukturgraphen ein und ermöglicht so einen iterativen Informationsaustausch zwischen beiden. FusionProt hat bei einer Vielzahl proteinbezogener biologischer Aufgaben Spitzenleistungen erzielt.
Die entsprechende Forschungsarbeit wurde auf bioRxiv unter dem Titel „FusionProt: Fusing Sequence and Structural Information for Unified Protein Representation Learning“ veröffentlicht.
Forschungshighlights:
* Das FusionProt-Framework durchbricht die Einschränkungen der bisherigen Strukturfragmentierungsverarbeitung, indem es eindimensionale und dreidimensionale Modalitäten effektiv integriert und die Genauigkeit der Erfassung von Proteinfunktionen und Interaktionseigenschaften verbessert.
* Eine neuartige modalübergreifende Fusionsarchitektur, die lernbare Fusionstoken verwendet, um einen iterativen Informationsaustausch zwischen dem Proteinsprachenmodell (PLM) und Protein-3D-Strukturgraphen zu ermöglichen.
* FusionProt erreicht bei mehreren Proteinaufgaben eine hochmoderne Leistung und demonstriert anhand von Fallstudien das Anwendungspotenzial des Modells in realen biologischen Szenarien.
Papieradresse:
Folgen Sie dem offiziellen Konto und antworten Sie mit „FusionProt“, um das vollständige PDF zu erhalten
Weitere Artikel zu den Grenzen der KI:
https://hyper.ai/papers
Erstellen Sie systematisch Datensätze mithilfe öffentlicher Proteindatenbanken
In dieser Studie nutzte das Forschungsteam öffentliche Proteindatenbanken in vollem Umfang und stellte die Wirksamkeit von FusionProt bei mehreren Aufgaben zum Verständnis von Proteinen durch systematische Datensatzkonstruktion, eine strikte Datenpartitionierungsstrategie und ein Multitasking-Auswertungsframework sicher.
In der Vortrainingsphase verwendete die Studie die Proteinstrukturdatenbank (AlphaFold DB) als Kerndatenquelle.Die Datenbank enthält 805.000 hochwertige dreidimensionale Proteinstrukturen, die von AlphaFold2 vorhergesagt wurden. Die Forscher wählten diesen Datensatz vor allem aus folgenden Gründen aus: Erstens gilt AlphaFold2 derzeit als hochmodernes Modell zur Vorhersage von Proteinstrukturen, und seine Vorhersagen sind äußerst zuverlässig, wodurch die Abhängigkeit von der Qualität und Verfügbarkeit externer experimenteller Strukturdaten effektiv reduziert wird. Zweitens gewährleistet die Verwendung einer einheitlichen vorhergesagten Struktur die Konsistenz über alle Datenquellen hinweg und erleichtert so einen fairen Vergleich mit früheren Arbeiten auf dem neuesten Stand der Technik wie SaProt und ESM-GearNet.
Das Forschungsteam bewertete die Leistung des Modells anschließend systematisch anhand von drei maßgeblichen nachgelagerten Aufgaben. Die Aufgaben verwendeten Datensätze von DeepFRI, das eine maßgebliche Datenpartitionierung ermöglicht und Fmax als einheitliche Bewertungsmetrik verwendet, um die Leistung des Modells bei der Annotation von Enzymfunktionen und der Genontologie-Inferenz umfassend zu messen. Die Aufgabe zur Vorhersage der Mutationsstabilität (MSP) verwendete denselben Datensatz und dasselbe Bewertungsprotokoll wie ESM-GearNet und nutzte AUROC als Bewertungsmetrik, um die Fähigkeit des Modells zu beurteilen, die Auswirkungen von Mutationen auf die Stabilität von Proteinkomplexen vorherzusagen.
Iterativer Informationsaustauschmechanismus, der durch „Fusion Token“ gesteuert wird
Das Design von FusionProt dreht sich um eine Kernidee:Durch lernbare Fusionstoken fungiert es als Brücke für eine bidirektionale, iterative kreuzmodale Interaktion zwischen Proteinsequenz und -struktur.Dadurch wird eine tiefe Verschmelzung und einheitliche Darstellung der beiden Informationsarten erreicht.
Erstens wird das Framework auf der Grundlage der dualmodalen parallelen Kodierungsarchitektur „Sequenzstruktur“ erstellt. Auf Sequenzebene wird ESM-2 als Proteinsprachenmodell verwendet, um die Aminosäuresequenz zu kodieren; auf Strukturebene wird der GearNet-Encoder als Strukturmodell verwendet, um das dreidimensionale Strukturdiagramm des Proteins zu modellieren. Das lernbare Fusionstoken wechselt während des Trainingsprozesses dynamisch zwischen den beiden Modalitäten und ermöglicht so einen iterativen Austausch und eine Fusion von Informationen. Auf Sequenzebene ist es mit der Proteinsequenz verbunden und die Aminosäure fragt das relevante eindeutige Fusionstoken ab, um wertvolle Informationen zu extrahieren und zu integrieren. Auf Strukturebene wird es dem dreidimensionalen Proteindiagramm hinzugefügt und als Knoten zusammengeführt und verbunden. Die Strukturebene wird von einem neuronalen Nachrichtenübermittlungsnetzwerk verarbeitet, wodurch das Fusionstoken globale räumliche Strukturinformationen integrieren kann.
Zweitens liegt die zentrale Antriebskraft dieses Frameworks im iterativen Fusionsalgorithmus.Der Prozess umfasst das Einfügen der Fusionstoken in die aktualisierte Sequenz, deren Weiterleitung an die Strukturebene und deren Einspeisung in das Strukturgraphennetzwerk als neue Knoten. Die aktualisierten Fusionstoken werden anschließend für die nächste Interaktionsrunde an die Sequenzebene zurückgeführt. Dieser iterative Prozess gleicht die verschiedenen Modalräume durch lernbare lineare Transformationen ab und passt sie an. Durch diesen iterativen Prozess werden die Modelldarstellungen zu einer einheitlichen und umfassenden Proteindarstellung kombiniert.
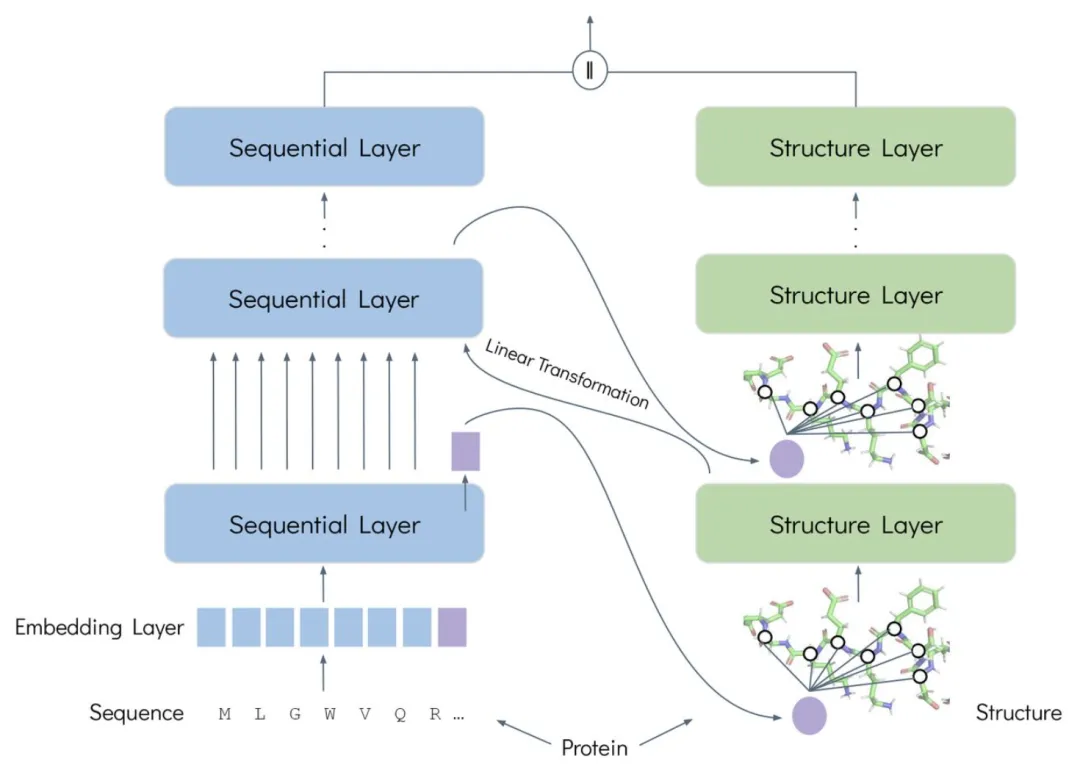
Schließlich verwendet FusionProt Multiview Contrastive Learning als Vortrainingsziel.Eine vielfältige Ansicht wird erstellt, indem aufeinanderfolgende Teilsequenzen zufällig ausgewählt und die Graphkanten von 15% ausgeblendet werden. Die InfoNCE-Verlustfunktion wird dann verwendet, um Darstellungen im latenten Raum auszurichten und dabei die Ähnlichkeit verwandter Proteinunterkomponenten bei der Abbildung auf den niedrigdimensionalen latenten Raum beizubehalten. Bei der Implementierung führte das Forschungsteam ein Vortraining mit der oben erwähnten AlphaFold DB-Datenbank durch. Während des Vortrainings verwendete FusionProt eine Lernrate von 2e-4, eine globale Batchgröße von 256 Proteinen und führte 50 Trainingsrunden durch. Die Eingabesequenz wurde auf 1.024 Token gekürzt, um lange Proteinsequenzen zu ermöglichen. Darüber hinaus wurde eine Feinabstimmung vorgenommen, indem aufgabenspezifische Klassifikationskopfvorhersagen hinzugefügt und dieselben Hyperparameter wie das neueste SaProt-Modell ausgewertet wurden. Alle Experimente wurden auf 4 x NVIDIA A100 80 GB GPUs durchgeführt, wobei eine einzelne Vortrainingssitzung ungefähr 48 Stunden dauerte.
Der Fusionsmechanismus übertrifft die bestehenden SOTA-Verfahren deutlich und hat erhebliche Auswirkungen
Die Studie wurde in mehreren nachgelagerten Aufgaben umfassend getestet.Die Ergebnisse zeigen, dass das FusionProt-Framework in mehreren Benchmarks eine SOTA-Leistung erreicht.Die experimentellen Ergebnisse sind in der folgenden Abbildung dargestellt.
Bei der Bewertung der EC-Zahlenvorhersage verglich das Forschungsteam die Leistung von FusionProt mit 11 Basismodellen. Die Ergebnisse zeigten, dass FusionProt den höchsten Fmax = 0,904 erreichte und damit Modelle, die ausschließlich auf Sequenzen basieren (wie ProtBERT-BFD, 0,838, ESM-2, 0,877), deutlich übertraf und auch GearNet (0,871) übertraf, das nur Strukturinformationen verwendet. Gleichzeitig liegt es im Vergleich zu anderen Methoden, die versuchen, diese beiden Arten von Informationen zu nutzen (wie MIF-ST, ESM-GearNet usw.), immer noch an der Spitze. Dieses Ergebnis zeigt, dass der iterative Fusionsmechanismus von FusionProt im Vergleich zur einfachen Verwendung einer Modalität als Kontext einer anderen Modalität wichtige dreidimensionale Strukturinformationen vollständiger beibehalten und dadurch die subtilen Strukturunterschiede, von denen die katalytische Aktivität abhängt, genauer erfassen kann.
Bei der Bewertung der GO-Term-Vorhersage erzielte FusionProt die besten Ergebnisse in den drei Unteraufgaben biologischer Prozess, molekulare Funktion und zelluläre Komponente und demonstrierte damit erneut die Wirksamkeit lernbarer Fusionstoken bei der gemeinsamen Modellierung von Sequenz und Struktur.
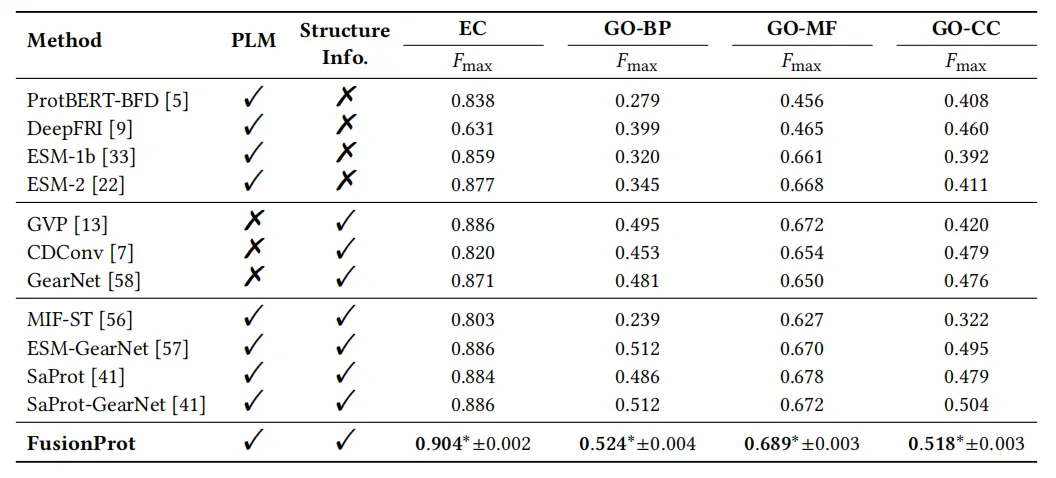
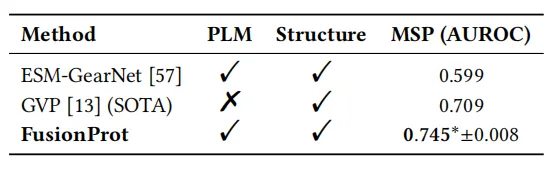
Das Forschungsteam führte zudem eine Evaluierung der Vorhersage mutationsbedingter Stabilität durch. Die experimentellen Ergebnisse zeigten, dass FusionProt von allen untersuchten Methoden den höchsten AUROC-Wert mit statistischer Signifikanz (p < 0,05) erreichte. Diese Leistung verbesserte sich im Vergleich zur aktuellen Spitzenmethode GVP um 5,11 TP3T deutlich und unterstreicht die Effektivität des iterativen Fusionsmechanismus bei der Integration weitreichender Sequenz-Struktur-Abhängigkeiten. Darüber hinaus ermöglicht FusionProt bidirektionale, modalübergreifende Interaktion durch lernbare Fusionstoken, wodurch die Proteindarstellung ausdrucksstärker und biologisch fundierter wird.
Um die Wirksamkeit der wichtigsten FusionProt-Designs zu bewerten, führte das Forschungsteam weitere Ablationsexperimente durch. Das Team testete das System bei verschiedenen Fusionsinjektionsfrequenzen und stellte fest, dass die optimale Leistung erreicht wurde, wenn die Fusionsmarker mehrere Interaktionsrunden zwischen den Sequenz- und Strukturcodierern bei einer Standardfrequenz durchführten. Eine Reduzierung der Interaktionsfrequenz hingegen schwächte die Leistung deutlich ab.Dies zeigt, dass ein häufiger Informationsaustausch für die Erfassung verkehrsträgerübergreifender Abhängigkeiten von entscheidender Bedeutung ist.
Schließlich konnte FusionProt in einer biologischen Fallanalyse die EC-Zahl des RNA-Polymerase-ω-Untereinheitsproteins erfolgreich vorhersagen, was mit herkömmlichen Methoden schwierig zu handhaben ist. Dieses Ergebnis schlug in Modellen wie ESM-2 völlig fehl, was ein weiterer Beweis dafür ist, dass die erlernte Darstellung komplexe Struktur-Funktions-Beziehungen erfassen kann und ein breites Anwendungspotenzial in der Arzneimittelentwicklung und Proteinfunktionsanalyse aufweist.
Cross-modale Fusion ist ein offensichtlicher Trend
FusionProt hat einen neuen Weg für das Lernen von Proteinrepräsentationen geebnet und gezeigt, dass die „Sprache“ und „Morphologie“ von Proteinen miteinander und nicht miteinander kommunizieren sollten. Mit der kontinuierlichen Weiterentwicklung der künstlichen Intelligenz in den Biowissenschaften hat sich die kreuzmodale Fusion zu einem klaren Trend entwickelt.
Die Westlake University schlug das Konzept eines strukturbewussten Vokabulars vor und kombinierte Aminosäurerest-Token mit Struktur-Token, um ein groß angelegtes universelles Proteinsprachenmodell namens SaProt anhand eines Datensatzes von rund 40 Millionen Proteinsequenzen und -strukturen zu trainieren. Dieses Modell übertraf etablierte Basismodelle bei zehn wichtigen nachgelagerten Aufgaben deutlich. Die zugehörige Forschung mit dem Titel „SaProt: Protein Language Modeling with Structure-aware Vocabulary“ wurde für ICLR 2024 ausgewählt.
Papieradresse:
https://openreview.net/forum?id=6MRm3G4NiU
Eine gemeinsame Studie mit dem Titel „Structure-Aligned Protein Language Model“, die von der Universität Montreal und Mila veröffentlicht wurde, schlägt ein strukturorientiertes Proteinsprachenmodell vor, das Strukturinformationen mittels kontrastivem Lernen einbezieht. Durch die Optimierung der vorhergesagten Strukturtoken des Modells verbessert das Modell die Leistung bei der Vorhersage von Proteinkontakten erheblich.
Papieradresse:
https://arxiv.org/abs/2505.16896
Erhalten Sie mit einem Klick hochwertige Papiere und ausführliche Interpretationsartikel im Bereich AI4S von 2023 bis 2024 ⬇️









