Command Palette
Search for a command to run...
ACL 2025: Die Universität Oxford Und Andere Schlagen Medizinisches GraphRAG Vor, Stellen Einen Neuen Rekord Für Die Genauigkeit Bei Der Beantwortung Von Fragen Auf Und Erzielen SOTA-Ergebnisse Bei 11 Datensätzen

Das Wissenssystem der Medizin basiert auf jahrtausendealten Entdeckungen und Wissensakkumulationen und umfasst eine enorme Menge an Prinzipien, Konzepten und praktischen Normen. Die wirksame Anpassung dieses Wissens an das begrenzte Kontextfenster aktueller großer Sprachmodelle ist mit unüberwindbaren technischen Hürden verbunden. Überwachtes Feintuning (Supervised Fine-Tuning, SFT) bietet zwar eine Alternative, doch aufgrund der Closed-Source-Natur der meisten kommerziellen Modelle ist dieser Ansatz nicht nur kostspielig, sondern in der Praxis auch äußerst unpraktisch. Zudem stellt die Medizin extrem hohe Anforderungen an die Genauigkeit der Terminologie und die Stringenz der Fakten. Für nicht professionelle Nutzer ist bereits die Überprüfung der Genauigkeit großer Modelle für medizinbezogene Antworten eine äußerst anspruchsvolle Aufgabe. Daher ist die Frage, wie große Modelle in die Lage versetzt werden können, große externe Datensätze für komplexe Schlussfolgerungen in medizinischen Anwendungen zu nutzen und genaue und glaubwürdige Antworten zu generieren, die durch überprüfbare Quellen gestützt werden, zu einem zentralen Thema der aktuellen Forschung in diesem Bereich geworden.
Das Aufkommen der Retrieval Enhanced Generation (RAG)-Technologie bietet einen neuen Ansatz zur Lösung der oben genannten Probleme.Es kann auf Benutzeranfragen mithilfe spezifischer oder privater Datensätze antworten, ohne dass das Modell weiter trainiert werden muss.Traditionelle RAGs sind jedoch immer noch unzureichend, wenn es darum geht, neue Erkenntnisse zu synthetisieren und Aufgaben zu bewältigen, die ein ganzheitliches Verständnis einer breiten Palette von Dokumenten erfordern. Das kürzlich vorgeschlagene GraphRAG übertrifft klassische RAGs bei komplexen Schlussfolgerungen deutlich, indem es LLMs nutzt, um einen Wissensgraphen aus Rohdokumenten zu erstellen und dann Wissen basierend auf dem Graphen abzurufen, um Antworten zu verbessern. Der Graphenkonstruktion von GraphRAG fehlt jedoch ein spezifisches Design, um die Authentifizierung und Glaubwürdigkeit der Antworten zu gewährleisten, und sein hierarchischer Community-Konstruktionsprozess ist aufgrund seines allgemeinen Charakters kostspielig, was eine direkte und effektive Anwendung im medizinischen Bereich erschwert.
Um dieser Situation zu begegnen, hat ein gemeinsames Team der Universitäten Oxford, Carnegie Mellon und Edinburgh eine graphenbasierte RAG-Methode speziell für den medizinischen Bereich vorgeschlagen – Medical GraphRAG (MedGraphRAG).Diese Methode verbessert die Leistung des LLM im medizinischen Bereich effektiv, indem sie evidenzbasierte Antworten und offizielle Erklärungen zur medizinischen Terminologie generiert, was nicht nur die Glaubwürdigkeit der Antworten erhöht, sondern auch die Gesamtqualität erheblich verbessert.
Die entsprechenden Forschungsergebnisse mit dem Titel „Medical Graph RAG: Towards Safe Medical Large Language Model via Graph Retrieval-Augmented Generation“ wurden für ACL 2025 ausgewählt.
Forschungshighlights:
* In dieser Studie wurde erstmals das Tukey RAG-Framework speziell für den Einsatz im medizinischen Bereich vorgeschlagen.
* In dieser Studie wurde eine einzigartige Dreifachgraphenkonstruktion und eine U-Retrieval-Methode entwickelt, die es dem Large Language Model (LLM) ermöglicht, die gesamten RAG-Daten effizient zu nutzen und beweisbasierte Antworten zu generieren.
* MedGraphRAG übertrifft andere Abrufmethoden und optimierte medizinspezifische große Sprachmodelle bei mehreren Benchmarks zur Beantwortung medizinischer Fragen.
Papieradresse:
Weitere Artikel zu den Grenzen der KI:
Forschung basierend auf drei Arten von Daten
Die in dieser Studie verwendeten Daten sind in drei Kategorien unterteilt, wobei die Merkmale jedes Typs seiner Rolle in der Studie entsprechen:
* RAG-Daten
Da Benutzer häufig aktualisierte private Daten verwenden (wie etwa elektronische Patientenakten), wurde für die Studie der öffentliche elektronische Patientendatensatz MIMIC-IV ausgewählt, der die sich dynamisch ändernden privaten Datenszenarien in tatsächlichen Anwendungen simulieren und eine Grundlage für die Überprüfung der Praktikabilität der Methode bieten kann.
* Repository-Daten
Dieser Datensatz dient dazu, vertrauenswürdige Quellen und maßgebliche Vokabeldefinitionen für die Antworten des großen Modells bereitzustellen. Die übergeordneten Repository-Daten sind MedC-K, das 4,8 Millionen biomedizinische wissenschaftliche Arbeiten, 30.000 Lehrbücher und alle Evidenzpublikationen von FakeHealth und PubHealth enthält. Der Inhalt deckt ein breites Spektrum ab und ist wissenschaftlich maßgeblich; die zugrunde liegenden Repository-Daten sind der UMLS-Graph, der maßgebliches medizinisches Vokabular und semantische Beziehungen enthält, um die Genauigkeit der medizinischen Terminologie sicherzustellen.
* Testdaten
Dieser Datensatz wird verwendet, um die Leistung der MedGraphRAG-Methode zu bewerten, einschließlich des Testteils von 9 biomedizinischen Multiple-Choice-Datensätzen in MultiMedQA (einschließlich MedQA, MedMCQA, PubMedQA, MMLU-klinischen Themen usw.), der verwendet wird, um die Leistung der Methode bei der Beantwortung routinemäßiger medizinischer Fragen zu testen; 2 Datensätze zur Überprüfung von Fakten im Bereich der öffentlichen Gesundheit, FakeHealth und PubHealth, werden verwendet, um die evidenzbasierte Antwortfähigkeit der Methode zu bewerten; außerdem hat die Studie auch den DiverseHealth-Testsatz gesammelt, der 50 echte klinische Fragen enthält, die ein breites Themenspektrum wie seltene Krankheiten und die Gesundheit von Minderheiten abdecken und sich auf Gesundheitsgerechtigkeit konzentrieren, was die Bewertungsdimensionen weiter bereichern kann.
MedGraphRAG: Basierend auf gleitender Fensterpartitionierung, Label-Clustering und U-Suche
Wie in der Abbildung unten gezeigt,Der gesamte Arbeitsablauf von MedGraphRAG umfasst hauptsächlich drei Kernverbindungen:Erstellen Sie einen Wissensgraphen auf der Grundlage von Dokumenten, organisieren und fassen Sie den Graphen zusammen, um das Abrufen zu unterstützen, und beantworten Sie Benutzeranfragen, indem Sie Daten abrufen.
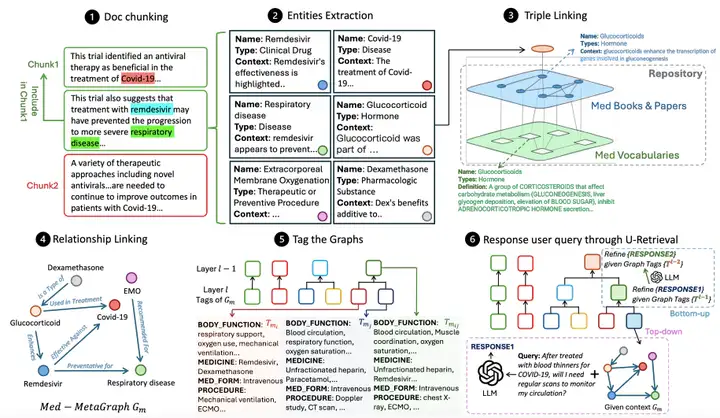
Medical Graph Construction führt zunächst eine semantische Dokument-Chunking-Methode durch, bei der das Dokument in Datenblöcke unterteilt wird, die den LLM-Kontextbeschränkungen entsprechen.Die Studie verwendet eine Hybridmethode, die Zeichentrennung und themenbezogene semantische Segmentierung kombiniert, d. h., zuerst werden Absätze durch Zeilenumbrüche getrennt und dann LLM LG durch das Diagramm erstellt, um die Themenrelevanz zwischen dem Absatz und dem aktuellen Block zu beurteilen und zu entscheiden, ob der Block aufgeteilt werden soll.Gleichzeitig wird ein 5-Segment-Schiebefenster zur Rauschreduzierung eingeführt und die LG-Tag-Einschränkung als harter Schwellenwert für die Blocksegmentierung verwendet, wobei sowohl die semantische Logik als auch die Modellkontextbeschränkungen berücksichtigt werden.
Nach der Blockteilung beginnt der Entitätsextraktionsprozess. Mithilfe von LG mit Eingabeaufforderungen zur Entitätsextraktion werden relevante Entitäten aus jedem Block identifiziert und eine strukturierte Ausgabe mit Name, Typ und Kontextbeschreibung generiert, die den Weg für die anschließende Entitätsverknüpfung ebnet.Dreifache Verknüpfung ist der Schlüssel zur Gewährleistung der Genauigkeit.Ein Repository-Diagramm (RepoGraph) wird erstellt, um RAG-Dokumente des Benutzers mit vertrauenswürdigen Quellen zu verknüpfen: Die unterste Ebene ist das UMLS-Diagramm (Med Vocabularies) mit medizinischem Vokabular und Beziehungen, und die obere Ebene besteht aus medizinischen Lehrbüchern und wissenschaftlichen Artikeln (Med Books & Papers). Als Nächstes definieren die Forscher aus den RAG-Dokumenten extrahierte Entitäten als E1. Basierend auf der Korrelation zwischen den Entitäten werden diese Entitäten mit Entitäten E2 verknüpft, die aus medizinischen Büchern oder Artikeln extrahiert wurden. E2 ist weiterhin mit UMLS-Entitäten E3 verknüpft, wodurch eine dreifache Struktur aus [RAG-Entität, Quelle, Definition] entsteht und sichergestellt wird, dass jede Entität auf eine eindeutige Quelle und Standarddefinition zurückgeführt werden kann. Anschließend wird die Beziehungsverknüpfung durchgeführt. Ein LG mit Hinweisen zur Beziehungserkennung identifiziert Beziehungen zwischen RAG-Entitäten basierend auf Entitätsinhalten und Referenzen und generiert Phrasen, die Quell-Entitäten, Ziel-Entitäten und Beziehungsbeschreibungen enthalten. Schließlich wird für jeden Datenblock ein gerichteter metamedizinischer Graph generiert.
Nachdem das Diagramm erstellt wurde, müssen Sie die Diagramme mit Tags versehen, um die Abrufeffizienz zu verbessern.Anders als GraphRAGs aufwändiger Ansatz zur Erstellung von Graph-Communitys nutzt diese Methode die strukturierte Natur medizinischer Texte und fasst jeden metamedizinischen Graphen mithilfe vordefinierter Bezeichnungen (wie Symptome, Krankengeschichte, körperliche Funktion und Medikamente) zusammen, um eine strukturierte Bezeichnungszusammenfassung zu erstellen. Diese Methode verwendet dann dynamisches, agglomeratives, hierarchisches Clustering mit Schwellenwerten basierend auf Bezeichnungsähnlichkeit, um die Graphen zu gruppieren und eine abstraktere, umfassendere Bezeichnungszusammenfassung zu erstellen. Zunächst wird jeder Graph als unabhängige Gruppe behandelt. Die Bezeichnungsähnlichkeit zwischen Clusterpaaren wird iterativ berechnet, und die 20%-Clusterpaare mit der höchsten Ähnlichkeit werden zusammengeführt, um eine neue Bezeichnungszusammenfassungsebene zu erstellen. Dieser Prozess ist auf 12 Ebenen begrenzt, wodurch ein Gleichgewicht zwischen Genauigkeit und Effizienz erreicht wird.
Die letzte U-Abrufphase erreicht eine effiziente Abfrageantwort, indem sie auf LLM LR reagiert.Zunächst generiert LR eine Beschriftungszusammenfassung für die Benutzerabfrage. Durch präzisen Top-down-Abruf, ausgehend von der Beschriftung der obersten Ebene, gleicht es die ähnlichsten Beschriftungen Schicht für Schicht ab und lokalisiert so den metamedizinischen Zielgraphen. Basierend auf der Einbettungsähnlichkeit zwischen der Abfrage und dem Entitätsinhalt ruft es die höchstrangigen Entitäten und ihre nächsten Triplett-Nachbarn ab und verwendet diese Entitäten und Beziehungen, um eine erste Antwort zu generieren. Anschließend beginnt die Bottom-up-Phase der Antwortverfeinerung. LR passt die Antwort basierend auf der Beschriftungszusammenfassung der vorherigen Ebene an. Dieser Prozess wiederholt sich, bis die Zielebene (normalerweise 4–6 Ebenen) erreicht ist, und generiert schließlich eine Antwort, die globale Kontextwahrnehmung und Abrufeffizienz in Einklang bringt.
MedGraphRAG: Validiert an 6 Modellen und 11 Datensätzen, um SOTA zu erreichen
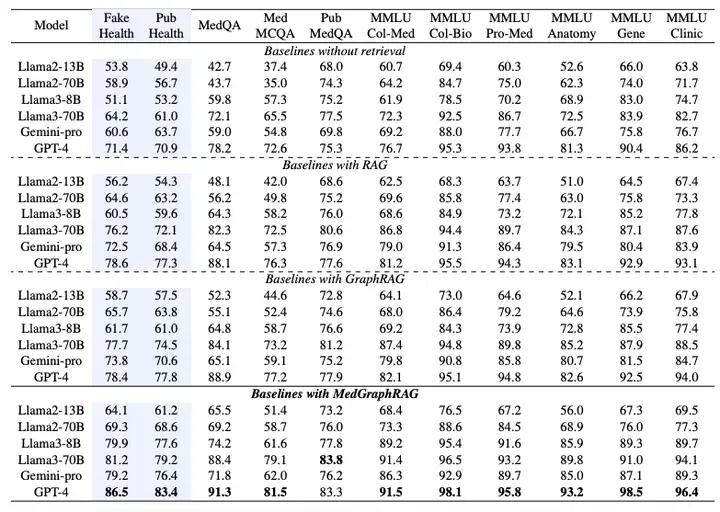
Um die Leistung von MedGraphRAG zu überprüfen, wurden im Rahmen der Studie sechs große Sprachmodelle ausgewählt und mehrere Versuchsreihen entworfen, darunter Llama2 (13B, 70B), Llama3 (8B, 70B), Gemini-pro und GPT-4.Der Hauptvergleich erfolgt mit dem von LangChain implementierten Standard-RAG und dem von Microsoft Azure implementierten GraphRAG.Alle Methoden werden mit denselben RAG-Daten und Testdaten ausgeführt.
Wie in der folgenden Tabelle gezeigt, wird die Leistung der Multiple-Choice-Bewertung anhand der Genauigkeit bei der Auswahl der richtigen Option gemessen.Experimentelle Ergebnisse zeigen, dass MedGraphRAG die Basislinie ohne Abruffunktion, Standard-RAG und GraphRAG deutlich übertrifft:Verglichen mit einem Basiswert ohne Suche erreichte es eine durchschnittliche Verbesserung von fast 101 TP3T bei der Faktenprüfung und 81 TP3T bei der Beantwortung medizinischer Fragen. Verglichen mit GraphRAG erreichte es eine Verbesserung von ungefähr 81 TP3T bei der Faktenprüfung und 51 TP3T bei der Beantwortung medizinischer Fragen. Die Verbesserung war bei kleineren Modellen (wie Llama2 13B) sogar noch signifikanter, was die effektive Integration von Modellschlussfolgerungsfähigkeiten und externem Wissen demonstriert. Bei Anwendung auf größere Modelle (wie Llama70B und GPT-4) erreichte es bei 11 Datensätzen eine hochmoderne Leistung und übertraf sogar auf medizinische Korpora optimierte Modelle wie Med-PaLM 2 und Med-Gemini, wodurch es eine neue hochmoderne Leistung auf der medizinischen LLM-Bestenliste erreichte.
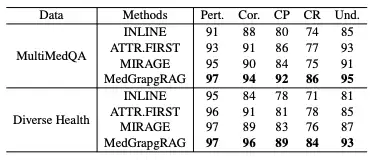
In der Langform-GenerationsbewertungIn dieser Studie wurde MedGraphRAG mit Modellen wie Inline Search und ATTR-FIRST hinsichtlich fünf Dimensionen verglichen: Relevanz, Richtigkeit, Zitationspräzision, Zitationserinnerung und Verständlichkeit anhand der MultiMedQA- und DiverseHealth-Benchmarks.Die Ergebnisse sind in der folgenden Tabelle dargestellt. MedGraphRAG erzielte dank seiner evidenzbasierten Antworten und klaren Erklärungen medizinischer Begriffe in allen Indikatoren bessere Ergebnisse, insbesondere bei der Zitationsgenauigkeit, der Erinnerung und der Verständlichkeit.
In der Fallstudie, die einen komplexen Fall von chronisch obstruktiver Lungenerkrankung (COPD) und Herzinsuffizienz behandelte, ignorierten die Empfehlungen von GraphRAG die Auswirkungen von Medikamenten auf die Herzinsuffizienz, während MedGraphRAG sichere Medikamente empfehlen konnte. Dies lag an der direkten Verknüpfung seiner Entitäten und Referenzen, wodurch das Weglassen wichtiger Informationen, das durch die Informationsverflechtung in GraphRAG entstanden wäre, vermieden wurde.
Integration von Wissensgraphen und großem Sprachmodell
An der Schnittstelle zwischen Medizin und künstlicher Intelligenz wird die Integration von Wissensgraphen und großen Sprachmodellen zu einer Schlüsselrichtung für die Förderung technologischer Durchbrüche und liefert neue Ideen zur Lösung komplexer Probleme im medizinischen Bereich.
Beispielsweise das KG4Diagnosis-Framework, das von einem gemeinsamen Team der Universitäten Cambridge und Oxford vorgeschlagen wurde,Es simuliert reale medizinische Systeme durch eine hierarchische Multiagentenarchitektur und kombiniert Wissensgraphen, um die diagnostischen Argumentationsfähigkeiten zu verbessern und umfasst die automatisierte Diagnose und Behandlungsplanung für 362 häufige Krankheiten.Ein Forschungsteam der Fudan-Universität hat das Proteom der menschlichen Gesundheit und Krankheit umfassend kartiert. Durch die eingehende Analyse der Plasmaproteomdaten von 53.026 Personen über einen durchschnittlichen Nachbeobachtungszeitraum von 14,8 Jahren deckt die Karte 2.920 Plasmaproteine und 406 Vorerkrankungen, 660 während der Nachbeobachtung neu aufgetretene Erkrankungen sowie 986 gesundheitsbezogene Phänotypen ab.Aufdeckung zahlreicher Assoziationen zwischen Proteinen und Krankheiten sowie zwischen Proteinen und Phänotypen,Bereitstellung einer wichtigen Grundlage für die Präzisionsmedizin und die Entwicklung neuer Medikamente.
Das von Google DeepMind eingeführte AMIE-System,Integrieren Sie die Fähigkeiten des großen Gemini-Modells zum Denken in langen Kontexten mit dem Wissensgraphen.Durch die dynamische Suche in klinischen Leitlinien und Medikamentendatenbanken kann ein konsistenter Behandlungsplan für mehrere Diagnosefälle erstellt werden. Beispielsweise können Patienten mit chronisch obstruktiver Lungenerkrankung (COPD) und Herzinsuffizienz kardioselektive Betablocker präzise empfohlen werden, wodurch die Wechselwirkungsrisiken herkömmlicher KI-Systeme vermieden werden.
Der von AstraZeneca erstellte biomedizinische Wissensgraph integriert 3 Millionen Dokumente und interne Forschungsdaten und beschleunigt das Screening neuer Arzneimittelkandidaten durch die Analyse des Arzneimittel-Ziel-Krankheits-Assoziationsnetzwerks.Die Karte umfasst nicht nur die Indikationen zugelassener Medikamente, sondern deckt auch die Daten zum „Off-Label-Use“ in klinischen Studien ab.Bereitstellung von Entscheidungshilfen für die Umwidmung etablierter Medikamente. Darüber hinaus integriert die Knowledge-Graph-Plattform von IBM Watson Health eine Milliarde Patientendaten mit evidenzbasierten Richtlinien, um personalisierte Behandlungspläne für Lungenkrebs zu erstellen, die genetische Tests, die Vorhersage der Medikamentenempfindlichkeit und Nachsorgepläne umfassen. Dadurch wird der prognostizierte Fehler beim Patientenüberleben auf ±2,3 Monate reduziert.
Diese innovativen Verfahren treiben nicht nur die schrittweise Weiterentwicklung der medizinischen KI-Technologie voran, sondern bieten auch enormes Potenzial für die Verbesserung der Diagnosegenauigkeit, die Beschleunigung der Arzneimittelentwicklung und die Optimierung klinischer Entscheidungen. Mit der Weiterentwicklung der Technologie wird die Integration von Wissensgraphen und großen Sprachmodellen Informationsbarrieren im medizinischen Bereich weiter abbauen und der Entwicklung der globalen Gesundheitsversorgung nachhaltige Impulse verleihen.
Referenzartikel:
1.https://mp.weixin.qq.com/s/WhVbnoso2Jf2PyZQwV93Rw
2.https://mp.weixin.qq.com/s/RWy4taiJCu3kMPfTzOWYSQ
3.https://mp.weixin.qq.com/s/lMLk








