Command Palette
Search for a command to run...
Ausgewählt Für AAAI 2025! Um Das Problem Der Weichen Grenzen Und Des Gleichzeitigen Auftretens Bei Der Segmentierung Medizinischer Bilder Zu Lösen, Schlugen Die China University of Geosciences Und Andere Das Bildsegmentierungsmodell ConDSeg vor.
Die Segmentierung medizinischer Bilder ist ein kritischer und komplexer Schritt im Bereich der medizinischen Bildverarbeitung. Es extrahiert hauptsächlich Teile mit besonderer Bedeutung aus medizinischen Bildern, um die klinische Diagnose, Rehabilitationsbehandlung und Krankheitsverfolgung zu unterstützen. In den letzten Jahren haben sich Deep-Learning-basierte Segmentierungsmethoden mit Unterstützung von Computern und künstlicher Intelligenz allmählich zur gängigen Methode der medizinischen Bildsegmentierung entwickelt, und auch die damit verbundenen Ergebnisse sind florierend.
Unter den ausgewählten Ergebnissen, die auf der 39. jährlichen AAAI-Konferenz für künstliche Intelligenz (AAAI 2025), der wichtigsten internationalen Konferenz für künstliche Intelligenz, bekannt gegeben wurden, zeigten einige Beiträge erneut die fruchtbaren Fortschritte bei der automatischen Segmentierung medizinischer Bilder.Eines der Ergebnisse, „ConDSeg: Ein allgemeines Framework zur Segmentierung medizinischer Bilder über kontrastgesteuerte Merkmalsverbesserung“, das gemeinsam von einem Team der China University of Geosciences und Baidu veröffentlicht wurde, erregte große Aufmerksamkeit.
Um die beiden größten Herausforderungen der „weichen Grenzen“ und der Ko-Auftrittsphänomene im Bereich der medizinischen Bildsegmentierung zu bewältigen, schlugen die Forscher ein allgemeines Framework namens ConDSeg für die kontrastgesteuerte medizinische Bildsegmentierung vor. Dieses Framework führt auf innovative Weise die Trainingsstrategie der Konsistenzverstärkung (CR), das Modul zur semantischen Informationsentkopplung (Semantic Information Decoupling, SID), das Modul zur kontrastgesteuerten Merkmalsaggregation (Contrast-Driven Feature Aggregation, CDFA) und den größenbewussten Decoder (Size-Aware Decoder, SA-Decoder) usw. ein, um die Genauigkeit des medizinischen Bildsegmentierungsmodells weiter zu verbessern.
Papieradresse:
https://arxiv.org/abs/2412.08345
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 200 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Die Genauigkeit der Segmentierung medizinischer Bilder steht vor zwei großen Herausforderungen
Im letzten Jahrzehnt hat der Aufstieg der künstlichen Intelligenz zur rasanten Entwicklung der automatischen Segmentierung medizinischer Bilder beigetragen und Ärzte und Forscher von mühsamen Aufgaben befreit. Angesichts der Komplexität und Professionalität medizinischer Bilder ist es jedoch noch ein weiter Weg, bis eine vollständig automatisierte Bildsegmentierung erreicht ist. Dabei stellt die Genauigkeit eine wichtige Herausforderung dar, die nicht ignoriert werden kann, denn wenn die Genauigkeit einmal verloren geht, ist eine Automatisierung nicht mehr möglich.
Aus heutiger SichtDie „weichen Grenzen“ und Koexistenzphänomene in medizinischen Bildern sind die Hauptprobleme, die die Verbesserung der Genauigkeit der Segmentierung medizinischer Bilder behindern.
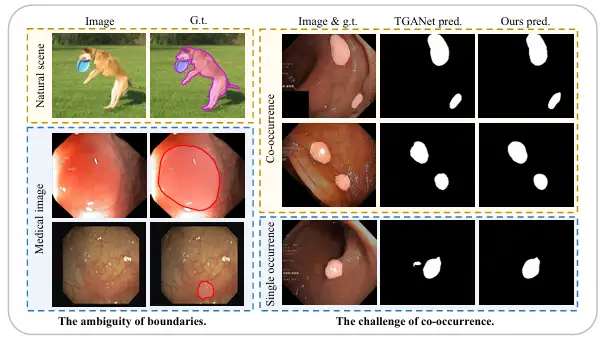
Erstens weisen medizinische Bilder im Vergleich zu natürlichen Bildern mit klaren Grenzen zwischen Vordergrund und Hintergrund häufig eine unscharfe „weiche Grenze“ zwischen Vordergrund (wie Polypen, Drüsen, Läsionen usw.) und Hintergrund auf. Der Hauptgrund hierfür besteht darin, dass es eine Übergangszone zwischen pathologischem Gewebe und umgebendem normalen Gewebe gibt, was die Definition der Grenze erschwert. Darüber hinaus führen die schlechten Lichtverhältnisse und der geringe Kontrast medizinischer Bilder in den meisten Fällen dazu, dass die Grenze zwischen krankhaftem und normalem Gewebe noch weiter verschwimmt, was die Unterscheidung der Grenzen erschwert.
Zweitens sind Organe und Gewebe in medizinischen Bildern im Gegensatz zu Objekten, die in natürlichen Szenen zufällig erscheinen, stark festgelegt und regelmäßig, sodass auch ein weit verbreitetes Koexistenzphänomen vorliegt, d. h., verschiedene Bildmerkmale, Gewebe oder Läsionen erscheinen in medizinischen Bildern gleichzeitig. Beispielsweise erscheinen in endoskopischen Polypenbildern kleine Polypen häufig zusammen mit Polypen ähnlicher Größe, was es dem Modell sehr leicht macht, bestimmte Koexistenzmerkmale zu erlernen, die nicht mit Polypen in Zusammenhang stehen. Wenn jedoch nur pathologisches Gewebe auftritt, kann das Modell häufig keine genauen Vorhersagen treffen.
Um die oben genannten Herausforderungen zu bewältigen, haben sich in den letzten Jahren immer mehr Forschungsmethoden darauf konzentriert. Beispielsweise hat das Team von Associate Professor Yue Guanghui von der School of Biomedical Engineering, School of Medicine, Shenzhen University ein Boundary Constraint Network BCNet veröffentlicht, das für eine genaue Polypensegmentierung verwendet werden kann. Es erwähnt ein bilaterales Grenzextraktionsmodul, das Grenzen erfassen kann, indem es oberflächliche Kontextmerkmale, hochrangige Positionsmerkmale und zusätzliche Polypengrenzüberwachung kombiniert. Dieses Ergebnis wurde im IEEE Journal of Biomedical and Health Informatics unter dem Titel „Boundary constraint network with cross layer feature integration for polyp segmentation“ veröffentlicht.
Papieradresse:
https://ieeexplore.ieee.org/document/9772424
Beispielsweise schlug das Team von Professor Dinggang Shen, Gründungsdekan der School of Biomedical Engineering an der ShanghaiTech University, und anderen ein ebenenübergreifendes Feature-Aggregationsnetzwerk CFA-Net vor, das zur Polypensegmentierung verwendet werden kann. Es entwirft ein Grenzvorhersagenetzwerk zur Generierung grenzbewusster Merkmale und verwendet eine hierarchische Strategie, um diese Merkmale in das Segmentierungsnetzwerk einzufügen. Dieses Ergebnis wurde in Pattern Recognition unter dem Titel „Cross-level Feature Aggregation Network for Polyp Segmentation“ veröffentlicht.
Papieradresse:
https://www.sciencedirect.com/science/article/abs/pii/S0031320323002558
Obwohl diese Methoden alle die Aufmerksamkeit des Modells auf Grenzen verbessern, indem sie explizit eine grenzenbezogene Überwachung einführen, konnten sie die Fähigkeit des Modells, Unsicherheiten in mehrdeutigen Bereichen spontan zu reduzieren, nicht grundlegend verbessern. Daher ist die Robustheit dieser Methoden in rauen Umgebungen immer noch schwach und es gibt immer noch Einschränkungen bei der Verbesserung der Leistung des Modells. Gleichzeitig bleibt die Unfähigkeit, genau zwischen Vordergrund und Hintergrund sowie zwischen verschiedenen Elementen in einem Bild zu unterscheiden, ein Problem, mit dem die meisten Modelle konfrontiert sind.
Anders als bei früheren Methoden,In einer von einem Team der China University of Geosciences und Baidu durchgeführten Studie schlugen Forscher ein allgemeines Framework namens ConDSeg für die kontrastgesteuerte medizinische Bildsegmentierung vor.Die konkreten Neuerungen sind:
* Als Reaktion auf den Robustheitstest in rauen Umgebungen schlugen die Forscher eine Vortrainingsstrategie zur Konsistenzverstärkung (CR) vor, um die Robustheit des Encoders zu verbessern und hochwertige Merkmale zu extrahieren. Gleichzeitig kann das Modul „Semantic Information Decoupling“ (SID) Feature-Maps in Vordergrund-, Hintergrund- und unsichere Bereiche entkoppeln und lernen, die Unsicherheit während des Trainings durch eine speziell entwickelte Verlustfunktion zu reduzieren.
* Das vorgeschlagene Modul „Contrast-Driven Feature Aggregation“ (CDFA) steuert die Fusion und Verbesserung mehrschichtiger Merkmale durch die von SID extrahierten Kontrastmerkmale. Der Size-Aware Decoder (SA-Decoder) zielt darauf ab, verschiedene Objekte in einem Bild besser zu unterscheiden und separate Vorhersagen für Objekte unterschiedlicher Größe zu treffen, um die Interferenz gemeinsamer Merkmale zu überwinden.
Die vier wichtigsten Innovationen von ConDSeg ermöglichen eine verbesserte Genauigkeit der medizinischen Bildsegmentierung
Gesamt,Das in dieser Studie vorgeschlagene ConDSeg ist ein allgemeines Framework zur medizinischen Bildsegmentierung mit einer zweistufigen Architektur.Wie in der folgenden Abbildung dargestellt:
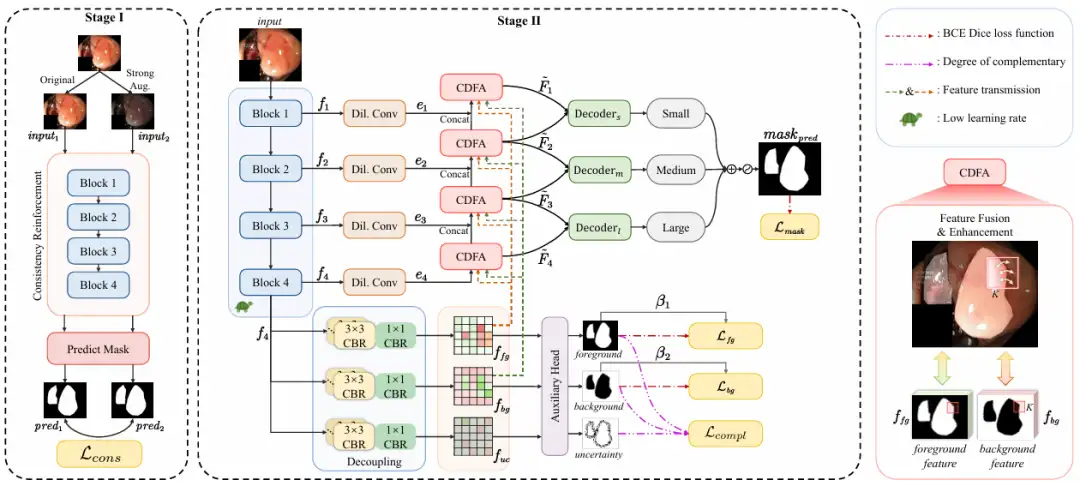
In der ersten PhaseZiel der Forschung ist es, die Fähigkeit zur Merkmalsextraktion und die Robustheit des Encoders in Szenen mit wenig Licht und geringem Kontrast zu maximieren.
Die Forscher führten die CR-Vortrainingsstrategie ein, um ein vorläufiges Training des Encoders durchzuführen, trennten den Encoder vom gesamten Netzwerk und entwarfen einen einfachen Vorhersagekopf (Predict Mask). Durch die Eingabe des Originalbilds (Original) und des verbesserten Bilds (Strong Aug.) in den Encoder wird die Konsistenz zwischen den vorhergesagten Masken maximiert, die Robustheit des Encoders bei unterschiedlichen Licht- und Kontrastanforderungen erhöht und seine Fähigkeit, qualitativ hochwertige Merkmale in rauen Umgebungen zu extrahieren, verbessert. Zu den Verbesserungsmethoden gehören das zufällige Ändern von Helligkeit, Kontrast, Sättigung und Farbton sowie die zufällige Konvertierung in Graustufenbilder und das Hinzufügen einer Gaußschen Unschärfe.
Erwähnenswert ist auch, dass der vom Forschungsteam vorgeschlagene Konsistenzverlust Lcons auf der Klassifizierungsgenauigkeit auf Pixelebene basiert. Es verwendet einfache Binärisierungsoperationen und die Berechnung des Verlusts durch binäre Kreuzentropie (BCE), um die Unterschiede auf Pixelebene zwischen vorhergesagten Masken direkt zu vergleichen. Diese Methode ist rechnerisch einfacher und vermeidet numerische Instabilitäten, sodass sie sich besser für große Datenmengen eignet.
In der zweiten PhaseDas gesamte Netzwerk ist fein abgestimmt und die Lernrate des Encoders ist auf ein niedriges Niveau eingestellt. Es ist in 4 Schritte unterteilt:
* Merkmalsextraktion: Der ResNet-50-Encoder extrahiert Merkmalskarten f₁ bis f₄ mit unterschiedlichen semantischen Informationen auf verschiedenen Ebenen.
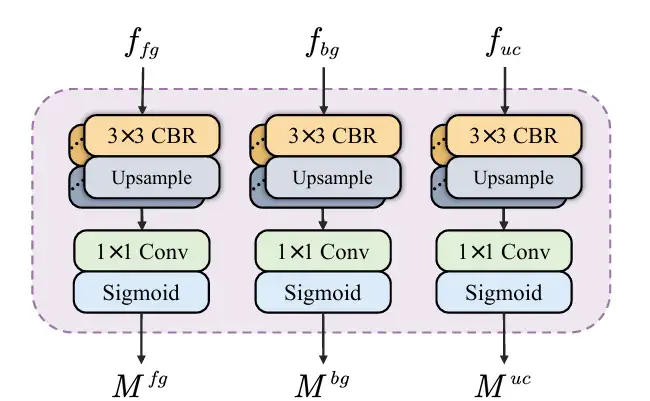
* Entkopplung semantischer Informationen: Die Feature-Map f₄ mit tiefen semantischen Informationen wird in SID eingegeben und in eine Feature-Map entkoppelt, die Informationen zu Vordergrund, Hintergrund und unsicheren Bereichen enthält. SID beginnt mit drei parallelen Zweigen, von denen jeder aus mehreren CBR-Modulen besteht. Nachdem die Merkmalskarte f₄ in die drei Zweige eingegeben wurde, werden drei Merkmalskarten mit unterschiedlichen semantischen Informationen erhalten, die jeweils mit Merkmalen für Vordergrund, Hintergrund und unsichere Bereiche angereichert sind. Anschließend prognostiziert ein Hilfskopf die drei Merkmalskarten und generiert Masken für den Vordergrund, den Hintergrund und den unsicheren Bereich. Durch die Einschränkungen der Verlustfunktion reduziert SID-Lernen die Unsicherheit und verbessert die Maskengenauigkeit zwischen Vordergrund und Hintergrund. Wie in der folgenden Abbildung dargestellt:
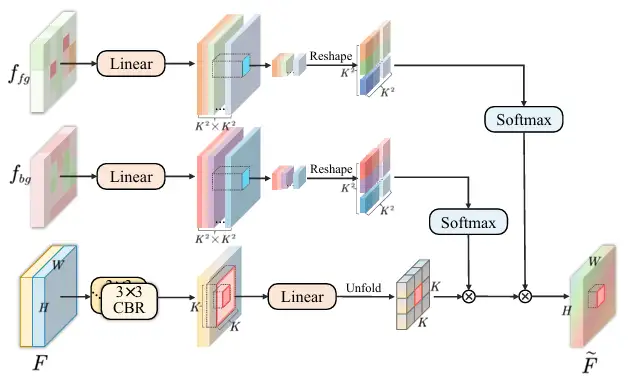
* Feature-Aggregation: Die Feature-Maps f₁ bis f₄ werden in das CDFA-Modul eingespeist, und mehrstufige Feature-Maps werden basierend auf den entkoppelten Feature-Maps schrittweise zusammengeführt, um die Darstellung von Vordergrund- und Hintergrund-Features zu verbessern. CDFA verwendet nicht nur die durch SID entkoppelten Kontrastmerkmale von Vordergrund und Hintergrund, um die mehrstufige Merkmalsfusion zu steuern, sondern hilft dem Modell auch dabei, besser zwischen den zu segmentierenden Entitäten und der komplexen Hintergrundumgebung zu unterscheiden. Wie in der folgenden Abbildung dargestellt:
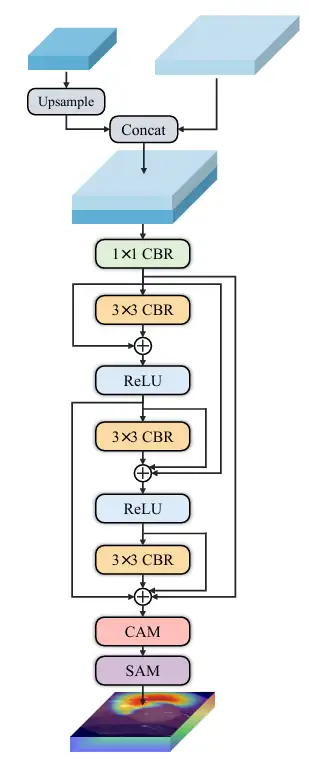
* Multiskalenvorhersage: Die Forscher haben drei Decoder kleiner, mittlerer und großer Größe eingerichtet – Decoder ₛ, Decoder ₘ und Decoder ₗ empfangen jeweils die Ausgabe von CDFA auf einer bestimmten Ebene und lokalisieren dann entsprechend der Größe mehrere Entitäten im Bild. Die Ausgabe jedes Decoders wird fusioniert, um die endgültige Maske zu erzeugen, sodass das Modell große Entitäten genau segmentieren und kleine Entitäten genau lokalisieren kann. Dadurch wird verhindert, dass Ko-Auftrittsphänomene falsch gelernt werden und das Skalensingularitätsproblem des Decoders gelöst. Wie in der folgenden Abbildung dargestellt:
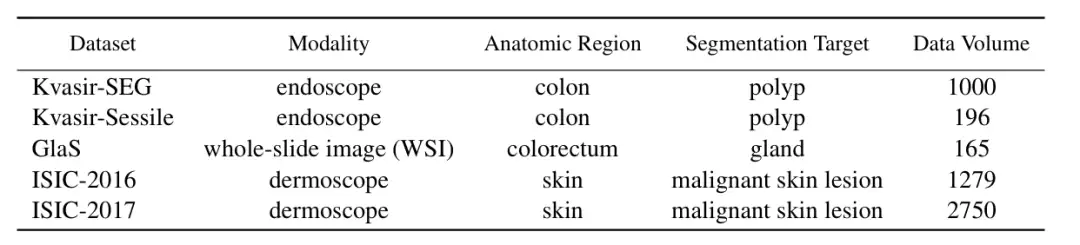
Um die Leistungsfähigkeit von ConDSeg im Bereich der medizinischen Bildsegmentierung zu überprüfen,Die Forscher wählten fünf öffentliche Datensätze (Kvasir-SEG, Kvasir-Sessile, GlaS, ISIC-2016, ISIC-2017, wie in der Abbildung unten dargestellt) aus, um drei medizinische Bildgebungsaufgaben (Endoskopie, Ganzschichtbilder und Dermatoskopie) zu testen. Die Forscher skalierten die Bilder auf 256 × 256 Pixel und legten die Batchgröße auf 4 fest. Zur Optimierung wurde der Adam-Optimierer verwendet.
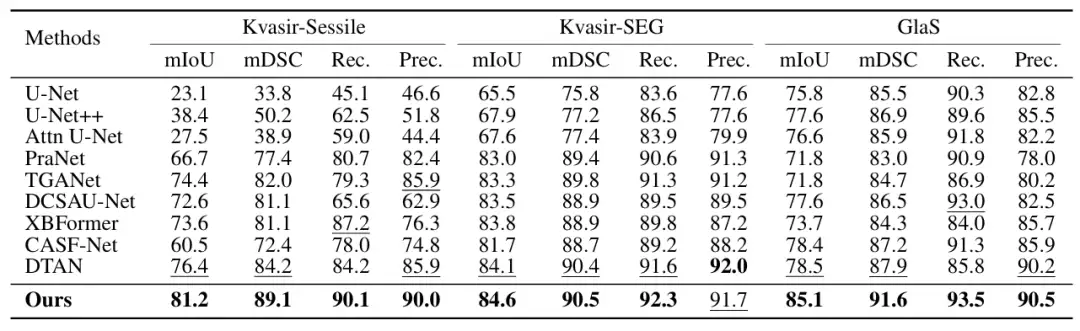
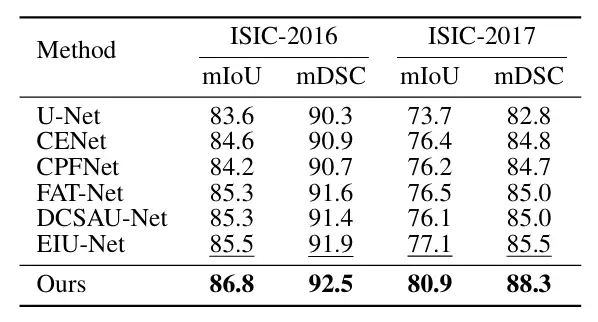
Zu den wichtigsten Vergleichsobjekten gehören die fortschrittlichsten Methoden wie U-Net, U-Net++, Attn U-Net, CENet, CPFNet, PraNet, FATNet, TGANet, DCSAUNet, XBoundFormer, CASF-Net, EIU-Net und DTAN.Die Ergebnisse zeigen, dass die vorgeschlagene Methode bei allen fünf Datensätzen die beste Segmentierungsleistung erzielt.Wie in der folgenden Abbildung dargestellt:
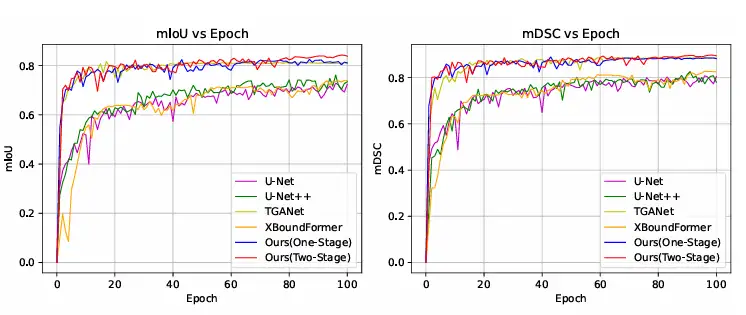
Darüber hinaus verglichen die Forscher die Trainingskonvergenzkurven mit anderen Methoden auf dem Kvasir-SEG-Datensatz. Die Ergebnisse zeigten, dass ConDSeg bereits mit einer einzigen Trainingsstufe fortgeschrittene Stufen erreichen kann und dass diese Methode bei Verwendung des vollständigen ConDSeg-Frameworks die schnellste Konvergenzgeschwindigkeit und die beste Leistung erzielte. Wie in der Abbildung unten gezeigt.
Die Segmentierung medizinischer Bilder ist zu einem heißen Thema für Kapital und Technologie geworden
Die Segmentierung medizinischer Bilder spielt sowohl in der klinischen Medizin als auch in der medizinischen Forschung eine wichtige Rolle. Speziell trainierte KI-Systeme haben mit ihrer hohen Effizienz und Intelligenz traditionelle medizinische Bildsegmentierungsmethoden revolutioniert und sie zu einem unverzichtbaren Hilfsmittel für medizinisches Personal und wissenschaftliche Forscher gemacht. Der Grund für die Entwicklung und die Ergebnisse der Segmentierung medizinischer Bilder liegt in der doppelten Kraft von Kapital und Technologie.
In Bezug auf Kapital ist das interdisziplinäre Feld der KI und Biomedizin in den letzten Jahren zu einem heißen Thema in der Investment-Community geworden, und in diesem Jahr hat die KI-gesteuerte medizinische Bildgebung die Führung übernommen und einen erfolgreichen Start hingelegt. Am 28. Januar gab das spanische Unternehmen für medizinische Bildgebung Quibim bekannt, dass es eine Serie-A-Finanzierung in Höhe von 50 Millionen US-Dollar (ca. 360 Millionen RMB) abgeschlossen habe. Erwähnenswert ist, dass die Kerntechnologie von Quibim auf der Analyse künstlicher Intelligenz auf der Grundlage medizinischer Bilddaten basiert und dass es sich bei QP-Liver um ein automatisiertes Segmentierungstool für die MR-Diagnose diffuser Lebererkrankungen handelt.
Technologisch gesehen ist die Kombination von KI und medizinischer Bildsegmentierung seit langem einer der Forschungsschwerpunkte großer Labore. So hat beispielsweise ein Team des Computer Science and Artificial Intelligence Laboratory (MIT CSAIL) des Massachusetts Institute of Technology in Zusammenarbeit mit Forschern des Massachusetts General Hospital und der Harvard Medical School ein allgemeines Modell für die interaktive biomedizinische Bildsegmentierung namens ScribblePrompt vorgeschlagen, das Annotatoren mithilfe verschiedener Annotationsmethoden wie Graffiti, Klicks und Begrenzungsrahmen dabei unterstützt, biomedizinische Bildsegmentierungsaufgaben flexibel durchzuführen, selbst für ungeschulte Beschriftungen und Bildtypen.
Die entsprechenden Ergebnisse mit dem Titel „ScribblePrompt: Schnelle und flexible interaktive Segmentierung für jedes biomedizinische Bild“ wurden von der führenden internationalen akademischen Konferenz ECCV 2024 angenommen.
Papieradresse:
https://arxiv.org/pdf/2312.07381
Darüber hinaus entwickelte das Team der Universität Oxford auf Grundlage von SAM 2, das von Meta veröffentlicht wurde, ein medizinisches Bildsegmentierungsmodell namens Medical SAM 2 (MedSAM-2), das medizinische Bilder wie Videos behandelt. Es eignet sich nicht nur gut für die Segmentierung medizinischer 3D-Bilder, sondern bietet auch eine neue Funktion zur Einzeleingabeaufforderungssegmentierung. Der Benutzer muss lediglich einen Hinweis für ein neues bestimmtes Objekt geben, und die Segmentierung ähnlicher Objekte in nachfolgenden Bildern kann vom Modell automatisch und ohne weitere Eingaben abgeschlossen werden.
Kurz gesagt: KI ist keine High-End-Technologie mehr. Die Entwicklung der automatischen Segmentierung medizinischer Bilder hat das Potenzial der KI im biomedizinischen Bereich bestätigt und ihre kommerzielle Machbarkeit wurde auch durch eine Kapitalgeschichte nach der anderen verifiziert. Als wichtigstes Bindeglied im Bereich der medizinischen Bildgebung wird die Segmentierung medizinischer Bilder in Zukunft sicherlich von der KI profitieren und sich auf der Hochbahn der Entwicklung befinden. Aufgrund der Erfolge im Bereich der Segmentierung medizinischer Bilder wird auch Kapital in den breiteren biomedizinischen Markt fließen, wodurch ein perfekter geschlossener Kreislauf aus Technologie, Kapital und Geschäft entsteht.








