Command Palette
Search for a command to run...
内含教程|MIT 等推出 BindCraft,直接调用 AF2,实现蛋白质结合体的智能化设计

在生命体系中,蛋白质很少单独发挥其生物学功能,而是依赖于蛋白质-蛋白质相互作用(PPI)来执行复杂的生物学过程。因此,设计能够特异性靶向和调控 PPI 的蛋白质结合物具有巨大的治疗和生物技术潜力。然而,传统的蛋白质结合物生成方法,例如免疫接种、抗体库筛选或定向进化,通常费力费时,并且对靶位的控制有限。
计算蛋白质设计提供了一种强大的替代方案,能够根据特定靶标和结合位点定制结合物。而早期计算方法(如 Rosetta)虽然尝试通过物理建模与侧链优化设计结合界面,但成功率往往不足 0.1% 。进入深度学习时代后,AlphaFold2 等结构预测模型彻底改变了这一局面。它不仅能精准预测单个蛋白的三维折叠结构,还能模拟蛋白复合物的空间互作方式。
但这仍非真正意义上的「智能设计」。现有方法,如 RFdiffusion 或 ProteinMPNN,依然需要人工设定骨架、对接界面,再用 AlphaFold 来验证是否合理。那么,既然 AlphaFold 已经能理解蛋白结构,那能否让它自己反向「思考」,直接生成一个与目标完美契合的新蛋白?
基于此,瑞士洛桑联邦理工学院(EPFL)与麻省理工学院(MIT)的团队提出了一个用于从头设计蛋白质结合物的开源自动化流程 BindCraft,其核心思想是通过 AlphaFold2 权重反向传播幻觉结合剂序列并计算误差梯度。研究团队将 AlphaFold2 的多体版本与神经网络相结合,通过梯度优化同时生成蛋白的结构、序列与界面。无需高通量筛选、实验迭代,甚至无需已知结合位点,即可在硅片中生成纳摩尔级亲和力的从头结合剂。
实验结果显示,BindCraft 在 12 个结构复杂、药理学意义重大的目标上均取得突破性成果,实验成功率涵盖 10%–100%,平均成功率达 46.3% 。这意味着,以往需要数百甚至上千次筛选的设计工作,如今只需一次计算即可获得可用的结合体。
目前,「BindCraft:蛋白质粘合剂设计」已上线至 HyperAI 超神经官网的「教程」板块,点击下方链接即可体验一键部署教程 ⬇️
教程链接:
Demo 运行
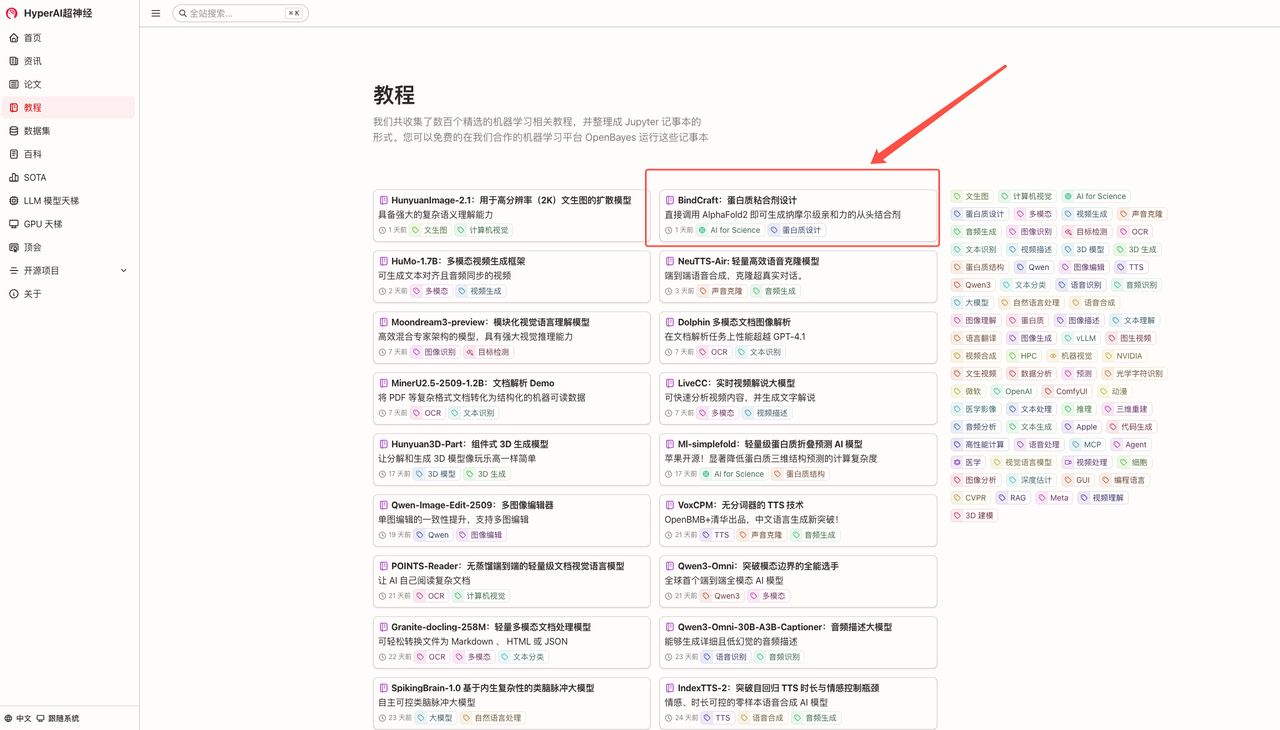
1. 进入 hyper.ai 首页后,选择「教程」页面,并选择「BindCraft:蛋白质粘合剂设计」,点击「在线运行此教程」。
2. 页面跳转后,点击右上角「克隆」,将该教程克隆至自己的容器中。
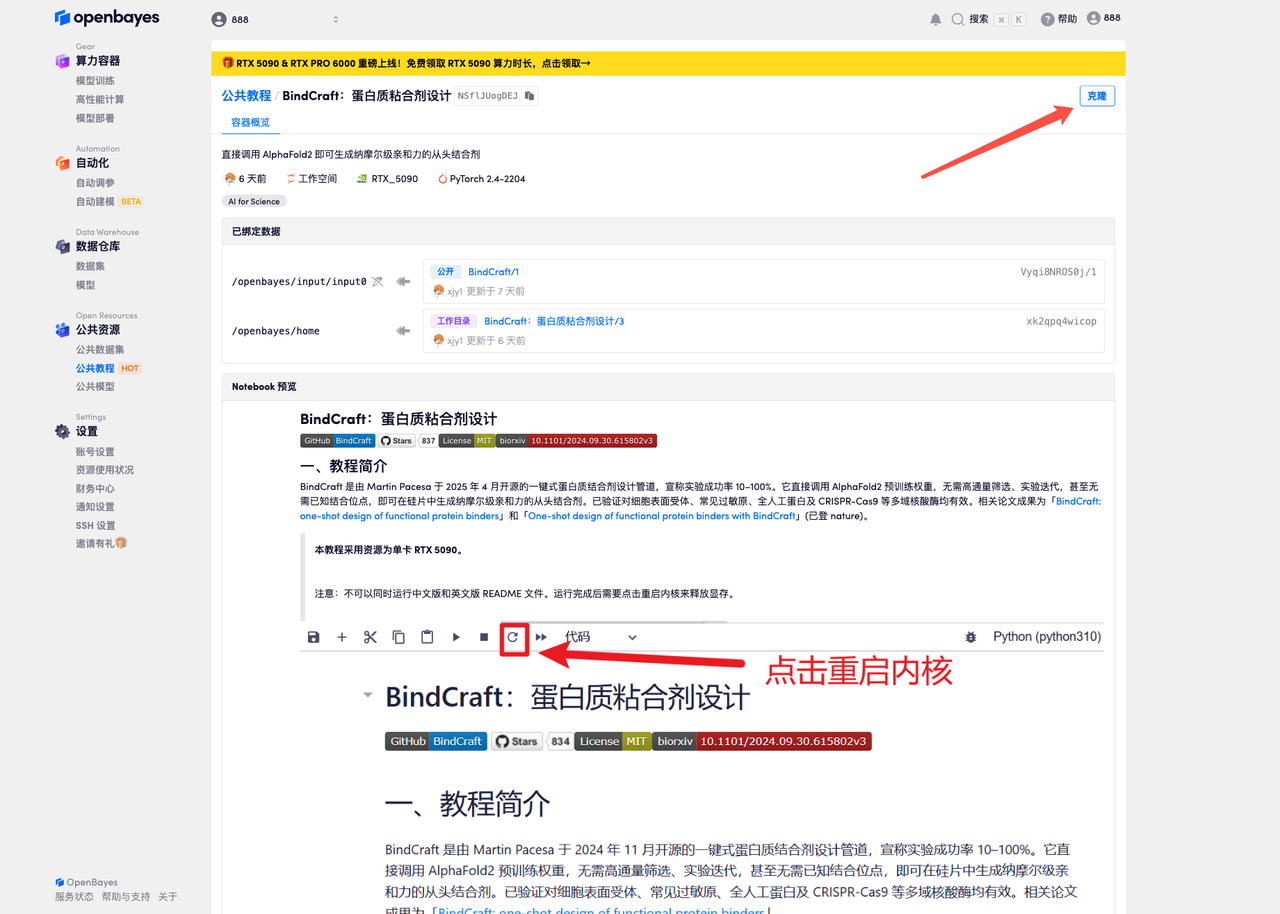
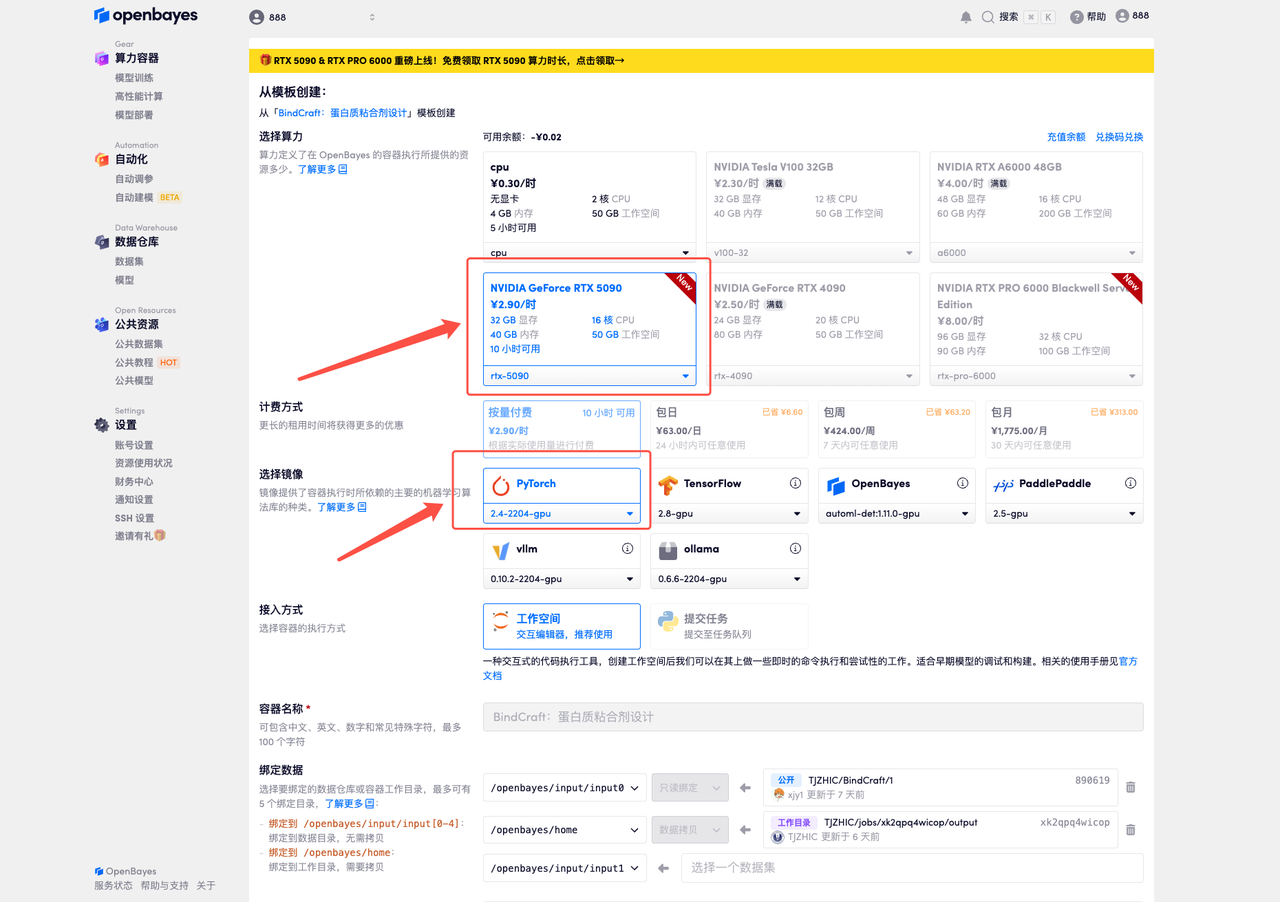
3. 选择「NVIDIA GeForce RTX 5090」以及「PyTorch」镜像,并点击「继续执行」。 OpenBayes 平台提供了 4 种计费方式,大家可以按照需求选择「按量付费」或「包日/周/月」。新用户使用下方邀请链接注册,可获得 4 小时 RTX 5090 + 5 小时 CPU 的免费时长!
HyperAI 超神经专属邀请链接(直接复制到浏览器打开):
https://openbayes.com/console/signup?r=Ada0322_NR0n
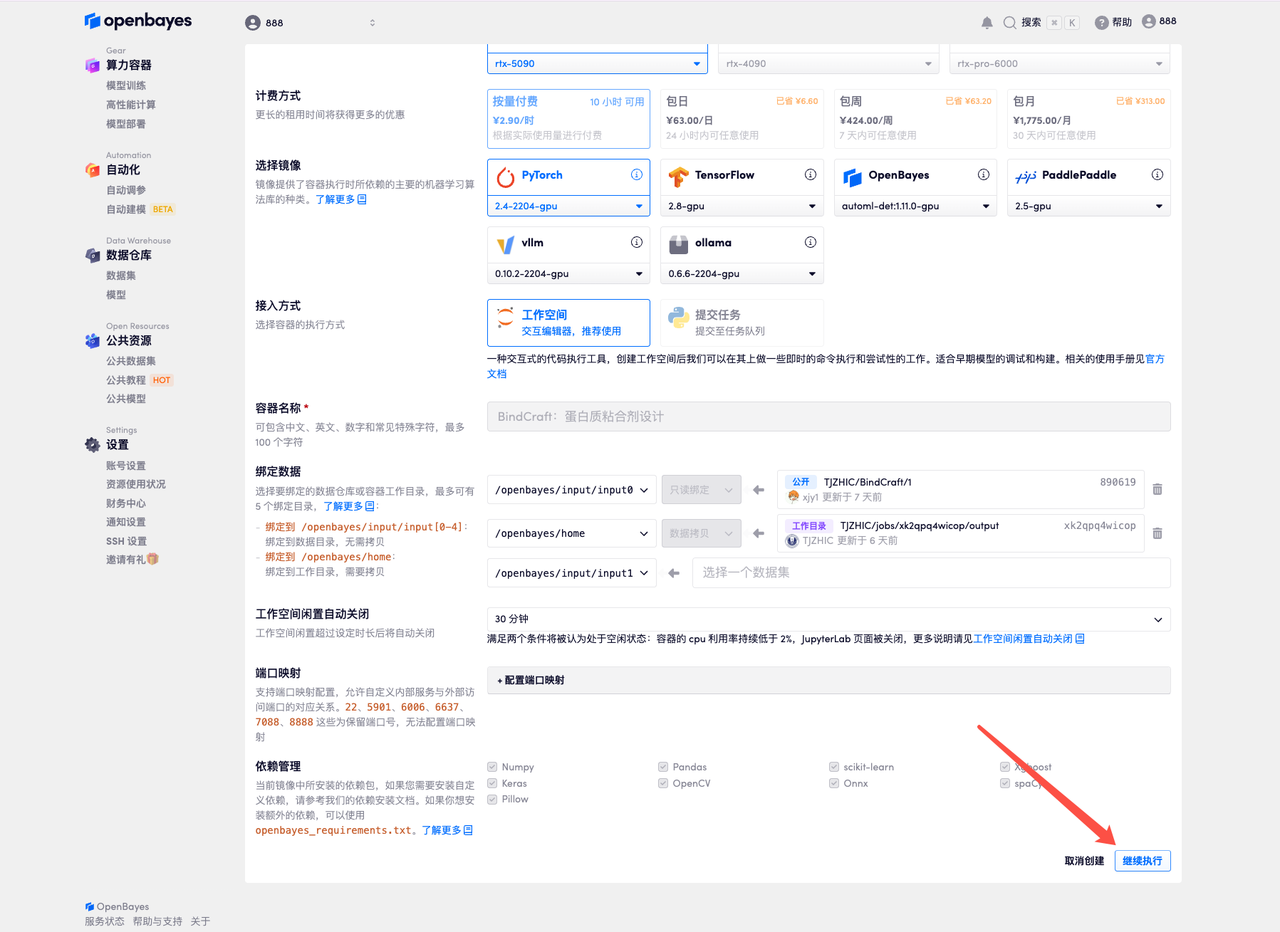
4. 等待分配资源,首次克隆需等待约 2 分钟左右的时间。当状态变为「运行中」后,点击「打开工作空间」,即可跳转至 Demo 页面。
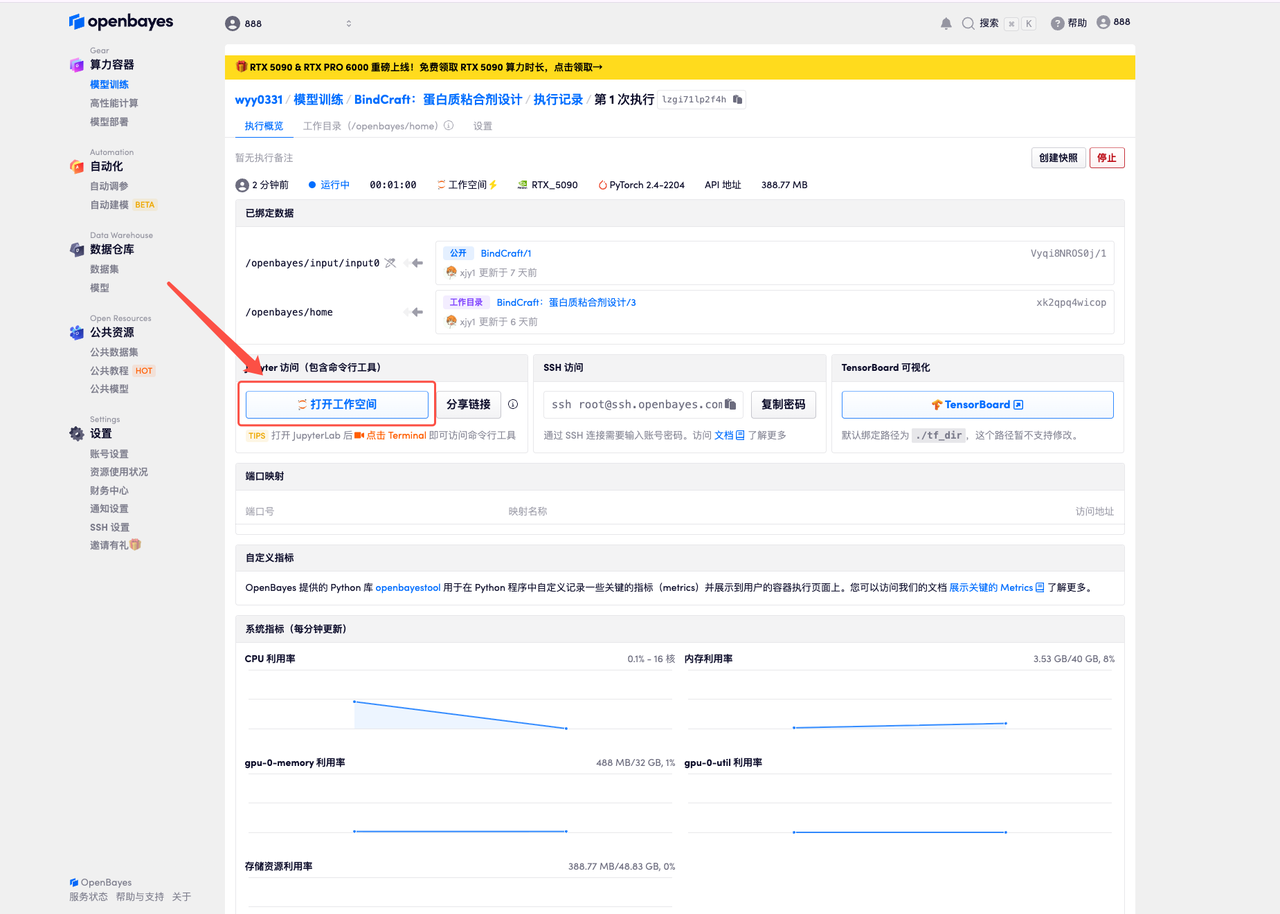
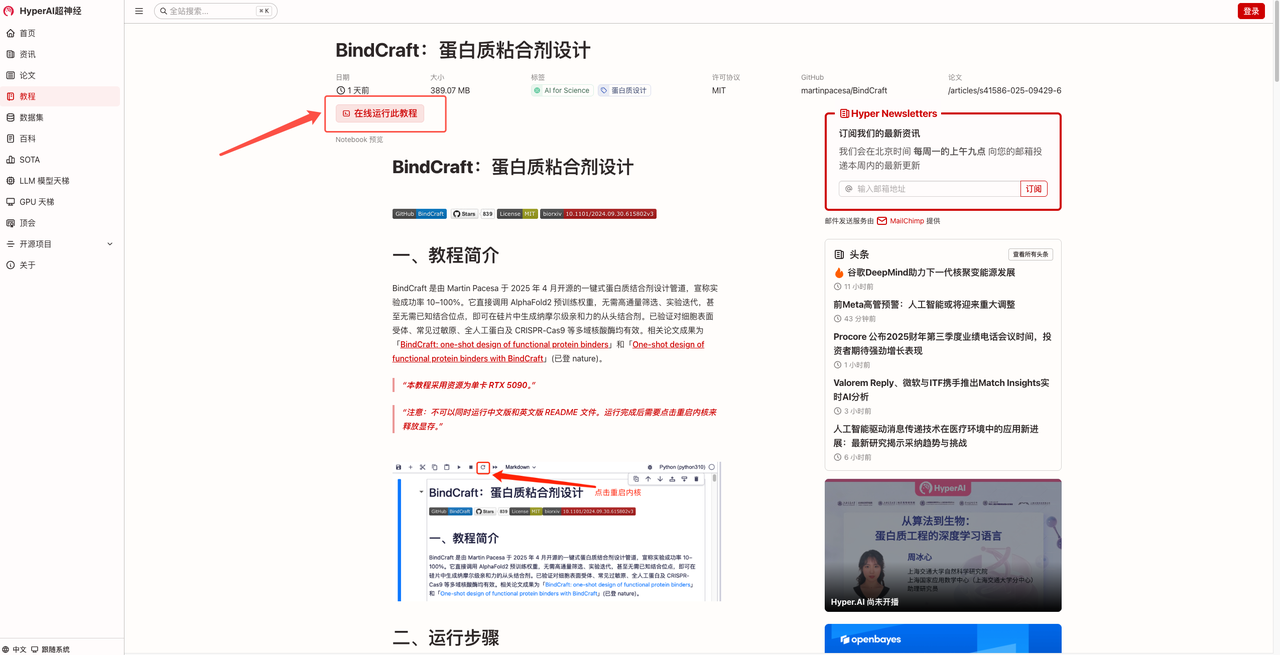
5. 以下为 BindCraft 使用页面,点击「README」即可跳转至生成界面。
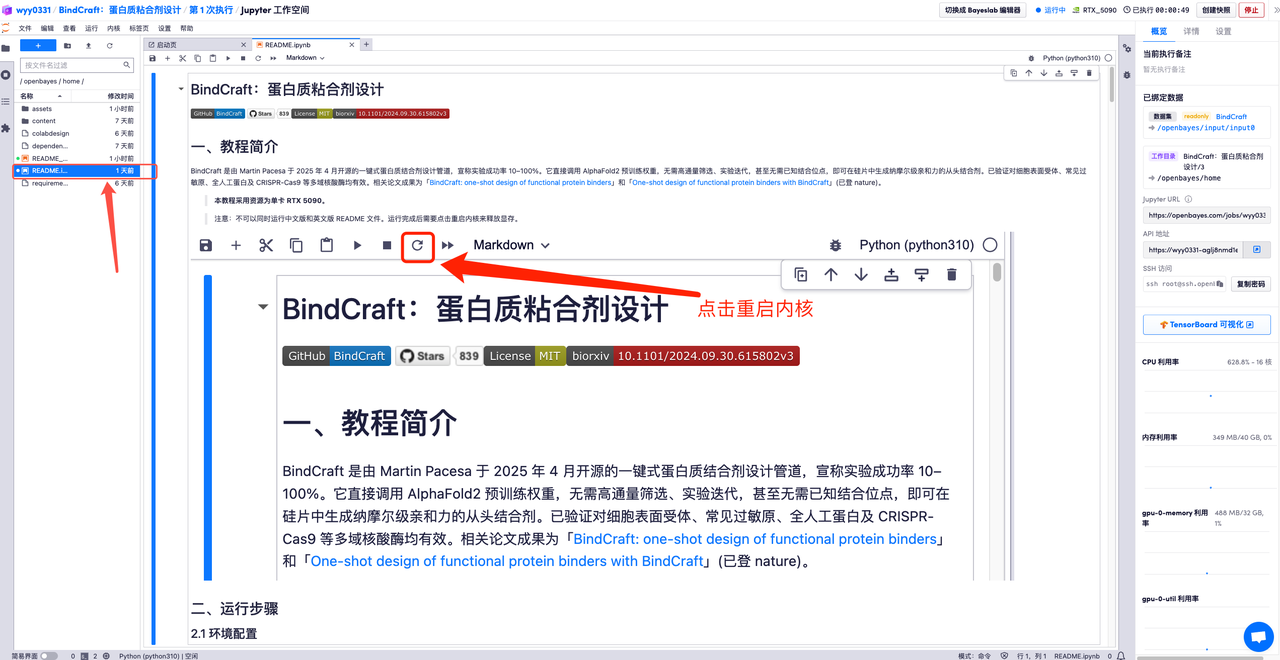
效果演示
1. 请根据 README 参数说明完成相关参数设置
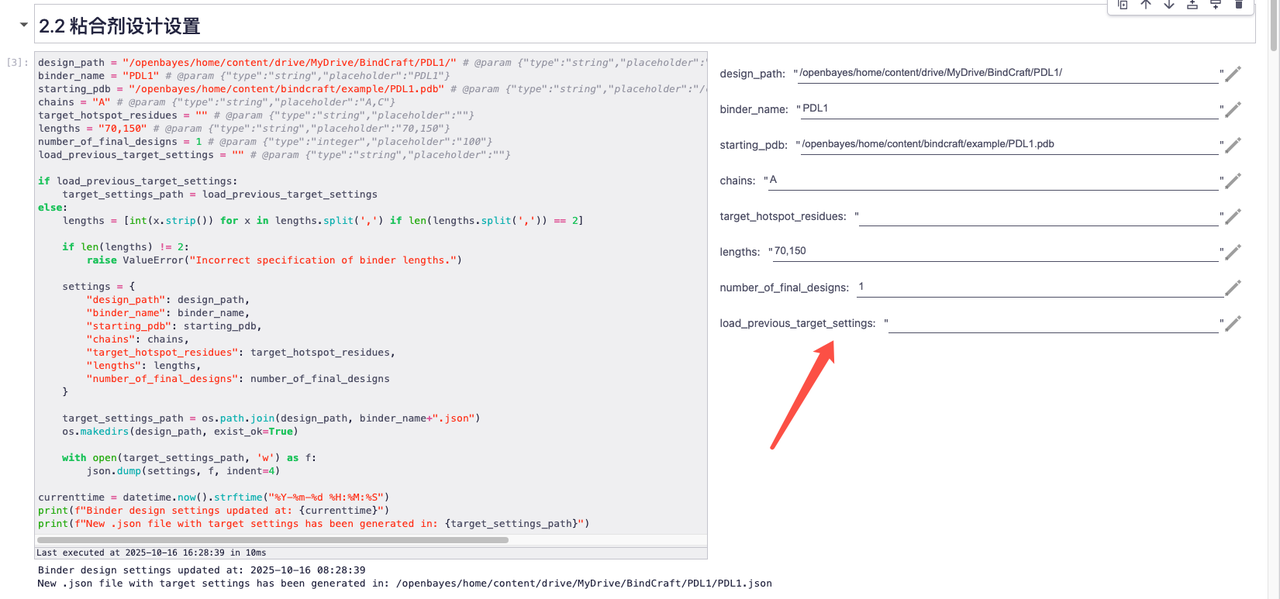
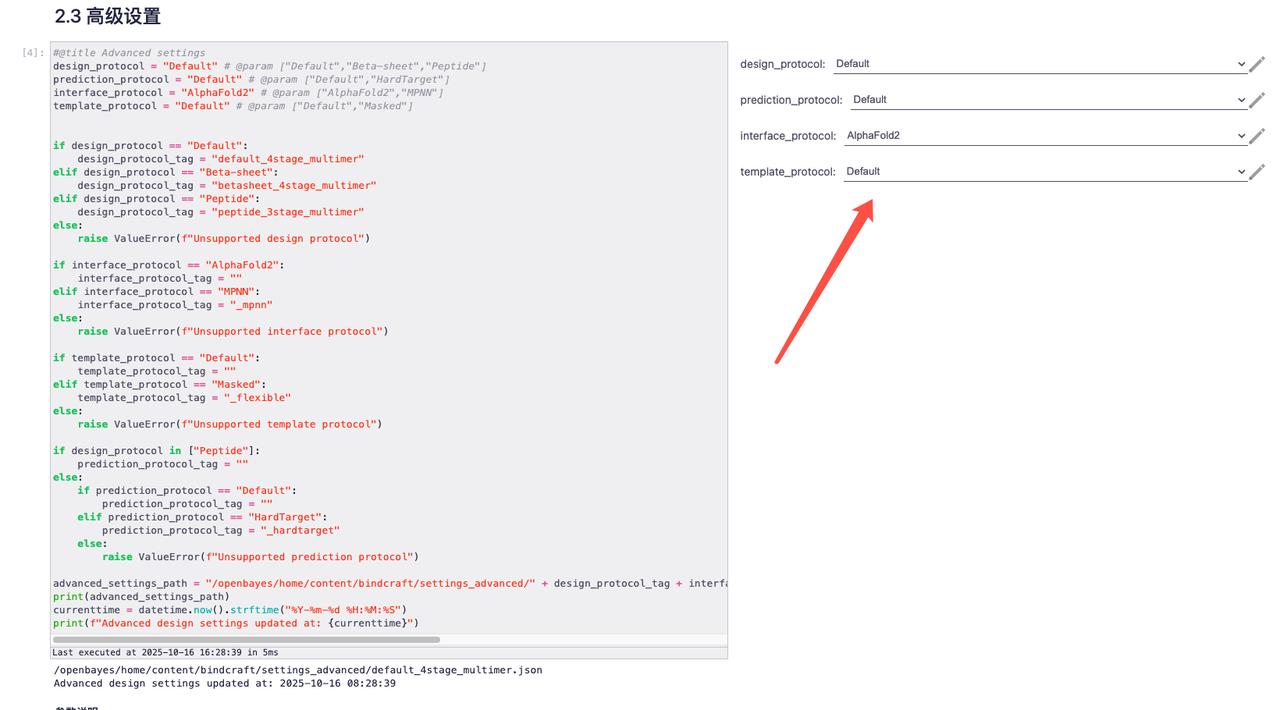
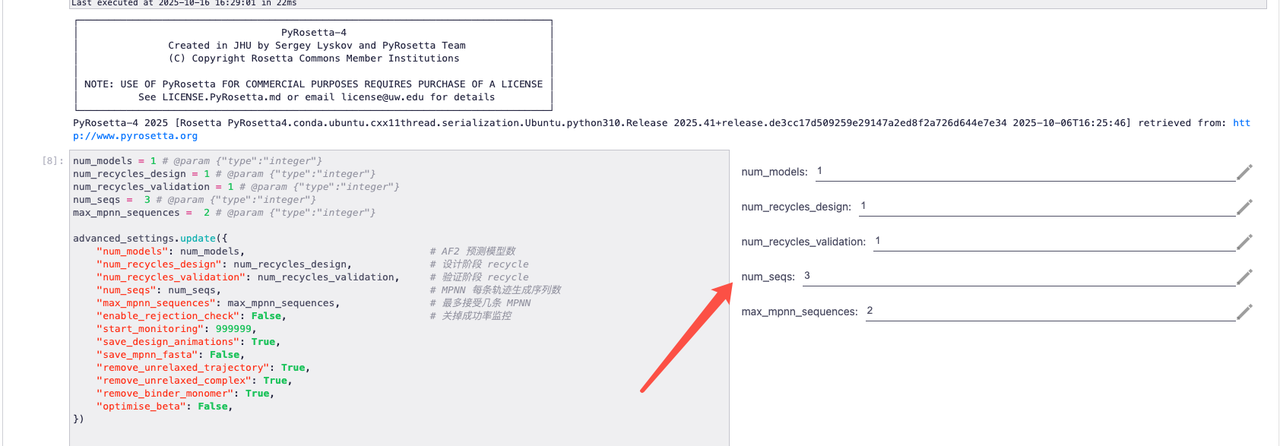
2. 以默认参数为例,相关效果如下所示
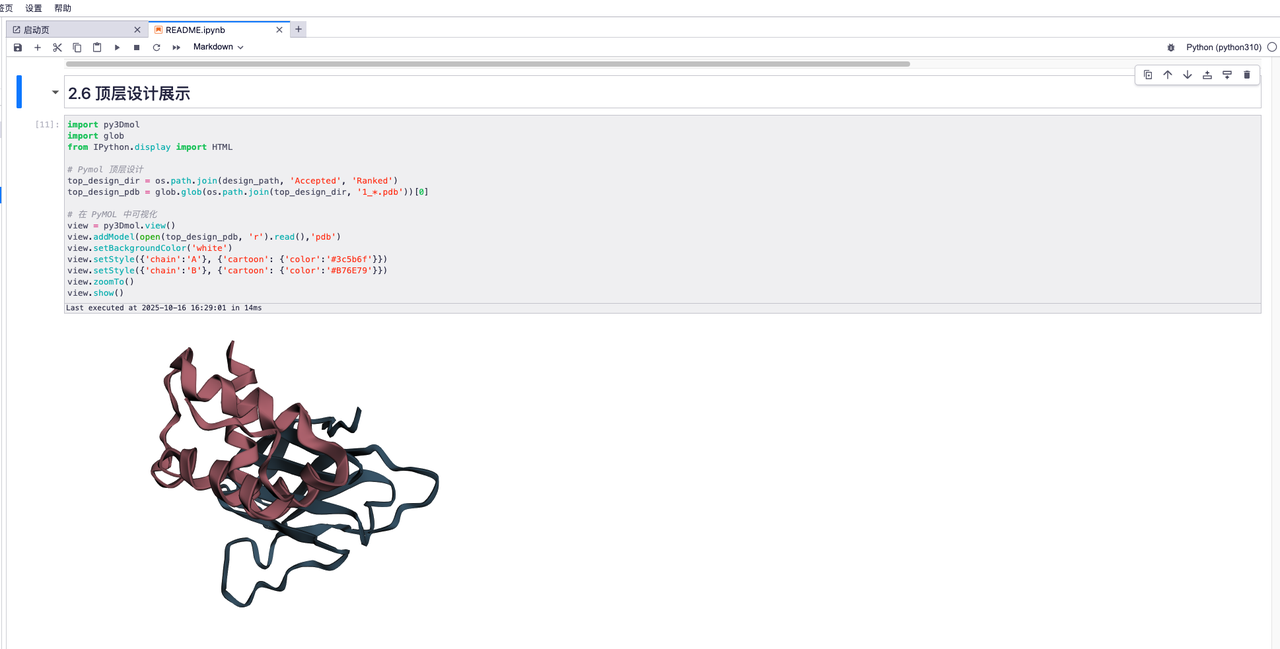
「顶层设计」效果,如下图:
「显示 animationy」效果,请看下方视频:
以上就是 HyperAI 超神经本期推荐的教程,欢迎大家前来体验!
教程链接: