Command Palette
Search for a command to run...
قام فريق جامعة طوكيو بتطوير إطار عمل التعلم العميق STAIG للقضاء على تأثيرات الدفعة دون محاذاة مسبقة، مما يكشف عن معلومات وراثية مفصلة في بيئة الورم.

الأنسجة البيولوجية عبارة عن شبكات معقدة مكونة من أنواع متعددة من الخلايا تؤدي وظائف مهمة من خلال تكوينات مكانية محددة. في السنوات الأخيرة، مكنت التطورات في تكنولوجيات النسخ المكاني مثل 10x Visium وSlide-seq وStereio-seq وSTARmap علماء الأحياء من رسم خرائط للبيانات الجينية داخل الهياكل المكانية، مما يوفر رؤى أعمق لمختلف الأمراض.
ومع ذلك، تعتمد تقنيات النقل التسلسلي بشكل كبير على تحديد المناطق المكانية ذات التعبير الجيني الموحد والسمات النسيجية. في الوقت الحالي،هناك طريقتان رئيسيتان للتعريف: التجميع غير المكاني وطرق التجميع المكانيتؤدي طرق التجميع غير المكاني التجميع بناءً على التعبير الجيني فقط، مما يؤدي غالبًا إلى نتائج تجميع غير متماسكة؛ تستخدم طرق التجميع المكاني نماذج التفاف الرسم البياني لدمج المعلومات الجينية والمكانية، ولكنها تعتمد على معايير المسافة المحددة بشكل مصطنع عند تحويل بيانات ST إلى هياكل بيانية، مما قد يؤدي إلى التحيز؛ وفي الوقت نفسه، تواجه الطرق التي تستخدم الصور النسيجية أيضًا تحديات لأنها عرضة للتغيرات في جودة التلوين؛ بالإضافة إلى ذلك، لا يزال التكامل الدفعي لمعظم الطرق الحالية يتطلب تدخلاً يدويًا، مثل المحاذاة اليدوية للإحداثيات أو الاعتماد على أدوات إضافية.
وللتغلب على هذه التحديات،اقترح فريق بحثي من معهد العلوم الطبية بجامعة طوكيو في اليابان إطار عمل للتعلم العميق يسمى STAIG (تحليل النسخ المكاني مع التعلم المقارن بالرسوم البيانية بمساعدة الصور).القدرة على دمج التعبير الجيني والبيانات المكانية والصور النسيجية دون محاذاة.
يقوم STAIG باستخراج الميزات من الصور الملطخة بالهيماتوكسيلين والإيوزين (H&E) من خلال نموذج خاضع للإشراف الذاتي دون الاعتماد على مجموعات بيانات علم الأنسجة واسعة النطاق للتدريب المسبق. بالإضافة إلى ذلك، يقوم STAIG بضبط بنية الرسم البياني بشكل ديناميكي أثناء التدريب ويستخدم معلومات الصورة النسيجية لاستبعاد العينات السلبية المتجانسة بشكل انتقائي، وبالتالي تقليل التحيز الناتج عن البناء الأولي.
أخيرًا، يحدد STAIG نقاط التشابه في التعبير الجيني من خلال المقارنة المحلية، مما يتيح تكامل الدفعة من البداية إلى النهاية دون الحاجة إلى محاذاة إحداثيات يدوية وتقليل تأثيرات الدفعة بشكل فعال. قام الباحثون بتقييم STAIG على مجموعات بيانات متعددة.وأظهرت النتائج أن لديها أداء جيدًا في تحديد المنطقة المكانية ويمكنها الكشف عن معلومات مكانية وجينية مفصلة في البيئة الدقيقة للورم، مما يعزز فهم الأنظمة البيولوجية المعقدة.
وقد نُشرت النتائج ذات الصلة في مجلة Nature Communications تحت عنوان "STAIG: تحليل النسخ المكاني عبر التعلم التبايني بمساعدة الصور لاستكشاف المجال والتكامل الخالي من المحاذاة".
أبرز الأبحاث:
* يتيح نموذج STAIG دمج أقسام الأنسجة دون محاذاة مسبقة ويزيل تأثيرات الدفعة
* نموذج STAIG قابل للتطبيق على البيانات المكتسبة من منصات مختلفة، سواء تم تضمين صور الأنسجة أم لا
* أظهر الباحثون أن STAIG يمكنه تحديد المناطق المكانية بدقة عالية وكشف رؤى جديدة حول البيئة الدقيقة للورم، مما يدل على إمكاناته الواسعة في تشريح التعقيد البيولوجي المكاني

عنوان الورقة:
https://www.nature.com/articles/s41467-025-56276-0
تنزيل عنوان مجموعة البيانات المستخدمة في هذه الدراسة:
https://go.hyper.ai/m5YC4
مجموعة البيانات: مجموعة من مجموعات بيانات ST وصور الأنسجة من منصات مختلفة
قام الباحثون بتنزيل مجموعات بيانات ST وصور الأنسجة المتاحة للجمهور من منصات مختلفة.كما هو موضح في الشكل أدناه. تتضمن مجموعة بيانات ST مجموعة بيانات القشرة الجبهية الظهرانية البشرية (DLPFC)، ومجموعة بيانات سرطان الثدي البشري، ومجموعة بيانات دماغ الفأر، ومجموعة بيانات Slide-seqV2، ومجموعة بيانات STARmap، وما إلى ذلك.
عنوان تنزيل مجموعة البيانات:
https://go.hyper.ai/m5YC4
* تحتوي مجموعة بيانات قشرة الفص الجبهي الظهراني البشري (DLPFC) من منصة 10x Visium على 12 شريحة من 3 أفراد، توفر كل منها 4 شرائح بفاصل 10 ميكرومتر و300 ميكرومتر، ويتراوح عدد النقاط في كل شريحة من 3498 إلى 4789. وقد تم شرح هذه الشرائح يدويًا على أنها طبقات قشرية L1–L6 والمادة البيضاء (WM)؛
* تحتوي مجموعة بيانات سرطان الثدي البشري على 3798 نقطة؛
* تتضمن مجموعة بيانات دماغ الفأر شريحتين، أمامية وخلفية، تحتويان على 2695 و3355 نقطة على التوالي؛
* بالنسبة لسرطان الجلد في سمكة الزرد، قام الباحثون بتحليل القسمين أ و ب، اللذين يحتويان على 2179 و 2677 بقعة على التوالي؛
* بالنسبة للتجربة الجماعية، تم استخدام مجموعات بيانات DLPFC ودماغ الفأر. تحتوي مجموعة بيانات Stereo-seq الخاصة بالبصلة الشمية للفأر على 19109 نقطة بدقة 14 ميكرومتر؛
* مجموعة بيانات Slide-seqV2 بدقة 10 ميكرومتر، بما في ذلك الحُصين لدى الفئران (18765 نقطة من ربع دائرة المركز) ومصباح الشم لدى الفئران (19285 نقطة)؛
* تحتوي مجموعة بيانات STARmap على 1,207 نقطة؛
* بالنسبة لمجموعة بيانات MERFISH، تحتوي منطقة MTG البشرية على 3970 بقعة، بينما تحتوي مناطق VIS للفأر 1 والفأر 2 على 5995 و2479 بقعة، على التوالي.
هندسة النموذج: تعلم تباين الرسم البياني بمساعدة الصور لتحليل النسخ المكاني
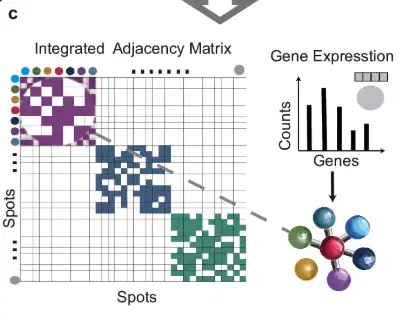
يوضح الشكل أدناه الإطار العام لـ STAIG، وهو إطار عمل للتعلم العميق يستخدم التعلم التبايني البياني جنبًا إلى جنب مع استخراج الميزات عالية الأداء لدمج التعبير الجيني والإحداثيات المكانية والصور النسيجية. يتكون من 6 وحدات:
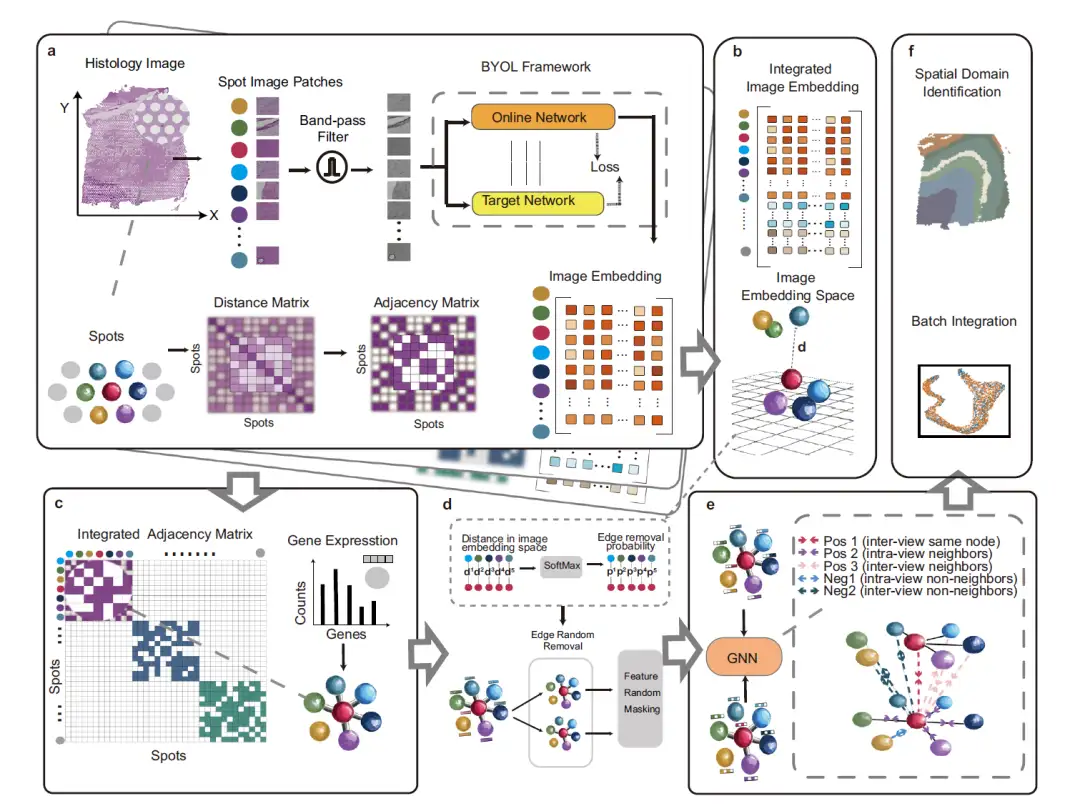
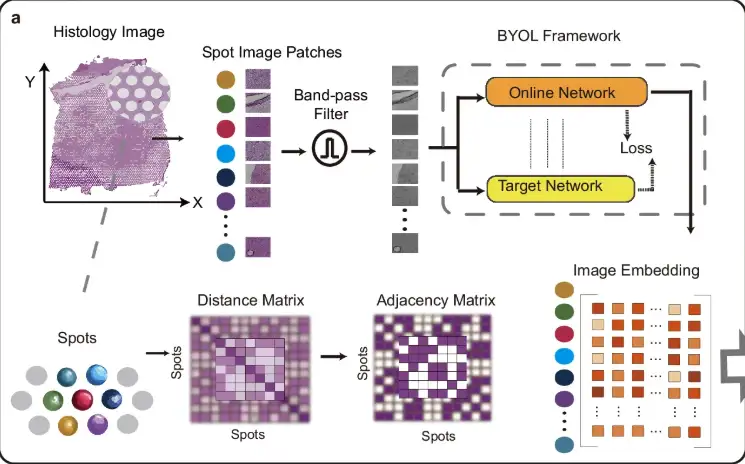
أولاً، كما هو موضح في الجزء (أ) من الشكل التالي، من أجل تقليل تأثير الضوضاء والتلوين غير المتساوي للأنسجة، يقوم STAIG أولاً بتقسيم الصورة النسيجية إلى بقع صغيرة (بقع الصور الموضعية) تتوافق مع المواضع المكانية لنقاط البيانات، ثم يستخدم مرشح تمرير النطاق لتحسين الصورة. يتم استخراج ميزات تضمين الصورة من خلال نموذج الإشراف الذاتي Bootstrap Your Own Latent (BYOL)، ويتم إنشاء مصفوفة مجاورة بناءً على المسافة المكانية بين نقاط البيانات.
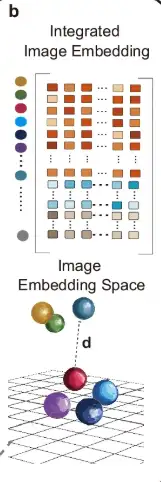
كما هو موضح في الجزء ب من الشكل التالي، من أجل دمج بيانات أقسام الأنسجة المختلفة، يستخدم STAIG طريقة التكديس الرأسي لتضمين ميزات أقسام الأنسجة المتعددة.
كما هو موضح في الجزء ج من الشكل أدناه، يتم دمج مصفوفات الجوار لكل شريحة باستخدام طريقة التنسيب القطري لتشكيل مصفوفة جوار متكاملة، والتي يتم استخدامها بعد ذلك لبناء بنية رسم بياني مع بيانات التعبير الجيني كمعلومات للعقدة.
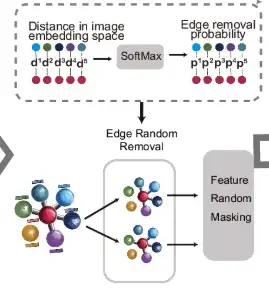
كما هو موضح في الجزء د من الشكل أدناه، بالنسبة لنقاط القياس المتصلة بالحواف، يتم حساب مسافاتها في مساحة تضمين الصورة، ويتم استخدام دالة SoftMax لتحويل هذه المسافات إلى احتمال إزالة الحافة العشوائية. على هذا الأساس، يخضع الرسم البياني الأصلي لجولتين من إزالة الحافة العشوائية (إزالة الحافة العشوائية) لتوليد عرضين معززين. بعد ذلك، يتم إخفاء ميزات العقدة في هذه العروض بشكل عشوائي.
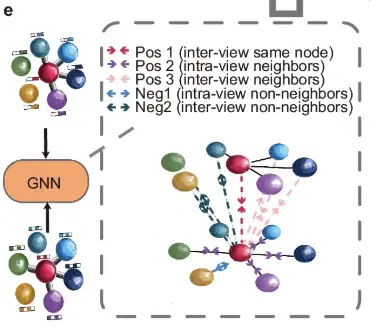
بعد ذلك، كما هو موضح في الجزء هـ من الشكل، تتم معالجة العرض الموسع لاحقًا بواسطة شبكة عصبية بيانية مشتركة (GNN) ويتم توجيهه بواسطة هدف تباين مجاور، والذي يهدف إلى تقريب العقد المجاورة من بعضها البعض أثناء تحريك العقد غير المجاورة بعيدًا في كلا عرضي الرسم البياني.
أخيرًا، كما هو موضح في الشكل f، تقوم GNN المدربة بتوليد تضمينات لتحديد المناطق المكانية وتقليل تأثيرات الدفعة بين أقسام الأنسجة المتتالية.

نتائج البحث: أظهرت STAIG أداءً متفوقًا في ظل ظروف مختلفة
أجرى فريق البحث تقييمًا معياريًا واسع النطاق لمقارنة STAIG مع تقنيات ST الحديثة الأخرى.وتظهر النتائج أن STAIG يظهر أداءً متفوقًا في ظل ظروف مختلفة.
تقييم أداء التعرف على مناطق الدماغ
لتقييم أداء STAIG في التعرف على مناطق الأنسجة، قارن الباحثون STAIG بالطرق الموجودة، بما في ذلك Seurat، وGraphST، وDeepST، وSTAGATE، وSpaGCN، وSEDR، وConST، وMuCoST، وstLearn. تتضمن مؤشرات تقييم الأداء ما يلي:
* مؤشر الراند المعدل (ARI) والمعلومات المتبادلة الطبيعية (NMI) (للمجموعات البيانات الموضحة يدويًا).
* معامل الصورة الظلية (SC) ومؤشر ديفيس-بولدينج (DB) (لمجموعات البيانات الأخرى).
① أداء مجموعة بيانات الدماغ البشري
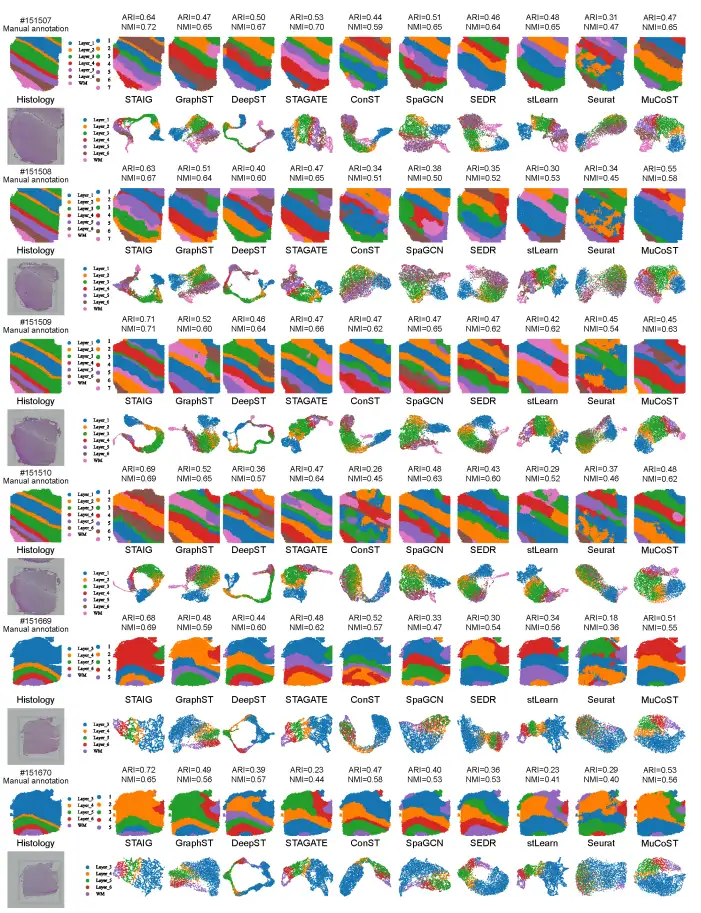
إجمالي،يحقق STAIG أفضل أداء على مجموعة بيانات الدماغ البشري.تم تحقيق أعلى متوسط ARI (0.69) و NMI (0.71)، كما هو موضح في الشكل التالي:
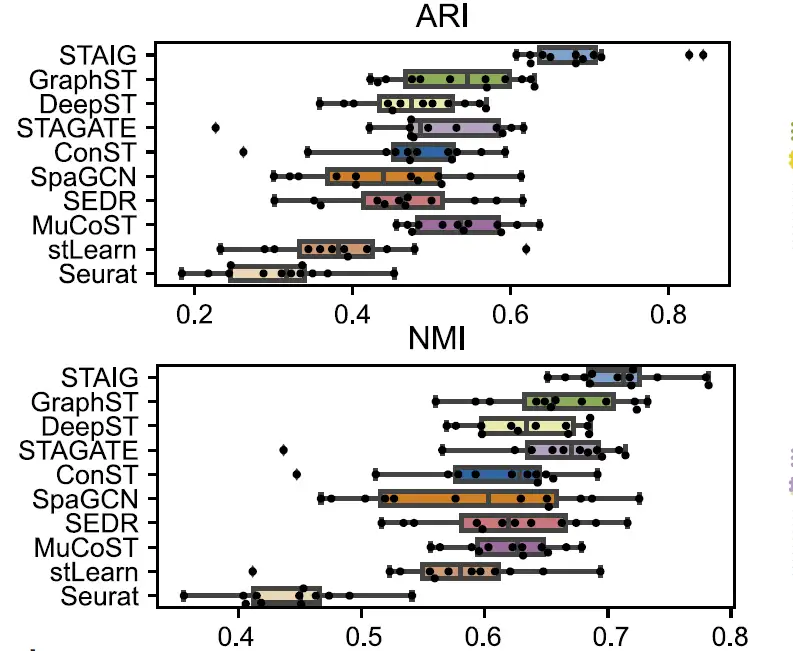
بالمقارنة، فإن الطرق الحالية تعمل بشكل سيئ: stLearn يخطئ في تقدير بعض النقاط ويفتقد بعض الطبقات؛ يحتوي GraphST على ARI بقيمة 0.64 و NMI بقيمة 0.73، ولكنه يحتوي على انحرافات كبيرة في مواضع طبقات L4 وL5؛ تتراوح مؤشرات ARIs للطرق الأخرى من 0.25 إلى 0.57 وتتراوح مؤشرات NMIs من 0.42 إلى 0.69، ويرجع ذلك أساسًا إلى عدم دقة تحديد نسبة الطبقة.
② أداء مجموعة بيانات دماغ الفأر
كما هو موضح في الشكل أدناه، في مجموعة بيانات الدماغ الخلفي للفأر،نجح STAIG في التعرف على القشرة المخيخية والحُصين، كما تمكن من التمييز بين قرن آمون (CA) والتلفيف المسنن.متوافق بشكل كبير مع شرح أطلس دماغ الفأر ألين؛ على الرغم من عدم وجود تعليقات توضيحية يدوية، لا يزال STAIG يحقق أعلى SC (0.31) وأدنى DB (1.11)، مما يشير إلى أداء التجميع المتفوق.
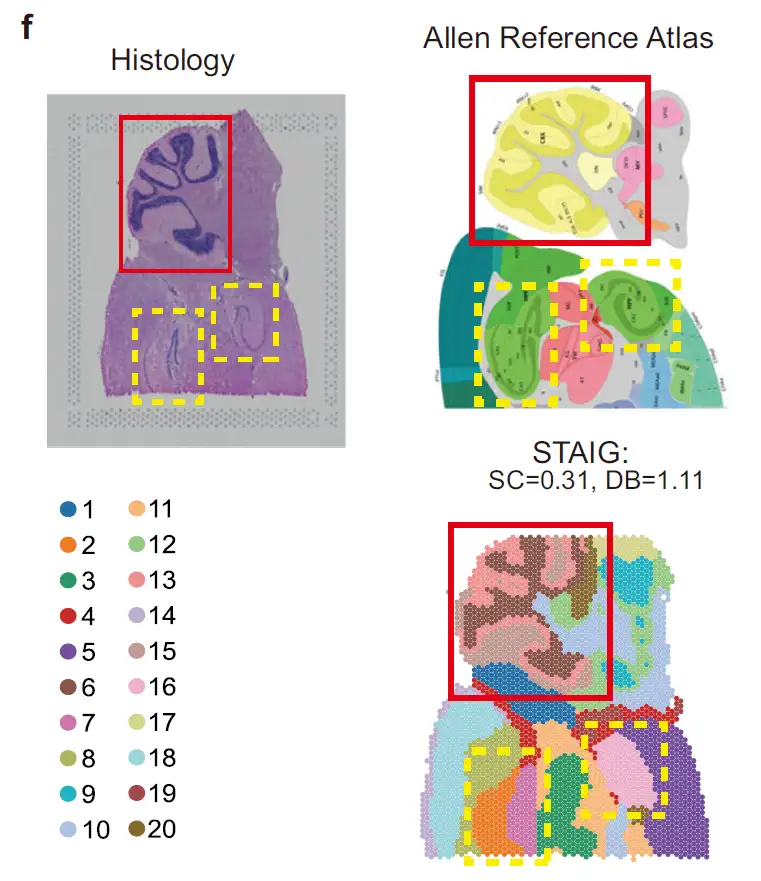
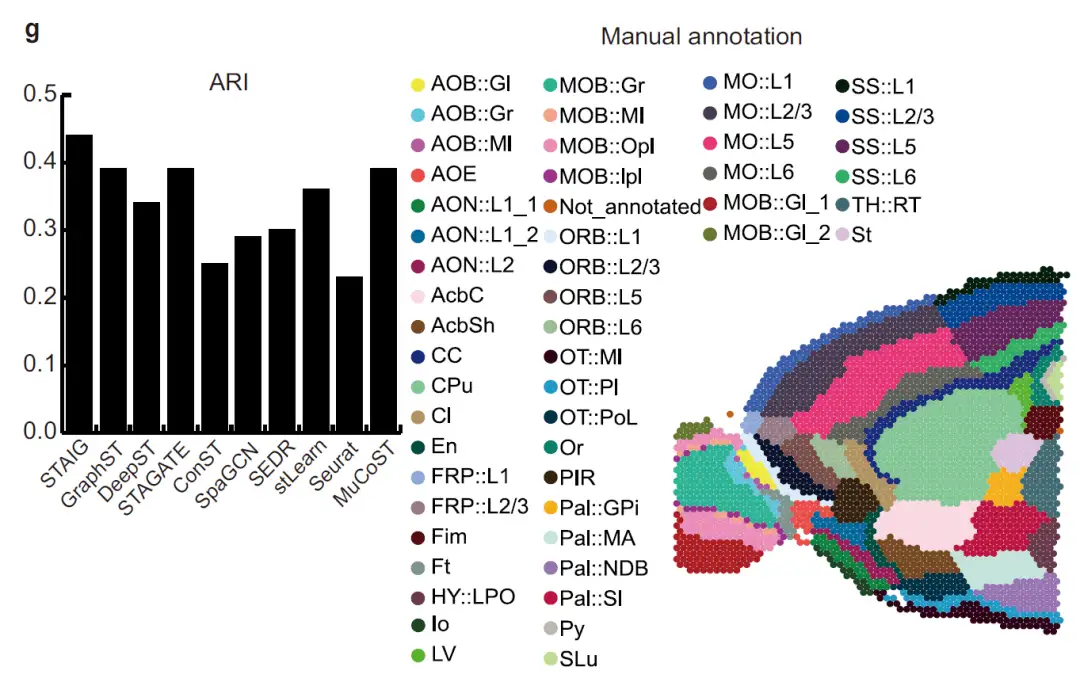
كما هو موضح في الشكل أدناه، في مجموعة بيانات الدماغ الأمامي للفأر،تمكنت شركة STAIG من تقسيم البصلة الشمية والباليوم الظهري بشكل دقيق.بعد الرجوع إلى الشرح اليدوي لـ Long et al.، وصل مؤشر ARI الخاص به إلى 0.44 ووصل مؤشر NMI إلى 0.72، وكلاهما أعلى القيم.
فعالية استخراج ميزات الصورة
لاستكشاف تأثير ميزات الصورة، استخدم الباحثون خوارزمية KNN لمقارنة ميزات الصورة المستخرجة بواسطة STAIG مع تلك المستخرجة بطرق أخرى (stLearn وDeepST وConST).
① تحليل شريحة أنسجة المخ
باستخدام الشريحة #151507 كمثال، كما هو موضح في الشكل أدناه، تتأثر ميزات الصورة الخاصة بـ stLearn بشكل خطير بشدة التلوين، مما يؤدي إلى عدم تطابق مع التعليق الهرمي الفعلي؛ على الرغم من أن DeepST وConST يستخدمان التعلم العميق، إلا أنهما يفشلان في التقاط ميزات النسيج المعقد لأنسجة المخ بدقة؛ تتوافق نتائج استخراج الميزات من STAIG بشكل كبير مع المستويات الموضحة يدويًا، وعلى الرغم من أن بعض الحدود لا تزال غير واضحة بعض الشيء، إلا أنها لا تتأثر تقريبًا باختلافات التلوين.

② تحليل صورة أنسجة سرطان الثدي
قام الباحثون باختبار قدرة استخراج ميزات الصورة بشكل أكبر باستخدام صور سرطان الثدي البشري الملطخة بالهرمونات والإيوزين، كما هو موضح في الشكل أدناه.
وأظهرت النتائج أن صورة stLearn تتميز بأورام مختلطة ومناطق طبيعية، مع تمييز ضعيف؛ يبدو أن ConST يقسم الصورة إلى مناطق مختلفة، ولكن بعد التكبير، انحرفت حدود المنطقة بشكل كبير عن التعليقات التوضيحية اليدوية؛ فشل DeepST في استخراج ميزات الصورة الفعالة؛يقوم STAIG بتحديد مناطق الورم بدقة. وتحافظ نتائج التجميع المكاني على درجة عالية من التماسك الإقليمي، وتتطابق المناطق المجزأة بشكل مثالي تقريبًا مع الخطوط الموضحة يدويًا.تم التحقق من قدرته الممتازة على استخراج ميزات الصورة.

تحديد البيئة الدقيقة للورم في سرطان الثدي البشري ST
في تحليل لمجموعة بيانات سرطان الثدي البشري،وجد الباحثون أن نتائج STAIG كانت متوافقة للغاية مع التعليقات اليدوية وحققت أعلى ARI (0.64) و NMI (0.70).ومن الجدير بالذكر أن STAIG يقترح تقسيمًا مكانيًا مختلفًا بعض الشيء ولكن أكثر دقة، وخاصة بالنسبة لمنطقة Healthy_1 الموضحة يدويًا (الشكل 2أ)، والتي يقسمها STAIG إلى مجموعات فرعية 3 و4 (الشكل 2ب).
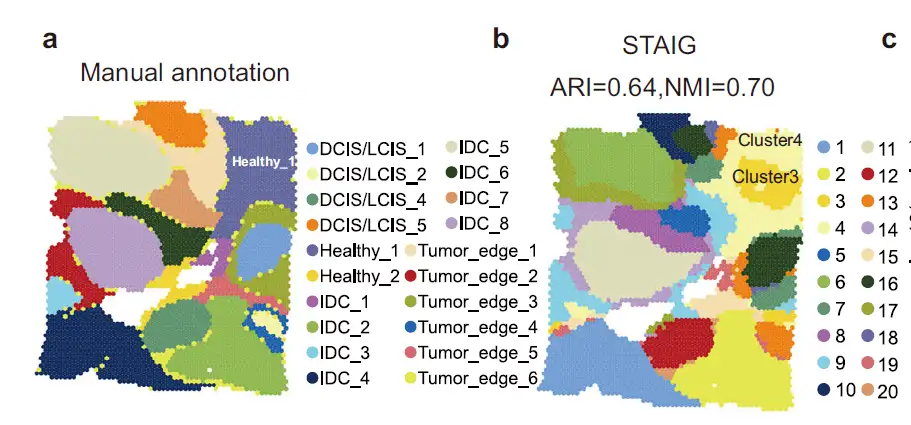
وفي الختام، من خلال التكامل المتعدد الوسائط لـ STAIG، وجدنا أن المجموعة الفرعية 3 شكلت بيئة ورمية كثيفة CAF وكشفت عن الخصائص الجزيئية للمناطق الغنية بـ CAF.
يوفر التعلم العميق أدوات قوية لتطوير تقنية ST
مع التطور السريع لعلم الجينوم وتكنولوجيا النقل الجيني، أصبح المجتمع الطبي الحيوي قادرًا على استكشاف التوزيع المكاني للتعبير الجيني داخل الأنسجة، وبالتالي الكشف عن الوظائف والهياكل المعقدة للكائنات الحية. لا توفر تقنية ST معلومات كمية عن التعبير الجيني فحسب، بل تحافظ أيضًا على العلاقة المكانية بين الخلايا في الأنسجة، مما يتيح للباحثين دراسة البيئة الدقيقة للأنسجة، وتفاعلات الخلايا، والخصائص المكانية لتطور المرض. لكن،نظرًا لأن بيانات ST عادةً ما تعاني من مشكلات مثل الأبعاد العالية والضوضاء القوية وتأثيرات الدفعة، فقد أصبح كيفية دمج هذه البيانات وتحليلها بشكل فعال تحديًا أساسيًا في الأبحاث الحالية.
يؤدي تقديم تقنيات التعلم العميق، وخاصة الشبكات العصبية الرسومية (GNNs) وطرق التعلم التبايني، إلى توفير أدوات قوية لتحليل بيانات ST. غالبًا ما تعتمد طرق التحليل التقليدية على تقليل الأبعاد والتجميع، في حين يمكن لطرق التعلم العميق استخراج ميزات متعددة المستويات تلقائيًا وتحسين تمثيل البيانات من خلال التدريب الشامل. كما ذكر أعلاه، يمكن لطريقة GNN استخدام معلومات الجوار المكاني لبناء بنية بيانية، بحيث لا يتمكن النموذج من التقاط التعبير الجيني فحسب، بل يتعلم أيضًا التبعيات المكانية بين الخلايا. ويؤدي تقديم التعلم التبايني إلى تعزيز قدرة النموذج على التعميم بشكل أكبر، مما يمكّنه من تعلم الميزات المكانية الرئيسية دون الحاجة إلى توضيح.
بالإضافة إلى ذلك، حققت الصناعة أيضًا الكثير من التقدم في الجمع بين تقنية التعلم العميق + ST:
نوفمبر 2024قام الفريق بقيادة يانغ يونغوي من المركز الوطني للمعلومات الحيوية في الصين والفريق بقيادة تشانغ شي هوا من معهد الرياضيات وعلوم النظم التابع للأكاديمية الصينية للعلوم بتطوير أداة توضيحية للخلايا النسخية المكانية تعتمد على التعلم العميق تسمى STASCAN.من خلال دمج ملفات تعريف التعبير الجيني وتعلم خصائص الخلايا من الصور النسيجية، فإننا نتوقع أنواع الخلايا في المناطق غير المعروفة من أقسام الأنسجة ونعلق على الخلايا داخل المنطقة الملتقطة، وبالتالي نعمل على تحسين الدقة الخلوية المكانية بشكل كبير. علاوة على ذلك، يمكن تطبيق STASCAN على مجموعات بيانات مختلفة من تقنيات ST المختلفة ويظهر مزايا كبيرة في فك تشفير توزيع الخلايا عالية الدقة وحل بنية الأنسجة المحسنة.
نُشرت هذه النتيجة في مجلة Genome Biology تحت عنوان "STASCAN يفك رموز خرائط توزيع الخلايا عالية الدقة في النسخ المكاني من خلال التعلم العميق".
* عنوان الورقة:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03421-5
23 يناير 2025قام فريق بحثي من جامعة برينستون في الولايات المتحدة بتطوير خوارزمية جديدة للتعلم العميق تسمى GASTON (تحليل التدرج للتنظيم النسخي المكاني باستخدام الشبكات العصبية). من خلال الجمع بين الشبكات العصبية العميقة غير الخاضعة للإشراف والخوارزميات القابلة للتفسير، اقترح GASTON بشكل مبتكر مفهوم "العمق المتساوي"، وهو مشابه للارتفاع في الخريطة الطبوغرافية ويُستخدم لقياس البنية الطوبولوجية المكانية للتعبير الجيني في أقسام الأنسجة. ،
من خلال العمق المتساوي وتدرجه، لا يستطيع الباحثون تقسيم المناطق المكانية المختلفة من الأنسجة فحسب، بل يمكنهم أيضًا تحديد اتجاهات التغيير المستمر والجينات الرئيسية للتعبير الجيني داخل الأنسجة. أظهرت الدراسة نجاح تطبيق GASTON في مجموعة متنوعة من العينات البيولوجية، بما في ذلك دماغ الفأر، وبصلة الشم لدى الفأر، وبيئة ورم سرطان القولون والمستقيم، وما إلى ذلك. تُظهر النتائج أن GASTON يمكنه تحليل بنية الأنسجة بدقة، والكشف عن التوزيع المكاني وأنماط التغيير لأنواع الخلايا، واكتشاف العديد من أنماط التعبير الجيني المكاني التي تتجاهلها الطرق الأخرى.
وقد نُشرت النتائج ذات الصلة في مجلة Nature Methods تحت عنوان "رسم خريطة تضاريس التعبير الجيني المكاني باستخدام التعلم العميق القابل للتفسير".
* عنوان الورقة:
https://www.nature.com/articles/s41592-024-02503-3
من الواضح أن الجمع بين التعلم العميق وتكنولوجيا ST لا يحسن فقط قدرة تكامل البيانات وتقليل الضوضاء، بل يعزز أيضًا التعدين العميق للمعلومات البيولوجية المكانية. في المستقبل، مع نمو موارد الحوسبة وتحسين الخوارزميات، سيلعب التعلم العميق دورًا أكثر أهمية في تحليل بيانات ST ويوفر دعمًا أقوى للطب الدقيق والعلاج الشخصي.
مراجع:
1.https://static-content.springer.com/esm/art%3A10.1038%2Fs41467-025-56276-0/MediaObjects/41467_2025_56276_MOESM1_ESM.pdf
2.https://www.bjqykxy.com/kexueyanjiu/dongwuzhiwu/7361.html
3.https://news.qq.com/rain/a/20250128A057OQ00?suid=&media_id=
4.https://www.medsci.cn/article/show_








