Command Palette
Search for a command to run...
كن أول من يجرب العرض التوضيحي! النموذج القائم على الجينوم Evo موجود على غلاف مجلة Science، مما يتيح التنبؤ والتوليد من المقاييس الجزيئية إلى الجينومية

لقد حقق الذكاء الاصطناعي في الآونة الأخيرة تقدمًا كبيرًا في مجال علم الأحياء.اقترح فريق بحثي من جامعة ستانفورد ومعهد آرك في الولايات المتحدة نموذج Evo يعتمد على الجينوم، والذي يمكنه تحقيق التنبؤ بالعينة الصفرية وتوليد الدقة العالية في المهام المتعددة الوسائط للحمض النووي والحمض النووي الريبي والبروتين.
نُشر البحث ذو الصلة في مجلة Science كمقالة رئيسية بعنوان "نمذجة التسلسل والتصميم من المستوى الجزيئي إلى مستوى الجينوم باستخدام Evo".
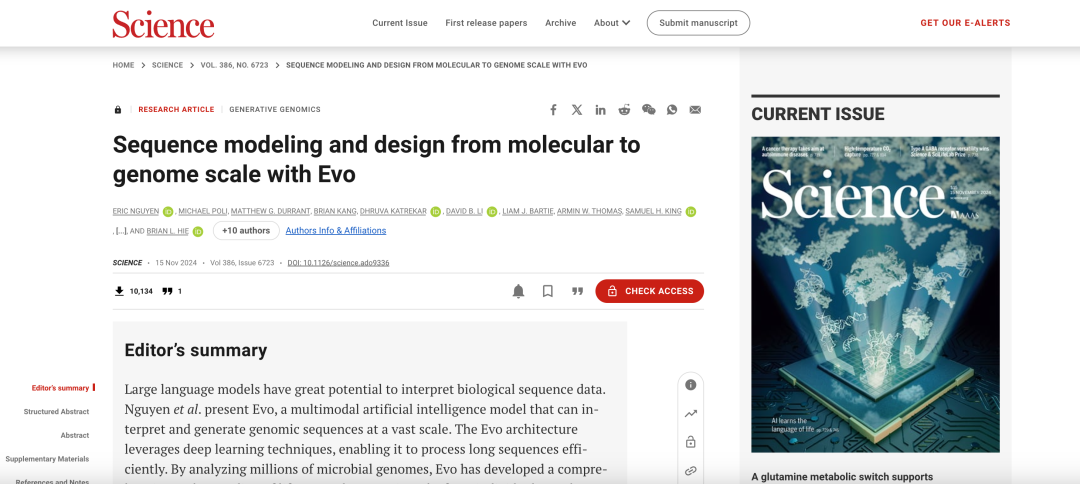
عنوان الورقة:
https://www.science.org/doi/10.1126/science.ado9336
قم بمتابعة الحساب الرسمي ورد "Evo" للحصول على ملف PDF كامل
وباعتباره المؤلف الأول للورقة البحثية، نشر إريك نجوين عدة تحديثات لتقديم إنجازات إيفو، وشكر أعضاء الفريق عدة مرات، وقال بصراحة: "إنه لشرف لي أن أعمل مع مثل هذا الفريق المذهل!"

وفقا للصحيفة، يستخدم Evo بنية StripedHyena ويتم تدريبه على مجموعة بيانات جينومية كبيرة تحتوي على أكثر من 80000 جينوم بكتيري وعتيق.وملايين من تسلسلات البكتيريا العاثية والبلازميدية المتوقعة، والتي تغطي 300 مليار رمز نوكليوتيد، قادرة على توليد تسلسلات الحمض النووي مع بنية جينوم معقولة يزيد طولها عن ميغا قاعدة واحدة.
أيضًا،بمقياس معلمات يبلغ 7 مليارات وطول سياق أقصى يبلغ 131072 رمزًا، يمكن لـ Evo الكشف عن التطور المشترك المعقد بين التسلسلات المشفرة وغير المشفرة وتصميم أنظمة بيولوجية معقدة.مثل مجمعات CRISPR-Cas والعناصر المنقولة IS200 و IS605.
باختصار، إن قدرة Evo على التنبؤ بتسلسلات الجينوم الكاملة وتوليدها وتصميمها لا توفر دعماً نظرياً جديداً للعلوم الحياتية فحسب، بل من المتوقع أيضاً أن يتم تطبيقها على تحرير الجينات واكتشاف الأدوية وتشخيص الأمراض والزراعة وغيرها من المجالات، مما يساعد على تطوير نتائج رائدة في مجالات متعددة.
لقد صدم العديد من مستخدمي الإنترنت من إطلاق Evo وكانوا يتطلعون إلى التطبيق المحدد لهذا النموذج.
من أجل السماح للجميع بتجربة الوظائف القوية لنموذج Evo لأول مرة،أصبح الآن برنامج HyperAI Hyperneuron التعليمي متاحًا على الإنترنت بعنوان "Evo: التنبؤ والتوليد من المقياس الجزيئي إلى الجينومي".لا حاجة لإدخال أي أوامر، فقط استنساخ بنقرة واحدة للحصول على تجربة سريعة!
رابط البرنامج التعليمي:
تشغيل تجريبي
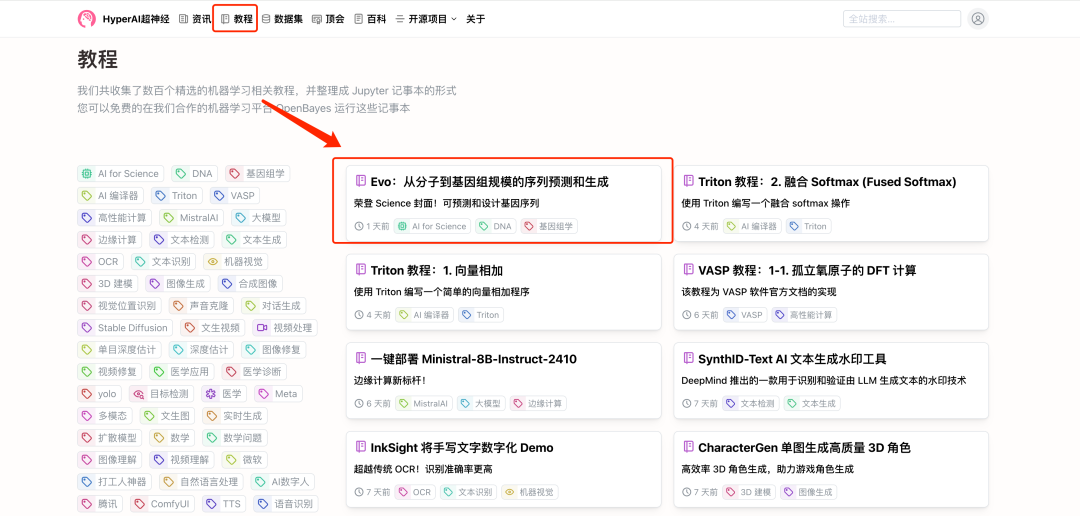
1. قم بتسجيل الدخول إلى hyper.ai، في صفحة البرامج التعليمية، حدد Evo: التنبؤ والتوليد من المقياس الجزيئي إلى الجينومي، ثم انقر فوق تشغيل هذا البرنامج التعليمي عبر الإنترنت.
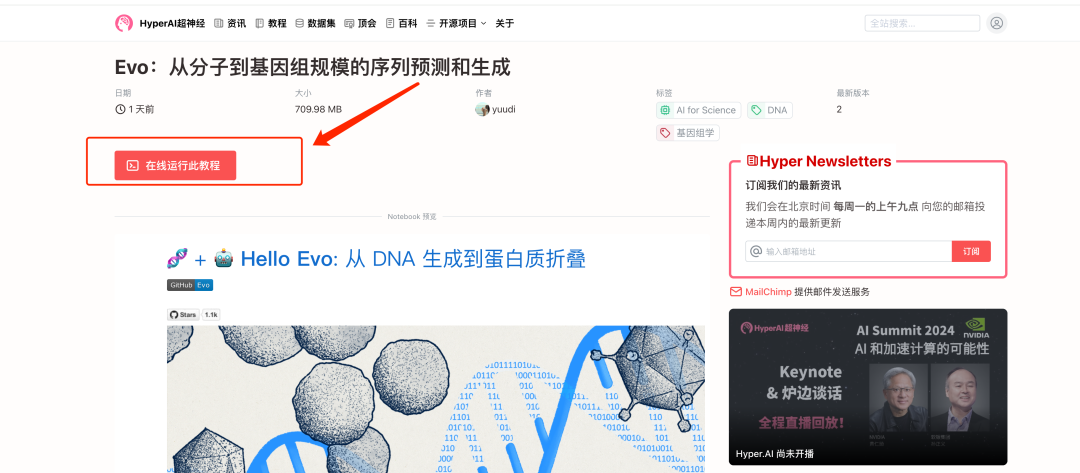
2. بعد الانتقال إلى الصفحة التالية، انقر فوق "استنساخ" في الزاوية اليمنى العليا لاستنساخ البرنامج التعليمي في الحاوية الخاصة بك.
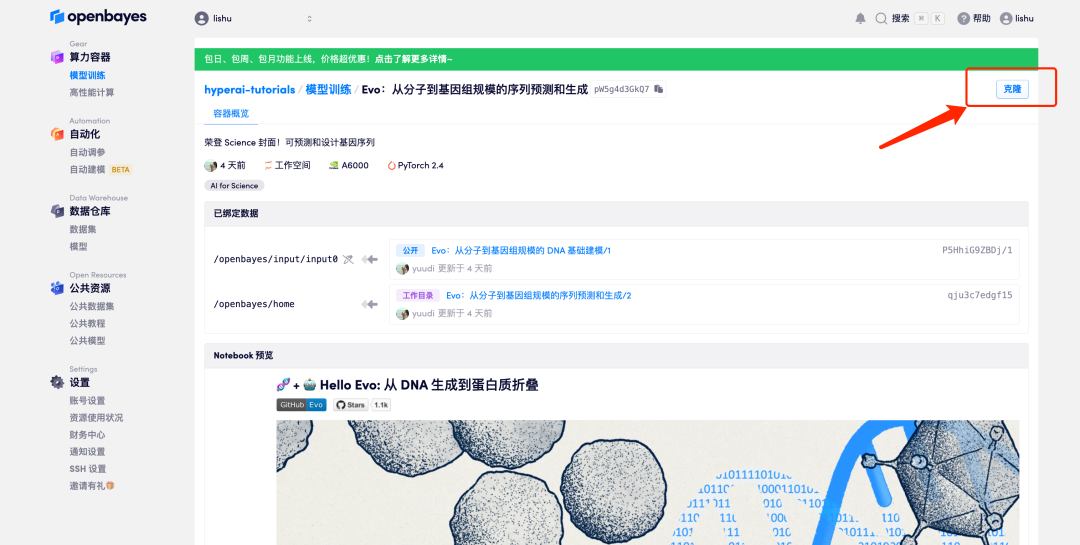
3. انقر فوق "التالي: حدد معدل التجزئة" في الزاوية اليمنى السفلية.
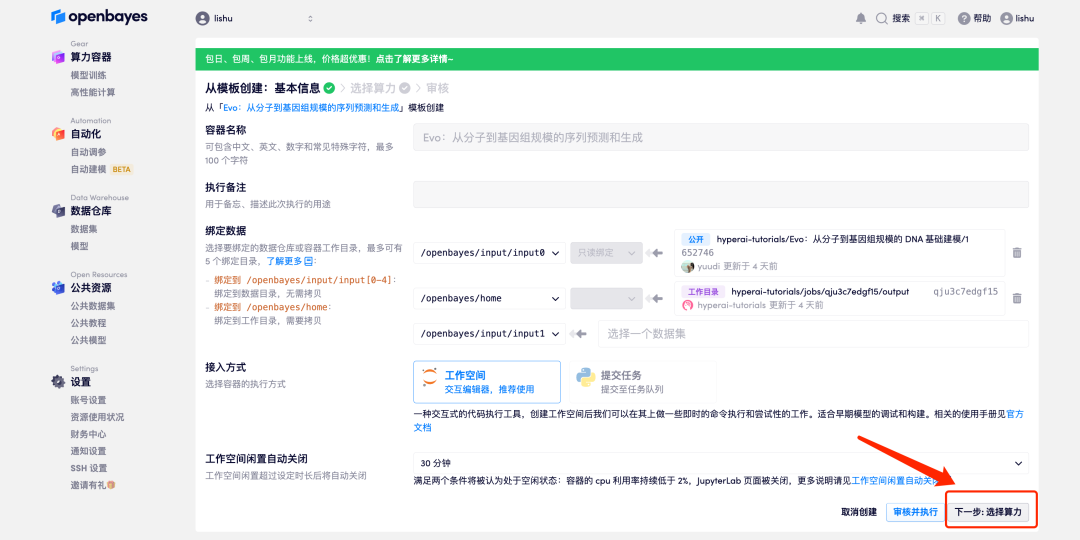
4. بعد الانتقال إلى الصفحة التالية، حدد "NVIDIA RTX A6000"، ثم حدد "الدفع حسب الاستخدام" أو "الحزمة اليومية/الأسبوعية/الشهرية" وفقًا لاحتياجاتك. بعد تحديد صورة "PyTorch"، انقر فوق "التالي: المراجعة". يمكن للمستخدمين الجدد التسجيل باستخدام رابط الدعوة أدناه للحصول على 4 ساعات من RTX 4090 + 5 ساعات من وقت فراغ وحدة المعالجة المركزية!
رابط دعوة حصرية لـ HyperAI (انسخ وافتح في المتصفح):
https://openbayes.com/console/signup?r=Ada0322_QZy7
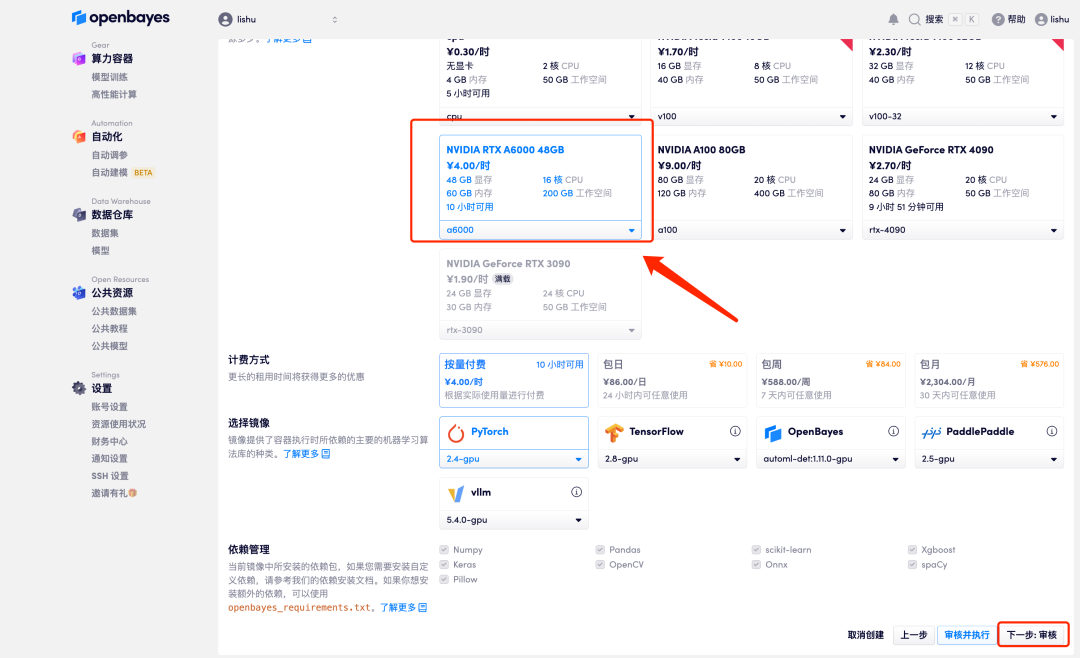
5. بعد التأكيد، انقر فوق "متابعة" وانتظر حتى يتم تخصيص الموارد. ستستغرق عملية الاستنساخ الأولى حوالي دقيقتين.
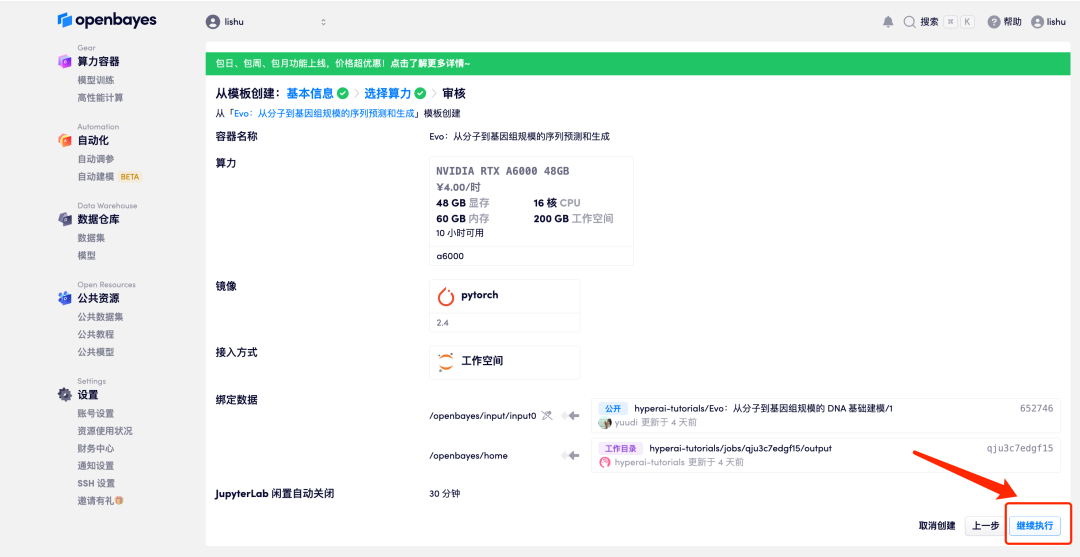
6. عندما تتغير الحالة إلى "قيد التشغيل"، انقر فوق خيار "فتح مساحة العمل".
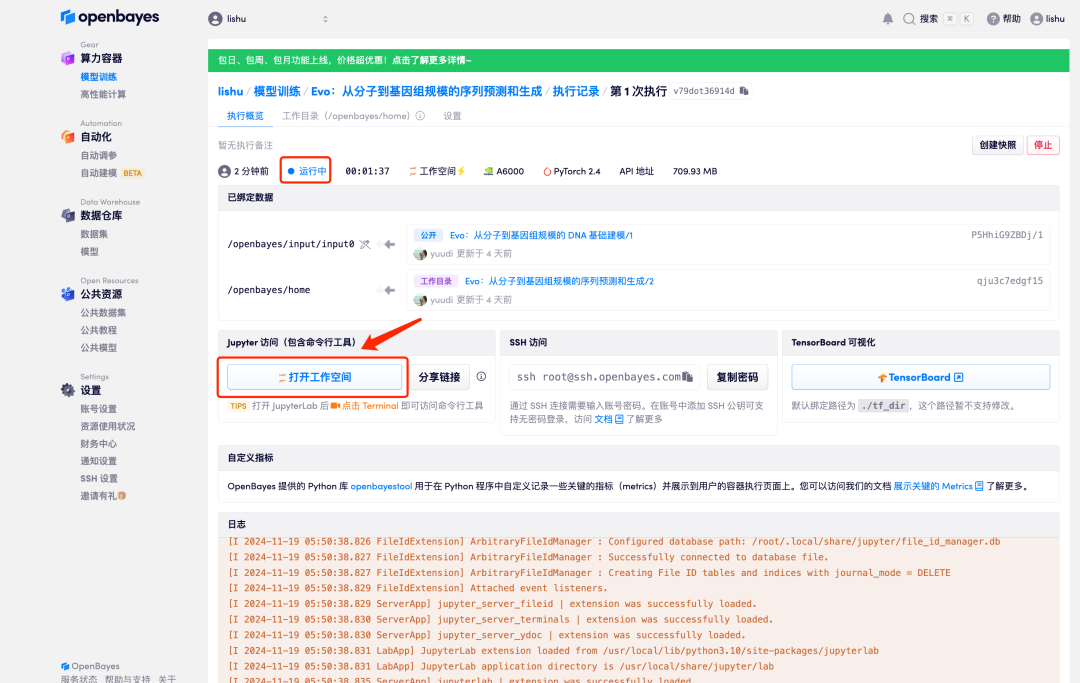
7. بعد الدخول إلى مساحة عمل Jupyter، انقر نقرًا مزدوجًا فوق ملف "README" للدخول رسميًا إلى صفحة تشغيل نموذج Evo.
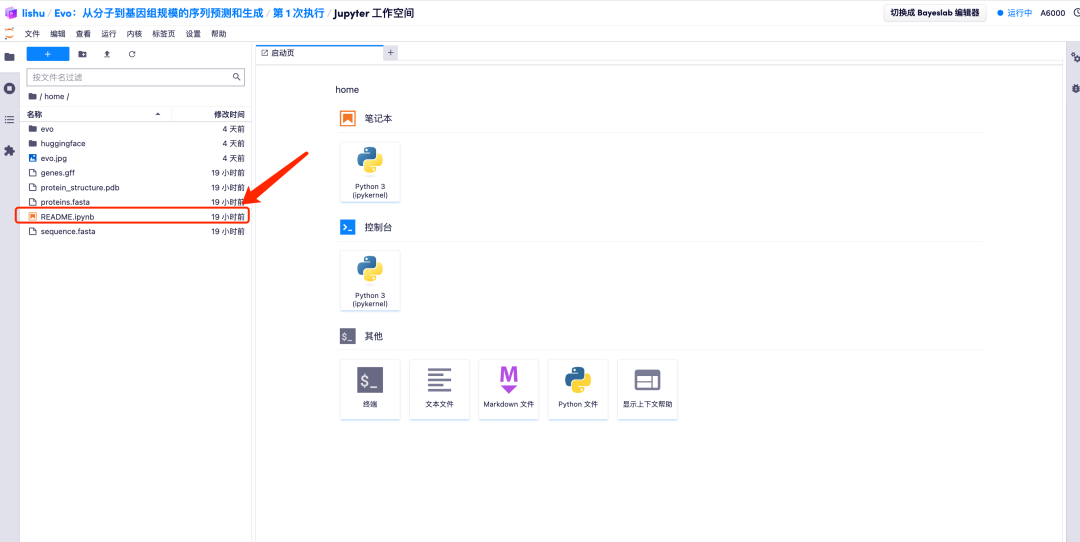
عرض التأثير
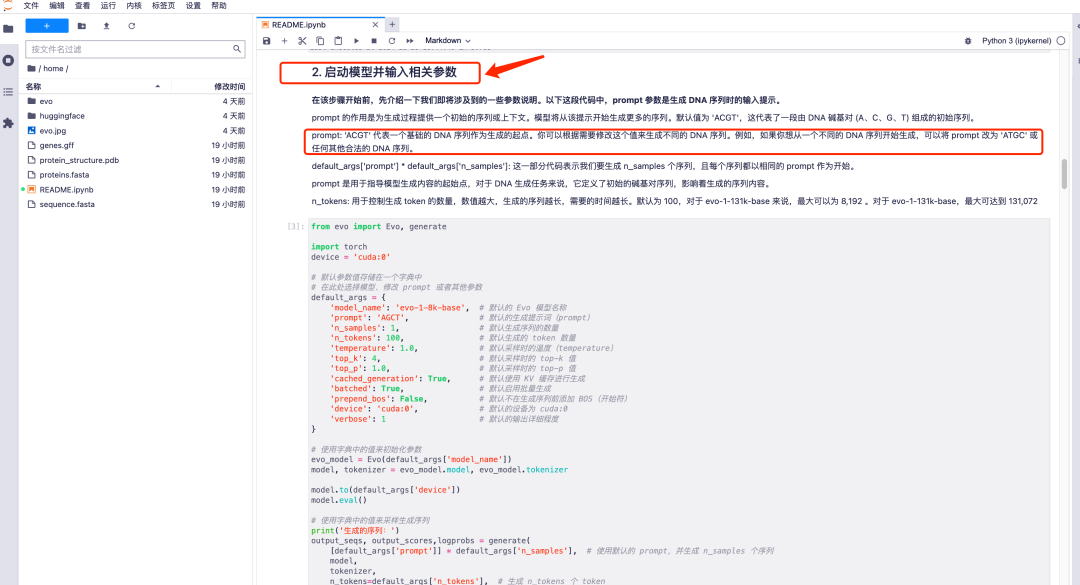
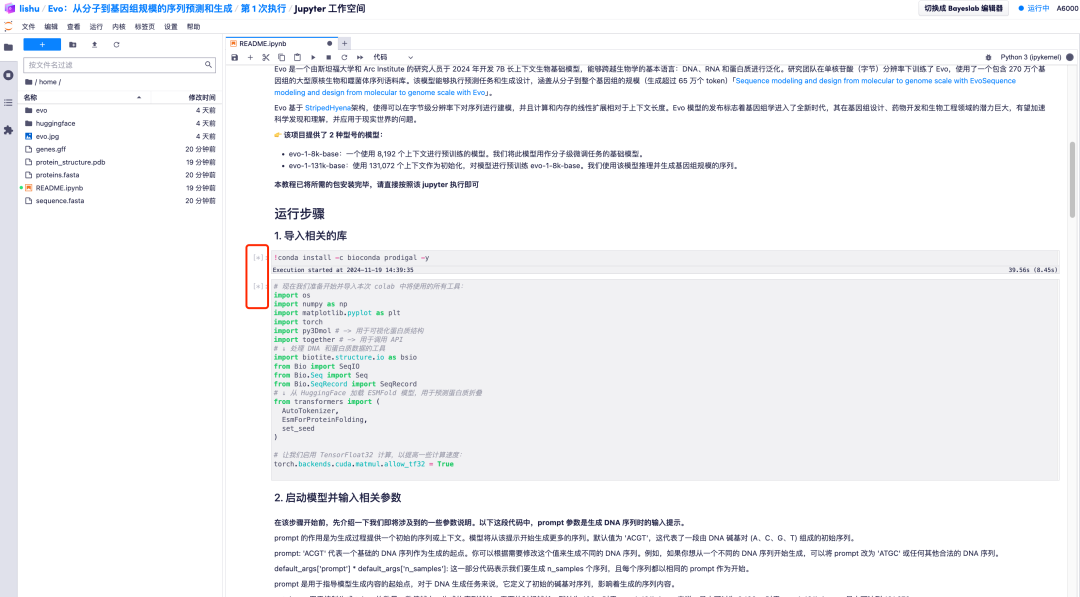
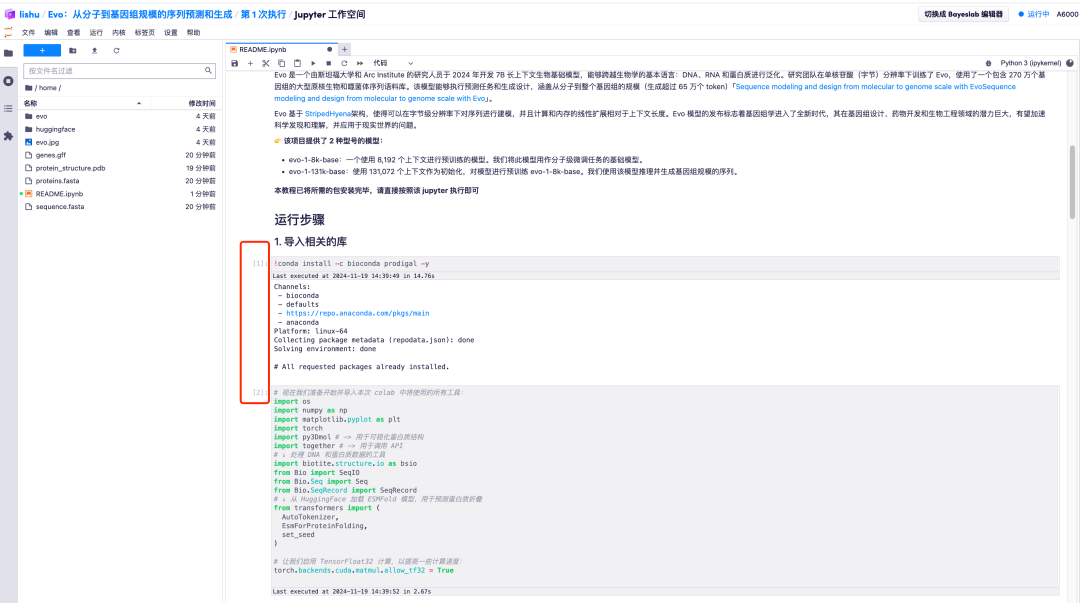
1. بعد الدخول إلى صفحة تشغيل نموذج Evo، تكون جميع المعلمات في الوضع الافتراضي. قم بالتمرير لأسفل إلى "2. ابدأ النموذج وأدخل المعلمات ذات الصلة" واضبط قيم المعلمات المطلوبة وفقًا لاحتياجاتك.تجدر الإشارة إلى أن القيمة الافتراضية للمطالبة هي "ACGT"، والتي تمثل تسلسلًا أوليًا يتكون من أزواج قواعد الحمض النووي (A، C، G، T).يمكنك تعديل هذه القيمة لتوليد تسلسلات DNA مختلفة حسب الحاجة.
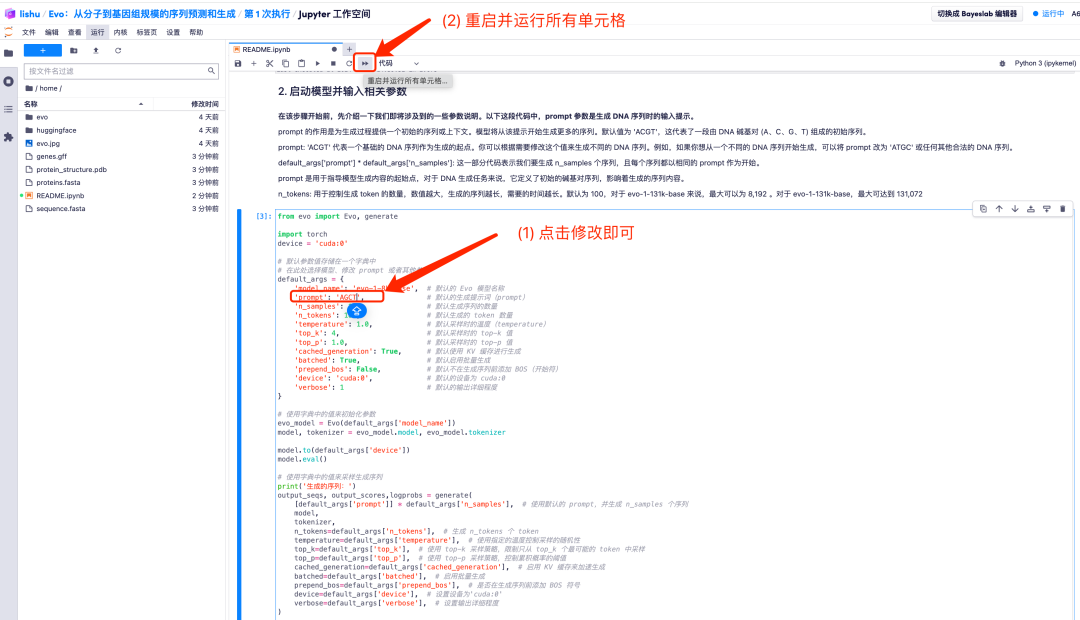
2. على سبيل المثال، قم بتغيير القيمة الافتراضية للمطالبة إلى AGCT. بعد ضبط قيم المعلمات الافتراضية، انقر فوق خيار "إعادة التشغيل وتشغيل كافة الخلايا" وحدد "إعادة التشغيل" للتشغيل.
3. انتظر لفترة من الوقت. عندما يتغير الرمز [*] إلى رقم، تكون العملية قد اكتملت. في أسفل "2. ابدأ النموذج وأدخل المعلمات ذات الصلة"، يمكنك رؤية التسلسل الناتج.
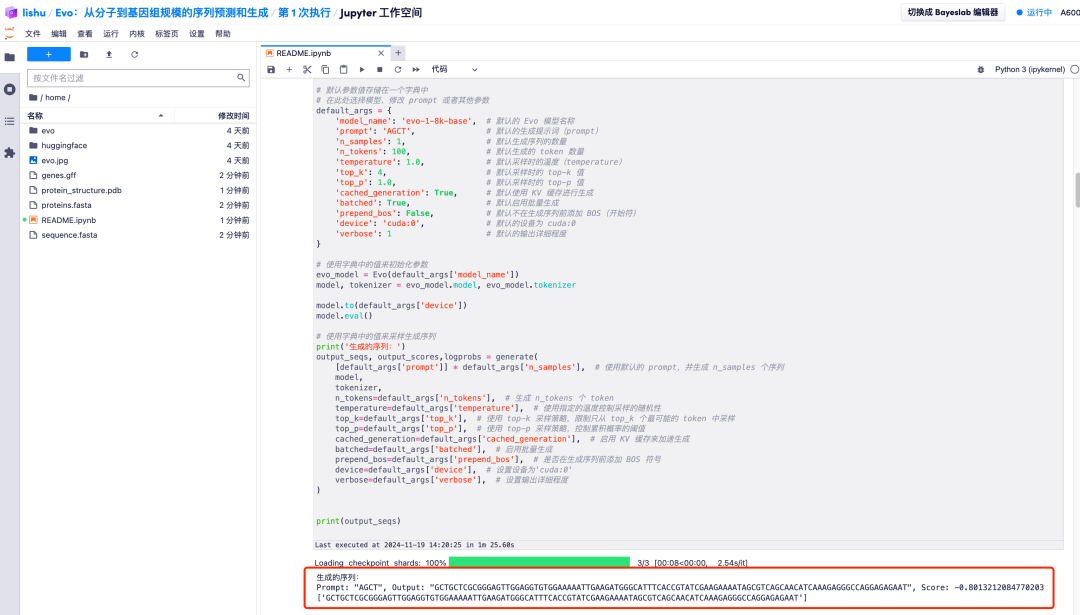
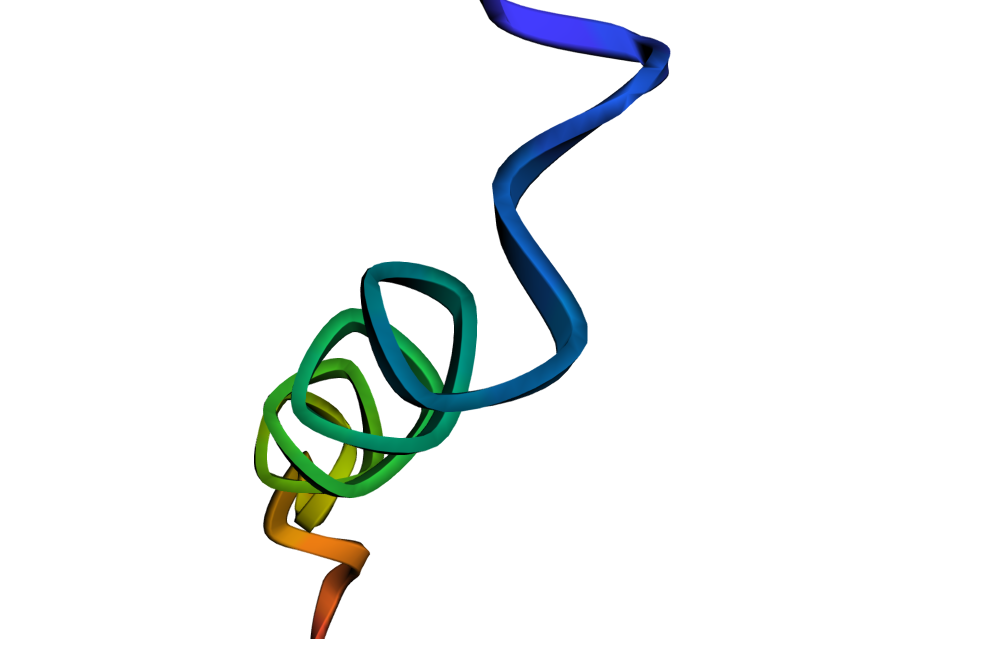
4. بالإضافة إلى ذلك، يمكن لنموذج Evo أيضًا تحليل تسلسلات الحمض النووي الناتجة ومعرفة العلاقة التطورية المشتركة بين التسلسلات المشفرة والتسلسلات غير المشفرة. ويمكنه أيضًا التنبؤ بالجينات المشفرة للبروتين من تسلسلات الحمض النووي، وترميز وتصميم أنظمة الحمض النووي الريبي، وبالتالي التنبؤ ببنية طي البروتين الناتجة، والتي يمكن تقديمها في النهاية في شكل صورة.
لقد قمنا بتأسيس "مجموعة تبادل الدروس حول الانتشار المستقر". نرحب بالأصدقاء للانضمام إلى المجموعة لمناقشة القضايا الفنية المختلفة ومشاركة نتائج التطبيق ~
قم بمسح رمز الاستجابة السريعة أدناه لإضافة HyperaiXingXing على WeChat (معرف WeChat: Hyperai01)، ولاحظ "SD Tutorial Exchange Group" للانضمام إلى الدردشة الجماعية.