Command Palette
Search for a command to run...
الذكاء الاصطناعي لعلم الجينوم | خوارزمية تمثيل بيانات النسخ المكاني SPACE، تطبيق الذكاء الاصطناعي في علم الجينوم

في الحلقة الثانية من سلسلة "Meet AI4S"، يشرفنا دعوة لي يوزي، زميل ما بعد الدكتوراه في مختبر تشانغ تشيانغفينغ في كلية العلوم الحياتية بجامعة تسينغهوا،ومختبره، تشانغ تشيانغفينج، تابع لكلية العلوم الحياتية بجامعة تسينغهوا. وهو أيضًا جزء مهم من المركز المشترك لجامعة تسينغهوا وبكين للعلوم الحياتية ومركز بكين للابتكار المتقدم في علم الأحياء البنيوي. تركز أبحاث المختبر على تقاطع علوم الحياة وخوارزميات الذكاء الاصطناعي، وتكنولوجيا المجموعة البنيوية للحمض النووي الريبي وتطوير الخوارزميات، وتكنولوجيا تسلسل جينوم الخلية الواحدة وتطوير الخوارزميات، ونمذجة بنية البروتين بناءً على بيانات المجهر الإلكتروني بالتبريد، وتطوير خوارزميات الذكاء الاصطناعي ذات الصلة.
هذه المشاركة،ألقى الدكتور لي يوزهي كلمة بعنوان "استكشاف تطبيقات الذكاء الاصطناعي في علم الجينوم: أخذ خوارزمية توصيف بيانات النسخ المكاني SPACE كمثال".وتمت مشاركة أحدث نتائج أبحاث الفريق، وتم تقديم أساليب الذكاء الاصطناعي في أبحاث النسخ المكاني وأبحاث أوميكس الخلية الواحدة.
قام HyperAI بتجميع وتلخيص المشاركة المتعمقة للدكتور لي يوزهي دون انتهاك النية الأصلية.
انقر هنا لمشاهدة الإعادة المباشرة الكاملة:
يُحدث الذكاء الاصطناعي في العلوم تغييرات هائلة في نموذج البحث في المجال العلمي
اليوم، الموضوع الذي سأشاركه هو الذكاء الاصطناعي للعلوم. أعتقد أن الذكاء الاصطناعي للعلوم قد أحدث تغييرات كبيرة في نموذج البحث في المجال العلمي بأكمله. وبعد ذلك سأشرح ذلك بالتفصيل مستخدمًا البحث في بنية البروتين كمثال.
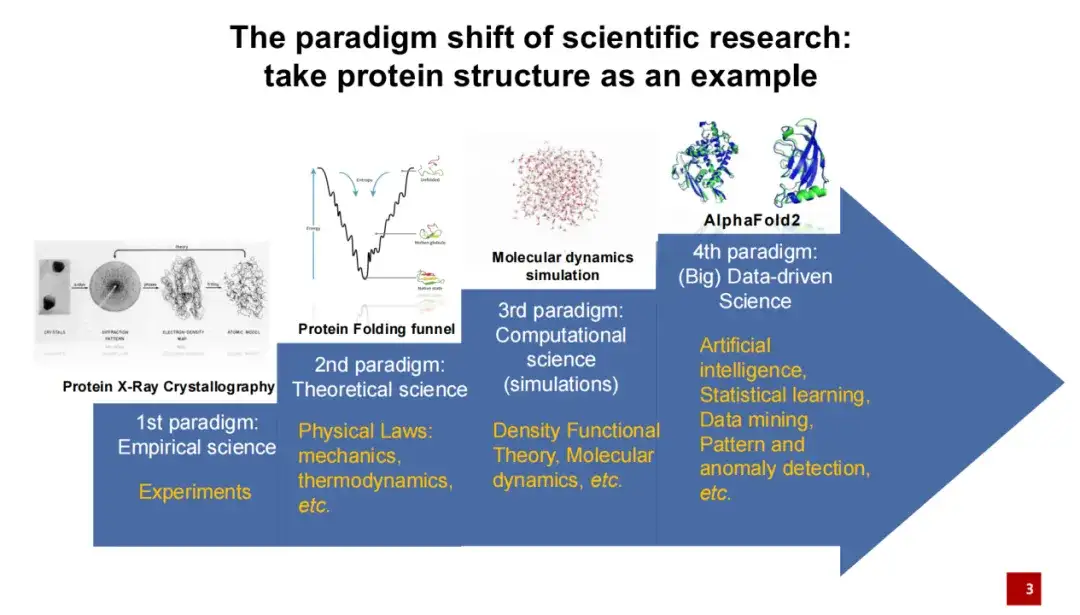
تم إجراء الجيل الأول من نموذج أبحاث بنية البروتين بشكل أساسي من خلال الوسائل التجريبية.وهذا يعني استخدام الأشعة السينية لتصوير البلورات التي يشكلها البروتين، ومن ثم إجراء النمذجة البنيوية.
كان الجيل الثاني من نموذج أبحاث بنية البروتين بقيادة علماء الفيزياء الذين أضافوا المعرفة النظرية إلى دراسة بنية البروتين.على سبيل المثال، إذا كانت طاقة طي البروتين منخفضة، فإن هذا الطي يكون مستقرًا نسبيًا.
يشير الجيل الثالث من نموذج أبحاث بنية البروتين إلى تسعينيات القرن العشرين، عندما تم تطبيق المحاكاة الحاسوبية تدريجياً على أبحاث بنية البروتين، مع تطور تكنولوجيا الكمبيوتر.وعلى وجه الخصوص، تم استخدام محاكاة الديناميكيات الجزيئية على نطاق واسع في السنوات الأخيرة. تساعدنا طرق المحاكاة هذه على حساب وتوقع بنية البروتين بشكل أفضل إلى حد ما. في السنوات الأخيرة، وخاصة في عام 2020، دخلت خوارزميات الذكاء الاصطناعي مجال بنية البروتين، مما أدى إلى تحقيق اختراق آخر. في مسابقة التنبؤ ببنية البروتين لعام 2020، كان AlphaFold 2 متقدمًا كثيرًا عن الطرق المنافسة الأخرى.
لقد أدى إدخال الذكاء الاصطناعي إلى إحداث تحول كبير في نموذج العلوم الحياتية ومجال البحث العلمي بأكمله. بالمقارنة مع طرق البحث التقليدية،يركز الذكاء الاصطناعي بشكل أكبر على البدء بالبيانات وإجراء البحوث العلمية القائمة على البيانات.وهذا يعني أننا لم نعد بحاجة إلى اقتراح فرضية علمية مسبقًا، بل أصبح بإمكاننا أن نتعلم ونكتشف قوانين الطبيعة مباشرة من البيانات.
تطور الذكاء الاصطناعي في مجال الجينوميات
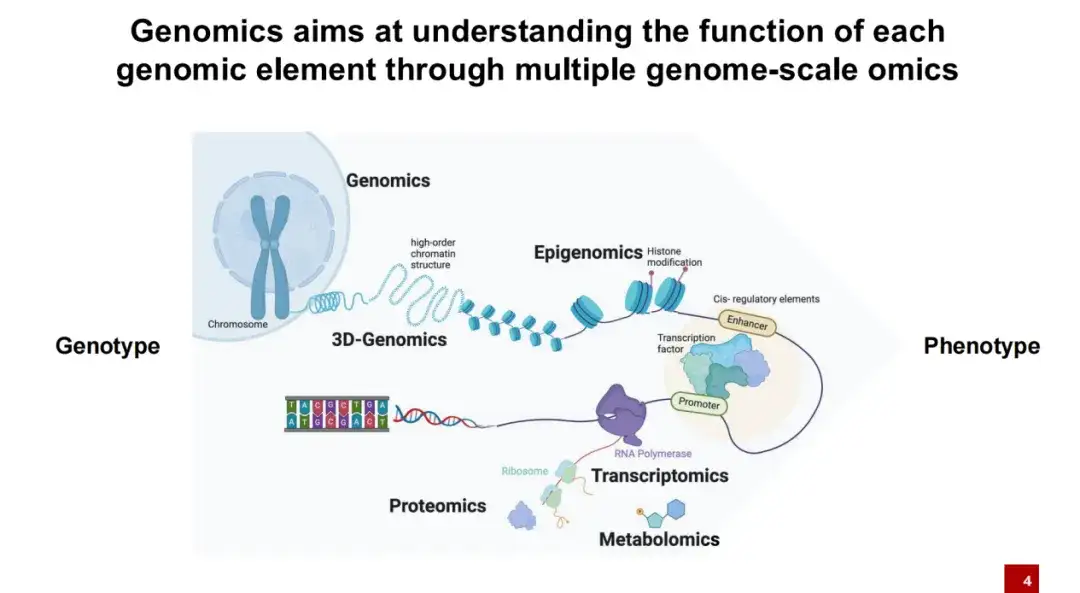
ستركز المشاركة التالية على تطبيق الذكاء الاصطناعي في مجال علم الجينوم. باختصار،يستكشف البحث في علم الجينوم بشكل أساسي العلاقة بين النمط الجيني (كل الحمض النووي في الجسم) والنمط الظاهري (الخصائص الفردية مثل الطول والوزن).
كما نعلم جميعًا، لا يوجد الحمض النووي (DNA) عاريًا في الخلايا، بل يكون ملفوفًا حول النيوكليوسومات. ترتبط النيوكليوسومات بالعديد من تعديلات الهيستون. في العادة، تكون هذه الحمض النووي مترابطة بشكل وثيق مع بعضها البعض. لا يمكن للحمض النووي أن يتعرض لتكوين فاصل مفتوح إلا في ظل ظروف معينة. في هذه المرحلة، يمكن للبروتينات مثل عوامل النسخ أن ترتبط بهذه المناطق المكشوفة من الحمض النووي.
وفي عملية النسخ اللاحقة، يمكن نسخ الحمض النووي الريبوزي (RNA) بواسطة بوليميراز الحمض النووي الريبوزي، ثم ترجمته إلى بروتين بواسطة الريبوسوم، ويلعب البروتين في النهاية دورًا في أنشطة الحياة.الهدف البحثي في علم الجينوم هو فهم كيفية تأثير عناصر الحمض النووي المختلفة على الأنشطة الحيوية.
نلخص الأحداث والتطورات المهمة في تطوير الذكاء الاصطناعي للعلوم منذ تصدع بنية الحلزون المزدوج للحمض النووي في الخمسينيات وحتى الفترة الأخيرة. يعود أصلها إلى اكتشاف البنية الحلزونية المزدوجة للحمض النووي في الخمسينيات من القرن العشرين وتطوير تقنية تسلسل سانجر في السبعينيات.
كما هو موضح في الشكل أدناه، يمثل الجزء الأزرق تطور تقنيات التسلسل المختلفة والتقنيات التجريبية؛ يمثل الجزء الأخضر الأساليب المهمة في مجال الذكاء الاصطناعي؛ ويمثل الجزء الأصفر إنشاء بعض خطط البحث وقواعد البيانات المهمة على نطاق واسع؛ يمثل الجزء الأحمر الأساليب والتطبيقات التمثيلية في مجال الذكاء الاصطناعي لعلم الجينوم.
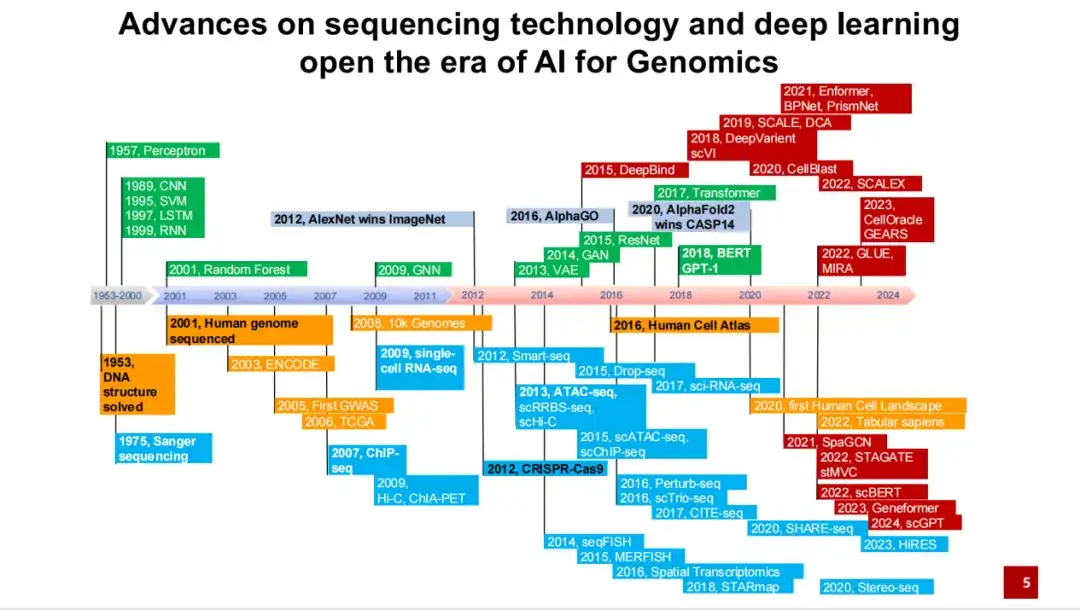
كما ترون،2001تم الانتهاء في البداية من مشروع الجينوم البشري، وتم تسلسل تسلسل الحمض النووي بأكمله لذكر أبيض. في عام 2012،لقد تفوقت AlexNet على البشر في مهام تصنيف الصور لأول مرة، مما أدى إلى التطور الهائل للذكاء الاصطناعي على مدى العقد الماضي. في عام 2016،تم اقتراح مشروع أطلس الخلايا البشرية، وتحول البحث تدريجيا من تسلسل الحمض النووي لفرد واحد إلى جميع الخلايا. وفي العام نفسه، هزم برنامج AlphaGo، الذي يعتمد على أساليب التعلم المعزز، البشر في لعبة Go.
بالنسبة للذكاء الاصطناعي في علم الجينوم أو الذكاء الاصطناعي في العلوم،كان الاختراق المهم هو أنه في عام 2020، احتل AlphaFold 2 المركز الأول في CASP 14 بفارق كبير.وقد أدى هذا إلى زيادة عدد أساليب الذكاء الاصطناعي المطبقة في مجال علم الجينوم.
في،يعد علم الجينوميات أحادية الخلية إنجازًا كبيرًا في مجال علم الجينوميات في السنوات الأخيرة.عادةً ما تتضمن أبحاث الجينوم التقليدية التسلسل الشامل. افترض أن كل خط في الشكل أدناه يمثل نوعًا من الخلايا، وأن الخطوط ذات الألوان المختلفة تمثل أنواعًا مختلفة من الخلايا. في الماضي، كانت طرق التسلسل تتضمن خلط وتسلسل الأنسجة بأكملها، مما يجعل من الصعب تحديد الخلية المحددة التي جاء منها كل DNA أو RNA. إن ظهور تقنية الخلية الواحدة يسمح لنا ليس فقط بالحصول على كامل الحمض النووي أو الحمض النووي الريبي في الأنسجة، بل وأيضاً بتحديد المصدر الخلوي المحدد لهذا الحمض النووي أو الحمض النووي الريبي. نظرًا لأن أنواع الخلايا المختلفة لها تعبيرات جينية مختلفة وتؤدي وظائف مختلفة، يمكننا فهم أنشطة الحياة بشكل أكبر.
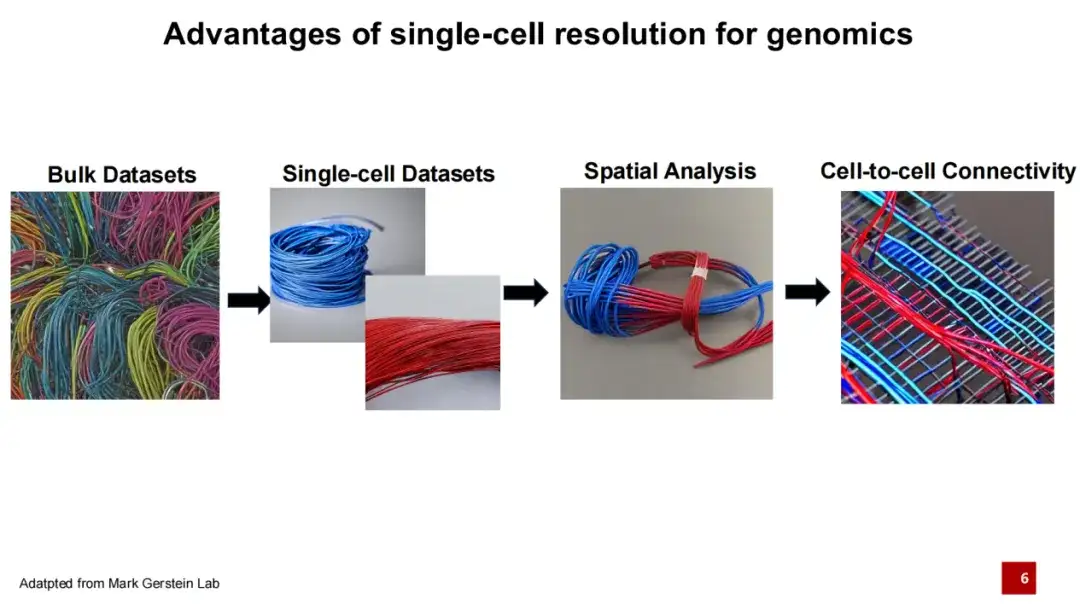
خلال السنوات الخمس الماضية، قطعت تقنية الجينوم المكاني المتمثلة في النسخ المكاني خطوة أبعد استنادًا إلى تقنية الجينوم أحادي الخلية.لا يمكننا فقط الحصول على معلومات حول كل نوع من الخلايا، بل يمكننا أيضًا تحديد توزيع هذه الخلايا في الفضاء.وبما أن التفاعلات بين الخلايا تشكل أساسًا مهمًا لتحقيق وظائفها، فإن الأبحاث اللاحقة تركز على كيفية ارتباط الخلايا ببعضها.
منذ إطلاق مشروع الجينوم البشري وحتى اقتراح مشروع أطلس الخلايا البشرية في عام 2016، كان هدفه استكمال الخريطة المرجعية لجميع الخلايا البشرية لمساعدتنا على فهم أنشطة الحياة بشكل أفضل وتقديم الدعم لعلاج وتشخيص أمراض محددة.
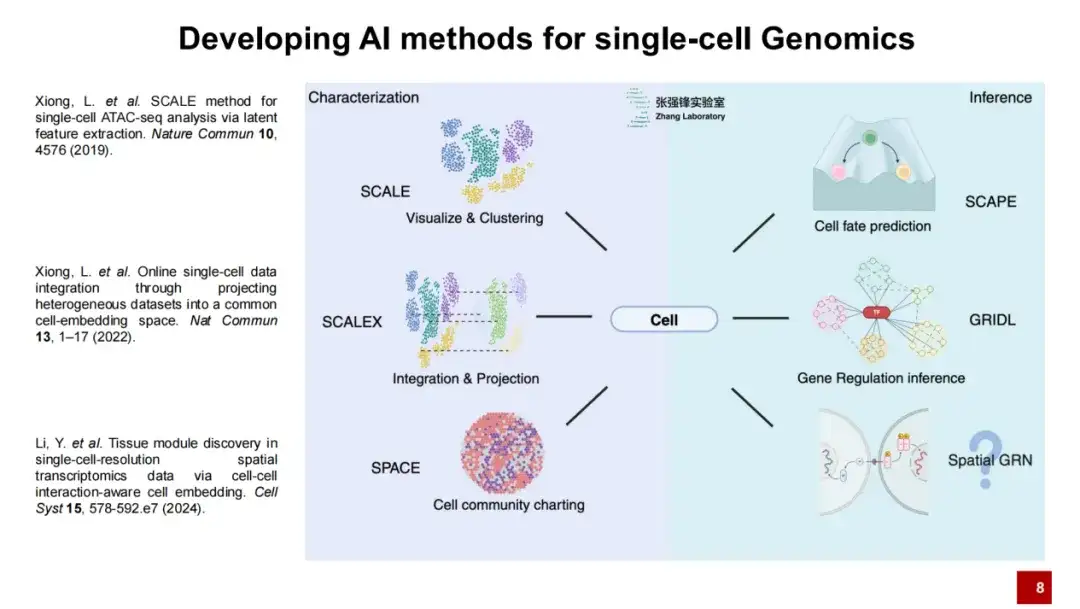
قام الفريق بتطوير ثلاث طرق: SCALE وSCALEX وSPACE لأبحاث الجينوميات أحادية الخلية
لقد قام مختبرنا بتطوير سلسلة من أساليب الذكاء الاصطناعي.نحن نعتقد أن علم الجينوم أحادي الخلية يتطلب خطوتين رئيسيتين: الأولى وصف الخلية، والثانية استنتاج الخلية.
لقد نشرنا ثلاثة أعمال لوصف الخلايا: SCALE، وSCALEX، وSPACE.SCALE مخصص بشكل أساسي للتصور والتجميع، وSCALEX مخصص لدمج البيانات وإسقاطها، وSPACE مخصص لوصف بيئة تنظيم بيانات النسخ المكاني بالكامل.سأقدم اليوم بشكل أساسي الطريقتين: SCALEX وSPACE.
طريقة SCALEX لإزالة تأثيرات الدفعة
طريقة SCALEX هي إزالة تأثيرات الدفعة.وهذه قضية مهمة للغاية في أبحاث الجينوميات. يشير تأثير الدفعة إلى الاختلافات في النتائج التجريبية بين دفعات مختلفة بسبب العوامل الفنية مثل الظروف التجريبية المختلفة.
كما هو موضح في الشكل أدناه، حتى لو قمنا بزراعة نسختين بيولوجيتين من الخلايا بشكل منفصل، فمن الناحية النظرية فإن تسلسل هاتين المجموعتين من الخلايا يجب أن يؤدي إلى تعبير جيني متشابه للغاية. ومع ذلك، نظرًا لأسباب تقنية، مثل الاختلافات في بيئة الثقافة، أو وقت إنشاء المكتبة، أو منصة التسلسل،قد تختلف ملفات تعريف التعبير الجيني النهائية بشكل كبير، مما يؤدي إلى إدخال قدر كبير من الضوضاء التقنية.لذلك، عند تحليل البيانات، من الضروري إزالة تأثير الدفعة هذا.

في البحث البيولوجي، لا يمكن في كثير من الأحيان جمع البيانات كلها مرة واحدة، ولكن يتم تجميعها تدريجيا من خلال تجارب متعددة. لذلك،إزالة تأثيرات الدفعة وتحليل البيانات بطريقة متكاملة للعثور على العوامل ذات الصلة البيولوجية الحقيقية.وهي خطوة أساسية في أبحاث الجينوميات أو جينوميات الخلية الواحدة.
وبناءً على ذلك، قمنا بتطوير طريقة SCALEX.إنه قادر على إسقاط البيانات التي تمت معالجتها من خلية واحدة في مساحة كامنة خلوية معممة. يعتمد إطار عمل SCALEX على المشفرات التلقائية المتغيرة (VAE).
المدخل الأول هو بيانات النسخ لخلية واحدة، والتي يتم إسقاطها بعد ذلك في مساحة كامنة خلوية معممة من خلال مشفر خالٍ من الدفعات.
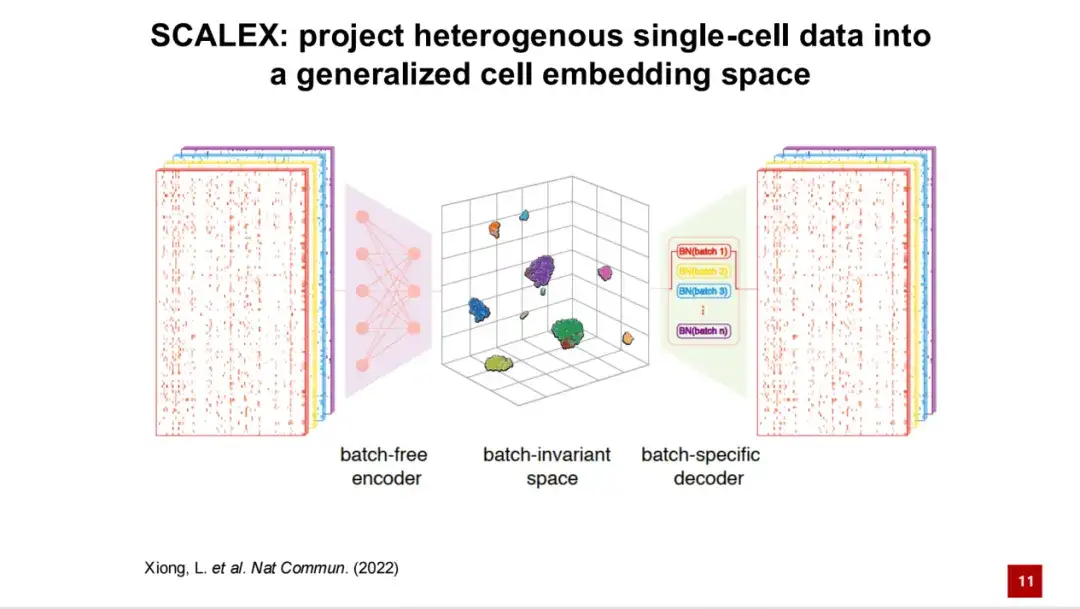
بعد ذلك، تتم إضافة معلومات الدفعة إلى النموذج من خلال تطبيع الدفعة الخاصة بالمجال عبر فك تشفير خاص بالدفعة. من خلال هذا التصميم غير المتماثل، فإن مساحة الخلية الكامنة الناتجة هي مساحة مستقلة عن الدفعة، والتي من الناحية النظرية لا تحتوي على أي ضوضاء تقنية مرتبطة بالدفعة. يتم إعادة بناء التعبير الجيني من خلال جهاز فك التشفير، ويتم حساب الخسارة باستخدام طيف التعبير الجيني المدخل الأصلي. في الوقت نفسه، بالتزامن مع تباعد KL، يتم إنشاء دالة الخسارة لنموذج SCALEX، وهو نموذج خاضع للإشراف الذاتي.
يتمتع تصميم المشفر وفك التشفير غير المتماثل هذا بميزتين رئيسيتين:أولاً، يكون المشفر الناتج عالميًا.وهذا يعني أنه يمكن إسقاط البيانات الجديدة مباشرة في مساحة الخلية الكامنة دون معلومات الدفعة من خلال المبرمج دون إعادة تدريب النموذج أو إعادة دمج البيانات الجديدة في البيانات الموجودة.
ثانيًا، تولي SCALEX اهتمامًا أكبر لتأثير الدفعة العالمية.الطريقة التقليدية لإزالة تأثيرات الدفعة تتمثل بشكل أساسي في العثور على خلايا مماثلة (أزواج خلايا) في دفعتين من البيانات وإقرانها للتصحيح لإزالة تأثير الدفعة. تعتبر هذه الطريقة في الأساس عبارة عن تصحيح تأثير الدفعة المحلي نسبيًا.
ومع ذلك، هناك مشكلة مع هذا النوع من الطرق، وهي أنه في تحليل البيانات الفعلي، قد لا تكون أنواع الخلايا في دفعتين مختلفتين متسقة تمامًا، وقد يكون هناك عدد قليل فقط من أنواع الخلايا المشتركة، والباقي خاص بالدفعة. إذا تم إجبار الخلايا على الاقتران، فقد يحدث تصحيح زائد لأنه لا يمكن العثور على خلايا مقترنة مناسبة، وسيتم إجبار أنواع الخلايا التي لا ينبغي محاذاتها على المحاذاة.
وفي هذا الصدد، سأشرح الميزتين الرئيسيتين لـ SCALEX بمزيد من التفصيل.
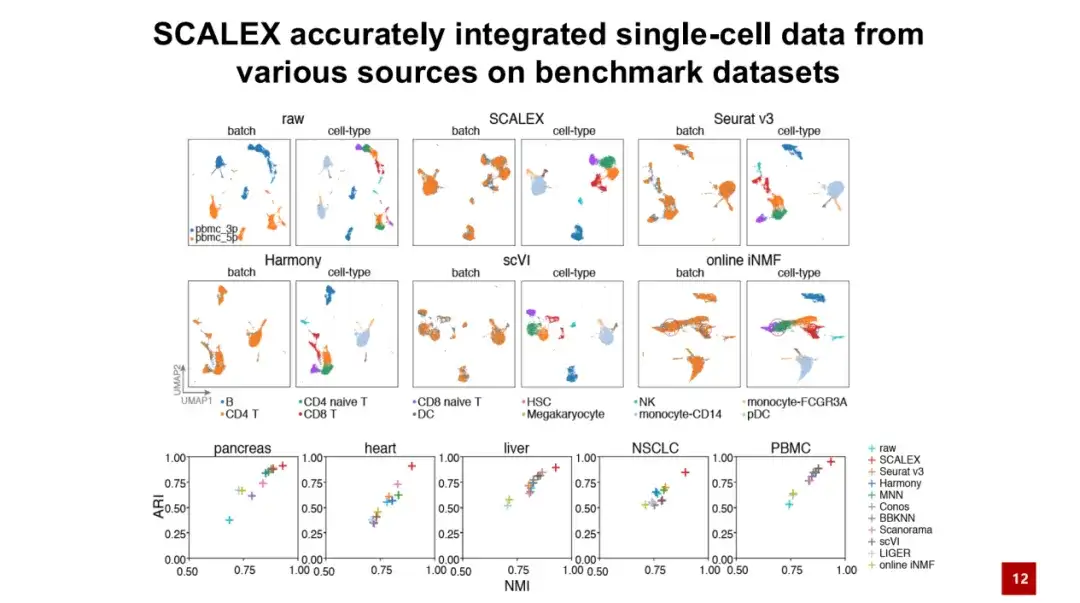
أولاً، قمنا بمقارنة أداء SCALEX على خمس مجموعات بيانات اختبار.وتظهر النتائج أن SCALEX يتفوق على الطرق الحالية من حيث الدقة.
كما هو موضح في الشكل أدناه، يمثل الرسم البياني للدفعة البيانات الأصلية وغير المصححة، ويمثل اللون الأزرق والبرتقالي دفعتين من البيانات على التوالي، ويمثل نوع الخلية نوع الخلية. يمكن ملاحظة أنه على الرغم من وجود أنواع خلايا متشابهة في هاتين الدفعتين، إلا أنه بسبب تأثير الدفعة الكبيرة، لا يمكن تجميع الخلايا التي تنتمي في الأصل إلى نفس نوع الخلية معًا، مما يؤدي إلى عوامل تقنية تغطي العوامل البيولوجية وتجعل البحث البيولوجي اللاحق مستحيلاً.
بعد دمج SCALEX، تمكنت الدفعتان من الخلايا من التجمع بشكل جيد وتم فصلهما بوضوح وفقًا لأنواع الخلايا، مما يوضح أهمية SCALEX في التطبيقات العملية.
من أهم مميزات SCALEX هو قدرته على معالجة دفعتين من البيانات بنفس أنواع الخلايا.وتسمى هذه البيانات بمجموعات البيانات المتداخلة الجزئية. كما هو موضح في الشكل أدناه، فإن التداخل 0 يعني أن أنواع الخلايا في الدفعتين مختلفة تمامًا، والتداخل 4 يعني أن هناك 4 أنواع مشتركة من الخلايا في الدفعتين.
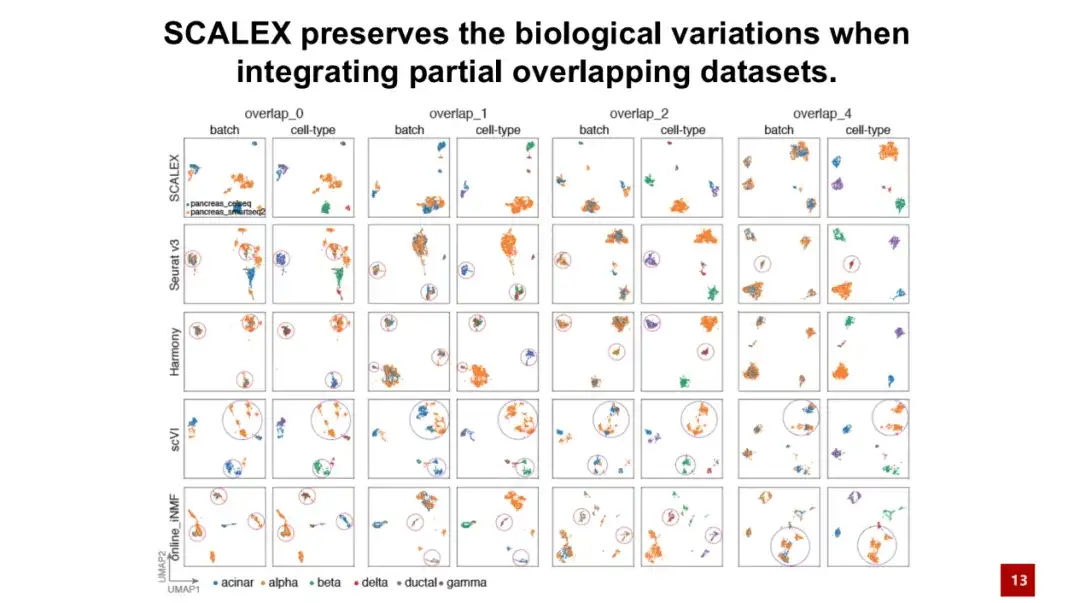
وتظهر النتائج أنه حتى عندما لا تحتوي الدفعتان من الخلايا على أنواع خلايا متطابقة على الإطلاق، فإن SCALEX لا يزال قادرًا على الحفاظ على الاختلافات البيولوجية بشكل جيد، أي أن SCALEX لن يدمج خلايا من أنواع خلايا مختلفة معًا بالقوة، في حين أن طرق أخرى مماثلة قد تعتمد على إيجاد أزواج الخلايا، مما يؤدي إلى الإفراط في التصحيح.
ميزة أخرى لـ SCALEX هي أن المشفر العالمي يمكنه إسقاط بيانات جديدة مباشرة في المساحة الكامنة الموجودة للخلايا دون تأثيرات الدفعة، ودون إعادة تدريب النموذج.كما هو موضح في الشكل أدناه، يتم تدريب أطلس الخلايا المرجعي أولاً باستخدام مجموعات بيانات البنكرياس، ثم يتم إسقاط البيانات الثلاثة الجديدة مباشرة في المساحة الكامنة للخلية من خلال المبرمج المدرب. تمثل الألوان في الشكل أنواع الخلايا، وتمثل النقاط الرمادية أنواع الخلايا المرجعية التي تم إنشاؤها. يمكن أن نجد أن أنواع الخلايا المختلفة منفصلة جيدًا في مواقعها الخاصة في الشكل.
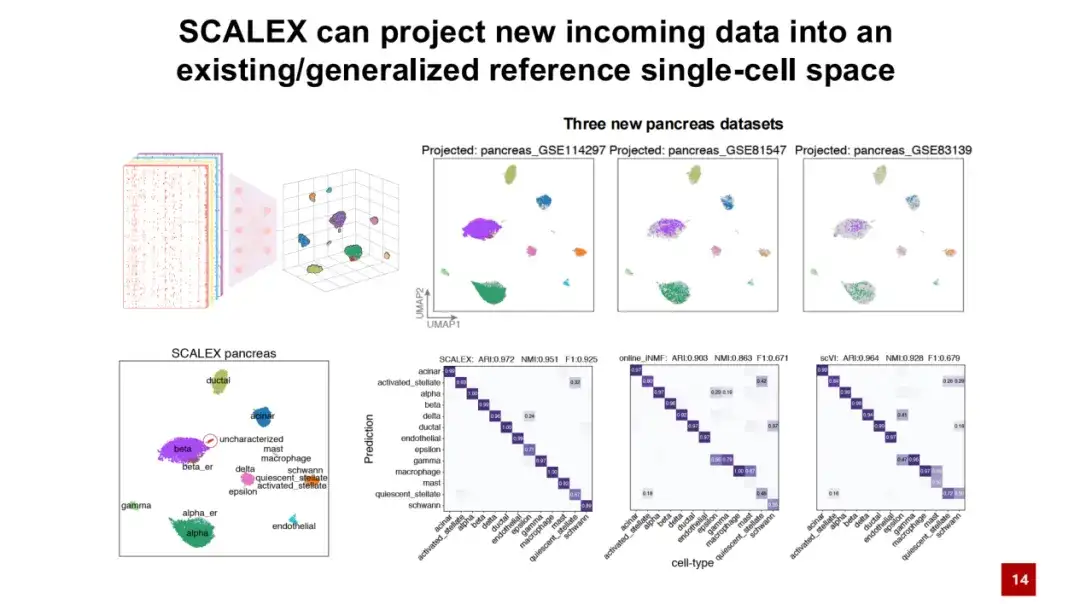
من خلال إسقاط تسميات الخلايا المرجعية حول موقع محدد على خلايا بيانات جديدة، يمكننا أن نرى أن SCALEX يعمل بشكل جيد في التعليق التلقائي على أنواع الخلايا. بالمقارنة مع الطرق الأخرى الموجودة، فإن SCALEX لديه تطبيق مهم للغاية.وهذا يعني أنه يمكن إسقاط البيانات الجديدة بشكل مباشر على البيانات المبنية، مما يساعدنا على إجراء تحليل مقارن بين البيانات.
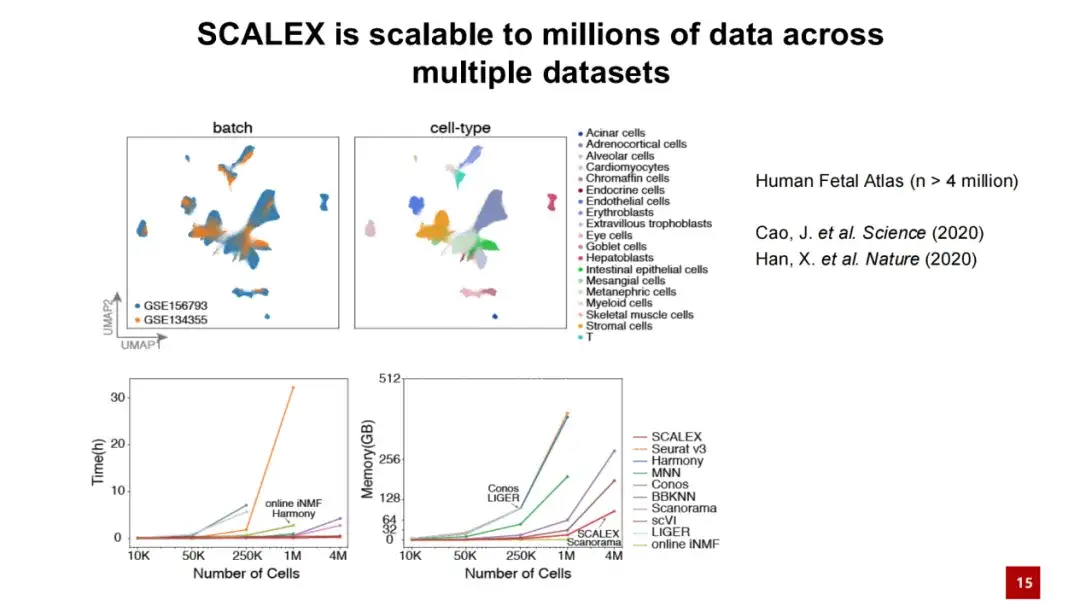
بالإضافة إلى ذلك، يتميز SCALEX أيضًا بأداء جيد في معالجة البيانات واسعة النطاق. يوضح الشكل أدناه أنه عندما تقوم SCALEX بمعالجة 4 ملايين بيانات خلية، فإن وقت الحساب لا يتجاوز عشرات الدقائق ويكون استهلاك الذاكرة أقل من 100 جيجابايت. يوضح هذا أن SCALEX يتمتع بقدرة جيدة على التوسع ويمكن استخدامه للتحليل المتكامل لبيانات الخلية الواحدة واسعة النطاق للغاية.
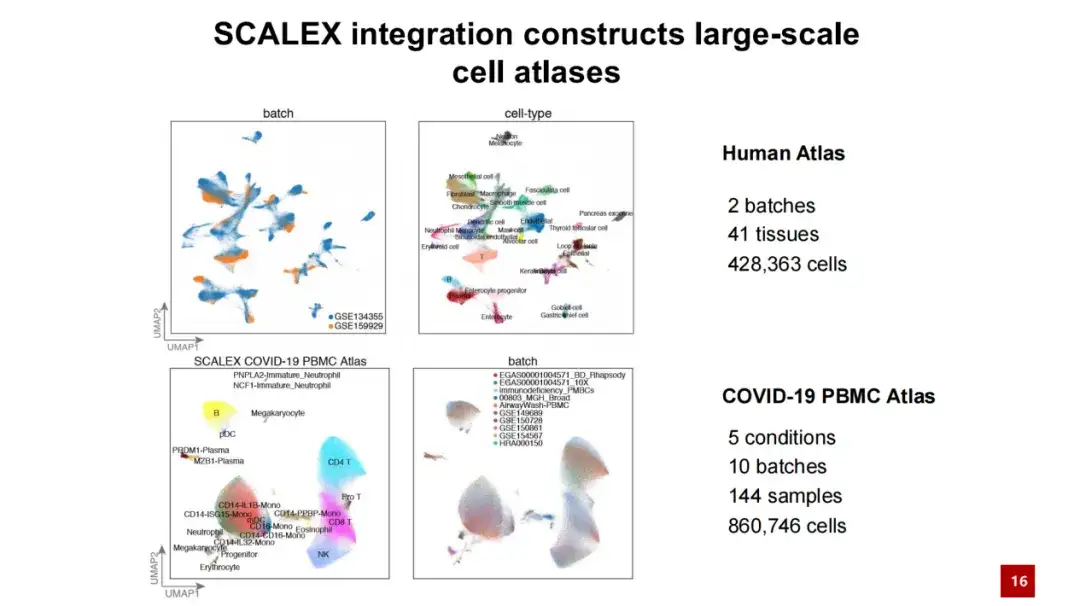
بالاستفادة من SCALEX، قمنا بإنشاء اثنين من أطلس الخلايا على نطاق واسع، الأول هو أطلس خلايا للأفراد من البشر، ويحتوي على أكثر من 400000 خلية؛ والآخر هو أطلس خلايا PBMC لـ COVID-19، والذي يحتوي على أكثر من 860.000 خلية وأكثر من 100 عينة.
SPACE: أداة تحليل الذكاء الاصطناعي لبيانات النسخ المكانية
بعد ذلك، سأقدم لك أداة تحليل النسخ المكاني SPACE التي نشرها فريقي مؤخرًا.
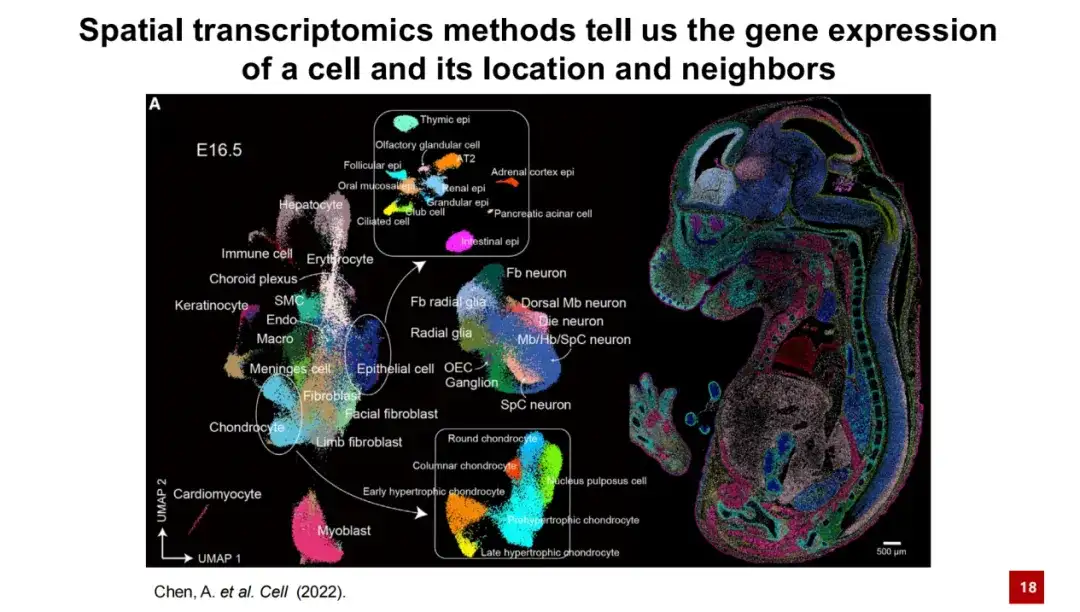
وبعبارة بسيطة، يمكن لتكنولوجيا النسخ المكاني أن توفر معلومات حول التعبير الجيني للخلايا وموقعها المحدد في الفضاء. يوضح الشكل أدناه نتيجة النسخ المكاني النموذجية. في الشكل الموجود على اليسار، تمثل كل نقطة خلية، ويشير اللون إلى نوع الخلية. تم تجميع هذه الخلايا عن طريق تقليل أبعاد التعبير الجيني لتشكيل خريطة UMAP. في اللوحة اليمنى، يظهر الموضع المكاني الفعلي لكل خلية في بيانات جنين الفأر E16.5. ومن الواضح أن التوزيع المكاني للخلايا يتمتع بخصوصية جيدة.
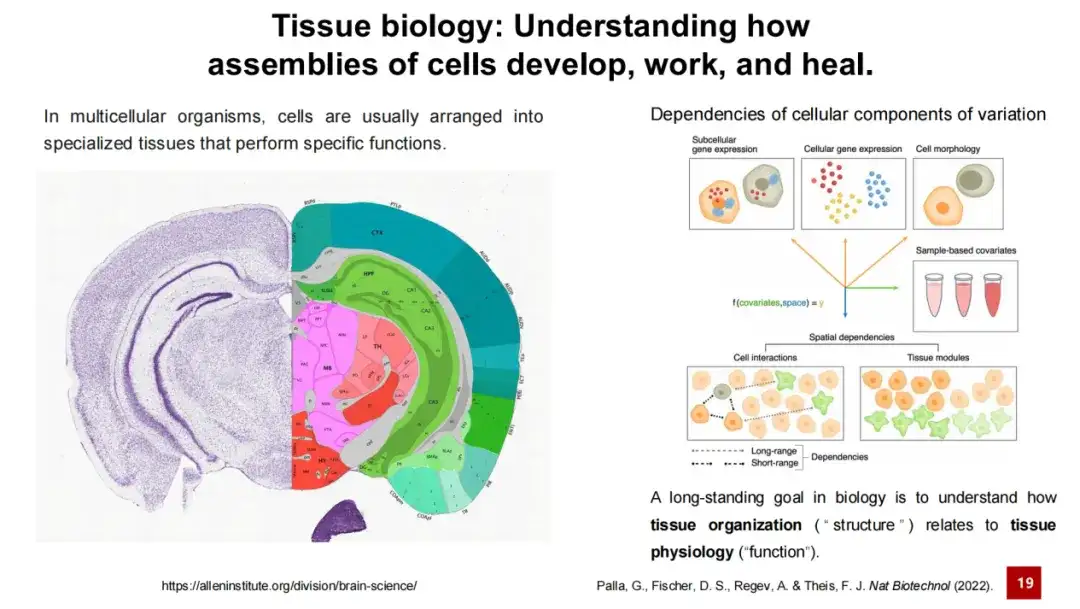
لقد كان البحث التنظيمي دائمًا أحد القضايا الأساسية في أبحاث علوم الحياة. يمكن القول أن أحد الأهداف طويلة المدى للبحث البيولوجي هو فهم العلاقة بين بنية المنظمة ووظيفتها. من السهل أن نفهم هذا. على سبيل المثال، تتكون مناطق الدماغ المختلفة من خلايا عصبية مختلفة وخلايا داعمة، والتي تؤدي وظائف مختلفة من خلال تفاعلات معقدة بين الخلايا. على سبيل المثال، بعض المناطق مسؤولة عن الذاكرة، وبعضها مسؤول عن التعلم، وبعضها مسؤول عن الاستجابات الحركية.
لذلك،إن إحدى القضايا الأساسية في تحليل النسخ المكاني هي تحديد أنواع مختلفة من الخلايا أو وحدات الأنسجة في الفضاء، وهي المهمة التي يشار إليها بشكل جماعي باسم التجميع المكاني.
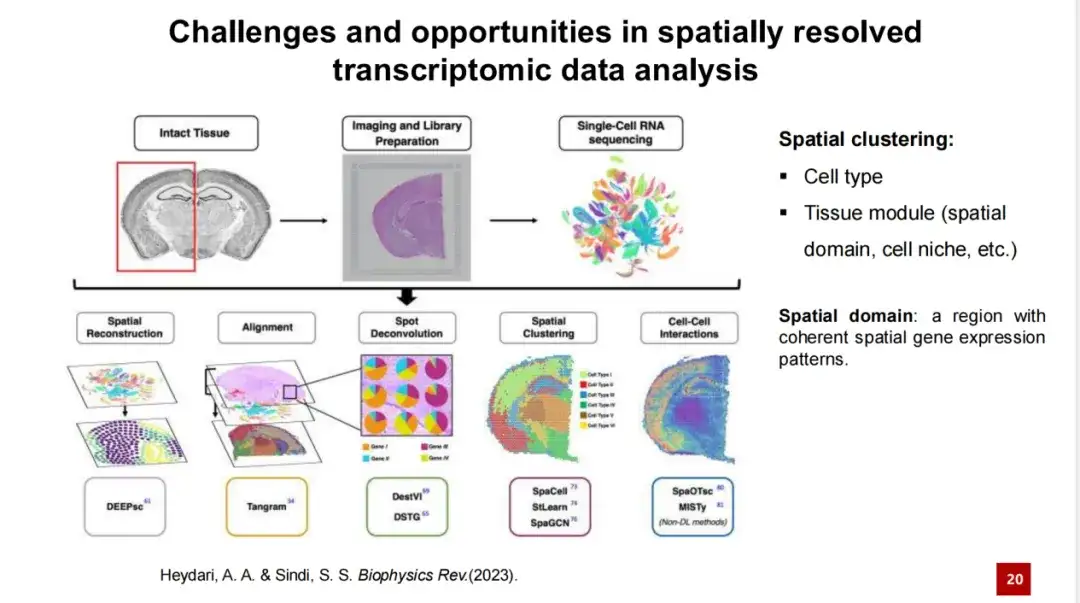
تتكون هذه المهمة من مهمتين فرعيتين: الأولى هي تحديد نوع الخلية، والثانية هي تحديد وحدة الأنسجة.. الطريقة الأولى أكثر بديهية، وهي تحديد أنواع مختلفة من الخلايا في بيانات النسخ المكاني، كما هو موضح في بيانات جنين الفأر؛ في حين أن الأخير مجرد نسبيًا، ويتضمن تحديد مناطق داخل الأنسجة أصغر من بنية الأنسجة، والتي قد تكون لها وظائف محددة أو تتكون من خلايا.
في دراسات مختلفة، يطلق الباحثون أسماء مختلفة على وحدات الأنسجة، مثل المجال المكاني أو مكانة الخلية، ومن بينها مصطلح المجال المكاني الأكثر استخدامًا. يعتقد بعض الباحثين أن تحديد الوحدات التنظيمية يعني تحديد المناطق ذات خصائص التعبير الجيني المكاني المتسقة.
ومع ذلك، فإن هذا المفهوم له حدود. على سبيل المثال، يوضح الشكل أ أدناه أن هناك فرقًا كبيرًا في التعبير الجيني بين منطقتين، ولكن في الشكلين ب وج، فإن توزيع التعبير الجيني بين المناطق ليس نظيفًا تمامًا وقد يكون مربكًا. يوضح الشكلان (ب) و(ج) المواقف التي لا يستطيع مفهوم المجال المكاني حلها.

ولمعالجة هذه المشكلة، نقترح طريقة SPACE.يتم التعامل مع مشكلة المجال المكاني من خلال تعلم تضمينات الخلايا الواعية للتفاعل.
يستخدم SPACE إطار عمل ترميز تلقائي للرسم البياني لتعلم تضمينات الخلايا منخفضة الأبعاد.
أولاً، نقوم بإدخال بيانات النسخ المكاني وإنشاء رسم بياني مجاور بناءً على الموضع المكاني لكل خلية، أي ربط الخلايا المجاورة الأقرب لكل خلية لتشكيل رسم بياني. في الشكل أدناه، تمثل العقد الخلايا، وخصائص العقد هي خصائص التعبير الجيني للخلايا. نقوم بإدخال الرسم البياني المجاور وملفات تعريف التعبير الجيني في مشفر SPACE، والذي يتكون من شبكة GAT ثلاثية الطبقات.
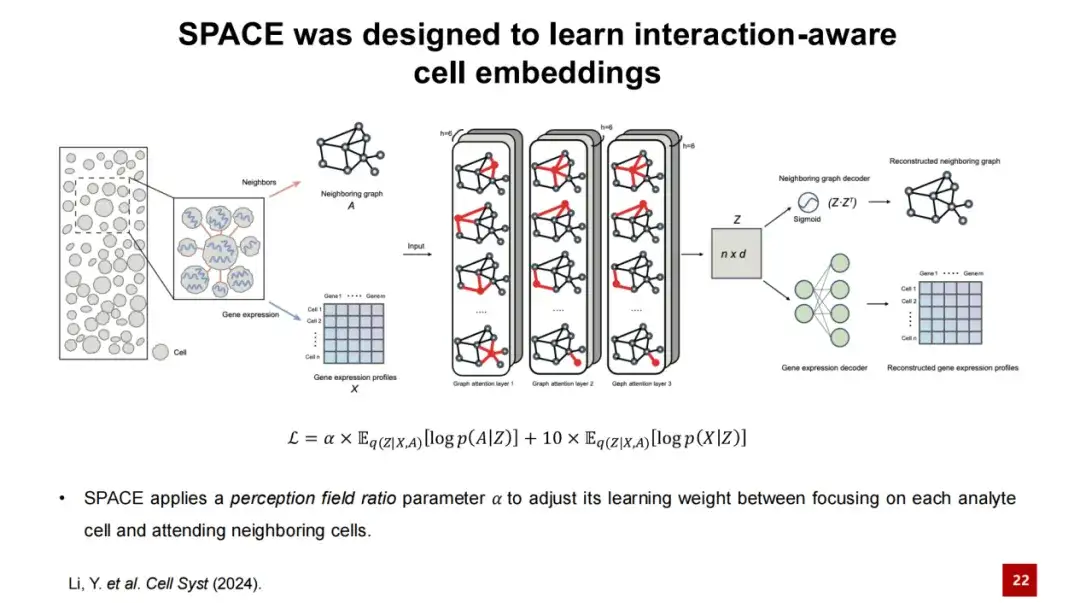
من خلال معالجة المشفر، يمكننا الحصول على التمثيل المضمن لكل عقدة وإعادة بنائه من خلال فكين مستقلين:يعمل أحد فكي التشفير على إعادة بناء تمثيل الطبقة المخفية للخلية ذات الأبعاد المنخفضة إلى رسم بياني مجاور، ويعمل فك التشفير الآخر على إعادة بناء ملف تعريف التعبير الجيني للخلية. دالة الخسارة في نموذج SPACE هي مجموع خسارتي إعادة البناء.
في هذه العملية،لقد قمنا بتصميم معلمة نسبة مجال الإدراك α لضبط أوزان دالتي الخسارة في النموذج.
عندما تكون قيمة α صغيرة،يركز النموذج بشكل أكبر على إعادة بناء التعبير الجيني للخلية نفسها، ويمكن استخدام تضمين الخلية الذي تم الحصول عليه لتحديد نوع الخلية؛عندما تكون قيمة α كبيرة،يركز النموذج بشكل أكبر على التفاعل بين الخلايا، ويمكن استخدام تضمين الخلايا الذي تم الحصول عليه في هذا الوقت لتحديد وحدات الأنسجة. نظرًا لأن تضمين الخلية منخفض الأبعاد Z يحتوي على معلومات حول تفاعلات الخلية، فإننا نطلق على تمثيل التضمين منخفض الأبعاد الذي تم الحصول عليه من خلال تضمينات خلية تفاعل SPACE-awara.
لتحديد الأنواع الفرعية للخلايا المكانية، قمنا بتطبيق SPACE على مجموعة بيانات القشرة الحركية الأساسية للفأر.
في الشكل أدناه، يوضح الركن الأيسر العلوي الموضع المكاني لكل خلية في الأنسجة الفعلية، حيث تمثل النقطة خلية ويشير اللون إلى نوع الخلية. هذه خريطة UMAP تم إنشاؤها بناءً على التعبير الجيني. الشكلان الموجودان في أسفل اليسار يوضحان الأنواع الفرعية للخلايا المكانية التي تم تحديدها بواسطة SPACE ومواقعها في الفضاء. لقد أجرينا تحليل مصفوفة الارتباك لهذه الأنواع الفرعية من الخلايا المكانية مع أنواع الخلايا المقدمة في الدراسة الأصلية (كما هو موضح في الشكل على اليمين)، وأظهرت النتائج أن الاثنين كانا متسقين بشكل عام، مع مؤشر راند المعدل (ARI) بقيمة 0.6. في نفس الوقت، يمكن لـSPACE التمييز بين الخلايا النجمية والخلايا القليلة التغصن بشكل أكثر دقة وتحديد المزيد من الأنواع الفرعية للخلايا.
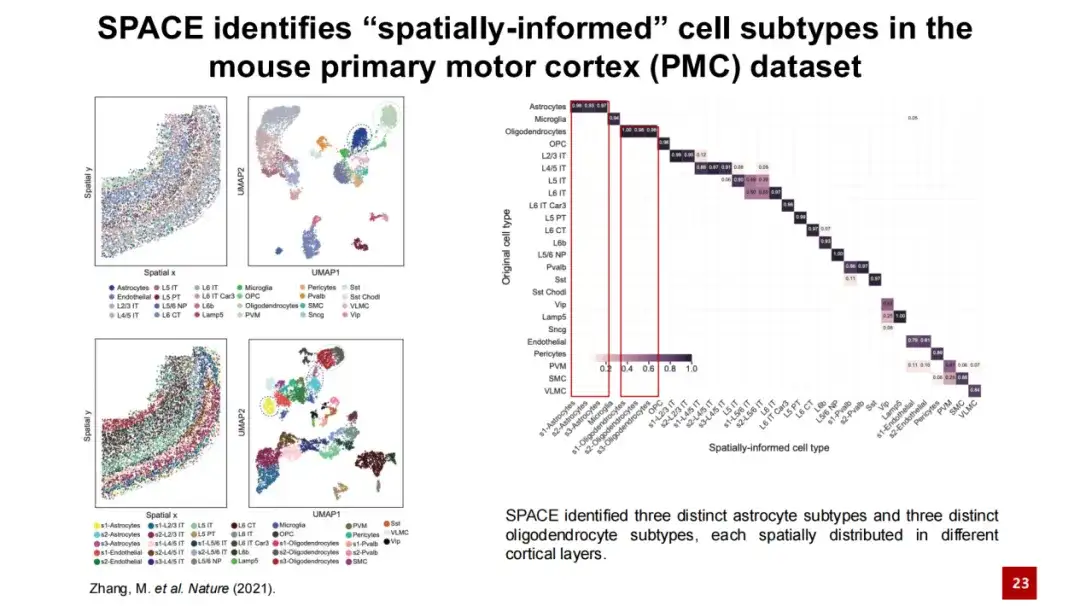
الصورة الموجودة في الأسفل إلى اليسار تظهر البنية التنظيمية للقشرة الحركية الأساسية للفئران. تمثل الطبقة البنية القشرية و WM تمثل المادة البيضاء. يمكن رؤية البنية الطبقية من الطبقة الأولى إلى المادة البيضاء بوضوح. كان من الصعب التمييز بين الأنواع الفرعية الثلاثة للخلايا النجمية التي حددها SPACE من خلال التعبير الجيني وحده، وتم خلطها معًا في خريطة UMAP.
ومع ذلك، تتميز هذه الأنواع الفرعية الثلاثة من الخلايا بوضوح في التوزيع المكاني: يتم توزيع النوع الفرعي للخلايا s1 بشكل أساسي في المنطقة من الطبقة 1 إلى الطبقة 4، ويتم توزيع النوع الفرعي للخلايا s2 بشكل أساسي في المنطقة من الطبقة 5 إلى الطبقة 6، ويتم توزيع النوع الفرعي s3 بشكل أساسي في المادة البيضاء. لقد قمنا بحساب نسب أنواع الخلايا المحيطة بهذه الأنواع الفرعية الثلاثة من الخلايا النجمية، وكانت النتائج متوافقة مع قاعدة التقسيم الطبقي هذه. على الرغم من أن الأنواع الفرعية الثلاثة من الخلايا كانت متشابهة في التعبير الجيني، إلا أنها لا تزال تظهر جينات التعبير العالي الخاصة بها.

إن الأنواع الفرعية الثلاثة للخلايا النجمية التي تم تحديدها بواسطة SPACE تتفق إلى حد كبير مع الدراسات السابقة. وأشارت دراسات سابقة إلى وجود تفاعل بين الخلايا النجمية والخلايا العصبية، وأن التقسيم الطبقي للخلايا النجمية يتوافق مع التقسيم الطبقي للخلايا العصبية. ومن خلال القضاء على العوامل الرئيسية في الخلايا العصبية، وجد الباحثون أن البنية الطبقية للخلايا العصبية قد دمرت، وأن البنية الطبقية للخلايا النجمية تغيرت أيضًا وفقًا لذلك. ويشير هذا إلى وجود تفاعل مكاني محدد وتنظيم جيني مكاني محدد بين الخلايا النجمية والخلايا العصبية.
ومن هذا المثال يمكننا أن نرى أنيمكن لـSPACE الاستفادة بشكل فعال من المعلومات المكانية وتحديد أنواع الخلايا البيولوجية المختلفة بدقة مع الخصائص المكانية.
قدمت المقالة السابقة أن SPACE يغير اتجاه تحسين النموذج عن طريق ضبط معلمة نسبة مجال الإدراك α: يمكنه إيلاء المزيد من الاهتمام لخصائص الخلايا نفسها لتحديد أنواع الخلايا، أو إيلاء المزيد من الاهتمام لمعلومات التفاعل بين الخلايا لاكتشاف وحدات الأنسجة.
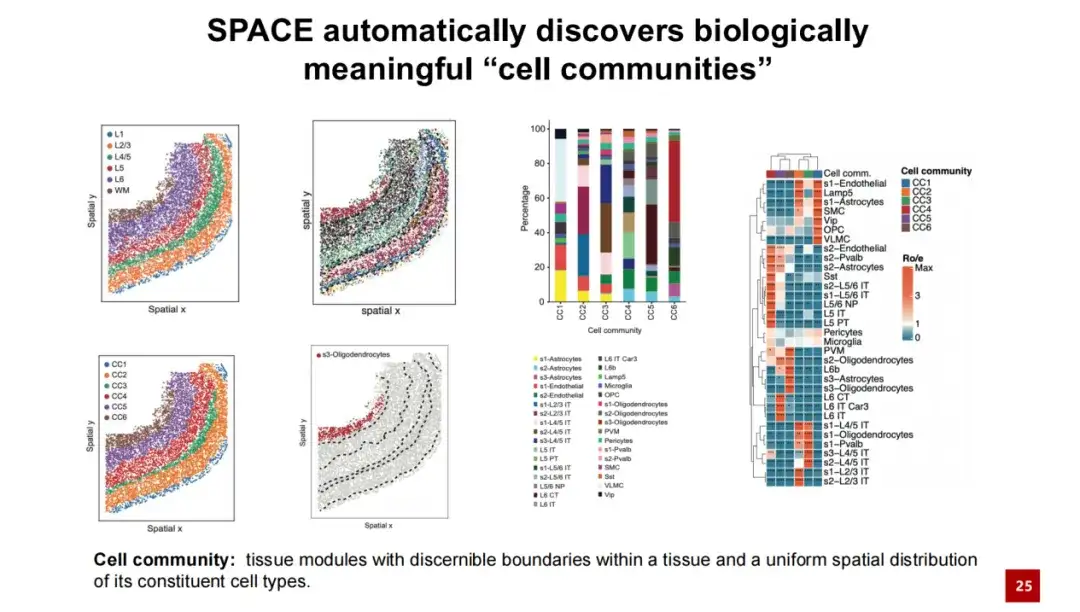
في نفس مجموعة البيانات،من خلال زيادة قيمة α، نجح SPACE في اكتشاف وحدة الأنسجة.أطلقنا عليها اسم مجتمعات الخلايا (CC للاختصار). نحن نعتقد أن وحدات الأنسجة التي اكتشفها SPACE لها حدود يمكن تحديدها وأن التوزيع المكاني لأنواع الخلايا داخلها موحد ومتسق نسبيًا. لقد قمنا بمقارنة مجتمعات الخلايا التي اكتشفها SPACE مع هياكل الأنسجة الموجودة ووجدنا أن الاثنين لديهما تطابق جيد واحد لواحد. تحتوي كل مستعمرة خلوية على أنواع مختلفة من الخلايا، والتوزيع المكاني لهذه الأنواع من الخلايا داخل مستعمرة الخلايا موحد نسبيًا.
لقد قمنا بمقارنة SPACE بالطرق الحالية لاكتشاف الوحدات التنظيمية وأجرينا اختبارات على خمس مجموعات بيانات.تظهر النتائج أن SPACE يتفوق على أفضل الطرق الموجودة في مجموعتين من البيانات ويؤدي بشكل مماثل لأفضل الطرق في مجموعات البيانات الثلاث الأخرى.لقد أجرينا أيضًا اختبارات وتحليلات على مجموعة بيانات الدماغ البشري Visium المستخدمة بشكل شائع، وأظهرت النتائج أن SPACE ينطبق أيضًا على بيانات النسخ المكاني دون دقة الخلية الفردية.
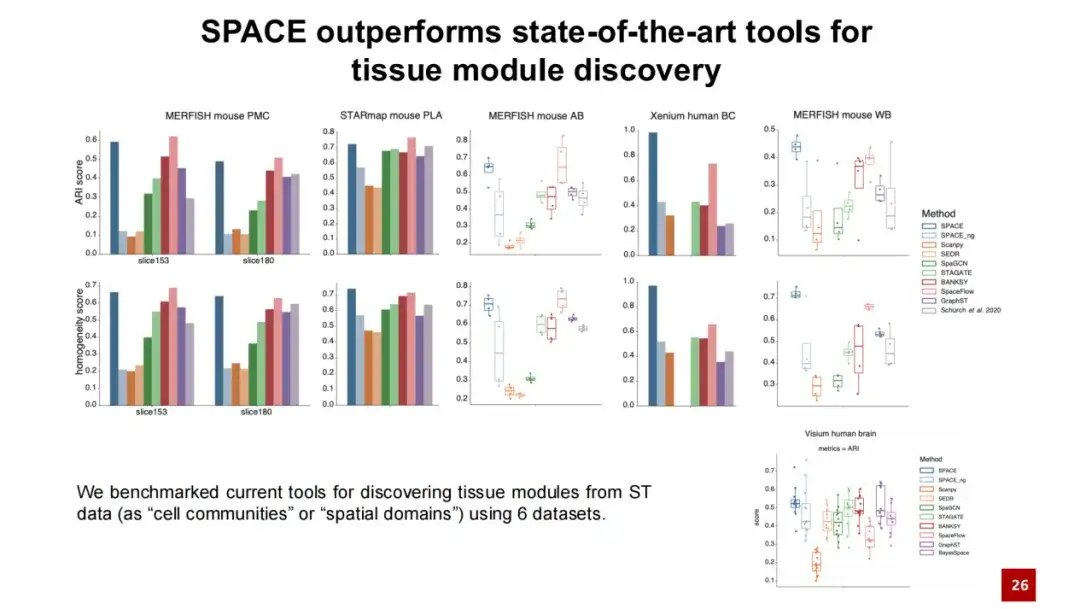
بالإضافة إلى ذلك، نقدم نموذج اختبار يسمى SPACE_ng، حيث يشير ng إلى أننا قمنا بإيقاف تشغيل خسارة إعادة بناء الرسم البياني المجاور في نموذج SPACE. تظهر النتائج أن أداء SPACE_ng أدنى بكثير من أداء SPACE.
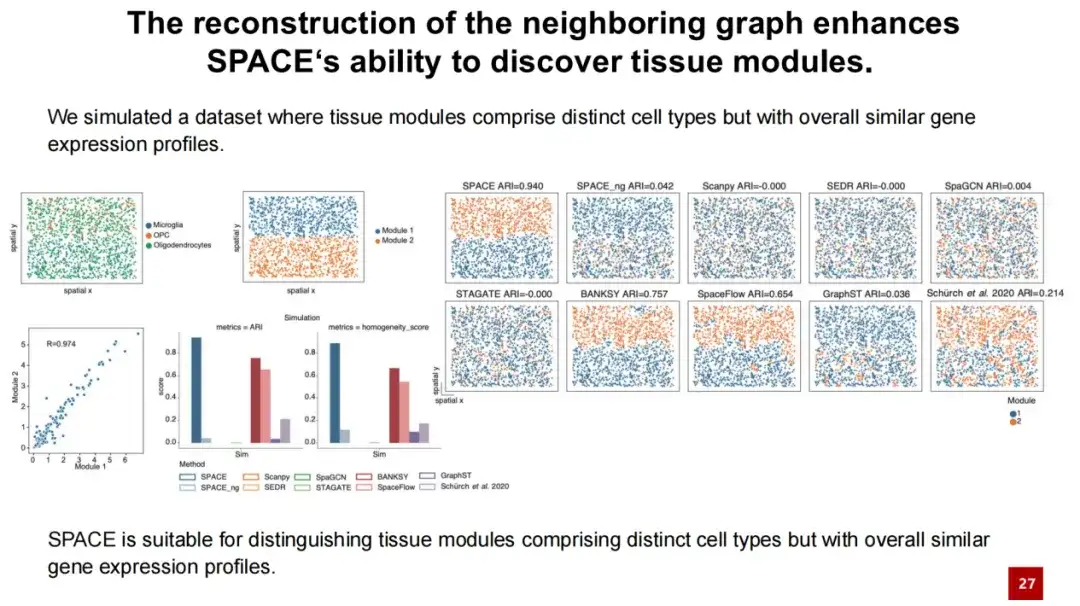
ولتوضيح أن SPACE يمكنه اكتشاف أداء الوحدات التنظيمية من خلال إعادة بناء الرسم البياني للحي، قمنا بتصميم تجربة محاكاة. لقد قمنا باختيار الخلايا القليلة التغصنات والخلايا الدبقية الصغيرة وخلايا OPCs الموزعة بالتساوي بين الخلايا القليلة التغصنات (انظر الصورة العلوية اليسرى أدناه)، لتشكيل وحدتين من الأنسجة.
نظرًا لأن معظم الخلايا في هاتين الوحدتين النسيجيتين هي خلايا قليلة التغصن ولديها تشابه كبير جدًا (التعاون = 0.97)، تظهر نتائج الاختبار أن SPACE متفوقة كثيرًا على الطرق الأخرى، في حين لا يمكن لـ SPACE_ng التمييز بين الوحدتين النسيجيتين.يشير هذا إلى أن أداء وحدة التعرف على الأنسجة SPACE ينبع من إعادة بناء الرسم البياني المجاور.
لقد لاحظنا ظاهرة مماثلة في التحليل النهائي، أي أن خصائص مجتمعات الخلايا التي تم تحديدها بواسطة SPACE لا تتجلى ببساطة في شكل تعبير جيني مكاني متسق كما هو الحال في المجالات المكانية، ولكنها تعكس تفاعلات مماثلة بين الخلايا المجاورة.
في خريطة الحرارة أدناه، يمثل كل عمود خلية، ويشير لونه إلى مجموعة الخلايا التي تنتمي إليها الخلية ونوع الخلية. يمثل كل صف نوع خلية ويظهر التردد النسبي للتفاعلات المجاورة بين هذا النوع من الخلايا والخلايا الأخرى. من خلال خريطة الحرارة هذه، يمكننا أن نرى أن الخلايا التي تنتمي إلى نفس مجتمع الخلايا تظهر أوجه تشابه في التفاعلات المجاورة، وهذا التشابه مستقل عن النوع المحدد من الخلايا. وعلى النقيض من ذلك، أظهرت الخلايا التي تنتمي إلى مجموعات خلوية مختلفة اختلافات أكبر في تفاعلاتها مع جيرانها.
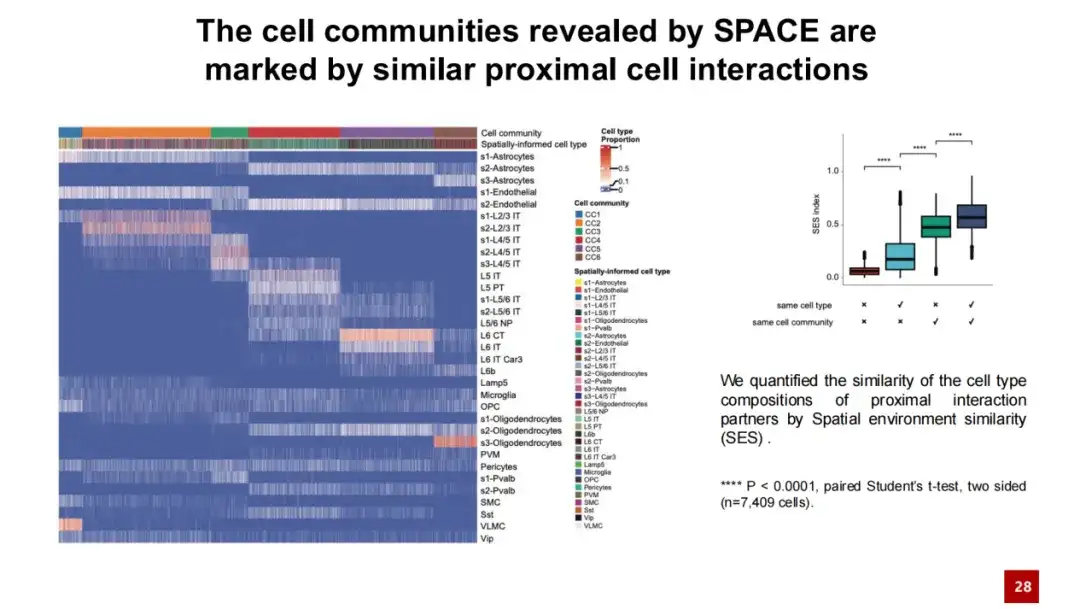
قمنا أيضًا بحساب التشابه في التفاعلات بين الخلايا كميًا باستخدام تشابه جيب التمام. وأظهرت النتائج أن الخلايا من نفس المجموعة الخلوية كانت متشابهة إلى حد كبير في تفاعلاتها مع الخلايا المجاورة، في حين أن الخلايا من مجموعات خلوية مختلفة كانت لها تفاعلات خارج الخلية مختلفة نسبيًا. وتشير هذه النتائج إلى أنإن المجتمعات الخلوية التي اكتشفها SPACE ليست مجرد نمط تعبير جيني مكاني، بل إنها تتأثر أيضًا بشبكة التفاعل بين الخلايا القريبة.
لقد أجرينا تحليلًا مشابهًا في مجموعة بيانات أخرى لمشيمة الفأر. تُظهر الصورة اليسرى الموقع المكاني لكل نوع من أنواع الخلايا في مجموعة البيانات، والصورة الموجودة في المنتصف الأيسر هي بنية أنسجة المشيمة للفأر التي تم شرحها يدويًا، وتُظهر الصورة الموجودة في المنتصف الأيمن مجموعات الخلايا الخمس التي اكتشفها SPACE. ومن الممكن ملاحظة وجود تطابق جيد واحد لواحد بين مجتمعات الخلايا التي اكتشفها SPACE والهياكل النسيجية التي تم شرحها يدويًا. لقد قمنا ببناء شبكة تفاعل خلوي قريبة مميزة لكل مجموعة من الخلايا، كما هو موضح في الشكل الموجود على اليمين، والذي يوضح التفاعلات الفريدة بين الخلايا داخل كل مجموعة من الخلايا.

إذا أخذنا CC1 كمثال، فإن المجتمع يقع بشكل أساسي في منطقة ديسيدوا الأمومية. وجدنا أنه في CC1، هناك تفاعل قوي بين الخلايا الأمومية المقطوعة الرأس S2 وخلايا الغلايكوتروفوبلاست S2. أظهرت دراسات سابقة أنه أثناء الحمل لدى الفئران، تغزو الخلايا الغاذية السكرية منطقة المشيمة الأمومية وتتفاعل مع خلايا المشيمة الأمومية الموجودة فيها، مما يؤدي إلى إعادة تشكيل الشرايين التي تحمل الدم الأمومي إلى المشيمة، وهي عملية بالغة الأهمية للحمل الطبيعي.
ومن التحليل أعلاه، يمكننا أن نستنتج أنيمكن لـSPACE تحديد التفاعلات بين الخلايا في العينات البيولوجية التي لها تأثيرات مهمة على العمليات الحيوية.لذلك فإننا نتكهن بأنيمكن استخدام شبكات التفاعل التي تم إنشاؤها بواسطة SPACE لتحسين تحليل الاتصالات الخلوية القائمة على الربيطة والمستقبل.
يعد تحليل الاتصال الخلوي القائم على الربيطة والمستقبل طريقة شائعة في تحليل بيانات الخلية الفردية، أي بناءً على التعبير الجيني للربيطة والمستقبلات في خليتين، يتم استنتاج إمكانية اتصال الخلايا من خلال أزواج الربيطة والمستقبل. لقد قمنا أولاً بتحليل الاتصالات بين الخلايا في CC1 باستخدام CellChat، وهي طريقة شائعة الاستخدام لتحليل الاتصالات بين الخلايا، في مجموعة بيانات المشيمة الخاصة بالفئران.
وجدت CellChat أن الخلايا الأمومية المقطوعة s3 يمكنها التواصل مع أنواع خلايا P-TGC من خلال مسارات الإشارة مثل الكولاجين FN1 وTHBS. ومع ذلك، تتطلب جميع مسارات الإشارة هذه اتصالاً جسديًا حتى تحدث فعليًا. ومع ذلك، وجدنا أن النوعين من الخلايا متباعدان للغاية في التوزيع المكاني (انظر الزاوية اليمنى السفلية من الشكل أدناه)، مما يجعل من غير المحتمل أن يلتقيا فعليًا.
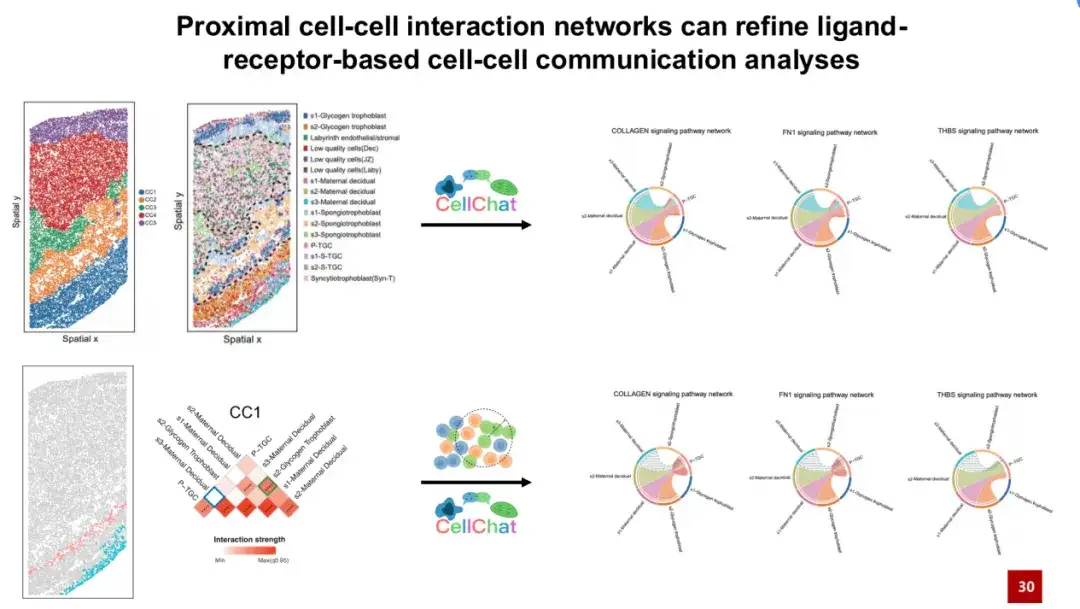
وقد تم تأكيد ذلك أيضًا من خلال شبكة التفاعل بين الخلايا القريبة التي تم إنشاؤها في CC1. تظهر المربعات الزرقاء أن التفاعلات بينهما غير محتملة.إن إدخال شبكة التفاعل الخلوية القريبة المميزة التي أنشأها SPACE في تحليل اتصالات الخلايا CellChat يمكن أن يساعدنا في استبعاد إشارات اتصالات الخلايا التي من المستحيل حدوثها فعليًا في الفضاء، وبالتالي تقليل الإشارات الإيجابية الخاطئة بشكل فعال.
الوظائف
أنشأت جامعة تسينغهوا والمختبر الرئيسي للدولة لعلم الأحياء الغشائي فرعًا لعلم بنية الغشاء وبيولوجيا الذكاء الاصطناعي في هانغتشو. ويقوم الفريق حاليًا بتجنيد متخصصين منخرطين في أبحاث متعددة التخصصات بين الذكاء الاصطناعي وعلم الأحياء. ونحن ندعو بصدق الباحثين المهتمين بهذا المجال للانضمام إلى فريقنا. لمزيد من تفاصيل التوظيف، يرجى مسح رمز الاستجابة السريعة أدناه للحصول على مزيد من المعلومات.









