Command Palette
Search for a command to run...
Selected for ACL 2024! To Achieve cross-modal Interpretation of Protein Data and Text Information, Wang Xiang's Team From USTC Proposed a protein-text Generation Framework ProtT3

Exploring the mysteries of protein dynamic structure is not only a key step in promoting the development of new drugs, but also an important cornerstone for understanding life processes. However, the complexity of proteins makes it difficult to directly capture and analyze their deep structural information. How to convert complex biological data into intuitive and easy-to-understand expressions has always been a major problem in the field of scientific research.
With the rapid development of language models (LM), an innovative idea came into being:Since language models can learn and extract text information from large amounts of data, can they learn to “read” protein information from protein data and directly convert dynamic protein structure information into text descriptions that are easy for humans to understand?
This promising idea has encountered many challenges in practical application. For example, language models pre-trained on a text corpus of protein sequences have strong text processing capabilities, but they are unable to understand the non-human "language" of protein structure. In contrast, protein language models (PLMs) pre-trained on a corpus of protein sequences have excellent protein understanding and generation capabilities.But its limitation is equally significant - lack of text processing capabilities.
If we can combine the advantages of PLMs and LM to build a new model architecture that can not only deeply understand protein structure but also seamlessly connect text information, it will have a profound impact on drug development, protein property prediction, molecular design and other fields.Protein structure and human language text belong to different data modalities, and it is not easy to break through the barriers and merge them.
In this regard,Wang Xiang from the University of Science and Technology of China, together with Liu Zhiyuan's team from the National University of Singapore and the research team from Hokkaido University, proposed a new protein-text modeling framework ProtT3.The framework combines the modality-differentiated PLM and LM through a cross-modal projector, where PLM is used for protein understanding and LM is used for text processing. To achieve efficient fine-tuning, the researchers incorporated LoRA into the LM, effectively regulating the protein-to-text generation process.
In addition, the researchers also established quantitative evaluation tasks for protein-text modeling tasks, including protein captioning, protein question answering (protein QA), and protein-text retrieval. ProtT3 achieved excellent performance in all three tasks.
The research, titled "ProtT3: Protein-to-Text Generation for Text-based Protein Understanding", was selected for the top conference ACL 2024.
Research highlights:
* ProtT3 framework can bridge the modality gap between text and protein and improve the accuracy of protein sequence analysis
* In the protein captioning task, ProtT3's BLEU-2 score on Swiss-Prot and ProteinKG25 datasets is more than 10 points higher than the baseline
* In the protein question answering task, ProtT3's exact matching performance on the PDB-QA dataset improved by 2.5%
* In the protein-text retrieval task, ProtT3's retrieval accuracy on Swiss-Prot and ProteinKG25 datasets is more than 14% higher than the baseline
Paper address:
https://arxiv.org/abs/2405.12564
Dataset download address:
https://go.hyper.ai/j0wvp
The open source project "awesome-ai4s" brings together more than 100 AI4S paper interpretations and provides massive data sets and tools:
https://github.com/hyperai/awesome-ai4s
Construction and optimization of three major datasets for protein research
The researchers selected three data sets: Swiss-Prot, ProteinKG25, and PDB-QA.
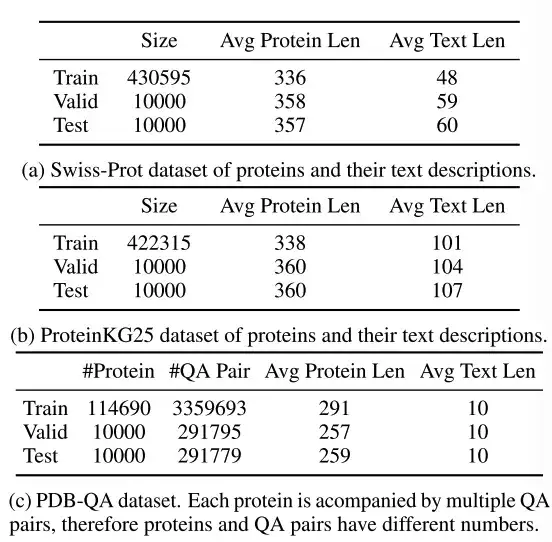
As shown in the table above,Swiss-Prot is a protein sequence database with text annotations.The researchers processed the dataset and excluded protein names from the text annotations to prevent information leakage. The generated text descriptions connect the annotations of protein function, location, and family.
ProteinKG25 is a knowledge graph derived from the Gene Ontology database.The researchers first aggregated triplets of the same protein and then filled the protein information into a predefined text template to convert its triplets into free text.
PDB-QA is a protein single-turn question answering dataset derived from RCSB PDB2.Contains 30 question templates about protein structure, properties, and supplementary information. As shown in the table below, for fine-grained evaluation, the researchers divided the questions into 4 categories based on the format of the answer (string or number) and content focus (structure/property or supplementary information).

ProtT3: An innovative protein-to-text generation model architecture
As shown in Figure a below,ProtT3 consists of a protein language model (PLM), a cross-modal projector (Cross-ModalProjector), a language model (LM), and a LoRA module.Effectively regulate the protein-to-text generation process.
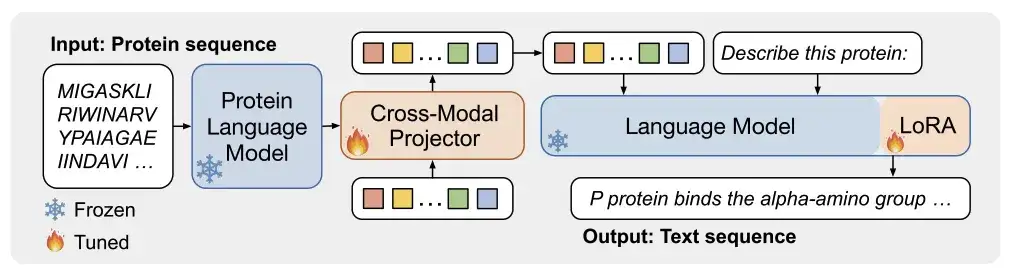
Among them, the protein language model selected by the researchers is ESM-2150M, which is used for protein understanding; the cross-modal projector selected is Q-Former, which is used to bridge the modal differences between PLM and language model LM, and then map the protein representation to the text space of LM; the language model selected is Galactica1.3B, which is used for text processing; in order to maintain the efficiency of downstream adaptation, the researchers also incorporated LoRA into the language model to achieve efficient fine-tuning.
As shown in Figure b,ProtT3 uses two training phases to enhance the effective modeling of protein text.They are Protein-Text Retrieval Training and Protein-to-Text Generation Training.
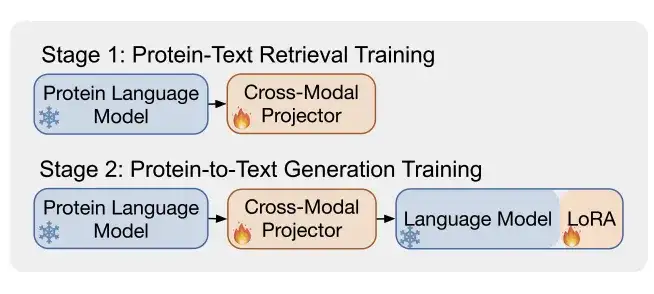
* Phase 1: Protein-text retrieval training
As shown in Figure a below, the cross-modal projector Q-Former consists of two transformers: Protein transformers for protein encoding and Text transformers for text processing. The two transformers share self-attention to achieve interaction between protein and text.
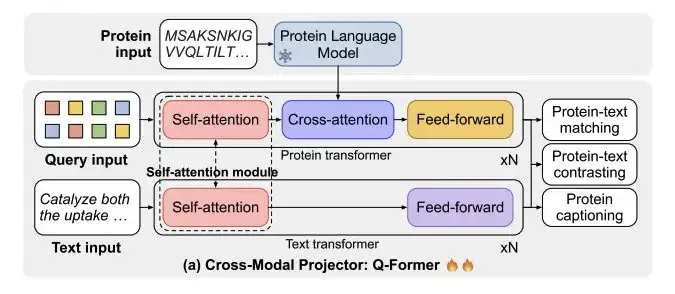
The researchers trained ProtT3 on a combined dataset of Swiss-Prot and ProteinKG25 for protein-text retrieval.It involves three tasks: protein-text contrasting, protein-text matching (PTM) and protein captioning (PCap).
* Phase 2: Protein-to-text generation training
The researchers connected the cross-modal projector to a language model (LM) and fed the protein representation Z into the language model in order to regulate the text generation process through protein information. Among them, the researchers used a linear layer to project Z to the same dimension of the language model input, trained ProtT3 for each generated dataset separately, and added different text prompts after the protein representation to further control the generation process.
In addition, the researchers introduced LoRA and fine-tuned it individually on 3 datasets on the protein-to-text generation task.
An all-rounder in the protein field, evaluating the performance of ProtT3 in 3 major tasks
To evaluate the performance of ProtT3,The researchers tested the system in three tasks: protein captioning, protein QA, and protein-text retrieval.
Closer to the true description of proteins, ProtT3 has a higher accuracy
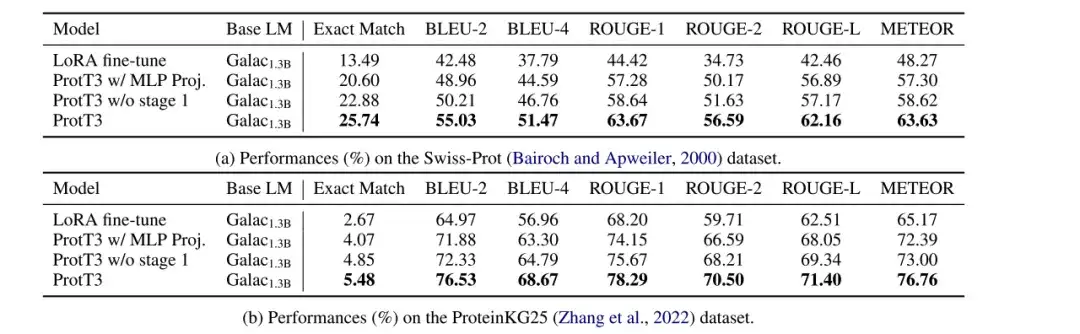
The researchers evaluated the performance of LoRA fine-tuned Galactica1.3B, ProtT3 w/ MLP Proj., ProtT3 w/o stage 1, and ProtT3 models on protein captioning tasks on the Swiss-Prot and ProteinKG25 datasets, and used BLEU, ROUGE, and METEOR as evaluation metrics.
* ProtT3 w/ MLP Proj.: A variant of ProtT3 that replaces ProtT3's cross-modal projector with MLP
* ProtT3 w/o stage 1: A variant of ProtT3 that skips the training stage 1 of ProtT3
As shown in the figure below, compared with LoRA fine-tuned Galactica1.3B,ProtT3 improves BLEU-2 score by more than 10 points.The importance of introducing protein language model and the effectiveness of ProtT3 in understanding protein input are intuitively demonstrated. In addition, different indicators of ProtT3 are better than its two variants, which shows the advantages of using Q-Former projector and training stage 1.
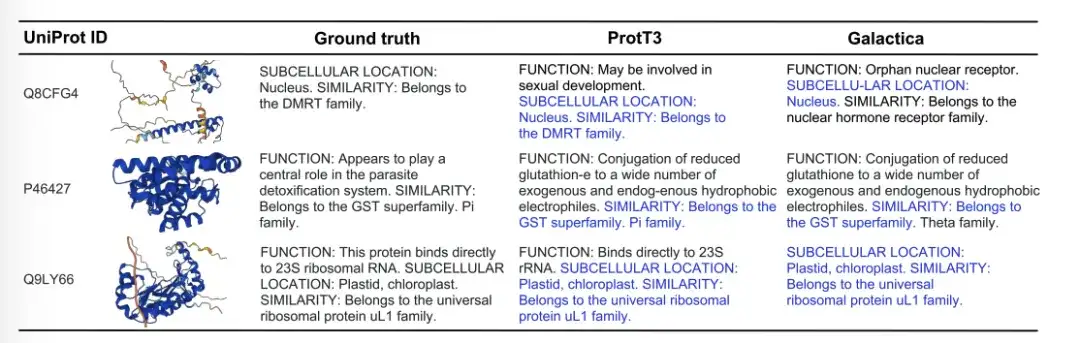
The figure below shows three examples of protein caption generation by Ground truth, ProtT3, and Galactica. In the Q8CFG4 example, ProtT3's annotation content more accurately identified the DMRT family, while Galactica did not. In the P46427 example, both models failed to identify the function of the protein, but ProtT3 predicted the protein family more accurately. In the Q9LY66 example, both models successfully predicted the subcellular location and protein family. ProtT3 goes a step further in predicting the function of proteins, which is closer to the true description.
The accuracy is 141% higher than the baseline model. TP3T, ProtT3 has better protein-text retrieval ability
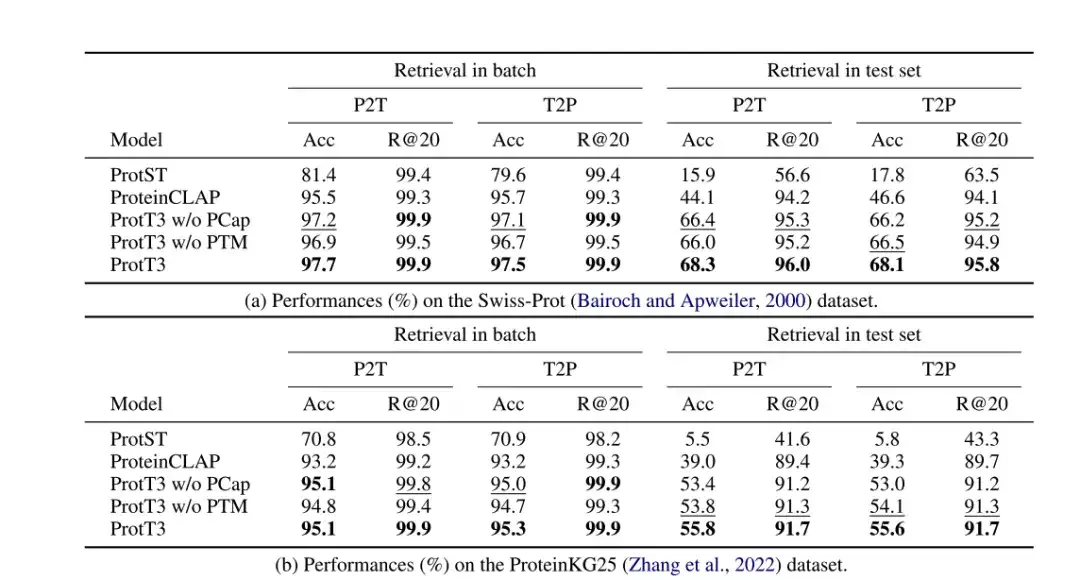
The researchers evaluated the performance of ProtT3 in protein-text retrieval on the Swiss-Prot and ProteinKG25 datasets, using accuracy and Recall@20 as evaluation metrics, and adopted ProtST and ProteinCLAP as baseline models.
As shown in the following table,The accuracy of ProtT3 is more than 14% higher than the baseline model.This indicates that ProtT3 is better at aligning proteins with their corresponding text descriptions.Protein-text matching (PTM) improved the accuracy of ProtT3 by 1%-2%,This is because PTM allows protein and text information to interact in the early layers of Q-Former, thus achieving a more fine-grained protein-text similarity measure.Protein captioning (PCap) improves the retrieval accuracy of ProtT3 by about 2%.This is because PCap encourages query tokens to extract the protein information most relevant to the text input, which helps protein-text alignment.
* ProtT3 w/o PTM: Skip the PTM stage of ProtT3
* ProtT3 w/o PCap: Skip the PCap stage of ProtT3
ProtT3 can predict protein structure and properties, and has better question-answering capabilities
The researchers evaluated the protein question answering performance of ProtT3 on the PDB-QA dataset, selecting exact match as the evaluation metric and using Galactica1.3B fine-tuned by LoRA as the baseline model (LoRA ft).
As shown in the figure below,The exact matching performance of ProtT3 is 2.51 higher than that of the baseline TP3T.It consistently outperforms the baseline in predicting protein structure and properties, demonstrating that ProtT3 has excellent multimodal capabilities for understanding protein and text questions.
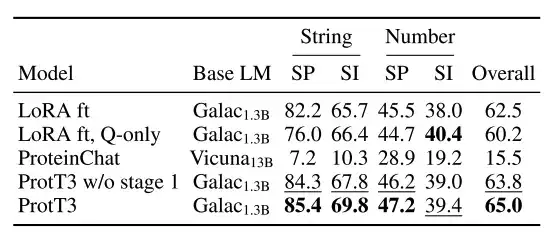
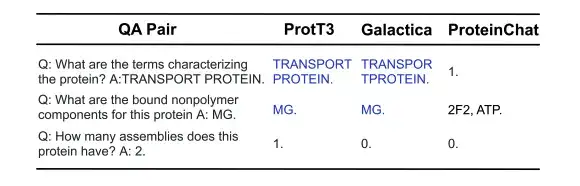
As shown in the figure below, in the following 3 protein question-answering examples, ProtT3 and Galactica both correctly answered the first two questions about protein properties/structure, but failed on the third question that required a numerical answer. ProteinChat had difficulty with all 3 questions and was unable to answer any of them.
Unlocking the language of proteins, LLM's cutting-edge exploration in life sciences
The researchers' exploration in the field of protein-to-text generation enables humans to unlock complex biological phenomena in an understandable way. The language model in the above study not only demonstrates a deep understanding of the "latent space" of proteins, but also serves as a bridge between biomedical tasks and natural language processing, opening up new paths for research such as drug development and protein function prediction. Furthermore,If large language models with billions or more parameters are used to process more complex language structures, it will be expected to enhance future exploration of life sciences at multiple levels.
for example,The team led by Zhang Qiang and Chen Huajun from Zhejiang University proposed an innovative large language model called InstructProtein.The model has the ability to generate both human language and protein language in both directions: (i) taking a protein sequence as input, predicting its textual functional description; and (ii) using natural language to prompt protein sequence generation.

Specifically, the researchers pre-trained LLM on protein and natural language corpora, and then used supervised instruction adjustment to promote the alignment of these two different languages. InstructProtein performs well in a large number of bidirectional protein text generation tasks. It has taken a pioneering step in text-based protein function prediction and sequence design, effectively narrowing the gap between protein and human language understanding.
The paper, titled "InstructProtein: Aligning Human and Protein Language via Knowledge Instruction", was selected for ACL 2024.
* Original paper:https://arxiv.org/pdf/2310.03269
also,The University of Technology Sydney team also joined forces with the Zhejiang University research team to jointly launch the large language model ProtChatGPT.The model learns and understands protein structure, allowing users to upload protein-related questions and engage in interactive conversations, ultimately generating comprehensive answers.
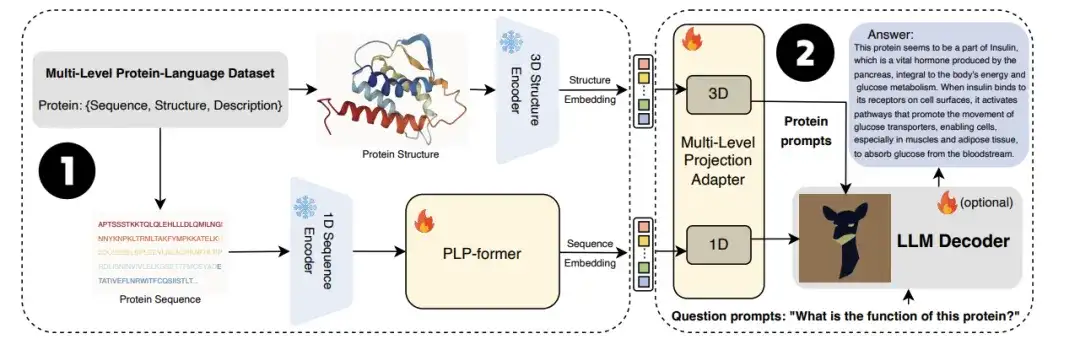
Specifically, proteins first pass through protein encoders and protein language pre-trained converters (PLP-former) to generate protein embeddings, and then these embeddings are projected to LLM through the Projection Adapter. Finally, LLM combines user questions with projected embeddings to generate informative answers. Experiments show that ProtChatGPT can generate professional responses to proteins and their corresponding questions, injecting new vitality into the in-depth exploration and application expansion of protein research.
* Original paper:https://arxiv.org/abs/2402.09649
In the future, when big language models are able to use massive and rich data to infer the underlying laws or deep structures of proteins that far exceed the limits of human cognition, their potential will be greatly released. We expect that with the continuous advancement of technology, big language models will lead protein research into a brighter future.








