Command Palette
Search for a command to run...
VenusFactory タンパク質工学設計プラットフォーム

1. チュートリアルの概要

VenusFactoryは、上海交通大学、上海人工知能研究所、華東科技大学の共同チームによって2025年に開発されました。関連する研究論文は以下の通りです。 VenusFactory: タンパク質工学データの取得と言語モデルの微調整のための統合プラットフォーム 。
VenusFactory は、タンパク質工学コミュニティ向けに特別に設計された統合プラットフォームであり、生物学的データの取得、標準化されたタスクのベンチマーク、事前トレーニング済みのタンパク質言語モデル (PLM) のモジュール式の微調整を統合することを目的としています。
このプラットフォームは、コマンドライン実行と Gradio ベースのコードフリー インターフェースをサポートし、40 を超えるタンパク質関連データセットと 40 を超える一般的な PLM を統合しているため、コンピューター サイエンスや生物学の研究者が簡単に使用できます。
チュートリアルでは 7 つの機能モジュールが提供されます。
- トレーニング: ゼロコード モデル トレーニング、40 を超える大規模モデルをサポートし、プライベート データセットを使用して独自のモデルをトレーニングします。
- 評価: タンパク質モデルの包括的なパフォーマンス評価のための使いやすいツール。
- 予測: トレーニング済みのモデルを使用して、新しいタンパク質配列の機能を予測します。
- VenusAgent: DeepSeek と連携して AI タンパク質計算を可能にするタンパク質エンジニアリング エージェント。
- クイック ツール: ゼロ サンプルの突然変異予測 (指向性進化) と教師あり予測 (機能またはプロパティの予測) をサポートする、使いやすいバージョンです。
- 高度なツール: ゼロサンプル突然変異予測 (指向性進化) と教師あり予測 (機能またはプロパティの予測) をサポートする高度なカスタマイズ バージョン。
- ダウンロード: タンパク質データに簡単にリンクし、主要なデータベース (RCSB、UniProt など) のマルチスレッド ダウンロードをサポートします。
このチュートリアルで使用するコンピューティングリソースは、RTX 4090カード1枚です。このチュートリアルで使用するモデルは、
/openbayes/input/input1すべてのデータはディレクトリに保存されます/openbayes/home/VenusFactoryディレクトリ。
2. 操作手順
1. コンテナを起動します
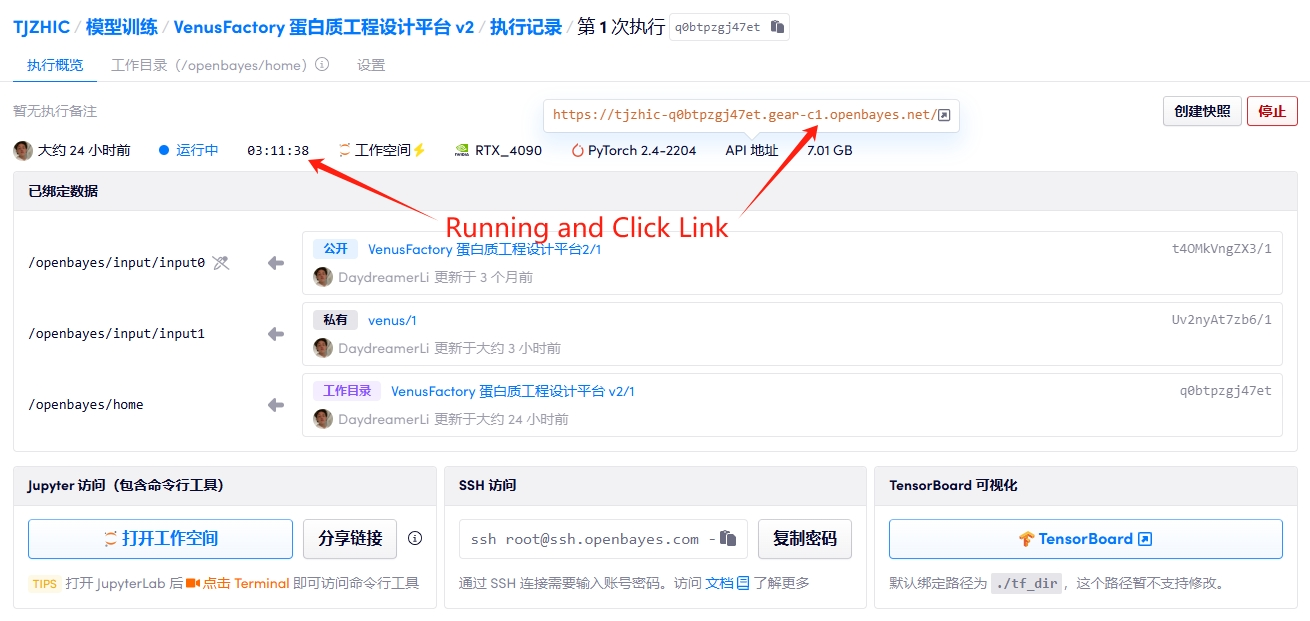
2. マニュアル
「Bad Gateway」と表示される場合は、プロジェクトが初期化中であることを意味します。1~2分ほど待ってからページを更新してください。
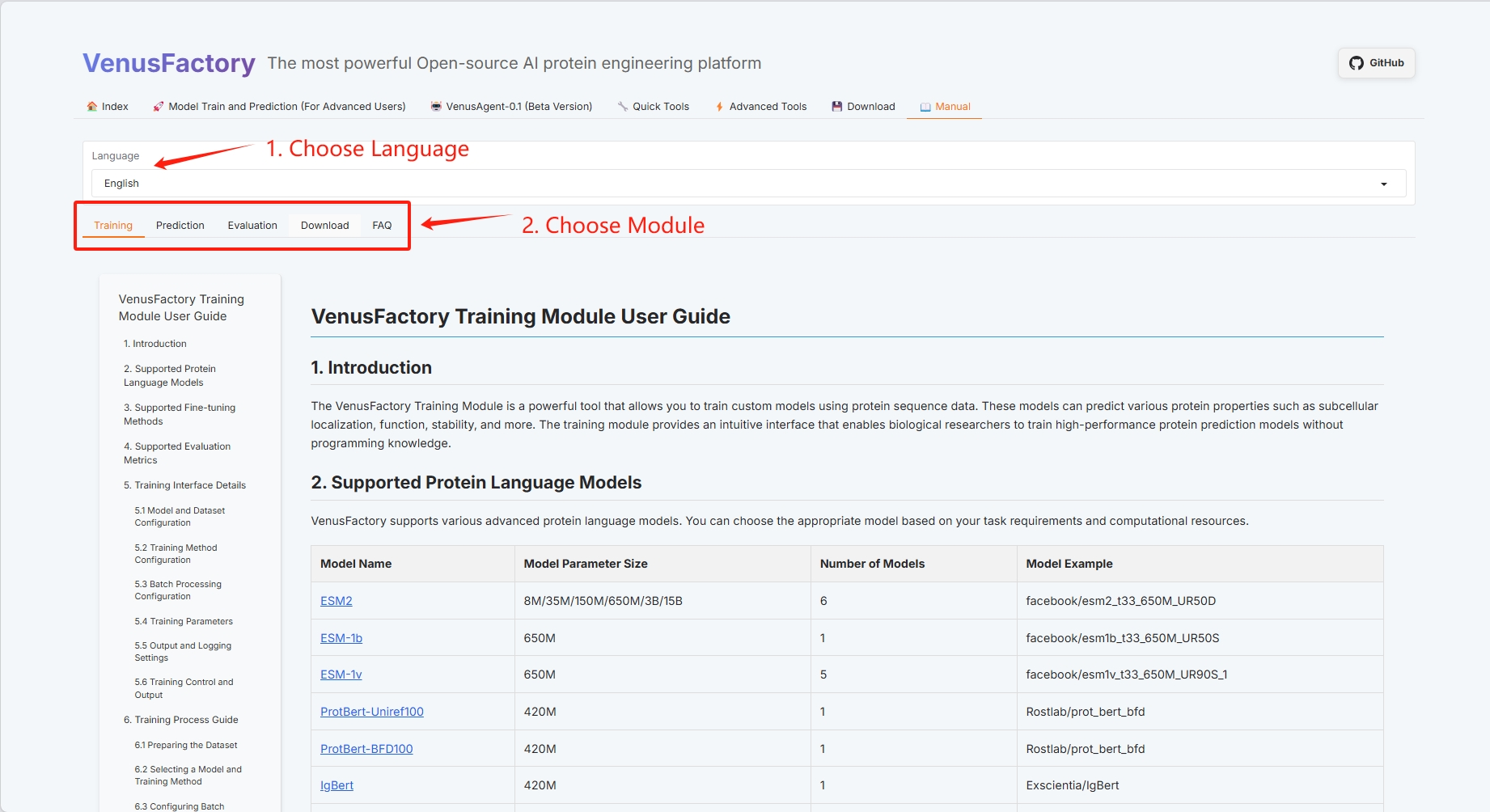
VenusFactory の「マニュアル」ユーザー ガイドには現在、トレーニング、評価、予測、ダウンロードの 4 つのモジュールが含まれています。
3. 具体的な機能のデモンストレーション
3.1 トレーニング
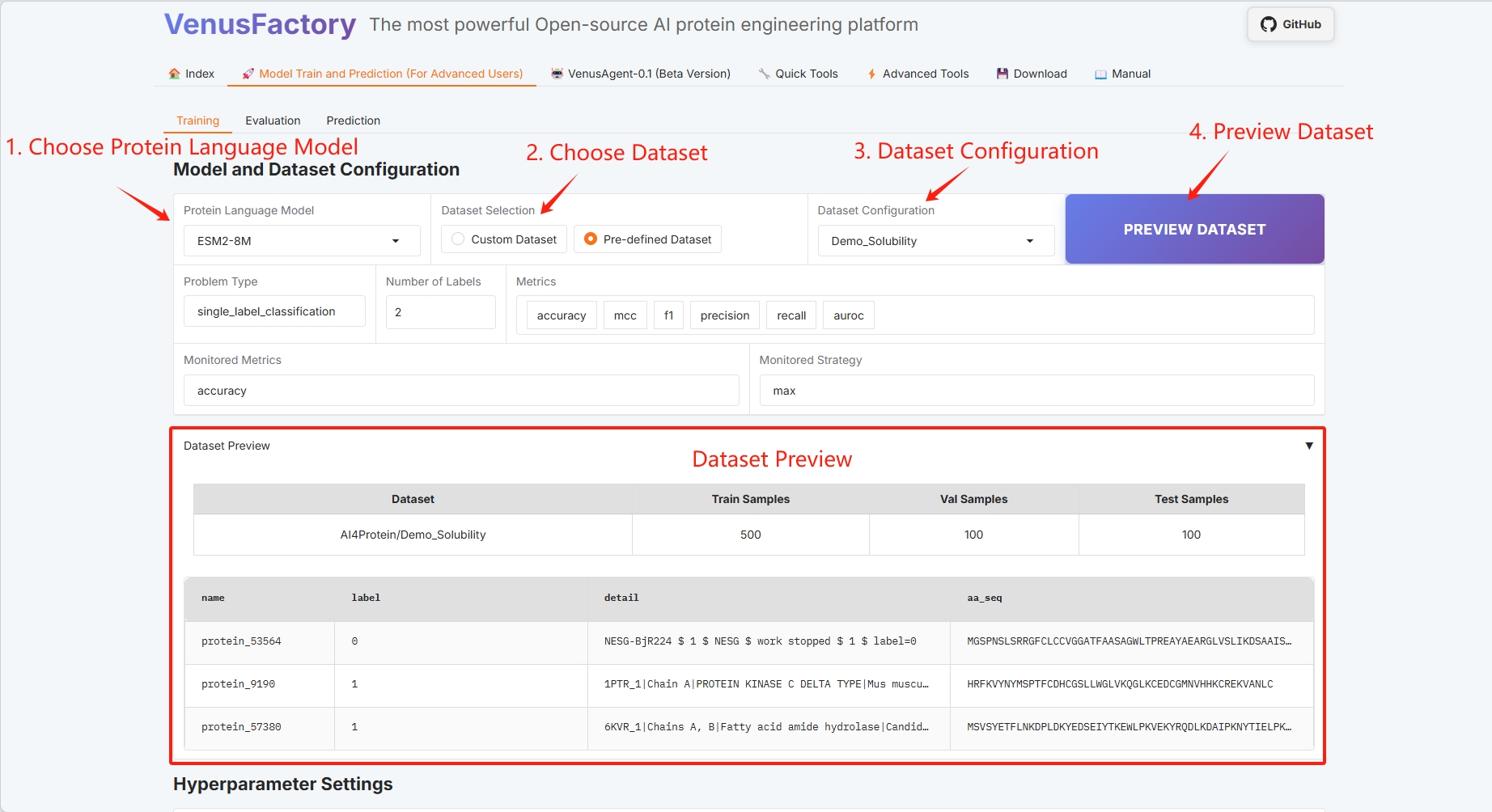
「モデルトレーニングと予測トレーニング」モジュールの「トレーニング」モジュールをクリックします。
- タンパク質言語モデルを選択
- データセットの選択
- データセットのプレビュー
- トレーニング方法の設定(具体的な情報についてはユーザーガイドを参照してください)
- バッチ構成(詳細はユーザーガイドを参照)
選択したモデルのパラメータが大きい場合は、グラフィック カードをより大きなサイズのものに交換してください。
トレーニング モデルの保存パスを設定し、「トレーニングを開始」をクリックしてトレーニングを開始します。
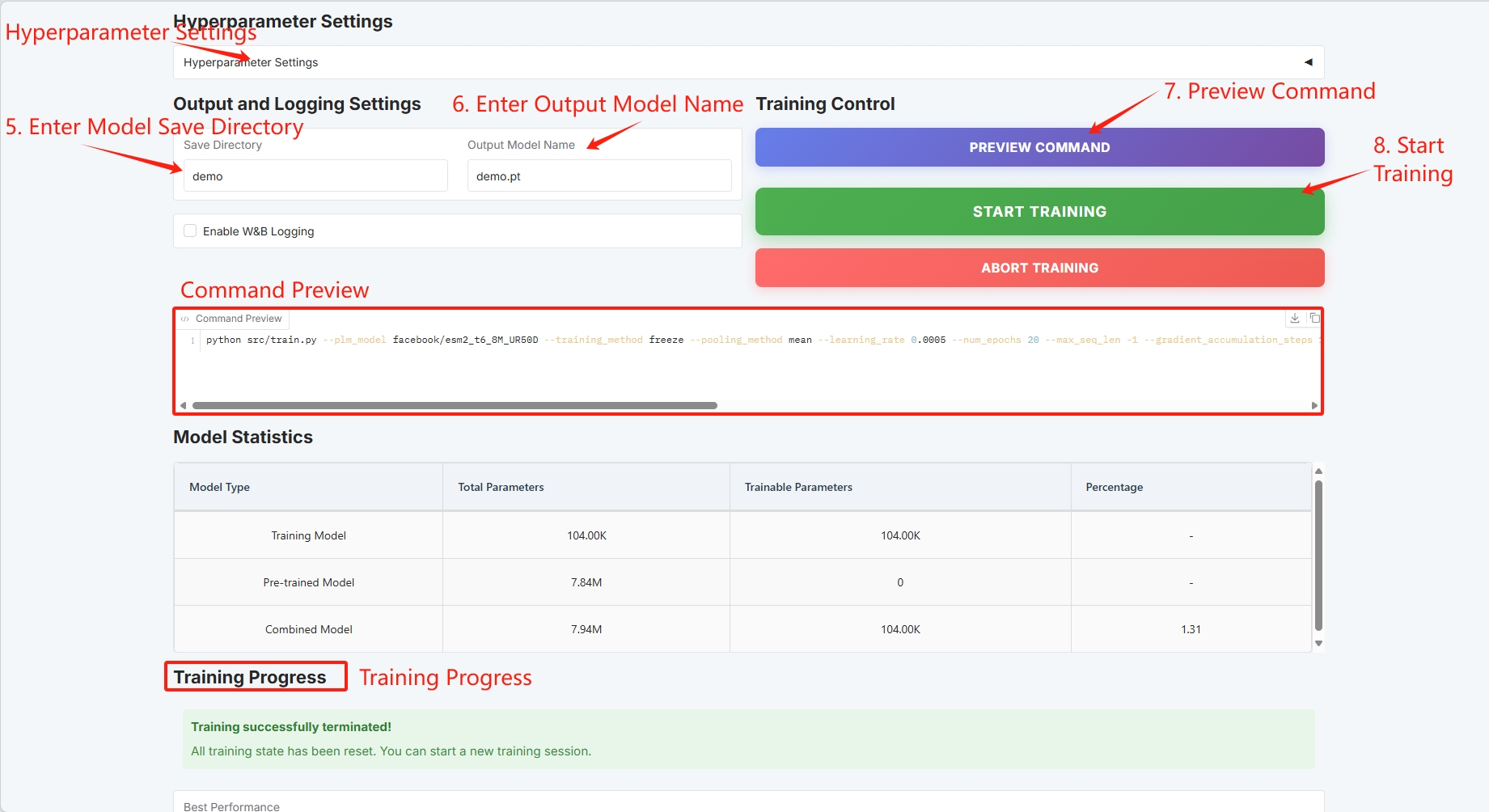
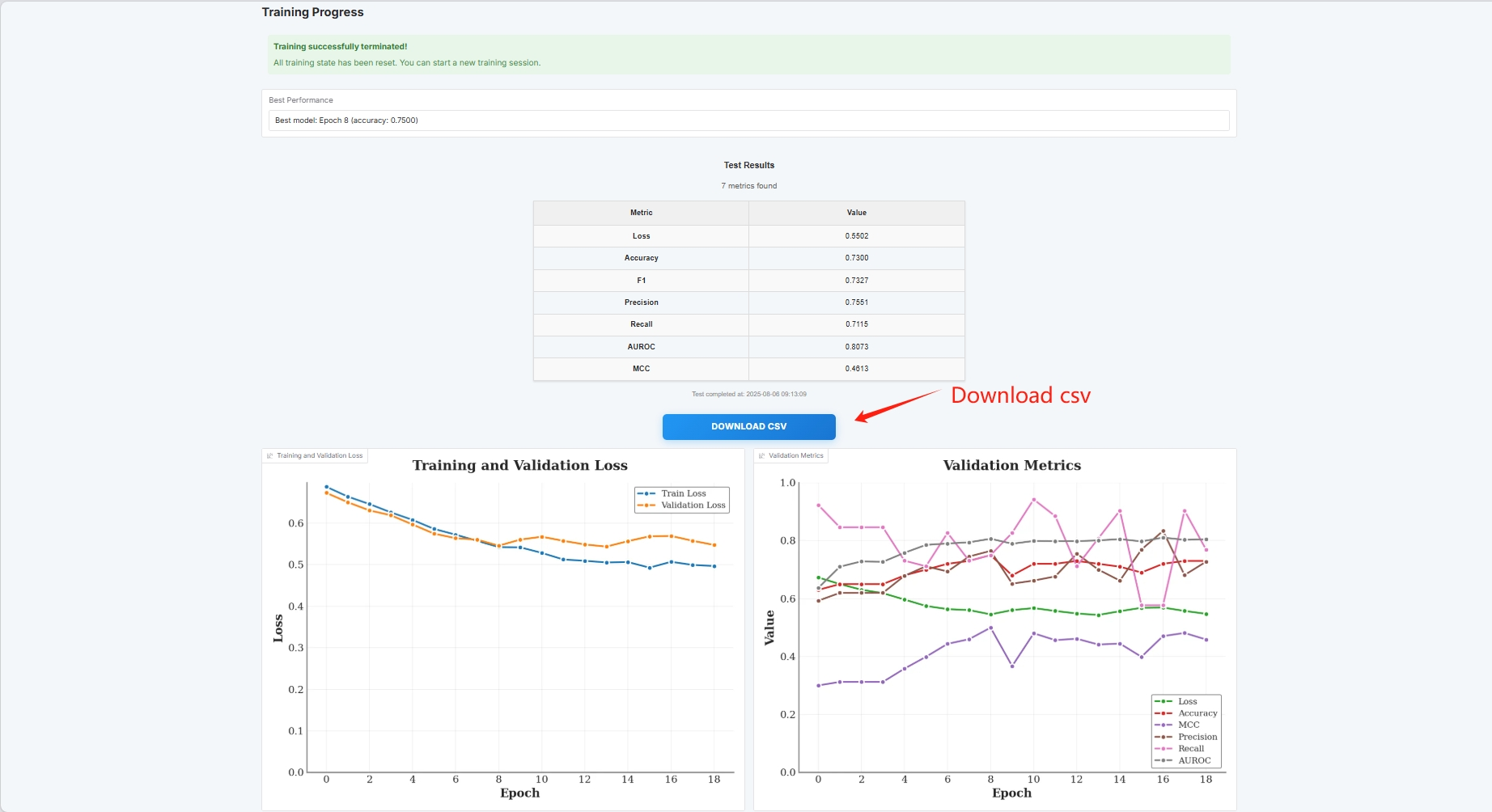
この時点で、トレーニングパラメータと損失曲線を見ることができます。
独自のデータセットを使用する場合は、カスタムデータセット設定を使用できます。データセットのパスを入力するだけです(詳細はマニュアルのドキュメントをご覧ください)。
3.2 評価
「モデルトレーニングと予測トレーニング」モジュールの「評価」モジュールをクリックします。
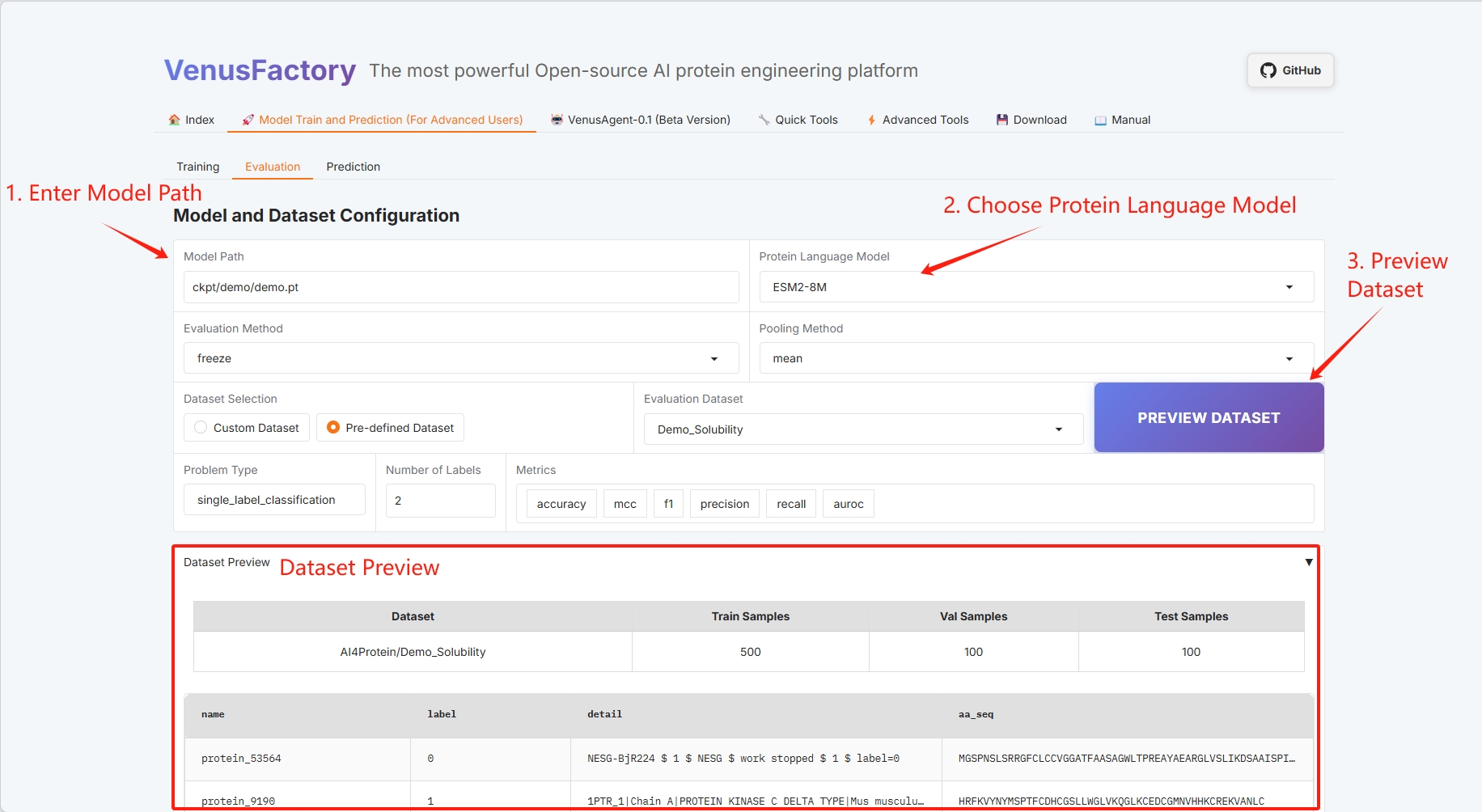
- モデルパスとタンパク質言語モデルの選択
- 評価方法とプーリング方法(具体的な情報についてはユーザーガイドを参照してください)
- データセットの選択
- データセットのプレビュー
- 質問の種類とタグ(詳細はユーザーガイドをご覧ください)
- バッチ構成(詳細はユーザーガイドを参照)
学習済みモデルを保存するパスを設定し、タンパク質言語モデルを選択します。
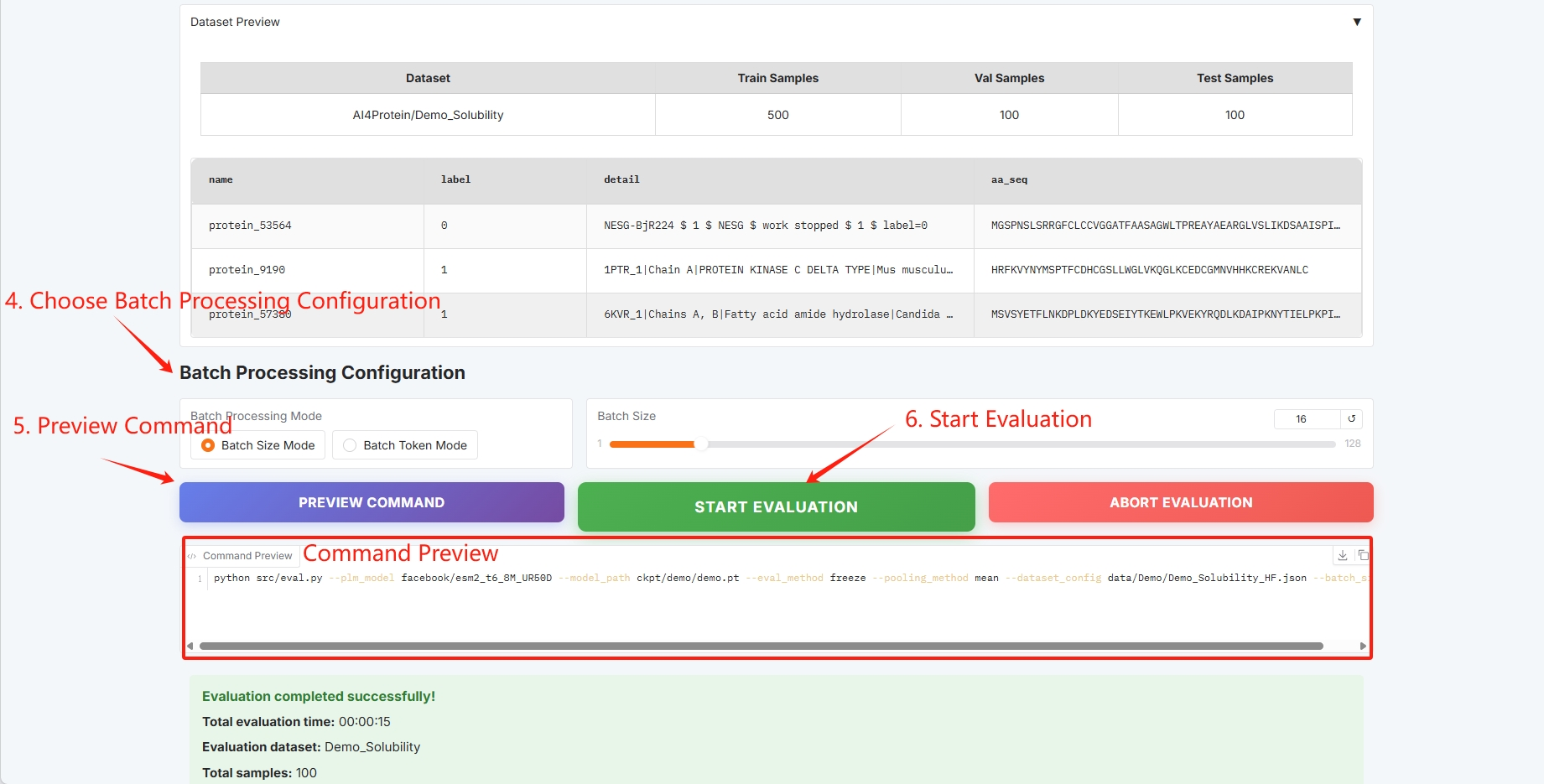
バッチ処理を設定し、「評価を開始」をクリックしてトレーニングを開始します。
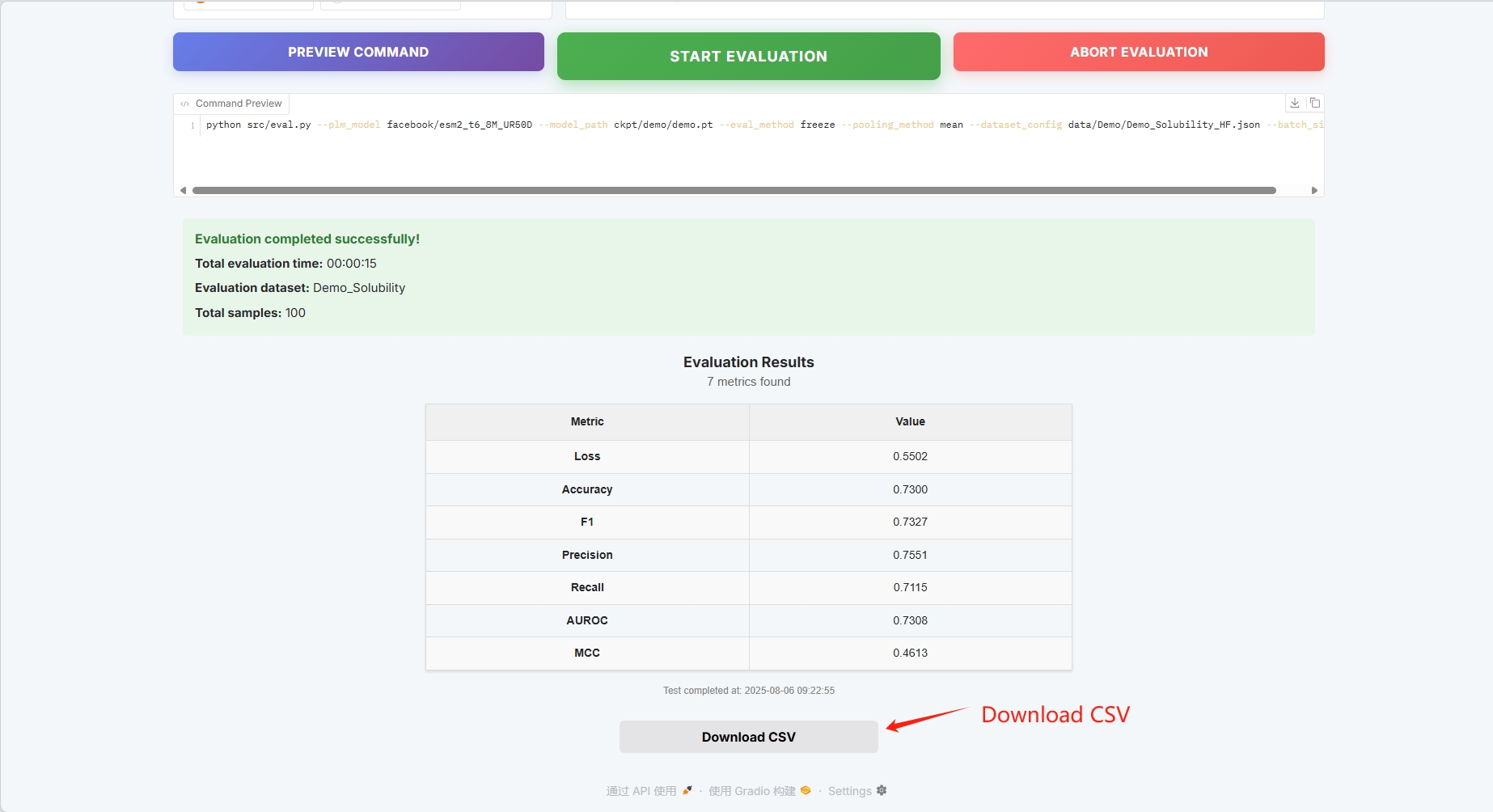
評価結果は以下のとおりで、CSV形式でダウンロードできます。
独自のデータセットを使用する場合は、カスタムデータセット設定を使用できます。データセットのパスを入力するだけです(詳細はマニュアルのドキュメントをご覧ください)。
3.3 予測
「モデル トレインと予測トレーニング」モジュールの「予測」モジュールをクリックして、単一シーケンス予測とバッチ予測を実行します。
- モデル構成
- 予測モジュールを選択します(詳細はユーザーガイドを参照してください)
トレーニングモデルの保存パスを設定し、タンパク質言語モデルを選択して、「予測を開始」をクリックしてトレーニングを開始します。
単一配列予測
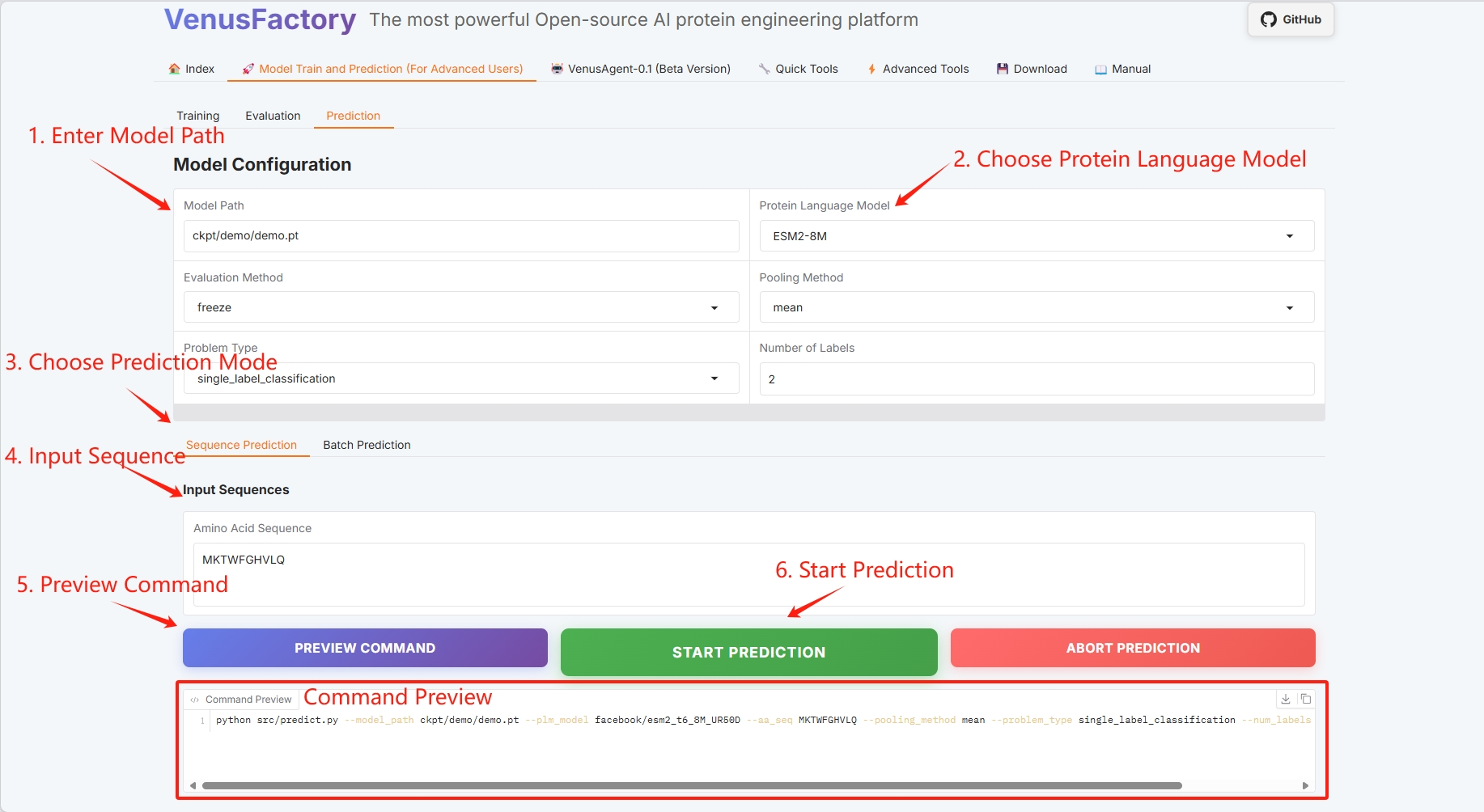
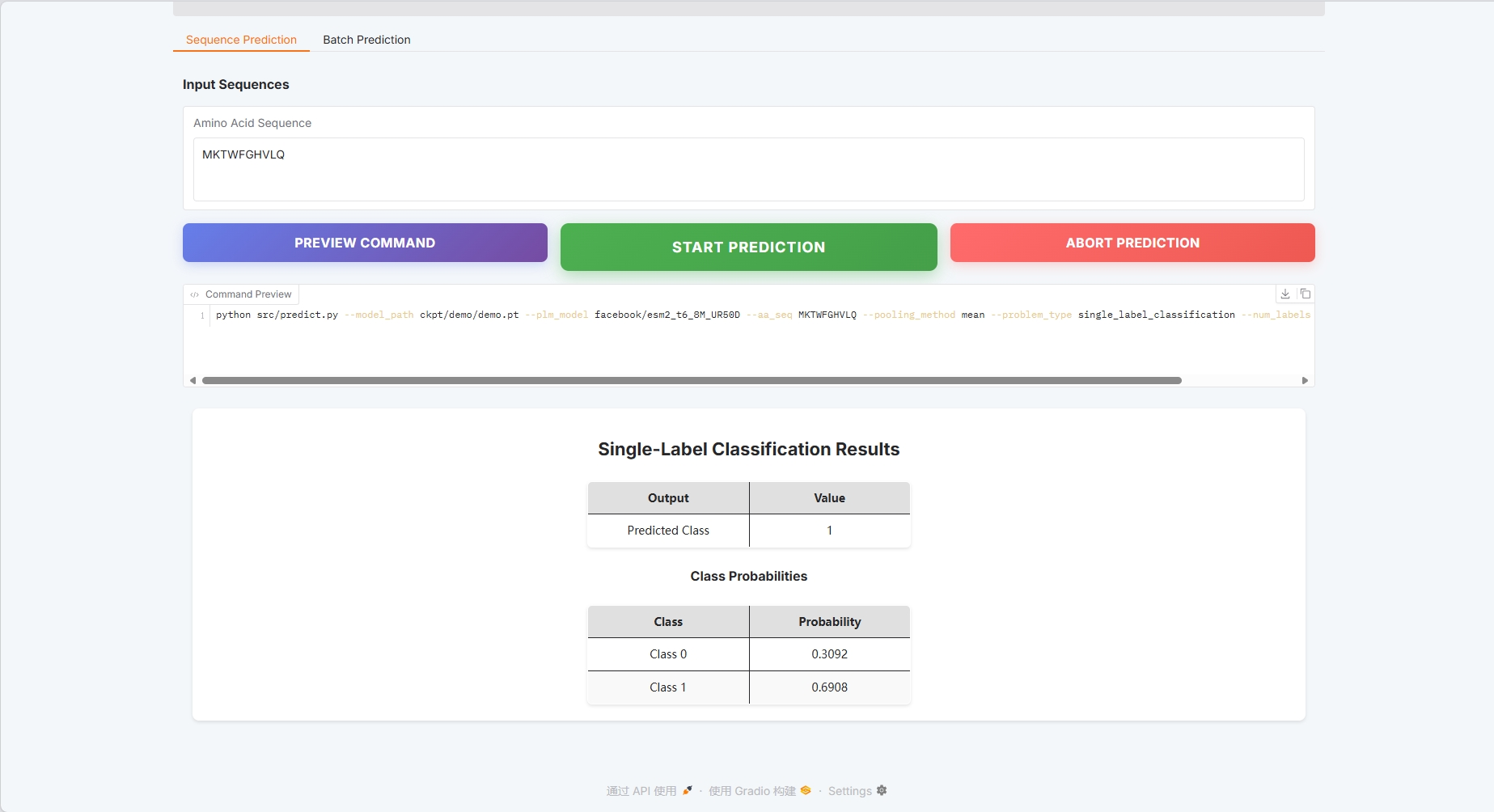
タンパク質配列の例: MKTWFGHVLQ
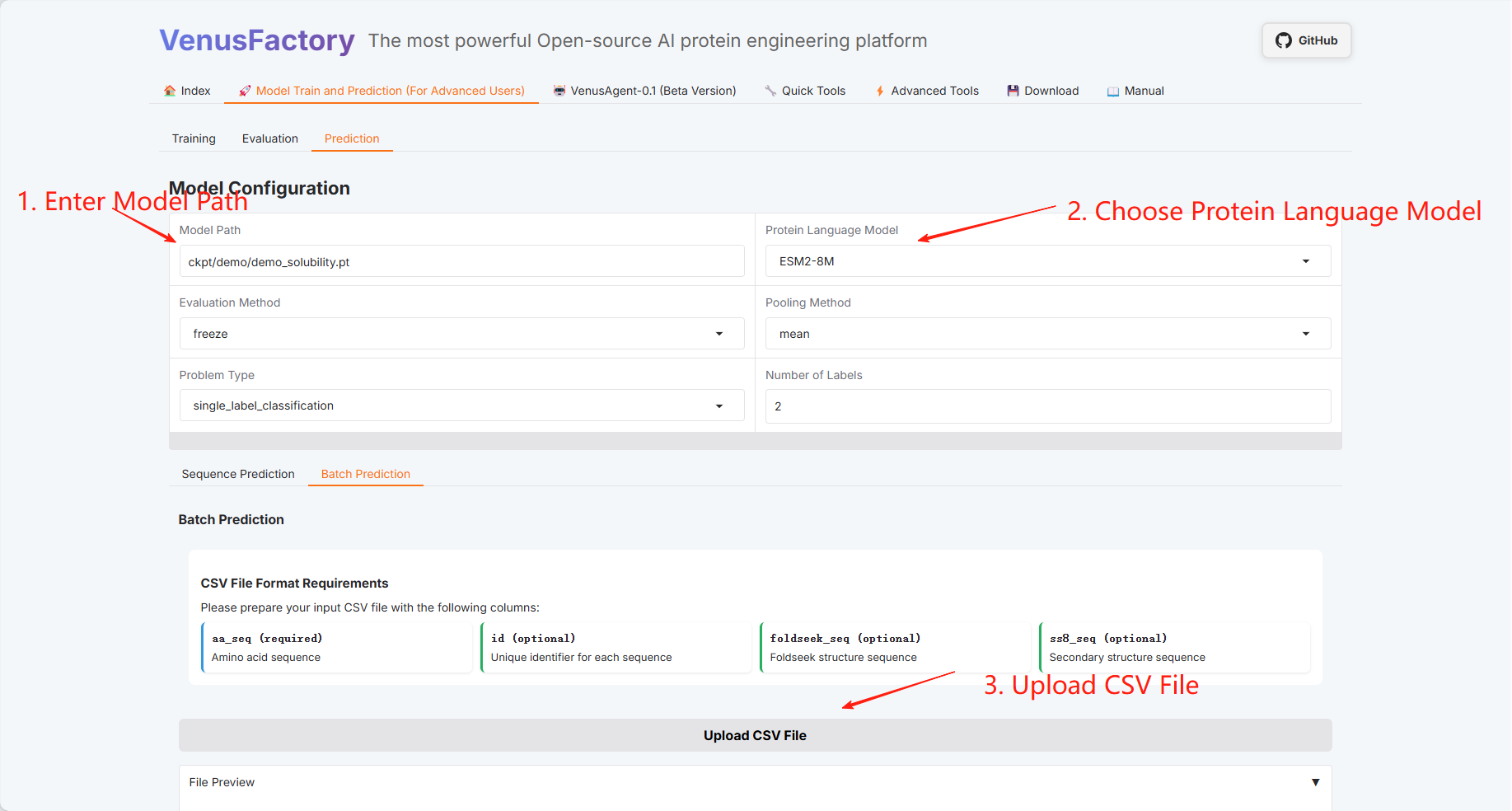
バッチ予測
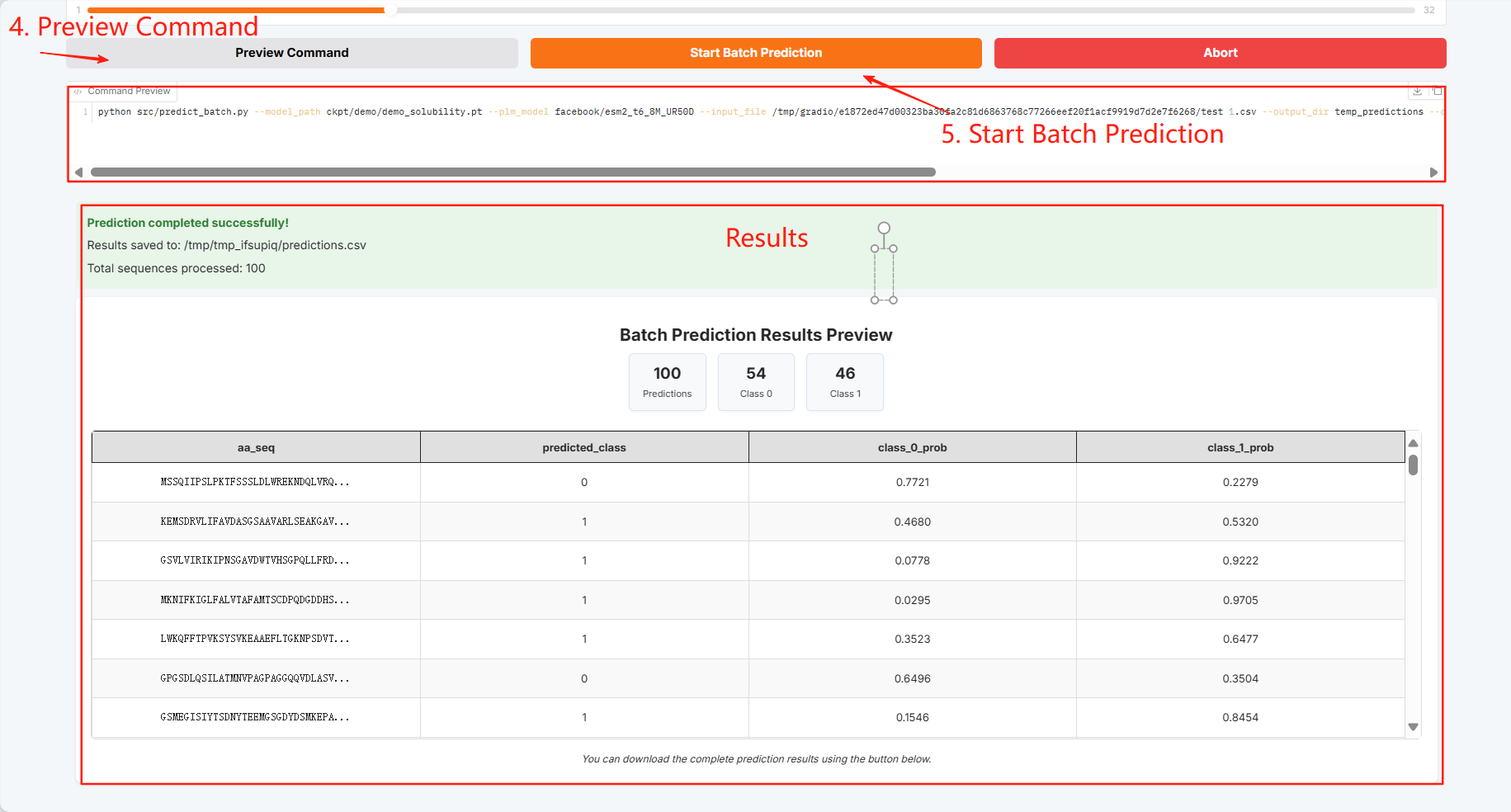
バッチ予測結果をダウンロードして保存できます
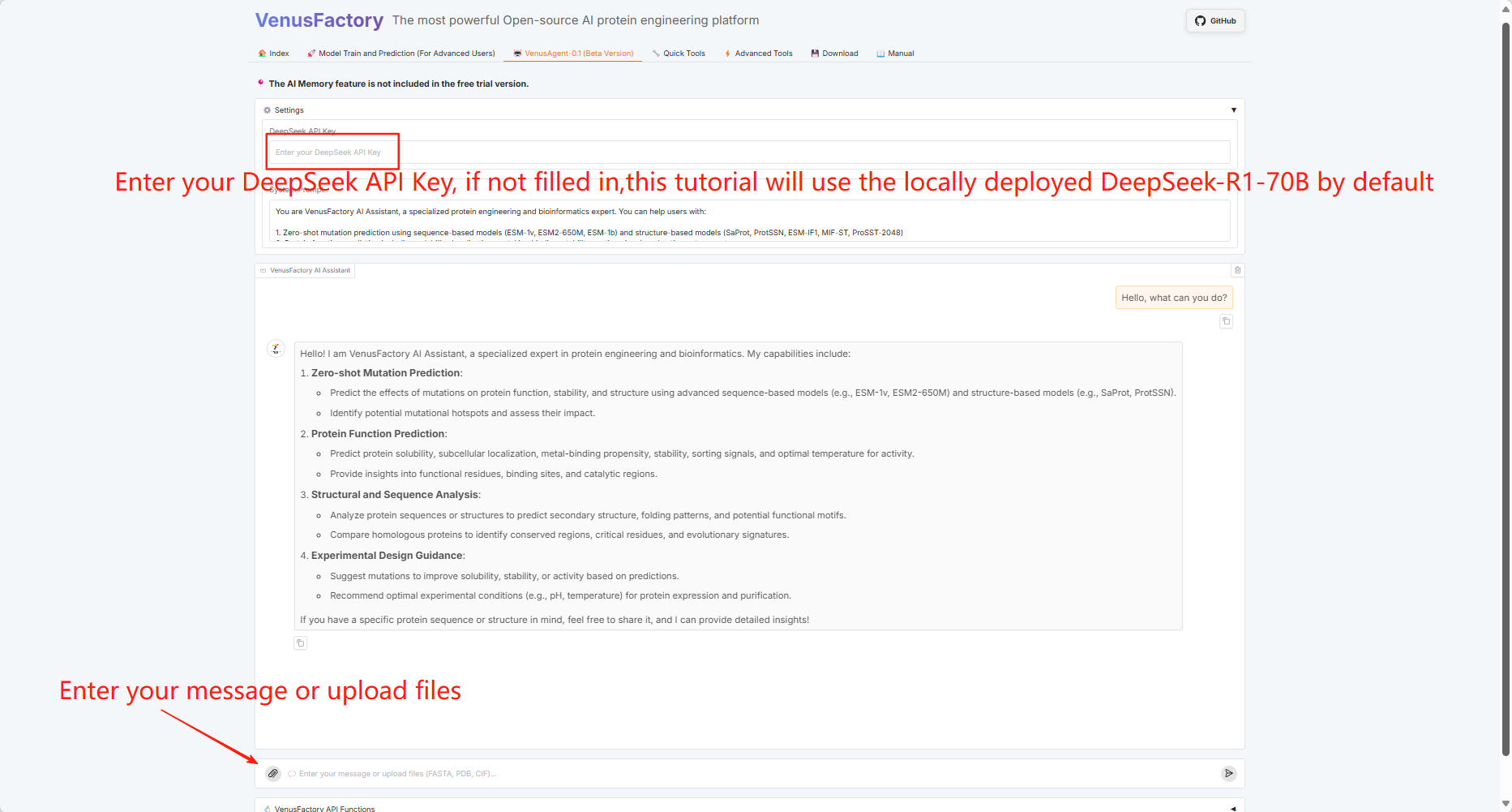
3.4 ヴィーナスエージェント
「VenusAgent」モジュールをクリックします
VenusAgent は DeepSeek の大規模モデルを呼び出す必要があるため、このチュートリアルでは、API キーを自分で入力するか、プラットフォームにデプロイされた DeepSeek-R1-70B モデルを使用するという 2 つの呼び出し方法を提供します。
必要な機能に応じて、さまざまなグラフィックカードを選択できます。カードの選択手順は次のとおりです。
単一の RTX 4090 グラフィック カードを使用する場合、VenusAgent 機能はローカルに展開された大規模モデル サービスの使用をサポートしません (DeepSeek API キーの使用は無制限です)。
デュアル RTX 4090 グラフィック カードを使用する場合、VenusAgent 機能を使用した後、すぐに (1 ~ 2 分後) 他の機能を使用することはできません (DeepSeek API キーを使用する場合は制限はありません)。
デュアル RTX A6000 グラフィック カードを使用すると、VenusAgent の機能は無制限になります。
このチュートリアルで使用するコンピューティングリソースは、RTX 4090カード1枚です。このチュートリアルで使用するモデルは、
/openbayes/input/input1すべてのデータはディレクトリに保存されます/openbayes/home/VenusFactoryディレクトリ。
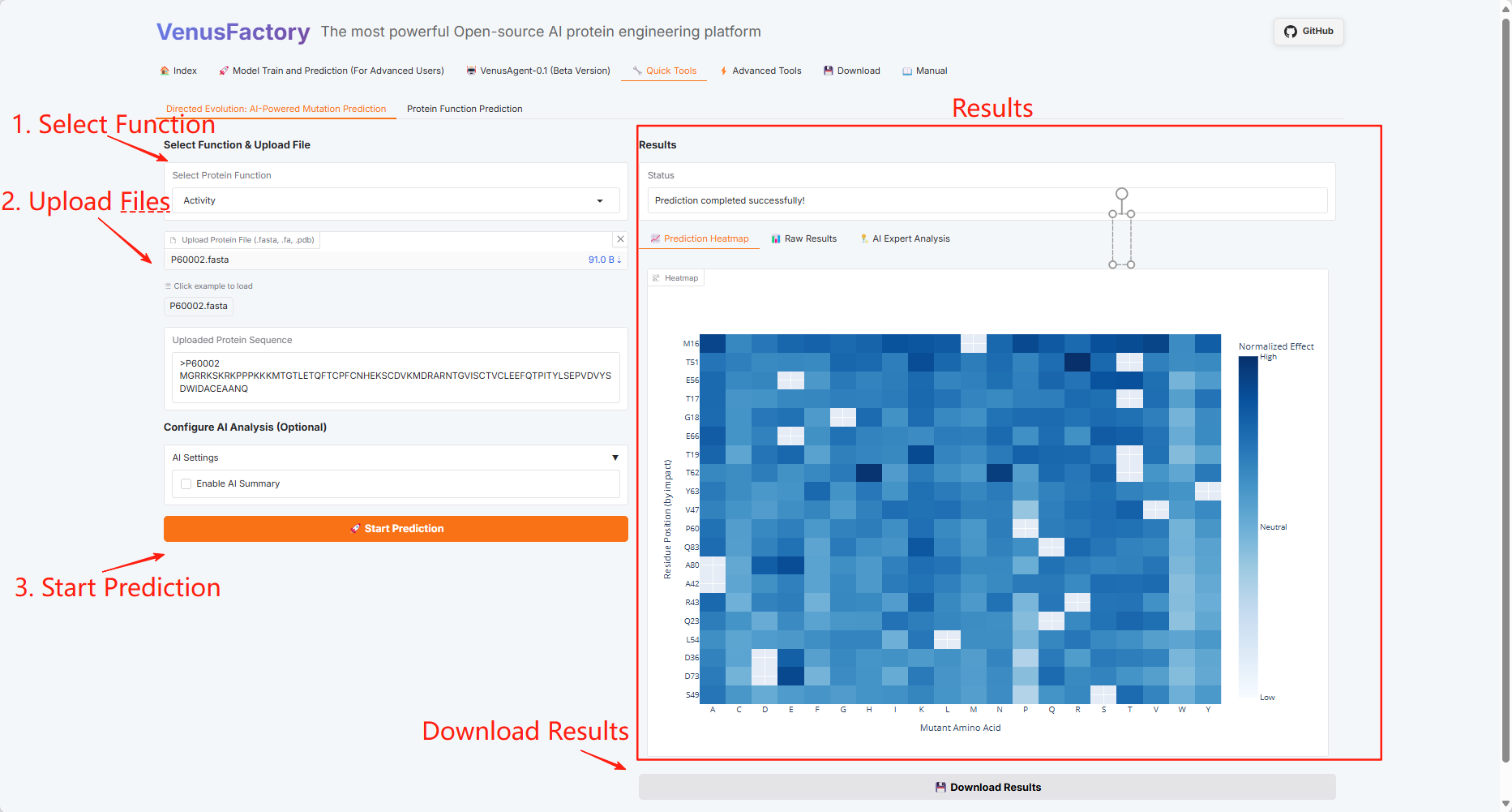
3.5 クイックツール
「クイック ツール」モジュールをクリックします。このモジュールには、「Directed Evolution: AI による突然変異予測」と「タンパク質機能予測」の 2 つの機能が含まれています。
指向性進化:AIを活用した突然変異予測
タンパク質機能予測
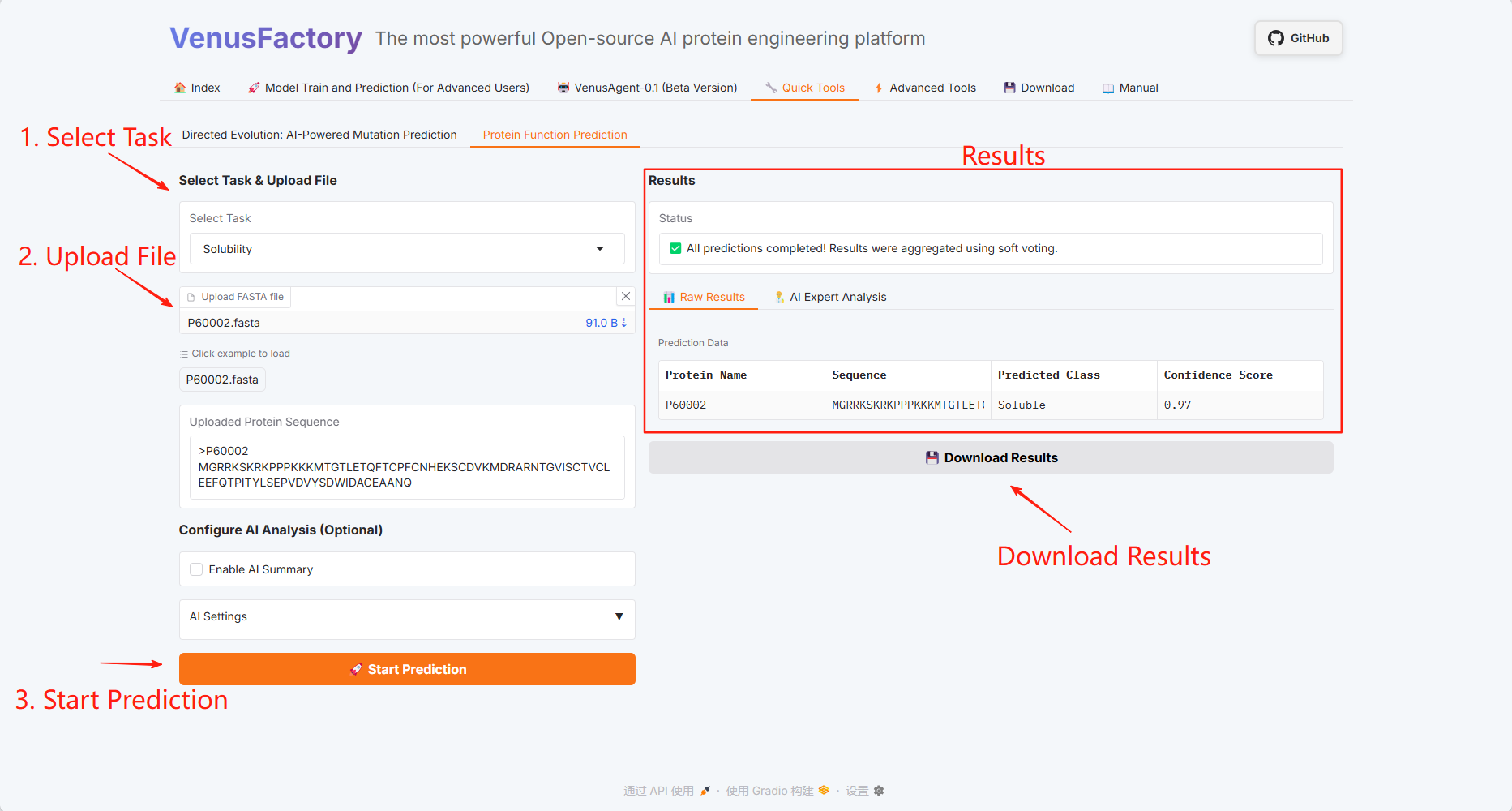
3.6 高度なツール
「高度なツール」モジュールをクリックします。このモジュールには、「Directed Evolution: AI による変異予測」と「タンパク質機能予測」の 2 つの機能が含まれています。
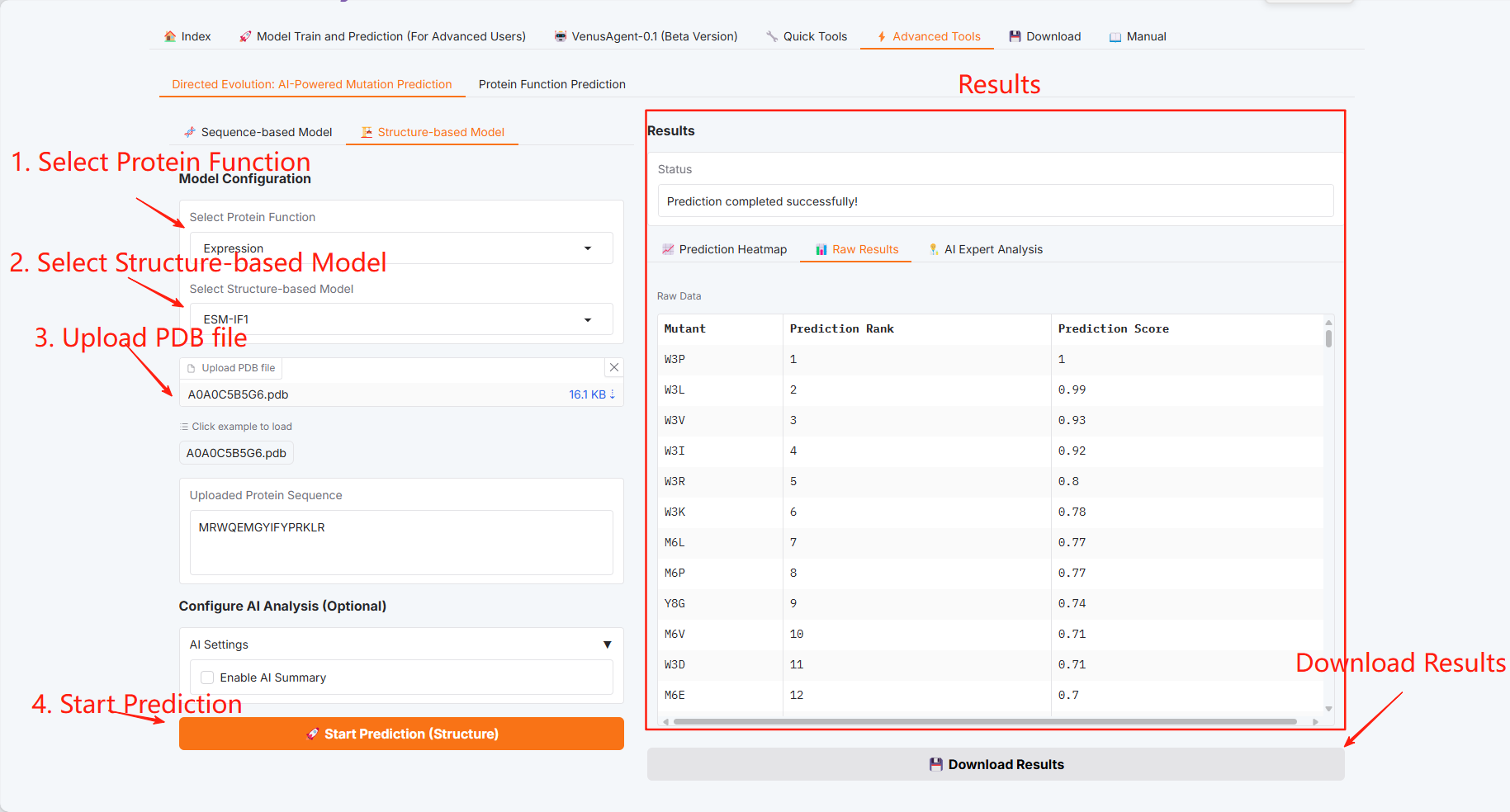
指向性進化:AIを活用した突然変異予測
シーケンスベースモデル
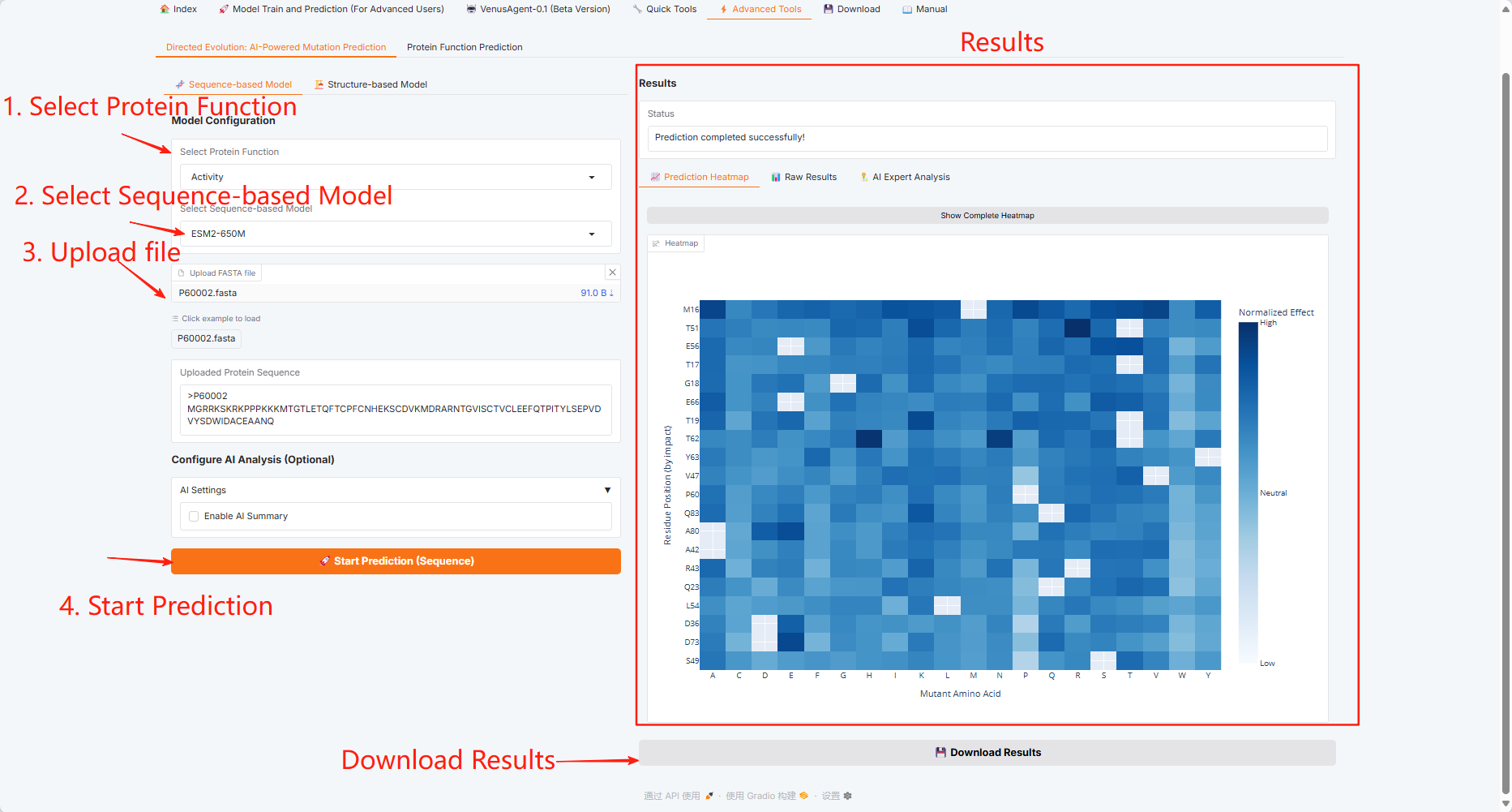
構造ベースモデル
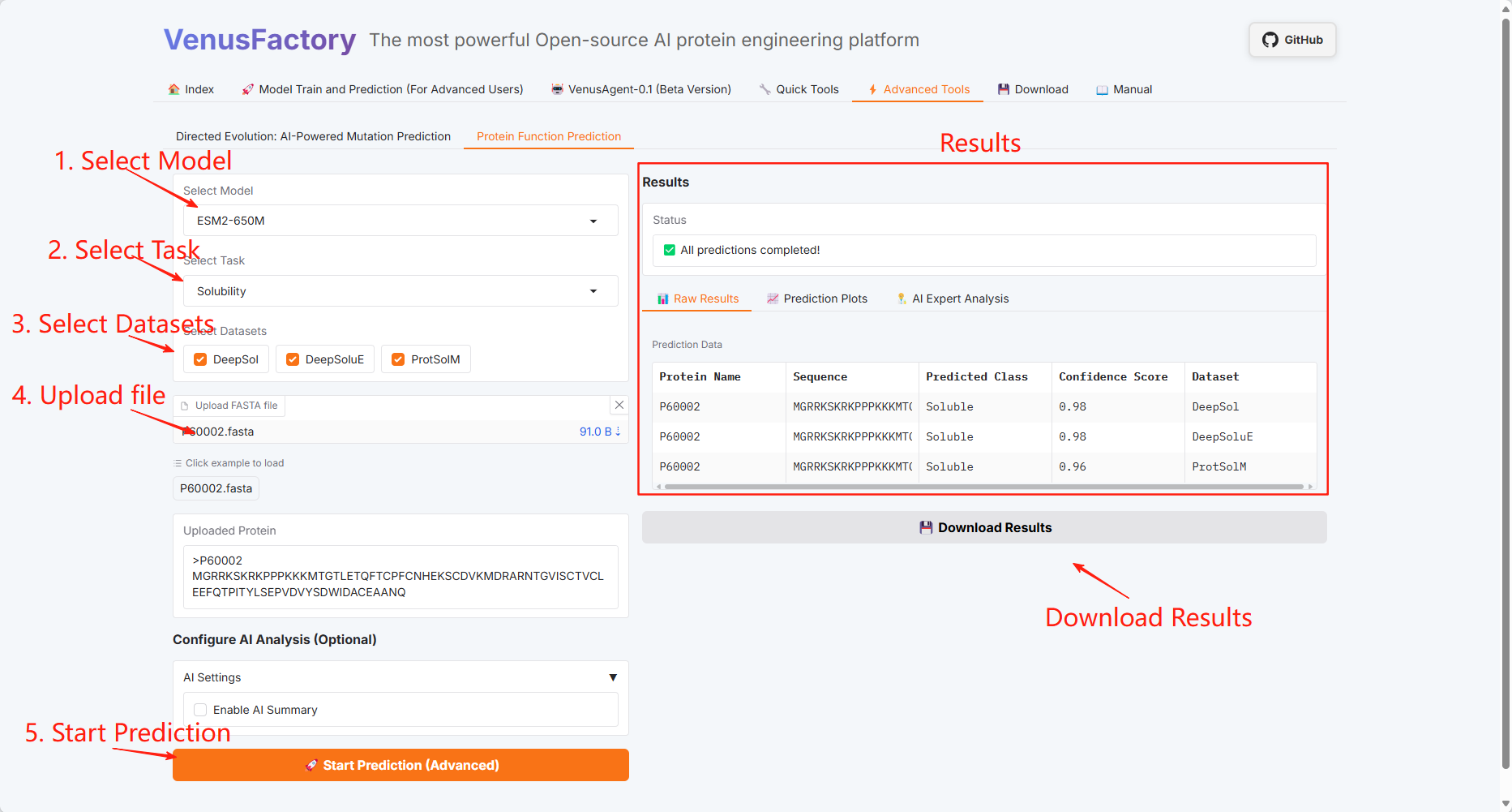
タンパク質機能予測
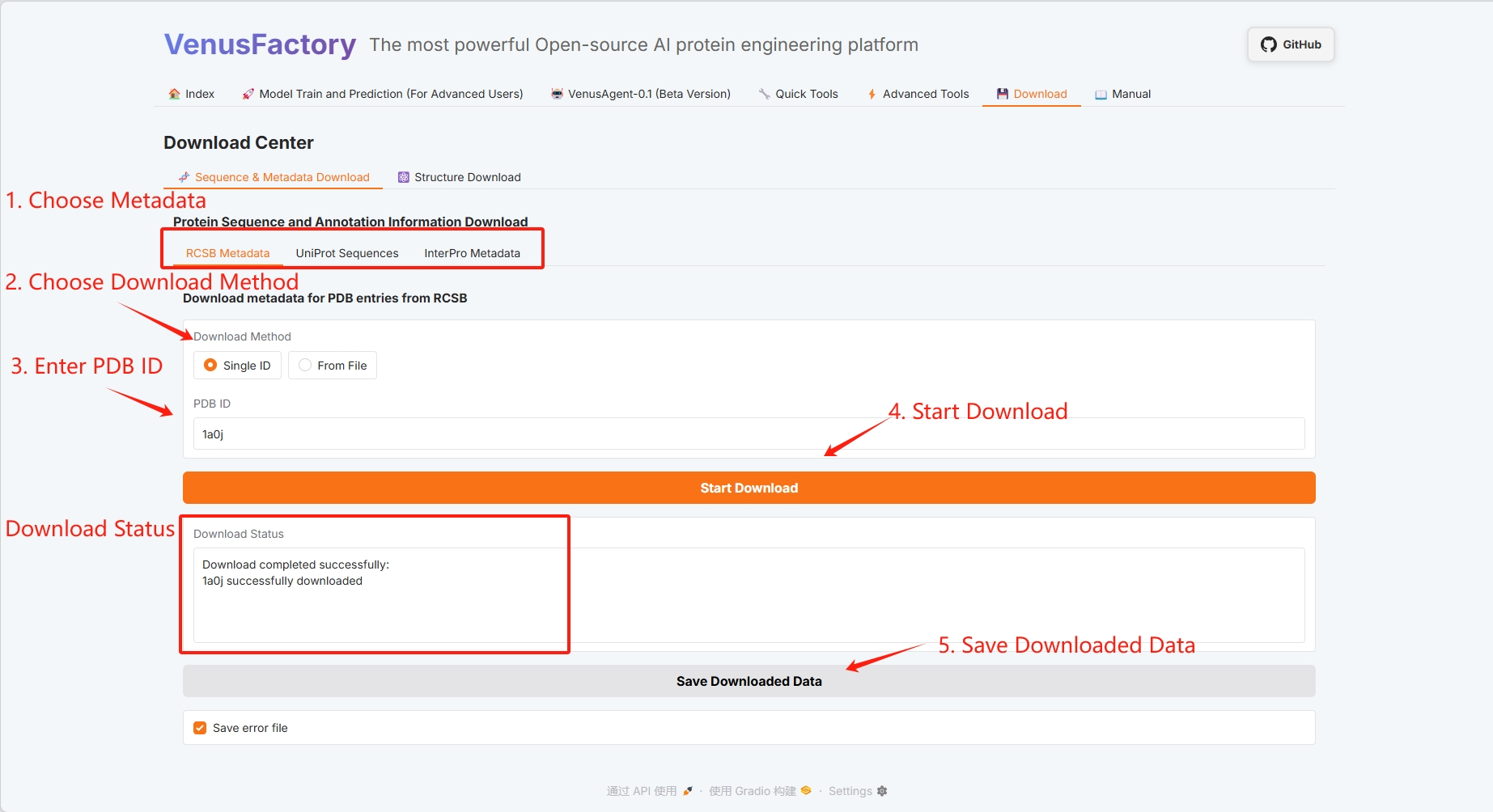
3.7 ダウンロード
このインターフェースでタンパク質データをダウンロードするには、ダウンロード モジュールをクリックします。
3. 議論
🖌️ 質の高いプロジェクトを見つけたら、ぜひバックグラウンドでメッセージを残して推薦してください!また、AI4S交流グループも設立しました。友人はQRコードをスキャンして[AI4S]とコメントし、グループに参加してさまざまな技術的な問題について話し合い、アプリケーションの結果を共有してください↓

引用情報
このプロジェクトの引用情報は次のとおりです。
@inproceedings{tan-etal-2025-venusfactory,
title = "{V}enus{F}actory: An Integrated System for Protein Engineering with Data Retrieval and Language Model Fine-Tuning",
author = "Tan, Yang and Liu, Chen and Gao, Jingyuan and Wu, Banghao and Li, Mingchen and Wang, Ruilin and Zhang, Lingrong and Yu, Huiqun and Fan, Guisheng and Hong, Liang and Zhou, Bingxin",
editor = "Mishra, Pushkar and Muresan, Smaranda and Yu, Tao",
booktitle = "Proceedings of the 63rd Annual Meeting of the Association for Computational Linguistics (Volume 3: System Demonstrations)",
month = jul,
year = "2025",
address = "Vienna, Austria",
publisher = "Association for Computational Linguistics",
url = "https://aclanthology.org/2025.acl-demo.23/",
doi = "10.18653/v1/2025.acl-demo.23",
pages = "230--241",
ISBN = "979-8-89176-253-4",
}