Command Palette
Search for a command to run...
David Baker 氏のチームは、Science 誌で、特に薬物治療が不可能なターゲットをターゲットとした、不規則領域結合タンパク質を設計するための新しいアプローチを提案しています。

ほとんどの疾患はタンパク質の機能異常と直接関連しているため、タンパク質は医薬品開発において重要な役割を果たします。新薬開発において、研究者はタンパク質を主要な薬剤標的として用いることが多く、薬剤が部分的に安定したタンパク質に結合して疾患プロセスに介入することを可能にします。しかし、構造、配列、および立体配座の選択性が明確に定義されていない本質的に無秩序なタンパク質 (IDP) に薬剤を標的とすることは、依然として困難です。
従来の抗体標的化法は、主に抗体が特定のタンパク質に高度に特異的に結合する能力を利用して標的タンパク質の認識と制御を実現するものです。しかし、この標的化経路は多くの実験操作を必要とするだけでなく、無秩序な抗原は注入後に容易に分解され、効果を発揮しなくなります。そのため、プロテオーム中の 50% 以上を占める本質的に無秩序な領域 (IDR) を持つタンパク質は、一般的に「創薬不可能な」ターゲットと判断され、医薬品開発に使用されたことはありません。
こうした中、2024年のノーベル化学賞を受賞し、ワシントン大学タンパク質設計研究所所長を務める優れた計算生物学者のデイビッド・ベイカー氏と彼のチームは、「Logos」と呼ばれるタンパク質設計戦略を提唱しました。誘導適合結合戦略に基づいて、39 個のターゲット不規則アミノ酸配列に適応できる結合タンパク質が設計されました。本研究では、特殊な拡張反復タンパク質バックボーンを生成し、RF拡散モデルを用いて一般化しました。このバックボーンには、反復ペプチド配列用に特別に設計されたポケットが組み込まれており、設計されたバインダーターゲットペプチドテンプレートが、タンパク質の不規則領域を普遍的に認識できるようになります。これにより、より多くのタンパク質が新薬開発の標的となり、がんやアルツハイマー病の研究を加速させる可能性が示唆されます。
関連する研究成果は、「本質的に無秩序な領域に結合するタンパク質の設計」というタイトルで『サイエンス』誌に掲載されました。
研究のハイライト:
*あらゆる標的配列に対する結合適応コンフォメーション誘導を実現するために、一般的な認識に適したテンプレート構造ライブラリを確立します。
* 幅広い多様性と治療の可能性を秘めた 18 個の合成ペプチド配列と 21 個の自然不規則領域 (IDR) に対応する結合タンパク質を設計し、がん関連細胞外受容体の不規則領域を標的とし、細胞内でのタンパク質の局在化を促進します。

用紙のアドレス:
https://www.science.org/doi/10.1126/science.adr8063
公式アカウントをフォローし、「Natural disordered protein」と返信すると、完全なPDFが手に入ります。
AIフロンティアに関するその他の論文:
テンプレートライブラリ生成:普遍的なペプチド同定
本研究では、IDR結合問題の解決策として、物理的設計手法と深層学習設計手法を組み合わせた。ペプチドユニットと異種標的配列の非適合性という制約により、この研究は、異なる反復タンパク質構造から始まり、拡散モデルを使用して、異なる反復単位内のアミノ酸結合ポケットを再編成し、それらを異なるアミノ酸と立体配座テンプレートに区別しました。これにより、シーケンスの認識範囲が広がります。
天然に不規則なタンパク質中のペプチドを同定するために、本研究ではまずバックボーンテンプレートライブラリを作成しました。このテンプレートライブラリには2つの特徴があります。
*各テンプレート構造は、伸長したペプチド鎖の立体構造を「包み込む」ことができ、水素結合やタイトパッキングなどの相互作用の機会を多数提供し、それによってターゲット配列の高度に特異的な認識を実現する必要があります。
* テンプレート構造は幅広く、あらゆるターゲット配列と一致できるため、少なくとも 1 つのテンプレートによって、定義された適切な結合構造を誘導できます。
バックボーン テンプレート ライブラリを生成するプロセスは、バックボーンの生成、タンパク質アクティブ ポケットの特殊化、およびタンパク質アクティブ ポケットのアセンブリの 3 つのステップに分かれています。
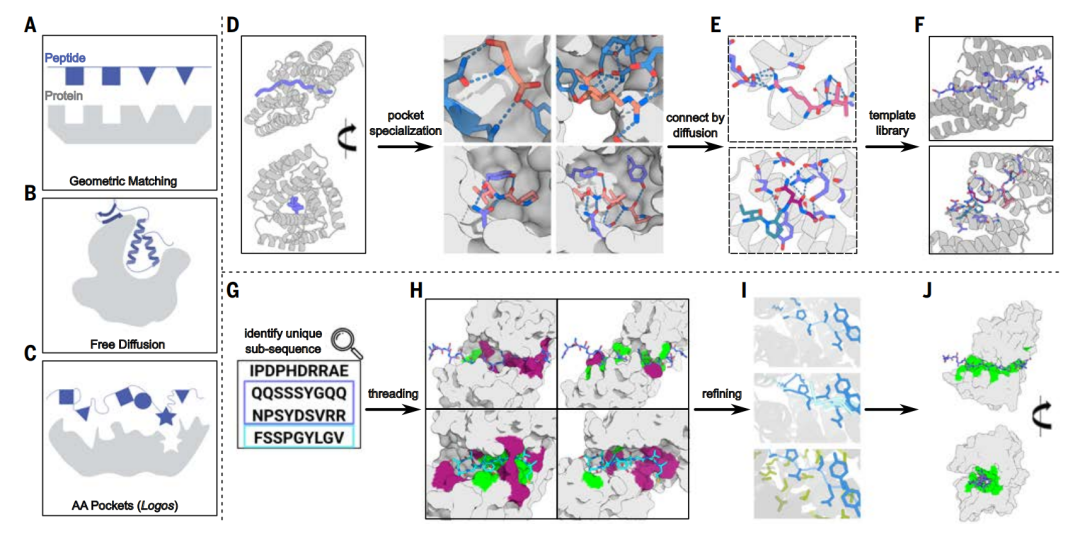
スキャフォールド生成
バックボーン生成段階では、ポリプロリン II 構造は主にプロリンを多く含むペプチドに発生するため、研究チームはポリプロリン II 構造に限定するのではなく、複数の拡張構造をターゲットにすることを選択しました。
伸長した構造では、アミノ酸の側鎖が交互に反対方向を向いており、これは 2 残基の繰り返しの特徴と一致しています。研究者らはロゼッタ設計法を使用して、一連のジペプチド繰り返し配列を設計した。LK、RT、YD、PV、GA(すべてアミノ酸の1文字略語)などがあり、さまざまな伸長構造でこれらのペプチドセグメントと絡み合って結合するように設計されているため、各繰り返し単位はジペプチド単位と相互作用します。
その後、研究者らは、設計された4反復ユニットバージョンの結合タンパク質を蛍光偏光実験により特性評価しました。その結果、LKおよびPV反復ペプチドに対してはナノモルレベルの結合能を示しましたが、より極性の高いRTおよびYDに対しては結合能が弱く、柔軟性の高いGAに対しては全く結合シグナルが検出されませんでした。
ポケット特化
タンパク質活性ポケットを指定するステップでは、研究者らは拡散モデリングを使用してポケットを微調整し、特定の標的ペプチド配列とのより正確な一致を実現しました。
テンプレートマッチング効率を向上させるため、研究者らは設計した結合ポケットを改良し、相互作用する反復ユニットの数を4から5に増やしながら、標的配列との適合性を向上させました。このアプローチは、標的構造間の親和性も向上させました。反復タンパク質とペプチド骨格間の側鎖二股水素結合を囲む4~9個のアミノ酸は固定したまま、設計した結合タンパク質間の疎水性相互作用を多様化しました。
この戦略の利点は、水素結合の幾何学的構成要件がより厳格であることです。これと比較して、非極性疎水性スタッキングは空間的自由度が高いため、設計においては、水素結合を最初から繰り返しサンプリングするよりも、テンプレート方式で水素結合を直接保持する方が効率的です。
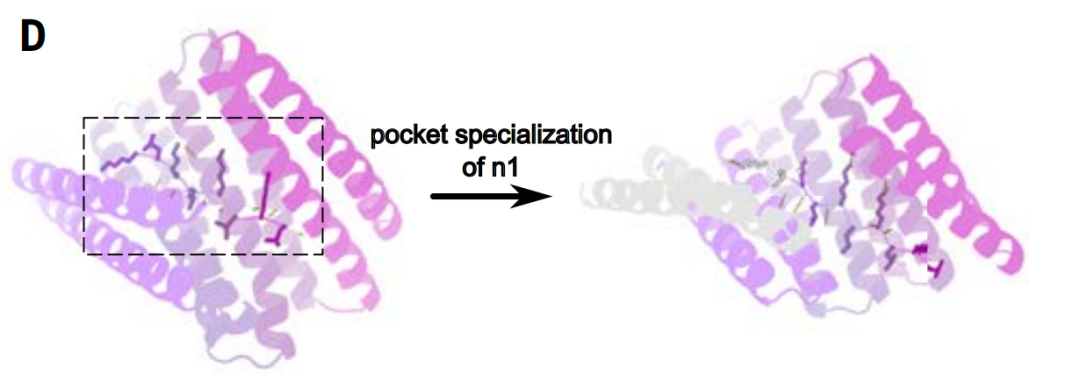
新たに拡張された5番目の繰り返し構造は薄い灰色で示されている。
ポケットアセンブリ
ポケット組み立て工程では、研究者らは、RF拡散モデルを使用してポケット間のインターフェースを作成し、全体的な剛性構造を実現し、結合ポケットを新しいバックボーンに組み立てるためのテンプレートを生成しました。テンプレート内のさまざまなポケットは、さまざまな順序と形状に従って配置され、一連の拡張されたコンフォメーションでペプチドターゲットと相互作用し、非反復配列のより一般的な認識を可能にします。
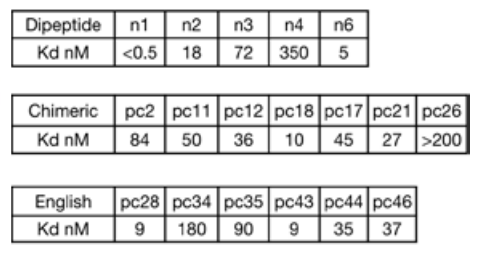
本研究では、キメラペプチド標的と相互作用するキメラタンパク質モデルを作製した後、結合ポケットをパラメータ的に配置して、高周波拡散によってそれらを接続しました。このアプローチを用いて、7つのキメラ標的について70の設計案を作成しました。スプリットルシフェラーゼ存在量とバイオレイヤー干渉法による特性評価の結果、標的ごとに平均わずか10の設計を試験しただけで、7つの標的のうち6つが2桁ナノモルの結合を達成したことが明らかになりました。
より広範囲の配列をカバーできるようにテンプレート ライブラリのサイズを拡大するため、この研究ではポケット アセンブリ技術を使用して、極性残基を認識するポケットを含む 36 個のキメラ バックボーンを構築し、設計された結合タンパク質と対応するペプチド バックボーンから構成される 1,000 個のテンプレートを生成しました。このテンプレートでは、ペプチド構造内のアミノ酸が結合タンパク質内の設計されたポケットと一致できます。
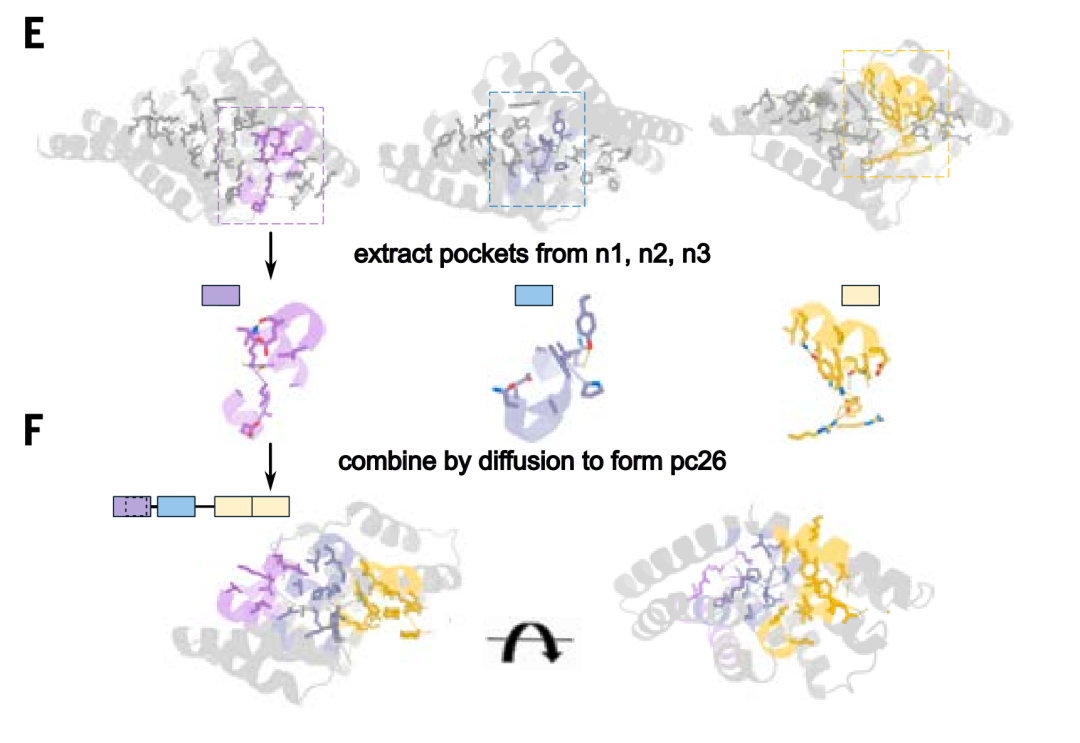
IDR結合タンパク質の設計と最適化
テンプレートライブラリを構築した後、研究者は自然に無秩序な領域をテンプレートライブラリに挿入し、非反復性の合成配列や自然に構造化されていないあらゆる標的に結合できる結合タンパク質を生成しました。このステップは、スレッドマッチングと構造改良の2つの部分に分かれています。
スレッドマッチング:最も適合性の高い配列フラグメントとテンプレートのペアを決定する
スレッド マッチングでは、ターゲット シーケンスが各テンプレートのバックボーンにスレッド化され、テンプレートとペアになる最も互換性のあるシーケンス フラグメントが識別されます。
一般的に、IDPまたはIDRには標的として使用できるペプチドが多数存在します。IDRにおいて最も標的となりやすいペプチドを見つけるには、この研究では、まず、配列の複雑性が低いペプチドと、プロテオーム内で複数の近似値を持つペプチドを除外し、そのようなターゲットの結合剤との交差反応を防止しました。残りのアミノ酸のユニークな配列フラグメントをテンプレートライブラリのターゲットバックボーンにマッピングすることによってローカルバックボーンを再サンプリングした後、本研究では、ディープラーニングベースのタンパク質配列設計ツールProteinMPNNを使用して結合タンパク質の配列を最適化し、設計された結合タンパク質と標的配列との適合性、およびAF2予測値とモデルとの整合性に基づいて評価しました。
AF2メトリクスが最適ではない場合、RFdiffusionを用いて特定のターゲットに合わせてバックボーンをカスタマイズしました。その後、スレッドマッチングを用いて、治療上重要なIDP、IDR、およびIDPフラグメントに対するバインダーを生成し、ターゲットごとに平均28個のデザインを作成しました。
構造最適化:結合タンパク質と標的ペプチドの適合性の向上
設計された結合タンパク質とターゲットペプチド間の適合性を高めるために、最適な一致も最適化されました。この研究では、DYNA_1b1結合タンパク質とダイノルフィンの解離定数をテスト用に選択し、合成ターゲットのヒット率が最も高くなるように無線周波数拡散を最適化しました。結果は、48 のデザインのうち 45 がスクリーニング テストで強い親和性を示し、6 つのデザインの解離定数のみが弱い親和性を示したことを示しました。
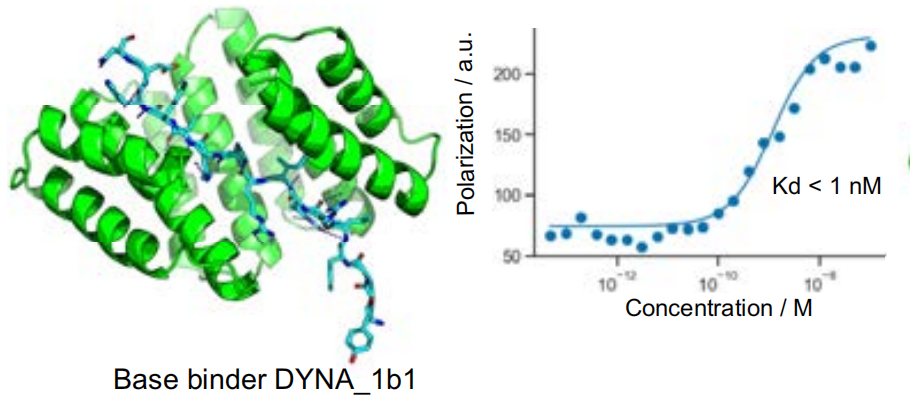
ダイノルフィン構造と結合タンパク質間の直交性の検証
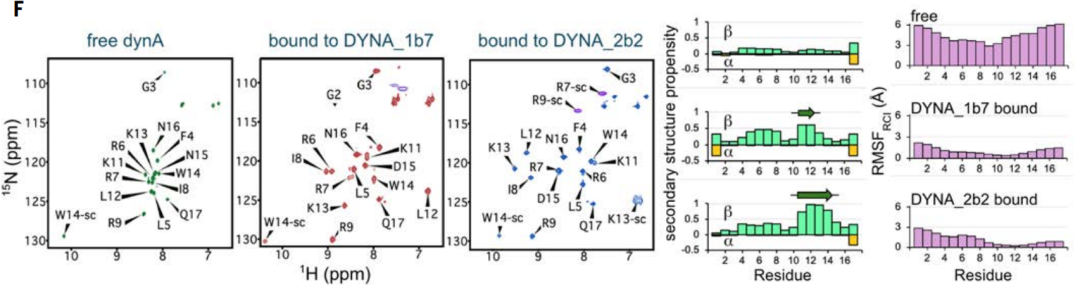
結合中のダイノルフィン構造の変化を確認するために、同位体標識ダイノルフィン A の核磁気共鳴 (NMR) スペクトルを、結合していないとき、DYNA_1b1 に結合しているとき、およびより高い親和性で DYNA_2b2 に結合しているときの溶液中で検出しました。
NMR の結果から、遊離のダイノルフィン A は本質的に無秩序であるが、設計されたバックボーンに含まれる領域は結合すると秩序化されることが示されました。両方の結合複合体について、NMR データにより、設計されたモデルと一致して、拡張された結合状態立体配座が明らかになり、無秩序なタンパク質およびペプチドを非ネイティブ立体配座に誘導するダイノルフィンの作用の有効性が確認されました。
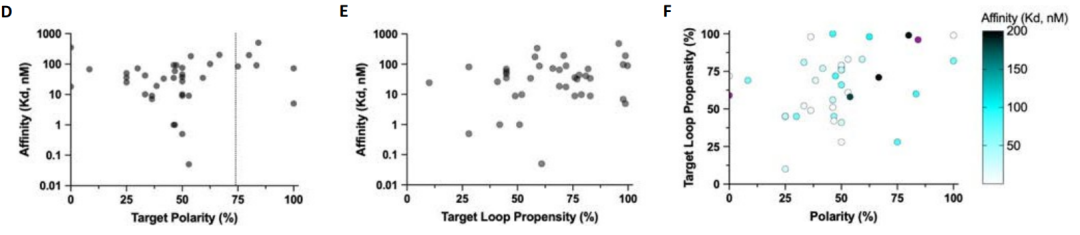
研究者らは、Logosの最適化の可能性を探るため、結合定数(Kd)が約1 nMのダイノルフィン結合剤DYNA_1b1を選択しました。研究者らは、RF拡散法を用いて上位にランクされた設計を最適化しました。48の設計のうち、45の設計がBLIスクリーニングにより5 nMの濃度で強い結合を示し、そのうち6つはBLI測定でKd値が100 pM以下でした。最適化された2つの設計(DYNA_2b1とDYNA_2b2)の蛍光偏光測定では、Kd値がそれぞれ60 pMと100 pM未満であることが明らかになりました(下図B)。
注: ダイノルフィンは、慢性疼痛に関連するカッパオピオイド受容体 (KOR) ペプチドリガンドです。
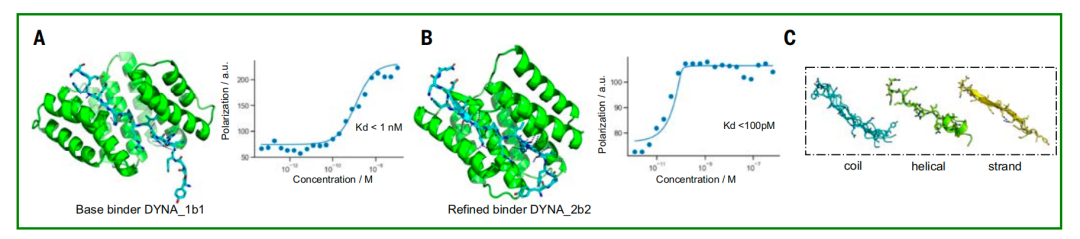
ダイノルフィンAのオリジナル設計および最適化設計では、ペプチドはランダムコイル、部分的なβストランド構造、部分的なαヘリックス構造など、複数の立体構造を示しました(上図C参照)。ダイノルフィンAとBは62%という配列相同性を共有していますが、それぞれの結合タンパク質は互いに交差せず、それぞれの標的にのみ結合します。同時に、ダイノルフィンAに結合した設計タンパク質DYNA_1b7の共結晶構造は、特にコア結合界面において、計算設計モデルと高い整合性を示しました(上図D〜E)。NMRデータによって、さらに以下のことが確認されました。もともと無秩序だったダイノルフィン A の骨格は、設計されたタンパク質に結合すると整列し、誘導適合メカニズムの有効性が再び確認されました (上図 F)。
結合タンパク質の機能性と直交性を検証する
本研究では、WASH複合体およびPER複合体の免疫沈降モデルを使用しました。WASH複合体は、WASH、FAM21、CCDC53、SWIP、およびWASHC2を含みます。試験の結果、FAM21_1b1は細胞溶解物からWASH複合体全体を抽出することが示されました。
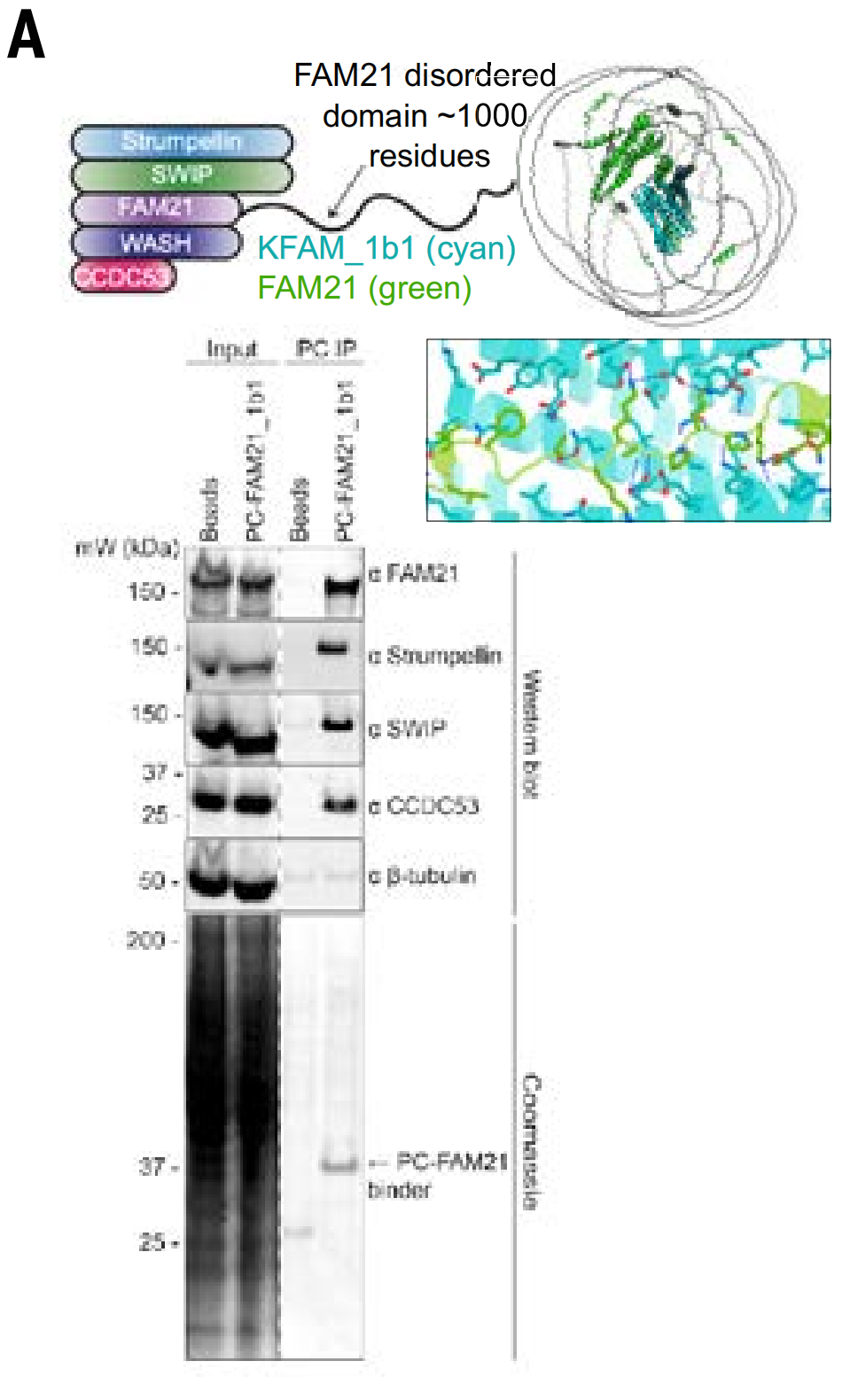
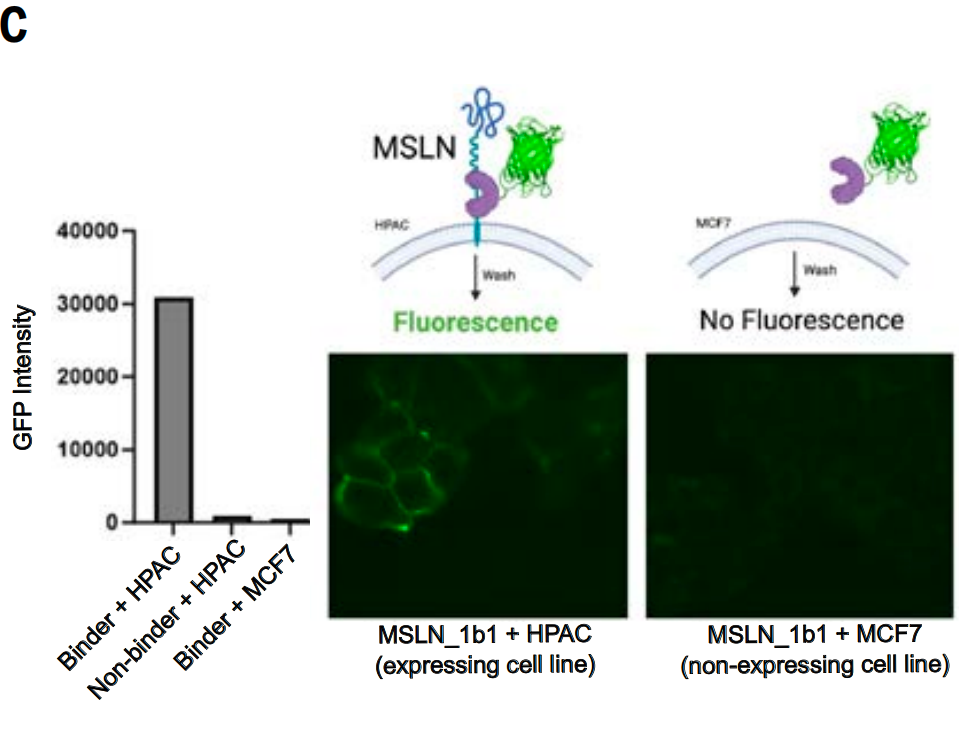
さらに、研究者らは、MSLN 膜近傍領域用に設計された結合タンパク質 (MSLN_1b1) が、この標的を発現している細胞に特異的に結合できるかどうかも研究しました (この領域でのプロテアーゼ切断により、より遠位の細胞外ドメイン領域が標的として適さなくなるため)。
注: メソテリン (MSLN) は、多くの癌においてアップレギュレーションされる細胞表面糖タンパク質であり、そのため腫瘍標的療法において大きな注目を集めています。
研究者らは緑色蛍光タンパク質(GFP)をMSLN_1b1に融合し、MSLNを発現する細胞(ヒト膵臓腺癌細胞株HPAC)およびMSLNを発現しない細胞株(ミシガン癌財団乳癌細胞株MCF7)とともにインキュベートした。また、コントロールとして、MSLNに結合しないGFP融合タンパク質も含めた。
蛍光顕微鏡観察により、GFP-MSLN_1b1融合タンパク質がHPAC細胞において細胞間結合部に凝集していることが明らかになりました。これはMSLNの局在特性と一致しています。この現象はMCF7対照細胞では観察されませんでした。さらに、図Cに示すように、対照結合タンパク質はHPAC細胞において結合シグナルを示さなかったことから、MSLN_1b1は細胞表面上のMSLNを特異的に認識し、結合すると考えられます。
AI主導でタンパク質標的の新たな展望を開く
現在、AIはタンパク質を標的とした研究にますます関与しており、研究は「マルチテクニック並列化」という新たな段階へと進んでいます。デビッド・ベイカー氏のチームに加え、ペンシルベニア大学のジョージ・M・バースレム氏とオフィール・シャレム氏のチームも、標的タンパク質研究において革新的なブレークスルーを達成しました。このチームは「タンパク質編集」技術を提唱し、スプリットインテインシステムを用いて、生きた哺乳類細胞内で合成されたタンパク質のアミノ酸配列を直接改変することに成功しました。これは、非標準アミノ酸や化学タグ(ビオチン、蛍光色素)を内因性タンパク質に精密に組み込むことに成功した初めての事例です。この研究成果は、「細胞内タンパク質編集により、内因性タンパク質への非標準残基の組み込みが可能になる」というタイトルでサイエンス誌に掲載されました。
用紙のアドレス:
https://www.science.org/doi/10.1126/science.adr5499
さらに、中国科学院遺伝学・発生生物学研究所の高彩霞氏と華中農業大学の李国田氏が率いる中国と国際研究チームは、デイビッド・ベイカー氏のチームのタンパク質設計研究を基に、逆フォールディングモデルに基づく汎用タンパク質工学手法「AiCE」を開発しました。このAI駆動型タンパク質設計戦略を用いて、研究チームはデアミナーゼやヌクレアーゼを含む8つのタンパク質クラスの最適化に成功し、新たな塩基エディターを開発しました。「構造的制約と進化的制約を統合した逆フォールディングモデルによるタンパク質進化の促進」と題されたこの研究論文は、Cell誌に掲載されました。
用紙のアドレス:
https://www.cell.com/cell/abstract/S0092-8674(25)00680-4
生細胞編集から神経保護療法、糖鎖修飾の革新からAIを活用したマルチチェーン設計まで、バイオメディシンにおけるAIの継続的な進歩に伴い、世界中のチームがこれまでにない多様なアプローチを用いて、自然変性タンパク質に潜むバイオメディカル課題に取り組んでいます。研究チームによる自然変性領域を標的とした研究は、がんやアルツハイマー病などの疾患に対する新たな治療法の創出につながるでしょう。
参考リンク:








