Command Palette
Search for a command to run...
ペンシルバニア大学は動物毒から386個の新しい抗菌ペプチドを発見し、潜在的な抗生物質候補を選別するためのディープラーニングモデル「APEX」を開発した。

1928年のペニシリンの発見は、細菌感染症との闘いにおける人類の新たな時代を切り開きました。これらの抗生物質は、感染症に対する人類の闘いの能力を飛躍的に向上させました。しかし、医療や農業などの分野で抗生物質が広く使用され、過剰に使用された結果、細菌耐性が出現し、驚くべき速度で悪化しています。
21世紀に入り、抗生物質耐性は世界の公衆衛生分野における大きな課題となっています。世界保健機関(WHO)のデータによると、薬剤耐性感染症によって毎年世界中で約500万人が直接的または間接的に死亡しており、この危機の主因は抗生物質耐性病原体の急速な増加と蔓延です。中でも、WHOが「優先病原体」に指定するグラム陰性細菌は特に厄介です。薬剤耐性を進化させる能力が強く、人類との「抗菌戦争」で優位に立っています。さらに深刻なのは、過去数十年間で、新規抗生物質の研究開発は停滞している。研究開発費の高騰と臨床試験サイクルの長期化により、多くの製薬企業がこの分野への参入をためらっている。新規抗生物質の数は激減し、薬剤耐性菌の進化に大きく遅れをとっている。
このジレンマに直面した科学者たちは、自然界の潜在的な「宝庫」、すなわち動物毒に注目し始めました。数百万年にわたる自然進化を経て、動物毒は生理活性ペプチドやタンパク質を含む、極めて豊かな分子多様性を生み出してきました。広範囲の生物学的標的と相互作用できるだけでなく、顕著な抗菌活性も示します。実際、生物医学における毒の応用は既に確固たる成功例となっています。イモガイ毒から抽出されたジコノチド(商品名:プリアルト®)は、慢性疼痛の治療に重要な鎮痛剤となっています。また、ヘビ毒から開発されたカプトプリルは、臨床現場で広く使用されている降圧剤です。これらの例は、抗菌分野における毒の探究に重要な知見をもたらします。
この文脈では、米国ペンシルベニア大学の研究チームは、世界中の毒タンパク質から抗菌候補ペプチドをハイスループット方式でスクリーニングし、世界中の動物毒から新しい構造を持つ抗菌ペプチド386個を発見しました。その中で、実験的に検証された分子91.4%は強力な抗菌活性を示し、新世代の抗生物質の発見に画期的なアイデアを提供しました。
関連する研究結果は、「Vennomics人工知能による抗菌剤発見のための地球規模の毒の計算探査」というタイトルでNature Communicationsに掲載されました。
研究のハイライト:
* この研究では、世界的な毒データベースを構築し、合計 16,123 個の毒タンパク質を含む暗号化ペプチドの候補ライブラリを生成し、4,000 万個を超える毒暗号化ペプチドを生成しました。
* この研究では、ディープラーニングモデル APEX を使用して、34 種類の細菌に対する各ペプチドの最小阻害濃度を予測し、最終的に抗菌力があり既知の AMP との配列類似性が低い 386 個の候補ペプチドをスクリーニングしました。
* 研究チームは386のVEPから58のペプチドを合成して試験したところ、そのうち91.4%(53)は少なくとも1つの病原体に対して顕著な阻害効果を示した。

用紙のアドレス:
https://www.nature.com/articles/s41467-025-60051-6[*](https://arxiv.org/pdf/2507.09466*)
公式アカウントをフォローし、「APEX」と返信すると完全なPDFが手に入ります。
データベース: 16,000種類の毒タンパク質のコレクション
新たな抗菌薬候補を同定するため、本研究ではConoServer、ArachnoServer、ISOB、VenomZoneの4つのデータベースから毒タンパク質配列を収集し、合計16,123個の毒タンパク質を抽出しました。ConoServerはコノペプチド、ArachnoServerはクモ毒、ISOBはバングラデシュ在来のヘビ毒のデータベースです。UniProtKBから編集されたVenomZoneは、ヘビ、サソリ、クモ、イモガイ、イソギンチャク、昆虫の6つの分類群(本研究ではUniProt)のタンパク質を網羅しています。
これらのデータセットはそれぞれ独自の特徴と用途を持っています。下図に示すように、異なるデータベース間での種の重複を分析したところ、UniProtには699種もの固有種が含まれており、タンパク質の多様性が広いことが示されています。一方、ConoServerとArachnoServerには比較的少数の固有種が含まれており、ISOBには固有種が全く含まれていません。この多様性の存在により、研究者は毒液中の抗菌ペプチドをさまざまな角度から調査し、さまざまな情報源からのタンパク質情報を統合し、新しい抗菌ペプチドを発見する可能性を高めることができます。
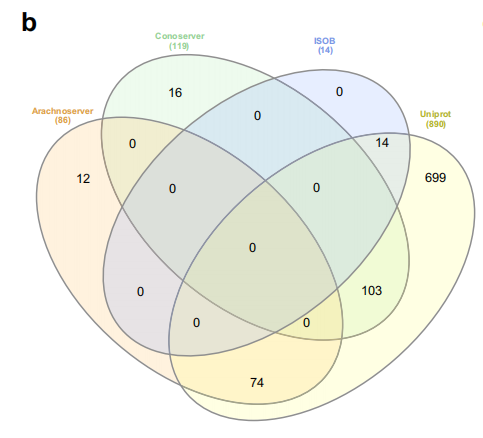
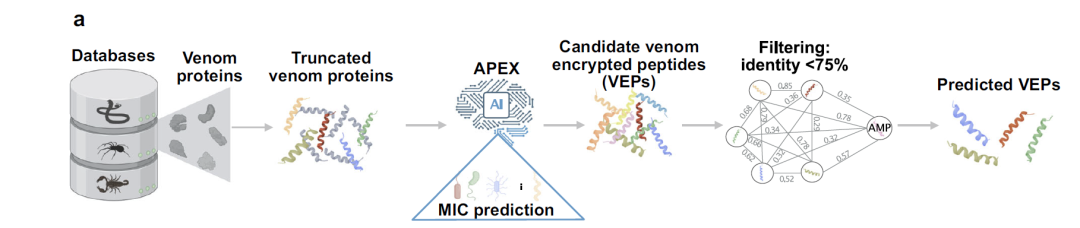
下図に示すように、研究者らはこれらの毒タンパク質を処理し、4,000万個を超える毒暗号化ペプチド(VEP)を生成しました。その後、APEXディープラーニングモデルを用いてこれらのペプチドの抗菌活性を予測し、細菌株に対する各ペプチドの最小発育阻止濃度(MIC)値を推定しました。抗菌活性の指標としてMICの中央値を使用しました。さらなるスクリーニングにより、研究者らは最終的に、MICの中央値が32 μmol L⁻¹以下の7,379個のVEPを特定しました。既知の抗菌ペプチドとの配列類似性をフィルタリングした後、386個の候補が特定されました。これらの候補は既知の抗菌ペプチドとは構造的および機能的に異なり、その後の実験検証のための貴重なターゲットとなります。
APEXモデル予測:複数ソースの毒プロテオームデータを統合し、抗菌候補ペプチドをスクリーニングするプロセス
APEX モデルは、抗菌ペプチドの潜在的なマイニングと予測に重点を置いたディープラーニング ツールです。このモデルの最大の強みは、細菌株特異的な抗菌活性を予測する能力にあります。体系的な学習により、このモデルは34種類の細菌株に対するペプチドの最小発育阻止濃度(MIC)値を正確に予測できるようになりました。MIC値は抗菌剤の有効性を測定するための重要なパラメータです。
モデルのトレーニング データセットは 2 つの部分で構成されます。内部的に蓄積されたペプチド配列データと、DBAASP データベースから取得された公開抗菌ペプチド (AMP) 情報。この多様なデータサポートにより、APEX はペプチド配列と抗菌活性の複雑なマッピング関係の解析において優れたパフォーマンスを発揮し、その後のスクリーニングのための強固なアルゴリズム基盤を築くことができます。
アーキテクチャ設計の面では、APEX は多目的タスク モデル フレームワークと「3 段階」戦略を採用して、毒素タンパク質から抗菌ペプチド候補分子をスクリーニングします。初め、スライディングウィンドウ法を使用してペプチドライブラリを構築し、8〜50アミノ酸の長さの断片を選択しました。第二に、APEX モデルを使用して、さまざまな細菌株に対するこれらのペプチドの最小阻害濃度 (MIC) を予測しました。やっと、配列の類似性に基づいてさらに選択を行った結果、新しい構造と優れた活性を備えた毒暗号化ペプチド (VEP) のグループが生まれました。
この設計により、大量の毒暗号化ペプチド(VEP)を処理する際に、抗菌活性を持つ候補分子を正確に特定することが可能になります。従来の実験スクリーニング法と比較して、APEXは抗菌ペプチドの発見効率を大幅に向上させると同時に、リソース集約型の生化学アッセイへの依存を軽減し、研究者にとってより迅速で経済的なスクリーニングパスを提供します。
APEXモデルを実際のアプリケーションで使用するには、一定の技術的準備が必要です。研究者は、Python 3.9、特定のバージョンのPyTorch、そしてnumpy、scipy、matplotlibなどの一連の依存ライブラリのインストールを含む、適切なランタイム環境をセットアップする必要があります。環境がセットアップされると、候補ペプチド配列がテキストファイルに保存されます。指定されたコマンドを実行すると、モデルはMIC予測結果を含むCSVファイルを生成します。これらの予測を分析することで、研究者は抗菌ポテンシャルの高いペプチドを迅速に特定し、さらなる実験検証を行うことができます。
VEPの抗菌活性の検証と作用機序の解析
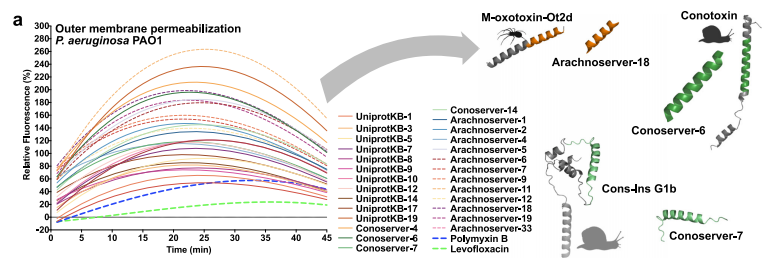
毒暗号化ペプチド(VEP)の抗菌活性を検証するために、研究チームは一連の実験を行った。抗菌活性試験では、58 種類の VEP をさまざまな病原体に対してテストしました。結果は下の図に示されており、そのうち 53 件 (91.4% を占める) が少なくとも 1 つの病原体に対する活性を示しました。ArachnoServerデータベースのすべてのVEPは抗菌活性を示し、このタイプのペプチドの強力な抗菌能力を強く裏付けました。。

しかし、UniProtデータベースに含まれる一部のVEPは、比較的効果が限られていました。例えば、UniprotKB-2は抗菌活性を示さず、UniprotKB-6とUniprotKB-11はEnterococcus faecalisに対して阻害効果しか示しませんでした。さらに解析を進めたところ、これらの低活性ペプチドは一般的に疎水性と正味電荷が低いことが明らかになりました。これは、疎水性と正味電荷は、ペプチドと細菌膜との相互作用を促進する上で重要な役割を果たし、それらのレベルは VEP の抗菌効能に直接影響を及ぼす可能性があります。
二次構造研究では、円二色性(CD)分析により、VEPの構造変化が環境に大きく依存することを観察しました。水溶液中では、VEPは主に無秩序構造で存在しますが、細菌膜を模擬した環境(SDSミセル系など)やらせん誘導媒体(トリフルオロエタノール/水混合溶液など)中では、VEPは顕著な構造変化を起こし、無秩序構造からαヘリックス構造へと折り畳まれます。この構造的特徴は、典型的な抗菌ペプチドの挙動と非常によく一致しています。これは、VEP がバイオフィルム関連の機能に特別に適応している可能性があることを示唆しており、抗菌メカニズムの重要な手がかりも提供します。
作用機序研究の観点から、研究チームは蛍光アッセイを用いてVEPの細菌外膜および細胞質膜に対する作用を評価した。結果は下図に示す通りである。23種類のVEPは細菌外膜を効果的に透過することができ、その中でもArachnoserver-18、ConoServer-6、ConoServer-7は特に優れた透過活性を示した。細胞質膜脱分極実験では、試験したほとんどのVEPの脱分極効果は対照群よりも優れていたが、いくつかの既知のペプチドファミリーよりもわずかに弱かった。全体として、VEPの抗菌作用は、主に細胞膜の脱分極を誘導することによって発揮されます。このメカニズムは、多くの既知の抗菌ペプチドや暗号化ペプチドの作用機序と一致しており、新規抗菌剤としてのVEPの合理性をさらに裏付けています。
学界と産業界が協力して、新しい低耐性抗菌薬の探索と開発に取り組んでいます。
薬剤耐性を誘発する可能性が低い新しい抗菌薬を開発するために、基礎研究から産業変革まで、学界と産業界が共同で探求することで、毒抗菌ペプチドの応用可能性が絶えず解き放たれています。
学術界では、様々な国の研究チームが毒抗菌ペプチドの発見、設計、そしてメカニズム解析について詳細な研究を行ってきました。シンガポールの南洋理工大学の穆玉光チームは、従来のモデルがタンパク質相互作用の予測においてデータ量と特徴次元の限界に直面するという問題に取り組んでいます。これまでで最大の構造ベースのタンパク質相互作用データセットを構築し、彼らはまた、ProAffinity-GNNディープラーニングフレームワークを開発しました。このモデルは、タンパク質言語モデルとグラフニューラルネットワークを革新的に統合し、配列情報と空間構造特徴を深く結び付けています。タンパク質間親和性予測の精度において既存の手法を凌駕するだけでなく、強力な一般化機能により、毒ペプチドと細菌標的との相互作用を効率的に予測するための技術的パラダイムを提供します。さらに、作用機序の解析は、抗菌ペプチドの最適化設計に対する理論的裏付けとなります。
山東大学斉魯医学院の研究チームは、人工知能分野の拡散モデルを用いて、自然界におけるペプチドの進化の「ランダム摂動誘導スクリーニング」過程をシミュレートした。40 個の新規ペプチド配列の生成に成功し、そのうち 25 個は明らかな抗菌または抗真菌活性を示しました。
ビジネスの世界では、イスラエルのBountica社が食品保存分野の課題に着目し、微生物発酵技術を用いて毒由来の抗真菌ペプチドを大量生産しています。同社のVenomShieldシリーズは、パンやジュースのカビや酵母の増殖を抑制するには、わずか 1 ppm の防腐剤で十分であり、保存期間が従来の防腐剤の 2 倍以上に延長されます。
中国上海ハイテクバイオエンジニアリング株式会社は復旦大学と協力し、私たちは協力して、世界で 3 つの比較的完全な抗菌タンパク質データベースを構築しました。これらのデータベースは、新しい生物学的抗菌酵素や抗菌ペプチドの研究開発に効率的なツールを提供するだけでなく、体系的なデータマイニングを通じて薬剤耐性微生物に対する新しい物質の研究を加速します。
これは、毒抗菌ペプチドの研究と応用が、基礎科学から産業応用まで、非常に活力に満ちていることを示しています。ディープラーニングモデルが配列と機能の関係をますます正確に分析し、合成生物学技術がペプチドの安定性と発現効率を継続的に最適化するにつれて、自然の「生存の知恵」に着想を得たこれらの分子は、抗生物質耐性との闘いにおいて重要な力となり、世界の公衆衛生に新たな安全策を提供する可能性を秘めています。
参考記事:
1. https://mp.weixin.qq.com/s/lwYWWIe9-Az22jlhiZ747A
2. https://mp.weixin.qq.com/s/sGHRf-ebaSRiqGiUxMMueA
3. https://mp.weixin.qq.com/s/0mJltGuaKTUwYDGK4wD9UA
4. https://mp.weixin.qq.com/s/R6Y_-38saZwqSSow0LNhzw








