Command Palette
Search for a command to run...
NVIDIA は原子レベルのタンパク質設計で画期的な進歩を達成し、最大 800 残基のタンパク質を高精度で生成しました。
特定の構造と機能を持つ新しいタンパク質を設計することは、創薬やバイオエンジニアリングなど、多くの分野において大きな応用の可能性を秘めていることはよく知られています。しかし、この目標を達成するのは容易ではなく、特にタンパク質の配列と構造の関係を捉えることは、タンパク質をゼロから設計する上で常に大きな課題となってきました。
過去には、ほとんどの方法ではタンパク質の配列と構造を別々に設計する傾向がありました。例えば、まず配列を生成してから折り畳む、あるいはまずバックボーンを設計してから配列を決定するといった方法があります。しかしながら、タンパク質配列と全原子構造の結合分布を正確にモデル化し、機能部位の微細制御を実現し、原子モチーフ骨格設計といった重要なタンパク質設計タスクを完了することは、依然として非常に困難な課題です。これには、離散配列と連続座標を扱うだけでなく、配列に応じて変化する側鎖の寸法の問題にも対処する必要があります。
この文脈では、NVIDIAの研究チームとカナダのケベック人工知能研究所MilaがLa-Proteinaを提案した。これは、部分的なポテンシャルフローマッチングに基づく原子レベルのタンパク質設計手法です。明示的なバックボーンモデリングと各残基の固定サイズのポテンシャル表現を効果的に組み合わせることで、配列と原子側鎖の情報を取得できます。これにより、タンパク質生成における明示的な側鎖表現の次元変動という重要な課題を解決し、タンパク質設計分野に新たなブレークスルーをもたらします。
関連する研究結果は、「La-Proteina: 部分的潜在フローマッチングによる原子論的タンパク質生成」というタイトルで arXiv に掲載されました。
研究のハイライト:
* 部分的に暗黙的なフローマッチングフレームワークLa-Proteinaが提案されています。これは、タンパク質配列と完全な原子レベルの構造を共同生成するために設計されています。明示的な主鎖モデリングと各残基の固定サイズの暗黙的表現を効果的に組み合わせることで、配列と原子レベルの側鎖の両方を捉えます。
* 広範なベンチマーク実験において、La-Proteina は無条件タンパク質生成において SOTA パフォーマンスを達成し、最大 800 残基の多様で共設計可能、かつ構造的に有効な完全な原子スケールのタンパク質を生成できます。
* この研究では、2 つの重要な条件付きタンパク質設計タスクであるインデックス付きおよび非インデックスの原子レベルのモチーフ スキャフォールド設計に La-Proteina を適用し、このモデルが従来の全原子ジェネレーターより優れていることを実証しました。

用紙のアドレス:
AIフロンティアに関するその他の論文:
https://go.hyper.ai/owxf6
データセット: 無条件モデル、タンパク質データの機能、関数のトレーニングに使用
この研究では、無条件モデルのトレーニングに 2 つのデータセットを使用しました。
1 つは、AlphaFold データベース (AFDB) のスクリーニングとクラスタリングから派生した、Foldseek によってクラスタ化された AFDB データセットです。クラスタリングでは、配列情報と構造情報を組み合わせ、約300万個の固有サンプルの初期セットを複数の基準を用いて最適化しました。これらの基準とは、平均pLDDTスコアが80以上、タンパク質長が32~512残基、コイル比が50%未満、連続コイル残基数が20以下といったものです。モデル生成タンパク質におけるβフォールド含有量の低さを補正するために、βフォールドの存在が特に求められました。最終的に約55万個のタンパク質サンプルが得られました。このデータセットは、モデルによって生成されたタンパク質の構造的特徴のバランスをより良くし、特にβフォールドの含有量を改善するために慎重に選別されています。
2 つ目は、長いシーケンスのトレーニング用にカスタマイズされた AFDB サブセットです。研究者らは、平均 pLDDT が少なくとも 70 で長さが 384 から 896 の AFDB サンプルをスクリーニングしました。クラスタリング後、トレーニング用に 400 万を超えるクラスターが得られました。より長いタンパク質サンプルに焦点を当て、長いシーケンスのトレーニングのニーズを満たします。
さらに、タンパク質データ自体には、配列(20種類の残基)と3D構造情報が含まれており、これらはAtom37表現を用いて統一的に保存されます。Atom37表現は、各残基について37個の潜在的な原子からなる標準化されたスーパーセットを定義します。L個の残基からなるタンパク質構造は、[L, 37, 3]の形状のテンソルとして保存でき、各残基の種類に応じて関連する座標サブセットが選択されます。
この標準化手法の特徴は、異なる残基の構造情報を統一的に保存および表現する方法を提供することであり、これによりモデルが異なる残基の構造情報を統一的に処理するための基盤が築かれます。AFDBの大規模データ特性は、モデルに豊富なサンプルを提供し、より広範なタンパク質配列と構造的特徴を学習し、性能と汎化能力を向上させるのに役立ちます。これらのデータを用いたトレーニングと実験を通じて、関連モデルはタンパク質配列と構造の関係をより適切に捉え、より正確な設計を実現できます。
La-Proteina: 原子レベルのタンパク質設計モデルの革新的なアーキテクチャとトレーニングメカニズム
La-Proteinaは、原子レベルのタンパク質設計のための革新的なモデルです。その中核となる設計は「部分的な暗黙的表現」を軸にしており、完全な原子レベルの構造を生成する際の複雑な課題を解決することを目指しています。
設計レベルでは、大規模なバックボーン、アミノ酸の種類、側鎖(側鎖の大きさはアミノ酸によって異なる)を考慮しながら全原子構造を生成するという課題を考えると、La-Proteina は、α 炭素座標による明示的なバックボーン モデリングを維持しながら、各残基の原子レベルの詳細と残基タイプを固定長の連続潜在空間にエンコードすることを提案します。
この設計は複数の利点をもたらします。モデルの主要生成コンポーネントにおける混合連続分類モデリングの難しさを回避し、完全連続フローマッチング法による効率的な隠れ変数生成を可能にするだけでなく、高性能なメインチェーンモデリングの進歩を基盤とすることができます。同時に、明示的なメインチェーンモデリングにより、グローバルα炭素骨格と残基の原子レベルの詳細に対して異なる生成スケジュールを設定できるようになり、これが高性能化の鍵となるだけでなく、スケーラビリティも向上し、最大800残基の大規模タンパク質へのモデル拡張が可能になります。このハイブリッドアプローチこそが、完全暗黙的モデリングフレームワークよりも優れている主な理由です。
構成構造から、下図に示すように、La-Proteina の中核は、エンコーダー、デコーダー、ノイズ除去の 3 つのニューラル ネットワークで構成されています。これら 3 つはすべて、バイアス アテンション メカニズムに基づくコア Transformer アーキテクチャを共有しています。
このうち、エンコーダーは、入力タンパク質(配列および構造情報を含む)を潜在変数にマッピングする役割を担います。初期配列表現には、元の原子座標、側鎖およびバックボーンのねじれ角、残基の種類が含まれ、初期ペア表現には、残基間の相対的な配列間隔、ペア間距離、相対方向が含まれます。デコーダーは、潜在変数とα炭素原子座標から完全なタンパク質を再構築し、各残基の8次元潜在変数とα炭素原子座標を処理します。ノイズ除去ネットワークは、サンプルを標準ガウス参照分布からターゲットデータ分布に転送する速度場を予測し、Transformerブロックで補間時間を直接調整します。
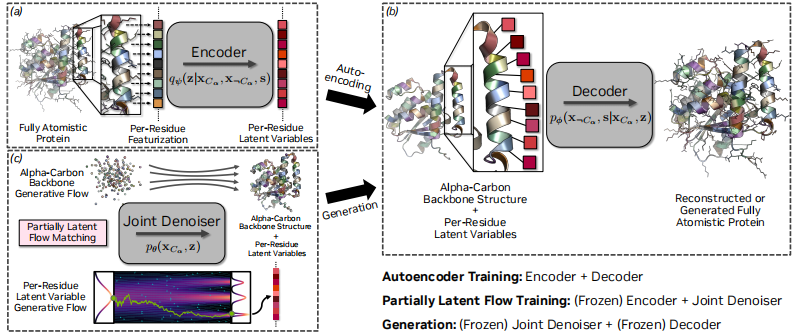
トレーニング方法に関しては、La-Proteina は 2 段階のトレーニング戦略を使用します。
最初の段階では、条件付き変分オートエンコーダ(VAE)を学習します。エンコーダは入力タンパク質を潜在変数にマッピングし、デコーダは潜在変数とα炭素原子座標に基づいてタンパク質を再構成します。VAE全体は、β重み付き証拠下限値(ELBO)を最大化することで最適化されます。上記のモデリングの選択により、再構成項は配列のクロスエントロピー損失と構造のL2損失の2乗に簡略化できます。
第2段階では、フローマッチングモデルを最適化し、目標分布を近似します。ノイズ除去ネットワークは、条件付きフローマッチング(CFM)の目的関数を最小化するように学習されます。この段階の鍵となる設計は、2つの異なる補間時間txとtzの使用です。この設定により、推論中にα炭素原子座標と潜在変数に対して異なる積分スケジュールを適用できるようになり、モデルのパフォーマンスが効果的に向上します。
このような設計とトレーニングを通じて、La-Proteina はタンパク質配列と全原子構造の結合分布を効率的に学習することができ、原子レベルのタンパク質設計に強力な技術的サポートを提供します。
実験結果:ラプロテイナは4つのテストすべてで大きな差をつけてリード
研究チームは、La-Proteina の性能を検証するために、さまざまなシナリオでのモデルの性能を総合的に考慮し、無条件の原子レベルのタンパク質生成と原子モチーフの足場設計という 2 つの主要な方向を中心に一連の実験を実施しました。
無条件原子スケールのタンパク質生成実験では、下図に示すように、研究チームはLa-Proteinaの2つのバリアント(三角形増殖層の有無)を、P(全原子)、APM、PLAIDなど、公開されている複数の全原子生成ベースライン手法と比較しました。評価指標は、全原子協調設計能力、多様性、新規性、標準設計能力です。
結果は、La-Proteina の 2 つのバリアントが、全原子共同設計機能、設計容量、多様性の点ですべてのベースライン手法よりも優れており、新規性の点でも非常に競争力があることを示しています。
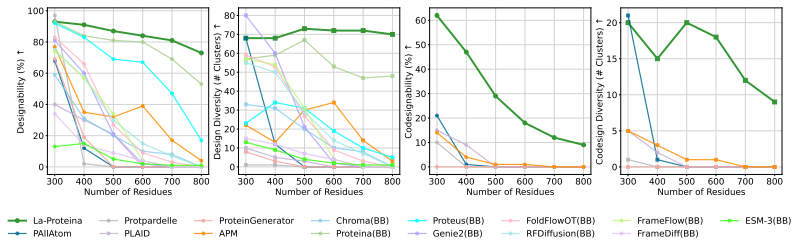
ラプロテイナの無条件長鎖生成能力
注目すべきは、三角形の乗算層を使用しない La-Proteina は、高いスケーラビリティを備えながら最先端のパフォーマンスを実現するのに対し、2 番目に優れたパフォーマンスの P (全原子) は、計算コストの高い三角形の更新層に依存しているため、短いタンパク質しか処理できないことです。
また、研究チームはまた、La-Proteina が大規模な全原子構造を生成する際の拡張性も実証しました。約 4,600 万のサンプルを含む AFDB データセットでトレーニングされたこのモデルは、残基の長さが 500 を超えるタンパク質を生成するタスクで最高のパフォーマンスを発揮します。一方、他の全原子ベースライン方法では、この長さの範囲で有効なサンプルを生成するのが困難な場合がよくあります。
生体物理学的分析では、MolProbity ツールを使用して構成妥当性を評価しました。結果は、La-Proteina によって生成された構造の方が高品質であることを示しました。スコアはすべてのベースライン手法よりも大幅に優れており、生成された構造は物理的レベルでより現実的で、実際のタンパク質に類似しています。同時に、側鎖の二面角の分布を視覚化し、PDBおよびAFDBの参照と比較することで、La-Proteinaはアミノ酸回転異性体の配座空間を正確にシミュレートすることができます。ベースライン法は基準から逸脱することが多く、パターンが欠落したり、非現実的な角度領域が埋められたりします。
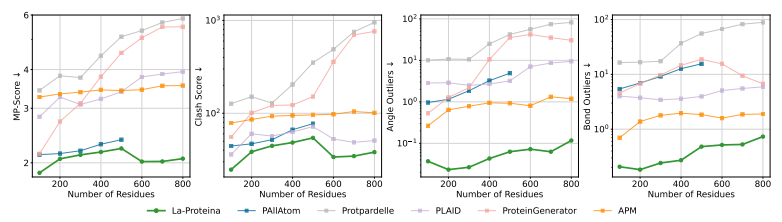
ラプロテイナは既存の全原子世代のベースラインよりも優れています
構成妥当性が高い
原子モチーフの足場設計実験では、研究チームは、原子モチーフ骨格設計タスクにおけるモデルの性能を評価しました。このタスクでは、モデルは事前に定義されたモチーフの原子構造に基づいて、そのモチーフを正確にサポートできるタンパク質構造を生成する必要があります。実験は、全原子および高度な原子骨格設計、インデックス付きバージョンおよびインデックスなしバージョンを含む4つの評価設定で実施されました。
結果は、4つの設定すべてにおいて、La-Proteina は、唯一の比較可能な全原子ベースライン手法である Protpardelle を大幅に上回り、ほとんどのベンチマーク タスクを正常に解決できます。特に、3 つ以上の異なる残基セグメントで構成されるモチーフの場合、La-Proteina の非インデックス バージョンの方がインデックス バージョンよりもパフォーマンスが優れています。これは、複数のセグメントの位置を固定すると、さまざまな構造ソリューションを探索するためのモデルの柔軟性が制限されるためと考えられます。
原子レベルのタンパク質設計分野における科学的ブレークスルーと革新的な実践
タンパク質設計分野において、La-Proteinaに代表される原子レベルのタンパク質設計研究の方向性は、学界と産業界から広く注目を集めており、多くの大学や企業がこの分野で重要な科学的ブレークスルーと革新的な実践を達成しています。
学術界では、いくつかの研究チームがタンパク質生成モデルの性能とスケーラビリティの向上に取り組んでいます。例えば、NVIDIAはMila、ケベック人工知能研究所、モントリオール大学、マサチューセッツ工科大学と提携し、開発された Proteina は、大規模な AlphaFold データベース (AFDB) でトレーニングされました。タンパク質構造生成のためのフローベースモデルのスケーラビリティを実証しました。
タンパク質設計において拡散モデルを用いた研究もいくつかあります。例えば、RFDiffusionやChromaといった初期の拡散ベースのタンパク質生成ツールは、バックボーン生成に重点を置いています。その後の研究では、SO(3)多様体上の拡散やユークリッドフローマッチング法など、タンパク質設計における拡散モデルの適用範囲がさらに拡大しています。
タンパク質の配列と構造の統合モデリングに焦点を当てた研究チームも存在します。例えば、NVIDIAとMITが共同で立ち上げたProtComposerは、補助的な統計モデルと3Dプリミティブを用いてタンパク質構造を生成します。また、タンパク質骨格と配列の統合モデリングや潜在変数モデルを用いることで、全原子構造を扱う研究も行われています。さらに、言語モデルもタンパク質設計に応用されており、タンパク質配列に着目した手法もあれば、構造情報をトークン化して配列と構造の統合モデリングを行う手法もあります。
ビジネスの世界では、オランダのバイオテクノロジー企業であるクレイドルは、人工知能を活用したタンパク質設計プロセスの簡素化に注力しています。同社はウェットラボを設立し、数十億ものタンパク質配列とデータを蓄積し、独自の生成型人工知能モデルを訓練することで、タンパク質の設計と最適化をより容易にしています。アメリカのAI医薬品サービスプロバイダーであるザイラ・セラピューティクスは、高度な機械学習研究、大規模データ生成、治療法開発における優位性を活かし、特定の適応症に適した分子の創出に注力しています。また、タンパク質設計技術と人工知能および機械学習を融合させ、タンパク質設計の効率と精度を向上させることに取り組んでいる企業もあります。
これらの大学の科学研究における画期的な成果と企業の革新的な実践は、タンパク質設計の発展に豊富な経験と技術的支援を提供し、この分野の継続的な発展を促進してきました。技術の継続的な進歩に伴い、タンパク質設計は今後、より多くの分野で重要な役割を果たすようになると考えられています。
参考記事:
1.https://mp.weixin.qq.com/s/7r69S3XpNMjemo3EiXzNeQ
2.https://mp.weixin.qq.com/s/DrZEdsb1SqSSkv_hbrp3TA








